Barta Endre: Funkcionális genomika
|
|
|
- Dezső Fodor
- 6 évvel ezelőtt
- Látták:
Átírás
1 Barta Endre: Funkcionális genomika Barta Endre Debreceni Egyetem, ÁOK, BMBI, Klinikai Genomikai Központ NAIK MBK, Mezőgazdasági Genomikai és Bioinformatikai Csoport
2 Témák Bioinformatika, genomika, funkcionális genomika meghatározása, rövid története Transzkripció meghatározása Funkcionális genomikai módszerek Microarray RNA-seq ChIP-seq GRO-seq ChIA-PET Barta Endre 12/6/16 2
3 Bioinformatikai részterületei Szekvencia analízis DNS szekvencia analízis Fehérje szekvencia analízis Összehasonlító szekvencia analízis(komparatív genomika) Génexpresszió, génszabályozás vizsgálata Genomika, genom annotáció Variáció vizsgálat (pl. GWAS) Funkcionális genomika Struktúrális bioinformatika Filogenetika (komputeres evolúcióbiológia) Biológiai adatbázisok fejlesztése Komputeres biológia (computational biology) = bioinformatikában használt algoritmusok fejlesztése, implementálása
4 Genomika, funkcionális genomika Genomika: A teljes örökítő anyag (magi, mitokondriális és ha van a kloroplasztisz DNS) szekvenálása, a szekvencia annotálása, elemzése Funkcionális genomika: A gének összességének, vagy egy-egy csoportjának a vizsgálata nagy áteresztőképességű kísérletekkel, amelyek vagy a genomszekvencia alapján lettek megtervezve (microarray, SNP), vagy szekvenálást használnak (ChIP-seq, RNA-seq stb.) A genotípus és a fenotípus közti összefűggések keresése genom szinten
5 Genomika története 90-es évek: genomprogramok beindulása, az első egyszerűbb modellszervezetek megszekvenálása (E. coli, élesztő, ecetmuslica, laboratóriumi fonálféreg, A. thaliana stb.), humán genomprojekt elindulása 2001: A draft humán genomszekvencia publikálása 2001-: További modellszervezetek (egér, patkány, csimpánz, fugu stb.), evolúciós szempontból fontos fajok (Ciona intestinalis, zebradánió, közönséges oposszum és vagy egyéb emlős faj) és a mezőgazdaságilag legfontosabb fajok (rizs, kukorica, marha, csirke, sertés stb.) megszekvenálása hagyományos (Sanger) módszerrel Az újgenerációs szekvenálási módszerek megjelenése, egyre több faj és fajta genomszekvenálása Első újgenerációs szekvenáláson alapuló funkcionális genomikai módszerek megjelenése Grandiózus genomszekvenálási projektek meghirdetése (humán 1000 genomprojekt, 100 Solanaceae projekt, Ezer, majd egymillió növényi és állati genom projekt stb.)
6 Funkcionális genomikai NGS technológiák ChIP-seq (ChIP-chip) DNS-fehérje kölcsönhatások genomszintű vizsgálata RNA-seq mrns-ek genomszintű vizsgálata GRO-seq transzkripció genomszintű vizsgálata ChIA-PET DNS hurkok vizsgálata A DNS-t vagy az RNS-t feldarabolják, majd a kis darabokból tag -eket szekvenálnak, és azokat illesztik a referencia genomhoz Az RNA-seq-et lehet használni referencia genom nélkül is, akkor a transzkriptomot rakjuk össze de novo Barta Endre 12/6/16 6
7 Különböző Omikák
8 Újgenerációs illumina szekvenáló könyvtárak készítése
9 A transzkripció leegyszerűsítve Három lépéses folyamat: 1. TF kötődik atfbs-hez (DNA) 2. TF katalízálja a polymerase II complex felépülését 3. A mrns szintézíse a transzkripciós kezdőpontról(tss) TF TFBS Pol-II TATA TSS
10 Eukarióta transzkripció ChIP-seq RNA-seq GROseq ChIA-PET
11 Mit vizsgálunk transzkiptomikában? Promóterek elhelyezkedése Transzkripciós starthely (TSS) elhelyezkedése Transzkripciós faktor kötőhelyek (TFBS) elhelyezkedése A transzkripció elindulásának a biológiai folyamata Legfontosabb kérdés: Hol, mikor, miért, mennyi mrns keletkezik egy gén melyik részéről (alternatív kivágódás/splicing) Mindez hol és hogyan van kódolva a DNS-ben 11
12 Microarray vagy DNS csip Hibridizáción alapszik Robot, csip vagy tintasugaras nyomtató technológia felhasználásával egy kísérletben akár hibridizációt lehet elvégezni Fő ereje a génexpresszió genom szintű vizsgálata (transzkriptomika) Különböző vizsgálatok, chip-ek: Expressziós (próbák mrns-hez) Exon array Promóter array Kromoszóma array SNP array Szekvenálás (complete genomics) Bioinformatikai kihívások: A csipekre felkerülő oligok (cdns-ek) megtervezése A kapott durva adatok feldolgozása, értelmezése 12 Transzkriptomika
13
14 A szkenner által szolgáltatott ábra 14 Transzkriptomika
15 Élesztő spóraképzéshez kapcsolódó négy gén vizsgálata Northern hibridizációval és microarray technológiával 15 Transzkriptomika
16 Két minta expressziós szintjének összehasonlítása Expressziós szint (2. minta) magas alacsony Expressziós szint (1. minta) 16 Transzkriptomika
17 Különböző génexpresszió a különböző szövet és sejt típusokban Brain Fibroblast Astrocyte Astrocyte 17 Transzkriptomika
18 Leíró statisztika: klaszterezés A klaszterező algoritmusok hasznos segítséget jelentenek az adatok megjelenítésében Lehet a géneket ( features ), a mintákat vagy mindkettőt klaszterezni Az általánosan alkalmazott módszer a hierarchikus klaszterezés, amely a filogenetikában használatos neighbor joining módszerhez hasonlít Az eredmény egy fa, amely tartalmaz ágakat és végpontokat 18 Transzkriptomika
19 Kétirányú klaszterezése a géneknek (ytengely), és a sejtvonalaknak (x-tengely) (Alizadeh et al., 2000)
20 GEO at the NCBI 20 Transzkriptomika
21 Array Express at EMBL 21 Transzkriptomika
22 Trancriptome analysis with RNA-seq Total RNA, rrna depleted or mrna isolation Fragmentation cdna synthesis Library preparation Sequencing one or both ends of the fragments Mortazavi et al Nat. Meth Wang et al Nat. Rew. Genet 2009.
23 Samples of interest RNA sequencing Isolate RNAs Generate cdna, fragment, size select, add linkers Condition 1 (normal colon) Condition 2 (colon tumor) Sequence ends Map to genome, transcriptome, and predicted exon junctions Downstream analysis 100s of millions of paired reads 10s of billions bases of sequence
24 RNA Illumina Tru-Seq library prep Size selection step 2 days for 8 samples Adaptor ligation and standard library preparation 5ug of total RNA ~$100 per sample Not strand-specific
25 Mire lehet az RNA-seq-et használni Teljes transzkriptom analízis Új transzkriptek (átíródó régiók) meghatározása Splice variánsok meghatározása Expressziós profil meghatározása (egyes transzkriptek abundanciájának a mérése) Genetikai polimorfizmusok meghatározása: SNPs, micro-indels CNVs
26 RNA-seq analízis pipeline Bowtie illesztő (tophat) + cufflinks + CummeRbound Ez a leggyakrabban használt csomag TRINITYRNASEQ de novo transzkriptóm összerakáshoz Nagy a memóriaigénye (90GB<: HPC a Pécsi Akkor hasznos, ha nincs referencia genom (és/vagy transzkriptóm) Normalizált számlálása a transzkriptekre eső read-eknek-> génexpresszió (probléma: egy génhez több transzkript is tartozhat) A kivágódási (splice) helyeket átfogó read-ek -> exon junctions -> alternatív splicing Alternatív promóterek Eredmény: A differenciáltan kifejeződő gének listája expressziós értékekkel, új transzkriptek listája (új splice formák) 26
27 RNA-Seq: Overview
28 RNA-seq: Assembly vs Mapping Reference-based RNA-seq Ref. Genome or Transcriptome RNA-seq reads De novo RNA-seq contig1 contig2
29 RNA-seq analysis pipeline Reads / tags (fastq format) TopHat Spliced read mapper (built on bowtie) Transcripts, splice variants (bed) Cufflinks Cuffdiff Transcript assembly, differential expression, differential regulation intersectbed, awk, sed Expression levels (fpkm), fold changes CummeRbund Exploration, analysis and visualization bed, fpkm table masigpro R, limma Chipster edger, limma Differential promoter usage Different splicing variants (junctions) Clusters, Heatmaps, Expression profiles, Plots, Gene lists, bed files Clusters, Heatmaps, Expression profiles, Plots, Gene lists, bed files
30 Software Short reads aligners Stampy, BWA, Novoalign, Bowtie (tophat), Data preprocessing (reads statistics, adapter clipping, formats conversion, read counters) Fastx toolkit fastqc Htseq MISO samtools Expression studies Cufflinks package RSEQtools R packages (DESeq, edger, bayseq, DEGseq, Genominator) Alternative splicing Cufflinks Augustus Commercial software Partek CLCBio
31 Bowtie-based TopHat: finds/maps to possible splicing junctions. Important to assemble transcripts later (cufflinks) TopHat: Spliced Reads Kim et al. Genome Biology 2013, 14:R36 METHOD Open Access TopHat2: accurate alignment of transcriptomes in the presence of insertions, deletions and gene fusions Daehwan Kim 1,2,3*, Geo Pertea 3, Cole Trapnell 5,6, Harold Pimentel 7, Ryan Kelley 8 and Steven L Salzberg 3,4
32 Cufflinks package Cufflinks: Expression values calculation Transcripts de novo assembly Cuffcompare: Transcripts comparison (de novo/genome annotation) Cuffdiff: Differential expression analysis
33 Cufflinks: transcript assembly Assembly: Reports the most parsimonious set of transcripts (transfrags) that explain splicing junctions found by TopHat Isoform A Isoform B Differential gene and transcript expression analysis of RNA-seq experiments with TopHat and Cufflinks Cole Trapnell 1,2, Adam Roberts 3, Loyal Goff 1,2,4, Geo Pertea 5,6, Daehwan Kim 5,7, David R Kelley 1,2, Harold Pimentel 3, Steven L Salzberg 5,6, John L Rinn 1,2 & Lior Pachter 3,8,9
34 Cufflinks: transcript abundance Quantification: Cufflinks implements a linear statistical model to estimate an assignment of abundance to each transcript that explains the observed reads with maximum likelihood. Likelihood Isoform A Isoform B kelihood Differential analysis of gene regulation at transcript resolution with RNA-seq Cole Trapnell 1,2,6, David G Hendrickson 1,2,6, Martin Sauvageau 1,2, Loyal Goff 1 3, John L Rinn 1,2,7 & Lior Pachter 4,5,7 100% Isoform A 100% Isoform B
35 Cufflinks: abundance output Cufflinks reports abundances as Fragments Per Kilobase of exon model per Million mapped fragments (FPKM) C: Number of read pairs (fragments) from transcript N: Total number of mapped read pairs in library L: number of exonic bases for transcript Normalizes for transcript length and lib. size
36 Cuffdiff: differential transcript expression Cudiff Tests for differential expression of a cufflinks assembly Differential analysis of gene regulation at transcript resolution with RNA-seq Cole Trapnell 1,2,6, David G Hendrickson 1,2,6, Martin Sauvageau 1,2, Loyal Goff 1 3, John L Rinn 1,2,7 & Lior Pachter 4,5,7
37 Cufflinks: Alternative splicing trans_id bundle_id chr left right FPKM FMI frac FPKM_conf_lo FPKM_conf_hi coverage length effective_length status ENST chr OK ENST chr OK ENST chr OK ENST chr OK ENST chr OK ENST chr OK ENST chr OK ENST chr OK ENST chr OK ENST chr OK ENST chr OK ENST chr OK ENST chr OK ENST chr OK ENST chr OK ENST chr OK ENST chr OK ENST chr OK
38 R/bioconductor Packages Based on raw read counts per gene/transcript/genome feature (mirna) Differential expression analysis DESeq Negative binomial distribution bayseq Bayesian approach Choice of Poisson and negative binomial distribution edger DEGSeq Genominator
39 Differential Gene Expression SYMBOL logfc PValue FDR counts.c1 counts.c2 counts.kd1 counts.kd2 HNRNPC & E& E& FAIM2 & E& E& AC & E& E& SSC5D & E& E& GGT5 & E& E& EXOC3L4 & E& E& FOXS1 & E& E& AQP5 & E& E& SLC27A3 & E& E& TIMP4 & E& E& Downstream Analyses Pathways/Gene Set (e.g. GOSeq) Regulatory Networks Machine Learning / Classifiers C1 C2 KD1 KD2 SCRN1 IL3RA ENSG LOC QRICH2 CNN2 ZNF684 ENSG PADI3 COL14A1 TSPAN1 PRMT3 PLXNB3 FAIM2 EXOC3L4 ENSG TLE6 SPINK4 VIT ENSG NDUFA4L2 CA9 SLC27A3 SLC23A1 CAMP SSC5D PLAU TIMP4 TNFRSF1A KCNK3 PLEKHA6 BAI2 INHA CFD ENSG BIRC7 AQP5 ANPEP COL16A1 ACAD10 CLCNKB ENSG EVA1A HNRNPC SRCRB4D FOXS1 NFATC4 MAP1A GGT5 ZNF KO C KD
40 Visualization: Genome Viewers Web based: Gbrowse ( UCSC Genome Browser ( Standalone Integrated Genome Viewer (
41 IGV: Differential Expression Visualization
42 Validation (qualitative) 33 of 192 assays shown. Overall validation rate = 85%
43 Comparing Microarray and RNA-Seq Wang, Zhong, Mark Gerstein, and Michael Snyder. "RNA-Seq: a revolutionary tool for transcriptomics." Nature Reviews Genetics 10.1 (2009):
44 Mi az a ChIP-szekvenálás? DNS-Fehérje kölcsönhatások vizsgálata kromatin immunoprecipitációval és NGS szekvenálással A génregulációban kulcsszerepe van a kromatin szerkezetének, a transzkripciós faktorok kötődésének A módszer a kromatin vagy a transzkripciós faktorok DNS-hez kötésén, majd specifikus antitesttel történő kicsapásán alapul. A ChIP-szekvenálás egy új forradalmi technológia, amellyel fehérje-dns interakciókat lehet térképezni genomszinten. ChIP-Seq A kromatin immunoprecipitáció (ChIP) kombinálása az újgenerációs szekvenálással
45 Egy Keresztkötés DNS-törés Kromatin sejt ImmunoPrecipitáció (ChIP) Sejtmag Hídképzés Együttes kötés Hídképzés Szekvenálás és Térképzés 45
46 ChIP-seq (újgenerációs szekvenálás kromatin immunoprecipitáció után) High-throughput sequencing Map sequence tags to genome Release DNA
47 ChIP-Seq analysis pipeline Reads / tags (fastq format) Functional annotation of binding and modification loci PeakAnnotator findpeaks bedgraphs (bed) motif logos metahistograms / metagenes Hypergeometric Optimization of Motif EnRichment Homer maketagdirectory makeucscfile findmotifsgenome annotatepeaks BWA Alignment files (bam) bam Differential Binding Analysis of ChIP- Seq peak data DiffBind edger, limma + GRO-Seq tags bed Clusters, Heatmaps, Plots, Profiles, Venn diagrams Peak lists, bed files Burrows-Wheeler Alignment Tool MACS2 bed bed + etr-s (GRO-Seq) bash, bedtools awk, mergebed intersectbed, awk, sed Overlaps, Peak lists, bed files Model Based Analysis for ChIP-Seq data rpkm-like table intersectbed, awk, sed bed Chipster edger, limma Clusters, Heatmaps, Profiles, Plots, Peak lists, bed files Barta E: Command line analysis of ChIP-seq results. EMBNET JOURNAL 17:(1) pp (2011)
48 A fehérjék DNS-kötése
49 Finding peaks The number of peaks depends on the methods used and the cutoff values applied. More reads doesn t mean necessarily more peaks Different methods give only 60-80% similar peaks! Different programs: HOMER, MACS, FindPeaks, PeakFinds etc. 49
50 MACS
51 HOMER
52 List of HOMER utilities Endre Barta 12/6/16 52
53 Annotation of the peaks (annotatepeaks.pl) Genomic localization Closest TSS Motif occurrences Enrichment in different ontologies Method: Generate a list of genes and compare the list statistically with the list of genes present in a given ontology Barta Endre 12/6/16 53
54 De novo motif finding with HOMER How findmotifsgenome.pl works: 1. Verify peak/bed file 2. Extract sequences from the genome corresponding to the regions in the input file, filtering sequences that are >70% "N 3. Calculate GC/CpG content of peak sequences. 4. Preparse the genomic sequences of the selected size to serve as background sequences 5. Randomly select background regions for motif discovery. 6. Auto normalization of sequence bias. 7. Check enrichment of known motifs 8. de novo motif finding Barta Endre 12/6/16 54
55 HOMER denovo motif finding result } RXR peaks overlapping with GRO-seq paired peaks } Enrichment = % of Targets / % of Background } The P-value depends on the size of the sample (not comparable between different samples) } Best match (HOMER has its own motif library coming from the JASPAR database and from ChIPseq analyses) does not mean perfect match!
56 HOMER known motif enrichment analysis Enrichment = % of targets sequences with Motif / % of Background sequences with motif
57 Downstream analysis Comparing different samples Overlapping regions (intersectbed) Occupancy analysis (diffbind) Generating profiles Re-analyze peak subsets for motif occurrences
58 RXR binding is accompanied by p300 recruitment and the release of PU.1 p300 sites overlapping with RXR PU.1 sites overlapping with RXR 0.5 BMDM_C_p300 BMDM_LG_p BMDM_C_PU1 BMDM_LG_PU1 0.4 Normalized read count Normalized read count Distance from the center of RXR peak (bp) Distance from the center of RXR peak (bp)
59 intersectbed Switches: - -a peakfile1.bed -b peakfile2.bed ((-abam => -bed)) - -u - -v - -c (count b on a) wo (fusing beds in a double bed table) - -f (minimum overlap %) -u -f r (reciprocal overlap) -u -f 0.6 -r - -s (strand specific match) 59
60 Peak overlaps in BMDM A listfile Unit (peak) Group A PU.1 A PU.1 A PU.1 A PU.1 A PU.1 A PU.1 A PU.1 A-1 RXR A-2 RXR A-3 RXR A-4 RXR A-5 RXR A-6 RXR A-7 RXR PU.1 VennMaster New list file format automatically from beds: RXR 280 p300 filename ê Peak (position; merge) Group chr2: PU1peaks chr2: PU1peaks chr2: PU1peaks chr2: PU1peaks chr2: PU1peaks chr2: PU1peaks chr2: PU1peaks 60
61 DiffBind R package
62 GRO-seq Global Run-On (Massive) Sequencing GRO-Seq detects all transcribing RNAs (coding RNAs, ncrnas, enhancer/e-transcripts) in the nuclei from the 5 to the 3 end (strand specific) Theoretically, one tag (read) in the GRO-seq data represents one RNA polymerase molecule RNA polymerase I synthesizes pre-rrna RNA polymerase II synthesizes precursors of mrnas most snrna and mirna RNA polymerase III synthesizes trnas, rrna5s and other small RNAs RNA plymerases IV and V are in plants, but no GRO-seq from plants so far Core et al. Science, 2008
63 Global Run-On sequencing (GRO-seq) Methodology Core et al. Science, 2008
64 Egy Sejtmag Elkötelezett Globális sejt magi izolálás polimerázok Run-On esszé Sejtmag Promóter mrns Enhanszer AAAAA gén térképezés 64
65 GRO-seq analízis pipeline Alapvetően hasonló megközelítés mind az RNA-seq analízisnél 1. A read-ek számolása transzkripteken Az intronokon is lesznek read-ek! A génen lévő enhenszerek (ott külön transzkripció van) torzíthatják az eredményt Hosszú géneknél befolyásolhatja az eredményt a transzkripció sebessége 2. Új transzkriptek keresése (mivel minden transzkripciót mutat) Lehetnek kódoló gének, génváltozatok Lehetnek nem-kódoló RNS-ek (ncrna, lncrna) Enhenszer transzkripció 65
66 GRO-Seq reads in IGV (visualization) Chr9: 62,190 kb 62,230kb ncrna? TSS(Transcription Start Site) Peaks/waves also show RNA polymerase II velocity
67 GRO-seq analysis pipeline Reads / tags (fastq format) maketagdirectory findpeaks makeucscfile (bed) (bedgraph) Alignment files (bam) bed bed (bam) BWA Homer PeakSplitter mergebed, awk, intersectbed, sed bam bed Burrows-Wheeler Alignment Tool Hypergeometric Optimization of Motif EnRichment Subdivision into discrete signal peaks Building transcripts Finding enhancers rpkm-like table, bed Functional annotation of binding and modification loci PeakAnnotator intersectbed, awk, sed Overlaps, Peak lists, bed files + ChIP peaks DiffBind edger, limma Clusters, Heatmaps, Plots, Profiles, Venn diagrams Peak lists, bed files Differential Binding Analysis of transcript and etranscript data Chipster edger, limma Clusters, Heatmaps, Profiles, Plots, Peak lists, bed files
68 Globális magi Run-On esszé Analízis Annotáció Enhanszer transzkripció Promóter / TSS régió A divergens transzkripciós helyek predikciója Transzkript predikció 68
69 Globális magi Run-On esszé Analízis 129kb Annotáció RefSeq TSS 3 túlnyúló vég átfedés X A divergens helyek predikciója Intronikus enhanszerek TSS Intergenikus enhanszerek Transzkript predikció 69
70 Defining transcribed regions overlappin g etranscripti on Not active New transcript Paused, active 3 extended transcriptio n Not paused, Paused, activenon- active Paused, not active 70
71 Transcription activation in real time 30 min 60 min 120 min ~160 kb / h 10,000 bp/min 160 bp/sec
72 GRO-seq, RNA-seq and ChIP-seq shows the transcription start site (TSS) GRO-seq: divergent transcription ChIP-seq: H3K4me3
73 Gének közös promóterrel 73
74 Az egér makrofág naszcens transzkriptóm Egér genom 11.4 % 100 % 2,707.7 Mb 10.7 % Transzkript lefedettség a pozitív szálon Transzkript lefedettség a negatív szálon 11 % awk -F"\t" '{if ($6 == "+") sum += $3-$2} END{print sum}' /molbio/projects/ogrochip/annotation/gro/alltrs.bed ~ % awk -F"\t" '{if ($6 == "-") sum += $3-$2} END{print sum}' /molbio/projects/ogrochip/annotation/gro/alltrs.bed ~ % mergebed -i /molbio/projects/ogrochip/annotation/gro/alltrs.bed awk -F"\t" '{sum += $3-$2} END{print sum}' ~ % Transzkripció mindkét szálon: ~ 0.8 % 74
75 Mi a közös a három funkcionális genomikai módszerben bioinformatikai szempontból Mind a genomi DNS-t (ChIP-seq), mind az RNS-t (RNA-seq), mind a naszcens RNS-t (GRO-seq) bp-os darabokra fragmentáljuk A könyvtárkészítés után hasonló DNS fragmenteket kapunk, amelyeket szekvenáló linkerek, primerek határolnak Egy (ChIP-seq, GRO-seq) vagy mindkét végét (RNA-seq) ezeknek a fragmenteknek megszekvenáljuk A következő lépés mindig a referencia genomra illesztés (kivéve RNA-seq de novo assembly) A ChIP-seq és GRO-seq esetében a cél csak a megszekvenált rövid read-ek pontos genomi localizációjának a meghatározása (elég bp-t meghatározni) Az RNA-seq esetében kiványcsiak lehetünk a különböző splicing variánsokra, ezért célszerű hosszabb szekvenciákat leolvasni a fragmentumok mindkét oldaláról
76 NGS szoftverek az ngsdeb (Klinikai Genomikai Központ) szerveren Programs used for ChIP-seq, GRO-seq and RNA-seq analysis Other programs used for sequence analysis HOMER SAMTOOLS BOWTIE ChIPSEEQER BEDTOOLS BAMTOOLS TOPHAT TRINITYRNASEQ BWA MEME MACS FASTX-TOOLKIT Head Node: 2x6 core, 144GB RAM, 20 Tbyte PICARD disk 6x computing nodes: 2x6 core, 48GB RAM 600GB disk VCF-TOOLS VCFUTILS TABIX EMBOSS BLAST BLAST+ CBUST LASTZ MULTIZ BLAT WEEDER DIALIGN SRMA CLUSTALW GLAM2 76
77 ChIA-PET protocol Wet-lab portion of protocol Dry-lab portion of protocol Linker filtering Guoliang Li et. al Genome Biology PET mapping to reference genome PET clustering 77
78 Analysis pipeline Download data (SRA/ ENCODE) Filter data (300kbp < interactions) Assign CTCF motifs to interactions CTCF peak CT CF INTERACTION 1 INTERACTION 2 CT CF Interaction analysis: Visualization Meta analysis Heatmap analysis Annotation etc CT CF CT CF P-value: 1e-1653 T: % B: 2.86% CT CF INTERACTION 3 CT CF 78
79 Köszönöm a figyelmet!
SOLiD Technology. library preparation & Sequencing Chemistry (sequencing by ligation!) Imaging and analysis. Application specific sample preparation
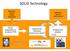 SOLiD Technology Application specific sample preparation Application specific data analysis library preparation & emulsion PCR Sequencing Chemistry (sequencing by ligation!) Imaging and analysis SOLiD
SOLiD Technology Application specific sample preparation Application specific data analysis library preparation & emulsion PCR Sequencing Chemistry (sequencing by ligation!) Imaging and analysis SOLiD
RNS szekvenálás a gyakorlatban január 29. február 2.
 RNS szekvenálás a gyakorlatban 2018. január 29. február 2. DEBRECEN VENTURE CATAPULT MIRŐL SZÓL A RENDEZVÉNY? Az RNS szekvenálás a jelenleg elérhető génexpressziós vizsgálatok között kiemelkedő helyet
RNS szekvenálás a gyakorlatban 2018. január 29. február 2. DEBRECEN VENTURE CATAPULT MIRŐL SZÓL A RENDEZVÉNY? Az RNS szekvenálás a jelenleg elérhető génexpressziós vizsgálatok között kiemelkedő helyet
Mapping Sequencing Reads to a Reference Genome
 Mapping Sequencing Reads to a Reference Genome High Throughput Sequencing RN Example applications: Sequencing a genome (DN) Sequencing a transcriptome and gene expression studies (RN) ChIP (chromatin immunoprecipitation)
Mapping Sequencing Reads to a Reference Genome High Throughput Sequencing RN Example applications: Sequencing a genome (DN) Sequencing a transcriptome and gene expression studies (RN) ChIP (chromatin immunoprecipitation)
discosnp demo - Peterlongo Pierre 1 DISCOSNP++: Live demo
 discosnp demo - Peterlongo Pierre 1 DISCOSNP++: Live demo Download and install discosnp demo - Peterlongo Pierre 3 Download web page: github.com/gatb/discosnp Chose latest release (2.2.10 today) discosnp
discosnp demo - Peterlongo Pierre 1 DISCOSNP++: Live demo Download and install discosnp demo - Peterlongo Pierre 3 Download web page: github.com/gatb/discosnp Chose latest release (2.2.10 today) discosnp
Supplementary Figure 1
 Supplementary Figure 1 Plot of delta-afe of sequence variants detected between resistant and susceptible pool over the genome sequence of the WB42 line of B. vulgaris ssp. maritima. The delta-afe values
Supplementary Figure 1 Plot of delta-afe of sequence variants detected between resistant and susceptible pool over the genome sequence of the WB42 line of B. vulgaris ssp. maritima. The delta-afe values
Supplementary materials to: Whole-mount single molecule FISH method for zebrafish embryo
 Supplementary materials to: Whole-mount single molecule FISH method for zebrafish embryo Yuma Oka and Thomas N. Sato Supplementary Figure S1. Whole-mount smfish with and without the methanol pretreatment.
Supplementary materials to: Whole-mount single molecule FISH method for zebrafish embryo Yuma Oka and Thomas N. Sato Supplementary Figure S1. Whole-mount smfish with and without the methanol pretreatment.
Phenotype. Genotype. It is like any other experiment! What is a bioinformatics experiment? Remember the Goal. Infectious Disease Paradigm
 It is like any other experiment! What is a bioinformatics experiment? You need to know your data/input sources You need to understand your methods and their assumptions You need a plan to get from point
It is like any other experiment! What is a bioinformatics experiment? You need to know your data/input sources You need to understand your methods and their assumptions You need a plan to get from point
Nan Wang, Qingming Dong, Jingjing Li, Rohit K. Jangra, Meiyun Fan, Allan R. Brasier, Stanley M. Lemon, Lawrence M. Pfeffer, Kui Li
 Supplemental Material IRF3-dependent and NF- B-independent viral induction of the zinc-finger antiviral protein Nan Wang, Qingming Dong, Jingjing Li, Rohit K. Jangra, Meiyun Fan, Allan R. Brasier, Stanley
Supplemental Material IRF3-dependent and NF- B-independent viral induction of the zinc-finger antiviral protein Nan Wang, Qingming Dong, Jingjing Li, Rohit K. Jangra, Meiyun Fan, Allan R. Brasier, Stanley
Flowering time. Col C24 Cvi C24xCol C24xCvi ColxCvi
 Flowering time Rosette leaf number 50 45 40 35 30 25 20 15 10 5 0 Col C24 Cvi C24xCol C24xCvi ColxCvi Figure S1. Flowering time in three F 1 hybrids and their parental lines as measured by leaf number
Flowering time Rosette leaf number 50 45 40 35 30 25 20 15 10 5 0 Col C24 Cvi C24xCol C24xCvi ColxCvi Figure S1. Flowering time in three F 1 hybrids and their parental lines as measured by leaf number
Gerinces és növényi ortológ promóter adatbázisok fejlesztése és elemzése. Eötvös Loránd Tudományegyetem Természettudományi Kar Biológia Doktori Iskola
 Doktori értekezés tézisei Gerinces és növényi ortológ promóter adatbázisok fejlesztése és elemzése Sebestyén Endre Eötvös Loránd Tudományegyetem Természettudományi Kar Biológia Doktori Iskola Vezetője:
Doktori értekezés tézisei Gerinces és növényi ortológ promóter adatbázisok fejlesztése és elemzése Sebestyén Endre Eötvös Loránd Tudományegyetem Természettudományi Kar Biológia Doktori Iskola Vezetője:
Supporting Information
 Supporting Information Cell-free GFP simulations Cell-free simulations of degfp production were consistent with experimental measurements (Fig. S1). Dual emmission GFP was produced under a P70a promoter
Supporting Information Cell-free GFP simulations Cell-free simulations of degfp production were consistent with experimental measurements (Fig. S1). Dual emmission GFP was produced under a P70a promoter
Cserző Miklós Bioinformatika és genomanalízis az orvostudományban. Integrált biológiai adatbázisok
 Bioinformatika és genomanalízis az orvostudományban Integrált biológiai adatbázisok Cserző Miklós 2018 A mai előadás A genom annotálás jelentősége Genome Reference Consortium Gene Ontology Az ensembl pipeline
Bioinformatika és genomanalízis az orvostudományban Integrált biológiai adatbázisok Cserző Miklós 2018 A mai előadás A genom annotálás jelentősége Genome Reference Consortium Gene Ontology Az ensembl pipeline
Forensic SNP Genotyping using Nanopore MinION Sequencing
 Forensic SNP Genotyping using Nanopore MinION Sequencing AUTHORS Senne Cornelis 1, Yannick Gansemans 1, Lieselot Deleye 1, Dieter Deforce 1,#,Filip Van Nieuwerburgh 1,#,* 1 Laboratory of Pharmaceutical
Forensic SNP Genotyping using Nanopore MinION Sequencing AUTHORS Senne Cornelis 1, Yannick Gansemans 1, Lieselot Deleye 1, Dieter Deforce 1,#,Filip Van Nieuwerburgh 1,#,* 1 Laboratory of Pharmaceutical
Trinucleotide Repeat Diseases: CRISPR Cas9 PacBio no PCR Sequencing MFMER slide-1
 Trinucleotide Repeat Diseases: CRISPR Cas9 PacBio no PCR Sequencing 2015 MFMER slide-1 Fuch s Eye Disease TCF 4 gene Fuchs occurs in about 4% of the US population. Leads to deteriorating vision without
Trinucleotide Repeat Diseases: CRISPR Cas9 PacBio no PCR Sequencing 2015 MFMER slide-1 Fuch s Eye Disease TCF 4 gene Fuchs occurs in about 4% of the US population. Leads to deteriorating vision without
ADATBÁNYÁSZAT I. ÉS OMICS
 Az élettudományi-klinikai felsőoktatás gyakorlatorientált és hallgatóbarát korszerűsítése a vidéki képzőhelyek nemzetközi versenyképességének erősítésére TÁMOP-4.1.1.C-13/1/KONV-2014-0001 ADATBÁNYÁSZAT
Az élettudományi-klinikai felsőoktatás gyakorlatorientált és hallgatóbarát korszerűsítése a vidéki képzőhelyek nemzetközi versenyképességének erősítésére TÁMOP-4.1.1.C-13/1/KONV-2014-0001 ADATBÁNYÁSZAT
Genomadatbázisok Ld. Entrez Genome: Összes ismert genom, hierarchikus szervezésben (kromoszóma, térképek, gének, stb.)
 Genomika Új korszak, paradigmaváltás, forradalom: a teljes genomok ismeretében a biológia adatokban gazdag tudománnyá válik. Új kutatási módszerek, új szemlélet. Hajtóerõk: Genomszekvenálási projektek
Genomika Új korszak, paradigmaváltás, forradalom: a teljes genomok ismeretében a biológia adatokban gazdag tudománnyá válik. Új kutatási módszerek, új szemlélet. Hajtóerõk: Genomszekvenálási projektek
Juhász Angéla MTA ATK MI Alkalmazott Genomikai Osztály SZEKVENCIA ADATBÁZISOK
 Juhász Angéla MTA ATK MI Alkalmazott Genomikai Osztály SZEKVENCIA ADATBÁZISOK Fehérjét kódol? Tulajdonságai? -Hol lokalizálódik? -Oldható? -3D szerkezete? -Accession #? -Annotációja elérhető? Már benne
Juhász Angéla MTA ATK MI Alkalmazott Genomikai Osztály SZEKVENCIA ADATBÁZISOK Fehérjét kódol? Tulajdonságai? -Hol lokalizálódik? -Oldható? -3D szerkezete? -Accession #? -Annotációja elérhető? Már benne
13. RNS szintézis és splicing
 13. RNS szintézis és splicing 1 Visszatekintés: Az RNS típusai és szerkezete Hírvivő RNS = mrns (messenger RNA = mrna) : fehérjeszintézis pre-mrns érett mrns (intronok kivágódnak = splicing) Transzfer
13. RNS szintézis és splicing 1 Visszatekintés: Az RNS típusai és szerkezete Hírvivő RNS = mrns (messenger RNA = mrna) : fehérjeszintézis pre-mrns érett mrns (intronok kivágódnak = splicing) Transzfer
10. Genomika 2. Microarrayek és típusaik
 10. Genomika 2. 1. Microarray technikák és bioinformatikai vonatkozásaik Microarrayek és típusaik Korrelált génexpresszió mint a funkcionális genomika eszköze 2. Kombinált megközelítés a funkcionális genomikában
10. Genomika 2. 1. Microarray technikák és bioinformatikai vonatkozásaik Microarrayek és típusaik Korrelált génexpresszió mint a funkcionális genomika eszköze 2. Kombinált megközelítés a funkcionális genomikában
Statistical Inference
 Petra Petrovics Statistical Inference 1 st lecture Descriptive Statistics Inferential - it is concerned only with collecting and describing data Population - it is used when tentative conclusions about
Petra Petrovics Statistical Inference 1 st lecture Descriptive Statistics Inferential - it is concerned only with collecting and describing data Population - it is used when tentative conclusions about
Bioinformatics: Blending. Biology and Computer Science
 Bioinformatics: Blending Biology and Computer Science MDNMSITNTPTSNDACLSIVHSLMCHRQ GGESETFAKRAIESLVKKLKEKKDELDSL ITAITTNGAHPSKCVTIQRTLDGRLQVAG RKGFPHVIYARLWRWPDLHKNELKHVK YCQYAFDLKCDSVCVNPYHYERVVSPGI DLSGLTLQSNAPSSMMVKDEYVHDFEG
Bioinformatics: Blending Biology and Computer Science MDNMSITNTPTSNDACLSIVHSLMCHRQ GGESETFAKRAIESLVKKLKEKKDELDSL ITAITTNGAHPSKCVTIQRTLDGRLQVAG RKGFPHVIYARLWRWPDLHKNELKHVK YCQYAFDLKCDSVCVNPYHYERVVSPGI DLSGLTLQSNAPSSMMVKDEYVHDFEG
Supporting Information
 Supporting Information Paired design of dcas9 as a systematic platform for the detection of featured nucleic acid sequences in pathogenic strains Yihao Zhang 1,2,8, Long Qian 4,8, Weijia Wei 1,3,7,8, Yu
Supporting Information Paired design of dcas9 as a systematic platform for the detection of featured nucleic acid sequences in pathogenic strains Yihao Zhang 1,2,8, Long Qian 4,8, Weijia Wei 1,3,7,8, Yu
Orvosi Genomtudomány 2014 Medical Genomics 2014. Április 8 Május 22 8th April 22nd May
 Orvosi Genomtudomány 2014 Medical Genomics 2014 Április 8 Május 22 8th April 22nd May Hét / 1st week (9. kalendariumi het) Takács László / Fehér Zsigmond Magyar kurzus Datum/ido Ápr. 8 Apr. 9 10:00 10:45
Orvosi Genomtudomány 2014 Medical Genomics 2014 Április 8 Május 22 8th April 22nd May Hét / 1st week (9. kalendariumi het) Takács László / Fehér Zsigmond Magyar kurzus Datum/ido Ápr. 8 Apr. 9 10:00 10:45
The nontrivial extraction of implicit, previously unknown, and potentially useful information from data.
 Budapesti Műszaki és Gazdaságtudományi Egyetem Méréstechnika és Információs rendszerek Tanszék Adatelemzés intelligens módszerekkel Hullám Gábor Adatelemzés hagyományos megközelítésben I. Megválaszolandó
Budapesti Műszaki és Gazdaságtudományi Egyetem Méréstechnika és Információs rendszerek Tanszék Adatelemzés intelligens módszerekkel Hullám Gábor Adatelemzés hagyományos megközelítésben I. Megválaszolandó
Expressziós microarray. Dr. Győrffy Balázs
 Expressziós microarray Dr. Győrffy Balázs Áttekintő 1. A technológia 2. Feldolgozás 2.1. Minőség-ellenőrzés 2.2. Jellemző kiválasztás 2.3. Vizualizálás 2.4. Kereszt-elemzés 2.5. Online diagnosztika 3.
Expressziós microarray Dr. Győrffy Balázs Áttekintő 1. A technológia 2. Feldolgozás 2.1. Minőség-ellenőrzés 2.2. Jellemző kiválasztás 2.3. Vizualizálás 2.4. Kereszt-elemzés 2.5. Online diagnosztika 3.
Expression analysis of PIN genes in root tips and nodules of Lotus japonicus
 Article Expression analysis of PIN genes in root tips and nodules of Lotus japonicus Izabela Sańko-Sawczenko 1, Dominika Dmitruk 1, Barbara Łotocka 1, Elżbieta Różańska 1 and Weronika Czarnocka 1, * 1
Article Expression analysis of PIN genes in root tips and nodules of Lotus japonicus Izabela Sańko-Sawczenko 1, Dominika Dmitruk 1, Barbara Łotocka 1, Elżbieta Różańska 1 and Weronika Czarnocka 1, * 1
Genome 373: Hidden Markov Models I. Doug Fowler
 Genome 373: Hidden Markov Models I Doug Fowler Review From Gene Prediction I transcriptional start site G open reading frame transcriptional termination site promoter 5 untranslated region 3 untranslated
Genome 373: Hidden Markov Models I Doug Fowler Review From Gene Prediction I transcriptional start site G open reading frame transcriptional termination site promoter 5 untranslated region 3 untranslated
Limitations and challenges of genetic barcode quantification
 Limitations and challenges of genetic barcode quantification Lars hielecke, im ranyossy, ndreas Dahl, Rajiv iwari, Ingo Roeder, Hartmut eiger, Boris Fehse, Ingmar lauche and Kerstin ornils SUPPLEMENRY
Limitations and challenges of genetic barcode quantification Lars hielecke, im ranyossy, ndreas Dahl, Rajiv iwari, Ingo Roeder, Hartmut eiger, Boris Fehse, Ingmar lauche and Kerstin ornils SUPPLEMENRY
Crash Course in Omics Terminology, Concepts & Data Types
 Crash Course in Omics Terminology, Concepts & Data Types 12 th Annual EuPathDB Workshop: 2017 Jessie Kissinger June 18, 2017 FungiDB MicrosporidiaDB AmoebaDB PlasmoDB ToxoDB CryptoDB PiroplasmaDB TrichDB
Crash Course in Omics Terminology, Concepts & Data Types 12 th Annual EuPathDB Workshop: 2017 Jessie Kissinger June 18, 2017 FungiDB MicrosporidiaDB AmoebaDB PlasmoDB ToxoDB CryptoDB PiroplasmaDB TrichDB
TÉMAKÖRÖK. Ősi RNS világ BEVEZETÉS. RNS-ek tradicionális szerepben
 esirna mirtron BEVEZETÉS TÉMAKÖRÖK Ősi RNS világ RNS-ek tradicionális szerepben bevezetés BIOLÓGIAI MOLEKULÁK FEHÉRJÉK NUKLEINSAVAK DNS-ek RNS-ek BIOLÓGIAI MOLEKULÁK FEHÉRJÉK NUKLEINSAVAK DNS-ek RNS-ek
esirna mirtron BEVEZETÉS TÉMAKÖRÖK Ősi RNS világ RNS-ek tradicionális szerepben bevezetés BIOLÓGIAI MOLEKULÁK FEHÉRJÉK NUKLEINSAVAK DNS-ek RNS-ek BIOLÓGIAI MOLEKULÁK FEHÉRJÉK NUKLEINSAVAK DNS-ek RNS-ek
Correlation & Linear Regression in SPSS
 Petra Petrovics Correlation & Linear Regression in SPSS 4 th seminar Types of dependence association between two nominal data mixed between a nominal and a ratio data correlation among ratio data Correlation
Petra Petrovics Correlation & Linear Regression in SPSS 4 th seminar Types of dependence association between two nominal data mixed between a nominal and a ratio data correlation among ratio data Correlation
Performance Modeling of Intelligent Car Parking Systems
 Performance Modeling of Intelligent Car Parking Systems Károly Farkas Gábor Horváth András Mészáros Miklós Telek Technical University of Budapest, Hungary EPEW 2014, Florence, Italy Outline Intelligent
Performance Modeling of Intelligent Car Parking Systems Károly Farkas Gábor Horváth András Mészáros Miklós Telek Technical University of Budapest, Hungary EPEW 2014, Florence, Italy Outline Intelligent
A genomikai oktatás helyzete a Debreceni Egyetemen
 A genomikai oktatás helyzete a Debreceni Egyetemen Bálint Bálint L. GNTP Oktatás és Tudásmenedzsment Munkabizottság, 2009. június 10. Tények Debreceni Egyetemről 21000 nappali és 33000 összes hallgató
A genomikai oktatás helyzete a Debreceni Egyetemen Bálint Bálint L. GNTP Oktatás és Tudásmenedzsment Munkabizottság, 2009. június 10. Tények Debreceni Egyetemről 21000 nappali és 33000 összes hallgató
Klaszterezés, 2. rész
 Klaszterezés, 2. rész Csima Judit BME, VIK, Számítástudományi és Információelméleti Tanszék 208. április 6. Csima Judit Klaszterezés, 2. rész / 29 Hierarchikus klaszterezés egymásba ágyazott klasztereket
Klaszterezés, 2. rész Csima Judit BME, VIK, Számítástudományi és Információelméleti Tanszék 208. április 6. Csima Judit Klaszterezés, 2. rész / 29 Hierarchikus klaszterezés egymásba ágyazott klasztereket
Biomassza alapú bioalkohol előállítási technológia fejlesztése metagenomikai eljárással
 Biomassza alapú bioalkohol előállítási technológia fejlesztése metagenomikai eljárással Kovács Zoltán ügyvezető DEKUT Debreceni Kutatásfejlesztési Közhasznú Nonprofit Kft. Problémadefiníció Első generációs
Biomassza alapú bioalkohol előállítási technológia fejlesztése metagenomikai eljárással Kovács Zoltán ügyvezető DEKUT Debreceni Kutatásfejlesztési Közhasznú Nonprofit Kft. Problémadefiníció Első generációs
A HUMÁN GENOM PROJEKT Sasvári-Székely Mária* Semmelweis Egyetem, Orvosi Vegytani, Molekuláris Biológiai és Pathobiokémiai Intézet
 A HUMÁN GENOM PROJEKT Sasvári-Székely Mária* Semmelweis Egyetem, Orvosi Vegytani, Molekuláris Biológiai és Pathobiokémiai Intézet *Levelezési cím: Dr. Sasvári-Székely Mária, Semmelweis Egyetem, Orvosi
A HUMÁN GENOM PROJEKT Sasvári-Székely Mária* Semmelweis Egyetem, Orvosi Vegytani, Molekuláris Biológiai és Pathobiokémiai Intézet *Levelezési cím: Dr. Sasvári-Székely Mária, Semmelweis Egyetem, Orvosi
Create & validate a signature
 IOTA TUTORIAL 7 Create & validate a signature v.0.0 KNBJDBIRYCUGVWMSKPVA9KOOGKKIRCBYHLMUTLGGAV9LIIPZSBGIENVBQ9NBQWXOXQSJRIRBHYJ9LCTJLISGGBRFRTTWD ABBYUVKPYFDJWTFLICYQQWQVDPCAKNVMSQERSYDPSSXPCZLVKWYKYZMREAEYZOSPWEJLHHFPYGSNSUYRZXANDNQTTLLZA
IOTA TUTORIAL 7 Create & validate a signature v.0.0 KNBJDBIRYCUGVWMSKPVA9KOOGKKIRCBYHLMUTLGGAV9LIIPZSBGIENVBQ9NBQWXOXQSJRIRBHYJ9LCTJLISGGBRFRTTWD ABBYUVKPYFDJWTFLICYQQWQVDPCAKNVMSQERSYDPSSXPCZLVKWYKYZMREAEYZOSPWEJLHHFPYGSNSUYRZXANDNQTTLLZA
Miben különbözünk az egértől? Szabályozás a molekuláris biológiában
 Az atomoktól a csillagokig, 2010. október 28., ELTE Fizikai Intézet Miben különbözünk az egértől? Szabályozás a molekuláris biológiában brainmaps.org Homo sapiens (Miroslav Klose) Mus musculus Farkas Illés
Az atomoktól a csillagokig, 2010. október 28., ELTE Fizikai Intézet Miben különbözünk az egértől? Szabályozás a molekuláris biológiában brainmaps.org Homo sapiens (Miroslav Klose) Mus musculus Farkas Illés
Crash Course in Omics Terminology, Concepts & Data Types
 Crash Course in Omics Terminology, Concepts & Data Types 13 th Annual EuPathDB Workshop Jessie Kissinger June 17th, 2018 FungiDB MicrosporidiaDB AmoebaDB PlasmoDB ToxoDB CryptoDB PiroplasmaDB TrichDB GiardiaDB
Crash Course in Omics Terminology, Concepts & Data Types 13 th Annual EuPathDB Workshop Jessie Kissinger June 17th, 2018 FungiDB MicrosporidiaDB AmoebaDB PlasmoDB ToxoDB CryptoDB PiroplasmaDB TrichDB GiardiaDB
Markerless Escherichia coli rrn Deletion Strains for Genetic Determination of Ribosomal Binding Sites
 Markerless Escherichia coli rrn Deletion Strains for Genetic Determination of Ribosomal Binding Sites Selwyn Quan *, Ole Skovgaard, Robert E. McLaughlin, Ed T. Buurman,1, and Catherine L. Squires * Department
Markerless Escherichia coli rrn Deletion Strains for Genetic Determination of Ribosomal Binding Sites Selwyn Quan *, Ole Skovgaard, Robert E. McLaughlin, Ed T. Buurman,1, and Catherine L. Squires * Department
Választási modellek 3
 Választási modellek 3 Prileszky István Doktori Iskola 2018 http://www.sze.hu/~prile Forrás: A Self Instructing Course in Mode Choice Modeling: Multinomial and Nested Logit Models Prepared For U.S. Department
Választási modellek 3 Prileszky István Doktori Iskola 2018 http://www.sze.hu/~prile Forrás: A Self Instructing Course in Mode Choice Modeling: Multinomial and Nested Logit Models Prepared For U.S. Department
GENOMIKA TÖBBFÉLE MAKROMOLEKULA VIZSGÁLATA EGYIDŐBEN
 GENOMIKA TÖBBFÉLE MAKROMOLEKULA VIZSGÁLATA EGYIDŐBEN Strukturális genomika Genomkönyvtárak DNS szekvenálás Genom programok Polimorfizmusok RFLP DNS könyvtár készítés humán genom 1. Emésztés RE-kal Emberi
GENOMIKA TÖBBFÉLE MAKROMOLEKULA VIZSGÁLATA EGYIDŐBEN Strukturális genomika Genomkönyvtárak DNS szekvenálás Genom programok Polimorfizmusok RFLP DNS könyvtár készítés humán genom 1. Emésztés RE-kal Emberi
Biológus MSc. Molekuláris biológiai alapismeretek
 Biológus MSc Molekuláris biológiai alapismeretek A nukleotidok építőkövei A nukleotidok szerkezete Nukleotid = N-tartalmú szerves bázis + pentóz + foszfát N-glikozidos kötés 5 1 4 2 3 (Foszfát)észter-kötés
Biológus MSc Molekuláris biológiai alapismeretek A nukleotidok építőkövei A nukleotidok szerkezete Nukleotid = N-tartalmú szerves bázis + pentóz + foszfát N-glikozidos kötés 5 1 4 2 3 (Foszfát)észter-kötés
Molekuláris genetikai vizsgáló. módszerek az immundefektusok. diagnosztikájában
 Molekuláris genetikai vizsgáló módszerek az immundefektusok diagnosztikájában Primer immundefektusok A primer immundeficiencia ritka, veleszületett, monogénes öröklődésű immunhiányos állapot. Családi halmozódást
Molekuláris genetikai vizsgáló módszerek az immundefektusok diagnosztikájában Primer immundefektusok A primer immundeficiencia ritka, veleszületett, monogénes öröklődésű immunhiányos állapot. Családi halmozódást
Crash Course in Omics Terminology, Concepts & Data Types
 Crash Course in Omics Terminology, Concepts & Data Types 14 th Annual EuPathDB Workshop Jessie Kissinger June 2, 2019 FungiDB MicrosporidiaDB AmoebaDB PlasmoDB ToxoDB CryptoDB PiroplasmaDB TrichDB GiardiaDB
Crash Course in Omics Terminology, Concepts & Data Types 14 th Annual EuPathDB Workshop Jessie Kissinger June 2, 2019 FungiDB MicrosporidiaDB AmoebaDB PlasmoDB ToxoDB CryptoDB PiroplasmaDB TrichDB GiardiaDB
Széchenyi István Egyetem www.sze.hu/~herno
 Oldal: 1/6 A feladat során megismerkedünk a C# és a LabVIEW összekapcsolásának egy lehetőségével, pontosabban nagyon egyszerű C#- ban írt kódból fordítunk DLL-t, amit meghívunk LabVIEW-ból. Az eljárás
Oldal: 1/6 A feladat során megismerkedünk a C# és a LabVIEW összekapcsolásának egy lehetőségével, pontosabban nagyon egyszerű C#- ban írt kódból fordítunk DLL-t, amit meghívunk LabVIEW-ból. Az eljárás
Új temékek az UD-GenoMed Kft. kínálatában!
 Új temékek az UD-GenMed Kft. kínálatában! Műanyag termékek: SARSTEDT műanyag termékek teljes választéka Egyszer használats labratóriumi műanyag eszközök szövet és sejttenyésztéshez Vérvételi és diagnsztikai
Új temékek az UD-GenMed Kft. kínálatában! Műanyag termékek: SARSTEDT műanyag termékek teljes választéka Egyszer használats labratóriumi műanyag eszközök szövet és sejttenyésztéshez Vérvételi és diagnsztikai
A tárgy címe: Bioinformatika
 A tárgy címe: Bioinformatika Kötelezően választható tárgy IV. és V. évfolyamos biológus hallgatók számára; heti 2+3 óra Előkövetelmény: Biokémia főkollégium; genetika főkollégium; alapszintű számítógépes
A tárgy címe: Bioinformatika Kötelezően választható tárgy IV. és V. évfolyamos biológus hallgatók számára; heti 2+3 óra Előkövetelmény: Biokémia főkollégium; genetika főkollégium; alapszintű számítógépes
Cluster Analysis. Potyó László
 Cluster Analysis Potyó László What is Cluster Analysis? Cluster: a collection of data objects Similar to one another within the same cluster Dissimilar to the objects in other clusters Cluster analysis
Cluster Analysis Potyó László What is Cluster Analysis? Cluster: a collection of data objects Similar to one another within the same cluster Dissimilar to the objects in other clusters Cluster analysis
Miskolci Egyetem Gazdaságtudományi Kar Üzleti Információgazdálkodási és Módszertani Intézet Nonparametric Tests
 Nonparametric Tests Petra Petrovics Hypothesis Testing Parametric Tests Mean of a population Population proportion Population Standard Deviation Nonparametric Tests Test for Independence Analysis of Variance
Nonparametric Tests Petra Petrovics Hypothesis Testing Parametric Tests Mean of a population Population proportion Population Standard Deviation Nonparametric Tests Test for Independence Analysis of Variance
Miskolci Egyetem Gazdaságtudományi Kar Üzleti Információgazdálkodási és Módszertani Intézet Factor Analysis
 Factor Analysis Factor analysis is a multiple statistical method, which analyzes the correlation relation between data, and it is for data reduction, dimension reduction and to explore the structure. Aim
Factor Analysis Factor analysis is a multiple statistical method, which analyzes the correlation relation between data, and it is for data reduction, dimension reduction and to explore the structure. Aim
Modular Optimization of Hemicellulose-utilizing Pathway in. Corynebacterium glutamicum for Consolidated Bioprocessing of
 [Supplementary materials] Modular Optimization of Hemicellulose-utilizing Pathway in Corynebacterium glutamicum for Consolidated Bioprocessing of Hemicellulosic Biomass Sung Sun Yim 1, Jae Woong Choi 1,
[Supplementary materials] Modular Optimization of Hemicellulose-utilizing Pathway in Corynebacterium glutamicum for Consolidated Bioprocessing of Hemicellulosic Biomass Sung Sun Yim 1, Jae Woong Choi 1,
ÚJ GENERÁCIÓS SZEKVENÁLÁS
 VÍZMIKROBIOLÓGUSOK XI. ORSZÁGOS KONFERENCIÁJA - 2012 ÚJ GENERÁCIÓS SZEKVENÁLÁS LEHETŐSÉG, VAGY NEHÉZSÉG? MÁRIALIGETI KÁROLY EÖTVÖS LORÁND TUDOMÁNYEGYETEM, BUDAPEST 1953 JAMES D. WATSON, FRANCIS CRICK:
VÍZMIKROBIOLÓGUSOK XI. ORSZÁGOS KONFERENCIÁJA - 2012 ÚJ GENERÁCIÓS SZEKVENÁLÁS LEHETŐSÉG, VAGY NEHÉZSÉG? MÁRIALIGETI KÁROLY EÖTVÖS LORÁND TUDOMÁNYEGYETEM, BUDAPEST 1953 JAMES D. WATSON, FRANCIS CRICK:
Supplementary Table 1. Cystometric parameters in sham-operated wild type and Trpv4 -/- rats during saline infusion and
 WT sham Trpv4 -/- sham Saline 10µM GSK1016709A P value Saline 10µM GSK1016709A P value Number 10 10 8 8 Intercontractile interval (sec) 143 (102 155) 98.4 (71.4 148) 0.01 96 (92 121) 109 (95 123) 0.3 Voided
WT sham Trpv4 -/- sham Saline 10µM GSK1016709A P value Saline 10µM GSK1016709A P value Number 10 10 8 8 Intercontractile interval (sec) 143 (102 155) 98.4 (71.4 148) 0.01 96 (92 121) 109 (95 123) 0.3 Voided
Statisztikai hipotézisvizsgálatok. Paraméteres statisztikai próbák
 Statisztikai hipotézisvizsgálatok Paraméteres statisztikai próbák 1. Magyarországon a lakosság élelmiszerre fordított kiadásainak 2000-ben átlagosan 140 ezer Ft/fő volt. Egy kérdőíves felmérés során Veszprém
Statisztikai hipotézisvizsgálatok Paraméteres statisztikai próbák 1. Magyarországon a lakosság élelmiszerre fordított kiadásainak 2000-ben átlagosan 140 ezer Ft/fő volt. Egy kérdőíves felmérés során Veszprém
AZ ALACSONY HŐMÉRSÉKLET HATÁSÁRA BEKÖVETKEZŐ REDOX ÉS GÉNEXPRESSZIÓS VÁLTOZÁSOK GABONAFÉLÉKBEN
 A martonvásári agrárkutatások hatodik évtizede AZ ALAONY HŐMÉRSÉKLET HATÁSÁRA BEKÖVETKEZŐ REDOX ÉS GÉNEXPRESSZIÓS VÁLTOZÁSOK GABONAFÉLÉKBEN KOY GÁBOR, VÁGÚJFALVI ATTILA, TÓTH BALÁZS, SZALAI GABRIELLA,
A martonvásári agrárkutatások hatodik évtizede AZ ALAONY HŐMÉRSÉKLET HATÁSÁRA BEKÖVETKEZŐ REDOX ÉS GÉNEXPRESSZIÓS VÁLTOZÁSOK GABONAFÉLÉKBEN KOY GÁBOR, VÁGÚJFALVI ATTILA, TÓTH BALÁZS, SZALAI GABRIELLA,
Gyakorlati bioinformatika
 Gyakorlati bioinformatika Szekvenciaillesztés PhD kurzus 2. Szekvenciaillesztés Bagossi Péter Fajtái: - egyszer ill. többszörös illesztés - globális ill. lokális illesztés Alkalmazása: - adatbázisokban
Gyakorlati bioinformatika Szekvenciaillesztés PhD kurzus 2. Szekvenciaillesztés Bagossi Péter Fajtái: - egyszer ill. többszörös illesztés - globális ill. lokális illesztés Alkalmazása: - adatbázisokban
A modern e-learning lehetőségei a tűzoltók oktatásának fejlesztésében. Dicse Jenő üzletfejlesztési igazgató
 A modern e-learning lehetőségei a tűzoltók oktatásának fejlesztésében Dicse Jenő üzletfejlesztési igazgató How to apply modern e-learning to improve the training of firefighters Jenő Dicse Director of
A modern e-learning lehetőségei a tűzoltók oktatásának fejlesztésében Dicse Jenő üzletfejlesztési igazgató How to apply modern e-learning to improve the training of firefighters Jenő Dicse Director of
Operációs rendszerek. UNIX fájlrendszer
 Operációs rendszerek UNIX fájlrendszer UNIX fájlrendszer Alapegység: a file, amelyet byte-folyamként kezel. Soros (szekvenciális) elérés. Transzparens (átlátszó) file-szerkezet. Link-ek (kapcsolatok) létrehozásának
Operációs rendszerek UNIX fájlrendszer UNIX fájlrendszer Alapegység: a file, amelyet byte-folyamként kezel. Soros (szekvenciális) elérés. Transzparens (átlátszó) file-szerkezet. Link-ek (kapcsolatok) létrehozásának
Génkifejeződési vizsgálatok. Kocsy Gábor
 Génkifejeződési vizsgálatok MTA Mezőgazdasági Kutatóintézete Növényi Molekuláris Biológia Osztály A génkifejeződés A sejtmag géneket tartalmaz; (fehérjéket, RNSeket kódoló); A gének átíródnak mrns; Pre-mRNS
Génkifejeződési vizsgálatok MTA Mezőgazdasági Kutatóintézete Növényi Molekuláris Biológia Osztály A génkifejeződés A sejtmag géneket tartalmaz; (fehérjéket, RNSeket kódoló); A gének átíródnak mrns; Pre-mRNS
Humán genom variációk single nucleotide polymorphism (SNP)
 Humán genom variációk single nucleotide polymorphism (SNP) A genom ~ 97 %-a két különböző egyedben teljesen azonos ~ 1% különbség: SNP miatt ~2% különbség: kópiaszámbeli eltérés, deléciók miatt 11-12 millió
Humán genom variációk single nucleotide polymorphism (SNP) A genom ~ 97 %-a két különböző egyedben teljesen azonos ~ 1% különbség: SNP miatt ~2% különbség: kópiaszámbeli eltérés, deléciók miatt 11-12 millió
Semmelweis Egyetem / Élettani Intézet / Budapest. Bioinformatika és genomanalízis az orvostudományban. Bevezetés. Cserző Miklós 2018
 Bioinformatika és genomanalízis az orvostudományban Bevezetés Cserző Miklós 2018 A mai előadás A kurzus menete Hol találkozunk bioinformatikával Mi a bioinformatika Miért van bioinformatika A számítógépekről
Bioinformatika és genomanalízis az orvostudományban Bevezetés Cserző Miklós 2018 A mai előadás A kurzus menete Hol találkozunk bioinformatikával Mi a bioinformatika Miért van bioinformatika A számítógépekről
FÖLDRAJZ ANGOL NYELVEN GEOGRAPHY
 Földrajz angol nyelven középszint 0623 ÉRETTSÉGI VIZSGA 2007. május 15. FÖLDRAJZ ANGOL NYELVEN GEOGRAPHY KÖZÉPSZINTŰ ÍRÁSBELI ÉRETTSÉGI VIZSGA INTERMEDIATE LEVEL WRITTEN EXAM JAVÍTÁSI-ÉRTÉKELÉSI ÚTMUTATÓ
Földrajz angol nyelven középszint 0623 ÉRETTSÉGI VIZSGA 2007. május 15. FÖLDRAJZ ANGOL NYELVEN GEOGRAPHY KÖZÉPSZINTŰ ÍRÁSBELI ÉRETTSÉGI VIZSGA INTERMEDIATE LEVEL WRITTEN EXAM JAVÍTÁSI-ÉRTÉKELÉSI ÚTMUTATÓ
Fehérje expressziós rendszerek. Gyógyszerészi Biotechnológia
 Fehérje expressziós rendszerek Gyógyszerészi Biotechnológia Expressziós rendszerek Cél: rekombináns fehérjék előállítása nagy tisztaságban és nagy mennyiségben kísérleti ill. gyakorlati (therapia) felhasználásokra
Fehérje expressziós rendszerek Gyógyszerészi Biotechnológia Expressziós rendszerek Cél: rekombináns fehérjék előállítása nagy tisztaságban és nagy mennyiségben kísérleti ill. gyakorlati (therapia) felhasználásokra
Ph.D. értekezés tézisei. Póka Nándor. Biokémia, Biofizika, Molekuláris és sejtbiológia doktori program
 A Pseudorabies vírus transzkriptom vizsgálata: a virális gének szelektíven szabályozódnak Ph.D. értekezés tézisei Póka Nándor Biokémia, Biofizika, Molekuláris és sejtbiológia doktori program Szegedi Tudományegyetem
A Pseudorabies vírus transzkriptom vizsgálata: a virális gének szelektíven szabályozódnak Ph.D. értekezés tézisei Póka Nándor Biokémia, Biofizika, Molekuláris és sejtbiológia doktori program Szegedi Tudományegyetem
Human genome project
 Human genome project Pataki Bálint Ármin 2017.03.14. Pataki Bálint Ármin Human genome project 2017.03.14. 1 / 14 Agenda 1 Biológiai bevezető 2 A human genome project lefolyása 3 Alkalmazások, kitekintés
Human genome project Pataki Bálint Ármin 2017.03.14. Pataki Bálint Ármin Human genome project 2017.03.14. 1 / 14 Agenda 1 Biológiai bevezető 2 A human genome project lefolyása 3 Alkalmazások, kitekintés
Új temékek az UD- GenoMed Kft. kínálatában!
 Új temékek az UD- GenoMed Kft. kínálatában! Szolgáltatásaink: Medical Genomic Technologies Kft. Betegtoborzás és biobanking Bioinformatika o Adatelemzés/adatbányászás o Integrált adatbázis készítés Sejtvonal
Új temékek az UD- GenoMed Kft. kínálatában! Szolgáltatásaink: Medical Genomic Technologies Kft. Betegtoborzás és biobanking Bioinformatika o Adatelemzés/adatbányászás o Integrált adatbázis készítés Sejtvonal
Egy új DNS motívum típus in silico jellemzése és szerepe a génszabályozásban Zárójelentés - OTKA # PD73575, BIOIN Cserző Miklós
 Egy új DNS motívum típus in silico jellemzése és szerepe a génszabályozásban Zárójelentés - OTKA # PD73575, BIOIN Cserző Miklós A kutatás első évében az előzetes tervnek megfelelően tudtunk haladni. Kidolgoztunk
Egy új DNS motívum típus in silico jellemzése és szerepe a génszabályozásban Zárójelentés - OTKA # PD73575, BIOIN Cserző Miklós A kutatás első évében az előzetes tervnek megfelelően tudtunk haladni. Kidolgoztunk
Az orvosi biotechnológiai mesterképzés megfeleltetése az Európai Unió új társadalmi kihívásainak a Pécsi Tudományegyetemen és a Debreceni Egyetemen
 Az orvosi biotechnológiai mesterképzés megfeleltetése az Európai Unió új társadalmi kihívásainak a Pécsi Tudományegyetemen és a Debreceni Egyetemen Azonosító szám: TÁMOP-4.1.2-08/1/A-2009-0011 Az orvosi
Az orvosi biotechnológiai mesterképzés megfeleltetése az Európai Unió új társadalmi kihívásainak a Pécsi Tudományegyetemen és a Debreceni Egyetemen Azonosító szám: TÁMOP-4.1.2-08/1/A-2009-0011 Az orvosi
Using the CW-Net in a user defined IP network
 Using the CW-Net in a user defined IP network Data transmission and device control through IP platform CW-Net Basically, CableWorld's CW-Net operates in the 10.123.13.xxx IP address range. User Defined
Using the CW-Net in a user defined IP network Data transmission and device control through IP platform CW-Net Basically, CableWorld's CW-Net operates in the 10.123.13.xxx IP address range. User Defined
A genetikai vizsgálatok jelene, jövője a Ritka Betegségek vonatkozásában
 Budapest, 2014. február 22. Ritka Betegségek Világnapja A genetikai vizsgálatok jelene, jövője a Ritka Betegségek vonatkozásában dr. Kósa János PentaCore Laboratórium, Budapest Semmelweis Egyetem I. sz.
Budapest, 2014. február 22. Ritka Betegségek Világnapja A genetikai vizsgálatok jelene, jövője a Ritka Betegségek vonatkozásában dr. Kósa János PentaCore Laboratórium, Budapest Semmelweis Egyetem I. sz.
DNS-szekvencia meghatározás
 DNS-szekvencia meghatározás Gilbert 1980 (1958) Sanger 3-1 A DNS-polimerázok jellemzői 5'-3' polimeráz aktivitás 5'-3' exonukleáz 3'-5' exonukleáz aktivitás Az új szál szintéziséhez kell: templát DNS primer
DNS-szekvencia meghatározás Gilbert 1980 (1958) Sanger 3-1 A DNS-polimerázok jellemzői 5'-3' polimeráz aktivitás 5'-3' exonukleáz 3'-5' exonukleáz aktivitás Az új szál szintéziséhez kell: templát DNS primer
Eladni könnyedén? Oracle Sales Cloud. Horváth Tünde Principal Sales Consultant 2014. március 23.
 Eladni könnyedén? Oracle Sales Cloud Horváth Tünde Principal Sales Consultant 2014. március 23. Oracle Confidential Internal/Restricted/Highly Restricted Safe Harbor Statement The following is intended
Eladni könnyedén? Oracle Sales Cloud Horváth Tünde Principal Sales Consultant 2014. március 23. Oracle Confidential Internal/Restricted/Highly Restricted Safe Harbor Statement The following is intended
A PSEUDORABIES VÍRUS TRANSZKRIPTOMIKAI ELEMZÉSE NAGY ÁTERESZTŐKÉPESSÉGŰ MÓDSZEREKKEL
 A PSEUDORABIES VÍRUS TRANSZKRIPTOMIKAI ELEMZÉSE NAGY ÁTERESZTŐKÉPESSÉGŰ MÓDSZEREKKEL Ph.D. Tézis Összefoglaló Oláh Péter Msc Orvosi Biológia Intézet Interdiszciplináris Orvostudományok Doktori Iskola Általános
A PSEUDORABIES VÍRUS TRANSZKRIPTOMIKAI ELEMZÉSE NAGY ÁTERESZTŐKÉPESSÉGŰ MÓDSZEREKKEL Ph.D. Tézis Összefoglaló Oláh Péter Msc Orvosi Biológia Intézet Interdiszciplináris Orvostudományok Doktori Iskola Általános
Tutorial 1 The Central Dogma of molecular biology
 oday DN RN rotein utorial 1 he entral Dogma of molecular biology Information flow in genetics:» ranscription» ranslation» Making sense of genomic information Information content in DN - Information content
oday DN RN rotein utorial 1 he entral Dogma of molecular biology Information flow in genetics:» ranscription» ranslation» Making sense of genomic information Information content in DN - Information content
11. Gyakorlat: Certificate Authority (CA), FTP site-ok
 11. Gyakorlat: Certificate Authority (CA), FTP site-ok 11.1. A CA szerver szerepkör telepítése a DC01-es szerverre 11.2. Az FTP szervíz telepítése a DC01-es szerverre 11.3. A szükséges DNS rekordok létrehozása
11. Gyakorlat: Certificate Authority (CA), FTP site-ok 11.1. A CA szerver szerepkör telepítése a DC01-es szerverre 11.2. Az FTP szervíz telepítése a DC01-es szerverre 11.3. A szükséges DNS rekordok létrehozása
Genetikai panel kialakítása a hazai tejhasznú szarvasmarha állományok hasznos élettartamának növelésére
 Genetikai panel kialakítása a hazai tejhasznú szarvasmarha állományok hasznos élettartamának növelésére Dr. Czeglédi Levente Dr. Béri Béla Kutatás-fejlesztés támogatása a megújuló energiaforrások és agrár
Genetikai panel kialakítása a hazai tejhasznú szarvasmarha állományok hasznos élettartamának növelésére Dr. Czeglédi Levente Dr. Béri Béla Kutatás-fejlesztés támogatása a megújuló energiaforrások és agrár
Energetika az építész kezében
 2013 Energetika az építész kezében Reicher Péter kereskedelmi vezető Natural resources are exploited What can architects do? 2 1 Environmental impact of buildings 40% of global raw materials is consumed
2013 Energetika az építész kezében Reicher Péter kereskedelmi vezető Natural resources are exploited What can architects do? 2 1 Environmental impact of buildings 40% of global raw materials is consumed
Molekuláris biológiai eljárások alkalmazása a GMO analitikában és az élelmiszerbiztonság területén
 Molekuláris biológiai eljárások alkalmazása a GMO analitikában és az élelmiszerbiztonság területén Dr. Dallmann Klára A molekuláris biológia célja az élőlények és sejtek működésének molekuláris szintű
Molekuláris biológiai eljárások alkalmazása a GMO analitikában és az élelmiszerbiztonság területén Dr. Dallmann Klára A molekuláris biológia célja az élőlények és sejtek működésének molekuláris szintű
Miskolci Egyetem Gazdaságtudományi Kar Üzleti Információgazdálkodási és Módszertani Intézet. Correlation & Linear. Petra Petrovics.
 Correlation & Linear Regression in SPSS Petra Petrovics PhD Student Types of dependence association between two nominal data mixed between a nominal and a ratio data correlation among ratio data Exercise
Correlation & Linear Regression in SPSS Petra Petrovics PhD Student Types of dependence association between two nominal data mixed between a nominal and a ratio data correlation among ratio data Exercise
Report on the main results of the surveillance under article 11 for annex II, IV and V species (Annex B)
 0.1 Member State HU 0.2.1 Species code 2633 0.2.2 Species name Mustela eversmanii 0.2.3 Alternative species scientific name 0.2.4 Common name molnárgörény 1. National Level 1.1 Maps 1.1.1 Distribution
0.1 Member State HU 0.2.1 Species code 2633 0.2.2 Species name Mustela eversmanii 0.2.3 Alternative species scientific name 0.2.4 Common name molnárgörény 1. National Level 1.1 Maps 1.1.1 Distribution
kpis(ppk20) kpis(ppk46)
 Table S1. C. elegans strains used in this study Strains carrying transgenes Strain Transgene Genotype Reference IT213 tcer-1 prom:tcer-1orf:gfp:tcer-1 3'utr unc-119(ed3) III; kpis(ppk6) This study IT283
Table S1. C. elegans strains used in this study Strains carrying transgenes Strain Transgene Genotype Reference IT213 tcer-1 prom:tcer-1orf:gfp:tcer-1 3'utr unc-119(ed3) III; kpis(ppk6) This study IT283
Report on the main results of the surveillance under article 11 for annex II, IV and V species (Annex B)
 0.1 Member State HU 0.2.1 Species code 4110 0.2.2 Species name Pulsatilla pratensis ssp. hungarica 0.2.3 Alternative species Pulsatilla flavescens scientific name 0.2.4 Common name magyar kökörcsin 1.
0.1 Member State HU 0.2.1 Species code 4110 0.2.2 Species name Pulsatilla pratensis ssp. hungarica 0.2.3 Alternative species Pulsatilla flavescens scientific name 0.2.4 Common name magyar kökörcsin 1.
CLUSTALW Multiple Sequence Alignment
 Version 3.2 CLUSTALW Multiple Sequence Alignment Selected Sequences) FETA_GORGO FETA_HORSE FETA_HUMAN FETA_MOUSE FETA_PANTR FETA_RAT Import Alignments) Return Help Report Bugs Fasta label *) Workbench
Version 3.2 CLUSTALW Multiple Sequence Alignment Selected Sequences) FETA_GORGO FETA_HORSE FETA_HUMAN FETA_MOUSE FETA_PANTR FETA_RAT Import Alignments) Return Help Report Bugs Fasta label *) Workbench
Diagnosztikai célú molekuláris biológiai vizsgálatok
 Diagnosztikai célú molekuláris biológiai vizsgálatok Dr. Patócs Attila, PhD MTA-SE Molekuláris Medicina Kutatócsoport, Semmelweis Egyetem II. sz. Belgyógyászati Klinika Laboratóriumi Medicina Intézet Genetikai
Diagnosztikai célú molekuláris biológiai vizsgálatok Dr. Patócs Attila, PhD MTA-SE Molekuláris Medicina Kutatócsoport, Semmelweis Egyetem II. sz. Belgyógyászati Klinika Laboratóriumi Medicina Intézet Genetikai
FÖLDRAJZ ANGOL NYELVEN
 Földrajz angol nyelven középszint 0821 ÉRETTSÉGI VIZSGA 2009. május 14. FÖLDRAJZ ANGOL NYELVEN KÖZÉPSZINTŰ ÍRÁSBELI ÉRETTSÉGI VIZSGA JAVÍTÁSI-ÉRTÉKELÉSI ÚTMUTATÓ OKTATÁSI ÉS KULTURÁLIS MINISZTÉRIUM Paper
Földrajz angol nyelven középszint 0821 ÉRETTSÉGI VIZSGA 2009. május 14. FÖLDRAJZ ANGOL NYELVEN KÖZÉPSZINTŰ ÍRÁSBELI ÉRETTSÉGI VIZSGA JAVÍTÁSI-ÉRTÉKELÉSI ÚTMUTATÓ OKTATÁSI ÉS KULTURÁLIS MINISZTÉRIUM Paper
A Multi Locus Sequence Typing (MLST) alkalmazhatósága az élelmiszermikrobiológiában
 A Multi Locus Sequence Typing (MLST) alkalmazhatósága az élelmiszermikrobiológiában Sipos Rita, Lukács Alena, Simon Janka, Szántó-Egész Réka, Micsinai Adrienn 2100 Gödöllő, Szent-Györgyi Albert út 4. info@biomi.hu,
A Multi Locus Sequence Typing (MLST) alkalmazhatósága az élelmiszermikrobiológiában Sipos Rita, Lukács Alena, Simon Janka, Szántó-Egész Réka, Micsinai Adrienn 2100 Gödöllő, Szent-Györgyi Albert út 4. info@biomi.hu,
BIOINFORMATIKA Ungvári Ildikó
 1 BIOINFORMATIKA Ungvári Ildikó Az elmúlt évtizedekben a molekuláris biológiai, genomikai technológiák robbanásszerű fejlődése a biológiai adatok mennyiségének exponenciális növekedéséhez vezetett. Ebben
1 BIOINFORMATIKA Ungvári Ildikó Az elmúlt évtizedekben a molekuláris biológiai, genomikai technológiák robbanásszerű fejlődése a biológiai adatok mennyiségének exponenciális növekedéséhez vezetett. Ebben
VIII. Magyar Sejtanalitikai Konferencia Fény a kutatásban és a diagnosztikában
 VIII. Magyar Sejtanalitikai Konferencia Fény a kutatásban és a diagnosztikában Budapest, 2015. szeptember 3 5. Semmelweis Egyetem II. sz. Belgyógyászati Klinika, valamint I. sz. Patológiai és Kísérleti
VIII. Magyar Sejtanalitikai Konferencia Fény a kutatásban és a diagnosztikában Budapest, 2015. szeptember 3 5. Semmelweis Egyetem II. sz. Belgyógyászati Klinika, valamint I. sz. Patológiai és Kísérleti
GENERATÍV TEST (VIRÁGOS NÖVÉNYEK)
 GENERATÍV TEST (VIRÁGOS NÖVÉNYEK) MITÓZIS IVAROS szaporító szervek hím női (antheridium) (archegonium) IVARTALAN szaporító szervek Sporangium 2n n MEIÓZIS (2n 2n) MITÓZIS IVARTALAN SZAPORÍTÓSZERVEK (2n
GENERATÍV TEST (VIRÁGOS NÖVÉNYEK) MITÓZIS IVAROS szaporító szervek hím női (antheridium) (archegonium) IVARTALAN szaporító szervek Sporangium 2n n MEIÓZIS (2n 2n) MITÓZIS IVARTALAN SZAPORÍTÓSZERVEK (2n
Csatlakozás a BME eduroam hálózatához Setting up the BUTE eduroam network
 Csatlakozás a BME eduroam hálózatához Setting up the BUTE eduroam network Table of Contents Windows 7... 2 Windows 8... 6 Windows Phone... 11 Android... 12 iphone... 14 Linux (Debian)... 20 Sebők Márton
Csatlakozás a BME eduroam hálózatához Setting up the BUTE eduroam network Table of Contents Windows 7... 2 Windows 8... 6 Windows Phone... 11 Android... 12 iphone... 14 Linux (Debian)... 20 Sebők Márton
Welcome! EuPathDB Workshop Crash Course in Omics Terminology, Concepts & Data Types
 Welcome! EuPathDB Workshop 2012 Crash Course in Omics Terminology, Concepts & Data Types Jessie Kissinger June 17, 2012 1 Protein Expression! Interactions! Genome and Annotation! RNA Sequencing! Structure!
Welcome! EuPathDB Workshop 2012 Crash Course in Omics Terminology, Concepts & Data Types Jessie Kissinger June 17, 2012 1 Protein Expression! Interactions! Genome and Annotation! RNA Sequencing! Structure!
A funkcionális genomikai eszköztár szerepe az onkológiai kutatásokban
 Összefoglaló közlemény 21 A funkcionális genomikai eszköztár szerepe az onkológiai kutatásokban Bálint Bálint L. 1, Nagy László 1,2 1 Debreceni Egyetem Orvos- és Egészségtudományi Centrum, Biokémiai és
Összefoglaló közlemény 21 A funkcionális genomikai eszköztár szerepe az onkológiai kutatásokban Bálint Bálint L. 1, Nagy László 1,2 1 Debreceni Egyetem Orvos- és Egészségtudományi Centrum, Biokémiai és
A HULLATÉK-ANALÍZIS ÉS A GYOMORTARTALOM ELEMZÉS ÖSSZE- HASONLÍTÁSA VÖRÖS RÓKA TÁPLÁLKOZÁS VIZSGÁLATA SORÁN
 A HULLATÉK-ANALÍZIS ÉS A GYOMORTARTALOM ELEMZÉS ÖSSZE- HASONLÍTÁSA VÖRÖS RÓKA TÁPLÁLKOZÁS VIZSGÁLATA SORÁN Szôcs Emese¹, Lanszki József², Heltai Miklós¹ és Szabó László¹ ¹Szent István Egyetem, Vadbiológiai
A HULLATÉK-ANALÍZIS ÉS A GYOMORTARTALOM ELEMZÉS ÖSSZE- HASONLÍTÁSA VÖRÖS RÓKA TÁPLÁLKOZÁS VIZSGÁLATA SORÁN Szôcs Emese¹, Lanszki József², Heltai Miklós¹ és Szabó László¹ ¹Szent István Egyetem, Vadbiológiai
Bakteriális identifikáció 16S rrns gén szekvencia alapján
 Bakteriális identifikáció 16S rrns gén szekvencia alapján MOHR ANITA SIPOS RITA, SZÁNTÓ-EGÉSZ RÉKA, MICSINAI ADRIENN 2100 Gödöllő, Szent-Györgyi Albert út 4. info@biomi.hu, www.biomi.hu TÖRZS AZONOSÍTÁS
Bakteriális identifikáció 16S rrns gén szekvencia alapján MOHR ANITA SIPOS RITA, SZÁNTÓ-EGÉSZ RÉKA, MICSINAI ADRIENN 2100 Gödöllő, Szent-Györgyi Albert út 4. info@biomi.hu, www.biomi.hu TÖRZS AZONOSÍTÁS
Weblog elemzés Hadoopon 1/39
 Weblog elemzés Hadoopon 1/39 Az előadás témái Egy Hadoop job életciklusa A Weblog-projekt 2/39 Mi a Hadoop? A Hadoop egy párhuzamos programozási séma egy implementációja. 3/39 A programozási séma: MapReduce
Weblog elemzés Hadoopon 1/39 Az előadás témái Egy Hadoop job életciklusa A Weblog-projekt 2/39 Mi a Hadoop? A Hadoop egy párhuzamos programozási séma egy implementációja. 3/39 A programozási séma: MapReduce
Bioinformatika és genomanalízis az orvostudományban. Biológiai adatbázisok. Cserző Miklós 2018
 Bioinformatika és genomanalízis az orvostudományban Biológiai adatbázisok Cserző Miklós 2018 A mai előadás Mi az adatbázis A biológia kapcsolata az adatbázisokkal Az adatbázisok típusai Adatbázis formátumok,
Bioinformatika és genomanalízis az orvostudományban Biológiai adatbázisok Cserző Miklós 2018 A mai előadás Mi az adatbázis A biológia kapcsolata az adatbázisokkal Az adatbázisok típusai Adatbázis formátumok,
Összefoglalás első fejezete
 Összefoglalás Az utóbbi években a transzkriptómikai vizsgálatokban alkalmazott technológiák fejlődésének köszönhetően a genom kutatás területén jelentős előrehaladás figyelhető meg. A disszertáció első
Összefoglalás Az utóbbi években a transzkriptómikai vizsgálatokban alkalmazott technológiák fejlődésének köszönhetően a genom kutatás területén jelentős előrehaladás figyelhető meg. A disszertáció első
Supplemental Table S1. Overview of MYB transcription factor genes analyzed for expression in red and pink tomato fruit.
 Supplemental Table S1. Overview of MYB transcription factor genes analyzed for expression in red and pink tomato fruit. MYB Primer pairs TC AtMYB Forward Reverse TC199266 MYB12 AGGCTCTTGGAGGTCGTTACC CAACTCTTTCCGCATCTCAATAATC
Supplemental Table S1. Overview of MYB transcription factor genes analyzed for expression in red and pink tomato fruit. MYB Primer pairs TC AtMYB Forward Reverse TC199266 MYB12 AGGCTCTTGGAGGTCGTTACC CAACTCTTTCCGCATCTCAATAATC
A cell-based screening system for RNA Polymerase I inhibitors
 Electronic Supplementary Material (ESI) for MedChemComm. This journal is The Royal Society of Chemistry 2019 Supporting Information A cell-based screening system for RNA Polymerase I inhibitors Xiao Tan,
Electronic Supplementary Material (ESI) for MedChemComm. This journal is The Royal Society of Chemistry 2019 Supporting Information A cell-based screening system for RNA Polymerase I inhibitors Xiao Tan,
