Bevezetés a bioinformatikába
|
|
|
- Marcell Antal Gál
- 8 évvel ezelőtt
- Látták:
Átírás
1 Bevezetésabioinformatikába őszifélév,biológiaBSC,levelezőképzés BálintBalázs szeged.hu/
2 Információakurzusról I.elméletialapok(azévvégivizsgaanyaga) II.azelméletirészheztartozógyakorlatimunka(nemszámonkért) (szakirodalmazás,adatbázis bányászatszekvenciahomológia keresés,fehérjetérszerkezetvizsgálat,stb.) Vizsga:írásban,tesztek/rövidválasztigénylőkérdések
3 Abioinformatikadefiníciója informatika biológia hardverek,szoftverek megválaszolandókérdés, biológiaiadat Bioinformatika:biológiaiadatokszámítógépesanalízise.
4 Centrális dogma és a bioinformatika főbb területei a molekuláris biológiában Gén genomika DNS transzkripció, RNS szerkesztés RNS transzkriptomika degradáció transzláció, poszttranszlációs módosítás proteomika fehérje degradáció biokémiai aktivitás metabolikus útvonalak metabolomika
5 adnsafőbiológiaiinformációhordozó F. Griffith Avery 1944 Streptococcus pneumoniae a transzformáló anyag DNS proteáz RNáz DNáz abaktériummódosítható(transzformálható) egér meghal egér meghal egér túlél
6 Hershey és Chase ,Escherichiacoli tfertőztekradioaktívanjelöltt2fággal adnsp32 vel,afehérjeburoks35 teljelölve (adns bennincss,afehérjébennincsp) 2.,Abaktériumhoztapadtkiürültfágburkokat rázássalleválasztották 3.,Abaktériumokatésaszabaddávált fágburkokatcentrifugálássalelkülönítették felülúszó,(s35fág fehérje) baktériumpellet,p32 (fágdns)
7 DNSszerkezete(1953) "Nemkerülteelafigyelmünketaz,hogyazáltalunkfeltételezettpárosítási szabályegymásolásimechanizmustissugallagenetikaianyagszámára." JamesWatsonésFrancisCrick
8 DNSreplikáció RNSszintézis(transzkripció)
9 Fehérjeszintézis(transzláció)
10 Azuniverzálisgenetikaikód Másodikkarakter Els ő karakter Harmadikkarakter DNS:ATG RNS:AUG AS:Metionin
11 Hogyannyerjükkiaszekvenciainformációt? Fehérje DNS Könnyentisztítható Nehezebbentisztítható Stabil Számosinstabilfehérjelétezik Könnyebben,szekvenálható Aközvetlenfehérjeszekvenálás igennehézfeladat AszekvenciainformációkatzömmelDNSmolekulárólnyerik,amiket azutánszámítógéppel(insilico)fehérjeszekvenciárafordítanak.
12 ADNSinformációtartalmánakmegismerése akezdetkezdete P s t I ( 1) V a r ia t io n 1 V a r ia t io n 2 V a r ia t io n 3 M is c F e a t u r e 2 V a r ia t io n 4 C D S 4 A pali (4780) A v a I ( 16 3 ) C D S 5 R e p O r ig in 1 C D S 6 m R N A 2 p h i- x M is c F e a t u r e 1 C D S bp C D S 8 C D S 9 FrederickSanger C D S 1 1 m R N A 1 C D S 1 0 azelsőpublikáltteljesgenom(1977) kb.5000nukleotid(!)
13 Sanger félednsszekvenáláselve P P P P P P P láncterminációddgtpjelenlétében P termékkeverék
14 Sanger félednsszekvenáláselve ddgtp ddttp ddatp ddctp P P P P P P P P P P P P P P P zseb poliakrilamidgél futtatásiránya:
15 Egyigaziszekvenálólétra Szekvencia: 5 tcaactttgtcggcttgagaaagacctgggatctgggtat... Atechnológiakorlátai: emberpróbáló,extrémmunkaigényeseljárás igenkicsileolvasásihossz atermékekdetektálásap32izotópsegítségével
16 ASanger félednsszekvenálásautomatizálása Anégyféledideoxynukleotidanalógegyreakcióbanadva mindanégyddntpegyedifluoreszcensfestékkeljelölve atermékekméretszerintielválasztásakapillárisoszlopon adetektorelőttelhaladófestékek sorrendje=>bázissorrend teljesenautomatizáltberendezés ~ nukleotidhosszúDNSdarabokolvashatóak szekvenogram:festékintenzitásokváltozásaazidőben
17 1995Haemophilusinfluenzaegenomszekvencia Shotgunmódszer DNS tisztítás darabolás futtatás ideálisméretűdarabok (1,5 2kb)kinyerése midenklónból plazmidtisztítás inszert szekvenálása aplazmidok bejuttatása E.coli ba contigassembly agenomifragmentek plazmidokbaligálása
18 ASanger szekvenálásraalapozottshotgunmódszerkorlátai Könyvtárkészítésszükséges munkaigényes,időigényes,költségesfolyamat egyenetlenlefedettség,agazdára(e.coli)toxikusrégiókteljesenkimaradnak agazdagenomnyomokbanszennyeződéskéntmegjelenhet különbözőprojektekközöttikereszt szennyeződéskönnyenelőfordulhat Alacsonyáteresztőképesség azújszálszintéziseésabázissorrendmeghatározásakülönlépés kapilláriselektroforézislépésszükséges kevéssépárhuzamosítható(max96kapilláris/berendezés) magasköltség/szekvenálásireakció
19 Újgenerációsszekvenálásitechnológiák Roche454FLX IlluminaSolexa ABISolid Nincsszükséghagyományosgenomikönyvtárra KözvetlenülatisztítottgenomiDNSkerülagépbe AgDNS tfizikailagtörik,afragmentekethordozóhozrögzítik,majdpcr segítségévelfelsokszorozzák Óriásiátereszőképesség nagyfokúpárhuzamosítás:akártöbbmilliószekvenálásireakcióegyszerre azújszálszintéziseésanukleotidsorrendmeghatározásaegyidejűlegtörténik kisebbköltség/szekvenálásireakció
20 Elkészültgenomszekvenciákstatisztikája2009 Bakteriálisgenom 1001(714*) Eukariótagenomok 74(22*) Caenorhabditiselegans(talajlakófonalféreg) Ecetmuslica(Drosophilamelanogaster) Egér(Musmusculus) Ember(Homosapiens) Kutya(Canislupusfamiliaris) Lúdfű(Arabidopsisthaliana) Méh(Apismellifera) Patkány(Rattusnorvegicus) Rizs(Oryzasativa) Sertés(Susscrofa) Szarvasmarha(Bostaurus) Szőlő(Vitisvinifera) *2008 asadat
21 Összefogásanukleinsavadatbankokközött NIH USA NCBI GenBan k EMBL CIB DDBJ NIG Japan EBI EMBL Europe NIH:NationalInstituteofHealth >NCBI:NationalCenterforBiotechnologyInformation >GenBank NIG:NationalInstituteofGenetics >CIB:CenterofInformationBiology >DDBJ EMBL:EuropeanMolecularBiologyLaboratory >EBI >EuropeanBioinformaticsInstitute >EMBL
22 Miazadatbázis? számítógépesfájlstrukturáltadattartalommal szabványosítottadatszerkezet gyorsösszetettkeresésekvégezhetőek /indexelés/ rendszeresenfrissített,naprakész /újkiadások/ kapcsolatokmásadatbázisokfelé /kereszthivatkozások/ Megfelelőszoftverekkellenek,melyekkel adatlekérdezés,adatfrissítés,adattörlés,adathozzáadásvégezhető
23 Hogyanépülfelegyadatbank? szabványosítás,szabványosítás,szabványosítás Feladat: adatoktárolása:jóldokumentáltszekvenciaformátumban anyersszekvenciákonkívültovábbifontoskiegészítőinformációkat tároljon(szekvencialeírása,eredete,típusa,hossza,stb.stb.) lehessenkeresniezekbena"kiegészítő"információkban kereshetőlegyenaszekvencia akutatókújszekvenciákatküldhessenekbeazadatbankba legyenlehetőségahibajavításra(update) nelegyenredundáns minélinkábbautomatizáltlegyen Adatbázisok
24 Azadatbázisoktípusai Elsődlegesadatbázisok Akísérletezőkeredetielküldöttadatai Közvetlenkísérletieredményekettartalmaznak Pl.GeneBank,GEO(génexpressziósadatbank) Származtatottadatbázisok Elsődlegesadatokanalízisévelnyerttöbbletinformációkattárol Hivatkozásokazelsődlegesadatbáziseredetibejegyzéseire Néhánypélda: RefSNP(pontmutációadatbank) CDD(konzerváltdomainadatbázis), PFAM(fehérjecsaládokadatbázisa)
25 AGeneBankadatbázis 1979 benalapítva(losalamos). 1992ótaazNCBIgondozza(Bethesda). azadatbázissajátszekvenciaformátumaagenebank szekvenciainformáció szekvenciákhozkapcsolódóegyébinformációk,annotációk kereszthivatkozásokmásadatbankokkapcsolódóbejegyzéseire azadatbázisdivíziókraosztott: PRIfőemlősszekvenciák ROD rágcsálókszekvenciái PLNnővényi,gombaésalga BCT bakteriális EST expresszáltszekvenciadarabkák(cdns) ENV környezetimintákbólnyertszekvenciák PAT szabadalmakhozkapcsolódószekvenciák ~taxonómia szekvenciajellege
26 AGeneBankadatbázisgyarapodása 2009október Nukleotid (milliárdbp) 50 Szekvencia (milliódb) Töretlen,közelexponenciálisnövekedés
27 EgyGeneBankbejegyzés LOCUS DEFINITION ACCESSION VERSION KEYWORDS SOURCE ORGANISM REFERENCE AUTHORS TITLE JOURNAL MEDLINE PUBMED REFERENCE AUTHORS TITLE JOURNAL REFERENCE AUTHORS TITLE JOURNAL REMARK COMMENT AF bp mrna linear INV 23-OCT-2002 Limulus polyphemus myosin III mrna, complete cds. AF AF GI: Limulus polyphemus (Atlantic horseshoe crab) Limulus polyphemus Eukaryota; Metazoa; Arthropoda; Chelicerata; Merostomata; Xiphosura; Limulidae; Limulus. 1 (bases 1 to 3808) Battelle,B.-A., Andrews,A.W., Calman,B.G., Sellers,J.R., Greenberg,R.M. and Smith,W.C. A myosin III from Limulus eyes is a clock-regulated phosphoprotein J. Neurosci. 18 (12), (1998) (bases 1 to 3808) Battelle,B.-A., Andrews,A.W., Calman,B.G., Sellers,J.R., Greenberg,R.M. and Smith,W.C. Direct Submission Submitted (29-APR-1998) Whitney Laboratory, University of Florida, 9505 Ocean Shore Blvd., St. Augustine, FL 32086, USA 3 (bases 1 to 3808) Battelle,B.-A., Andrews,A.W., Calman,B.G., Sellers,J.R., Greenberg,R.M. and Smith,W.C. Direct Submission Submitted (02-MAR-2000) Whitney Laboratory, University of Florida, 9505 Ocean Shore Blvd., St. Augustine, FL 32086, USA Sequence update by submitter On Mar 2, 2000 this sequence version replaced gi:
28 EgyGeneBankbejegyzés lókusz LOCUS AF bp mrna linear INV 23-OCT-2002 DEFINITION Limulus polyphemus mrna, complete LOCUS AF myosin bp III mrna linear cds.inv 23-OCT-2002 ACCESSION AF VERSION AF GI: KEYWORDS. SOURCE Limulus polyphemus (Atlantic horseshoe crab) ORGANISM Limulus polyphemus Eukaryota; Metazoa; Arthropoda; Chelicerata; Merostomata; Xiphosura; Limulidae; Limulus. REFERENCE 1 (bases 1 to 3808) AUTHORS Battelle,B.-A., Andrews,A.W., Calman,B.G., Sellers,J.R., Greenberg,R.M. and Smith,W.C. TITLE A myosin III from Limulus eyes is a clock-regulated phosphoprotein JOURNAL J. Neurosci. 18 (12), (1998) MEDLINE PUBMED REFERENCE 2 (bases 1 to 3808) AUTHORS Battelle,B.-A., Andrews,A.W., Calman,B.G., Sellers,J.R., Greenberg,R.M. and Smith,W.C. TITLE Direct Submission JOURNAL Submitted (29-APR-1998) Whitney Laboratory, University of Florida, 9505 Ocean Shore Blvd., St. Augustine, FL 32086, USA REFERENCE 3 (bases 1 to 3808) AUTHORS Battelle,B.-A., Andrews,A.W., Calman,B.G., Sellers,J.R., Greenberg,R.M. and Smith,W.C. TITLE Direct Submission JOURNAL Submitted (02-MAR-2000) Whitney Laboratory, University of Florida, 9505 Ocean Shore Blvd., St. Augustine, FL 32086, USA REMARK Sequence update by submitter COMMENT On Mar 2, 2000 this sequence version replaced gi: Hossz Lókusz név Molekula típus Divízió Módosítás Dátum
29 AGeneBankazonosítók LOCUS DEFINITION ACCESSION VERSION KEYWORDS SOURCE ORGANISM AF bp mrna linear INV 23-OCT-2002 Limulus polyphemus myosin III mrna, complete cds. AF AF GI: Limulus polyphemus (Atlantic horseshoe crab) Limulus polyphemus Eukaryota; Metazoa; Arthropoda; Chelicerata; Merostomata; Xiphosura; Limulidae; Limulus. 1 (bases 1 to 3808) Battelle,B.-A., Andrews,A.W., Calman,B.G., Sellers,J.R., Greenberg,R.M. and Smith,W.C. A myosin III from Limulus eyes is a clock-regulated phosphoprotein J. Neurosci. 18 (12), (1998) (bases 1 to 3808) Battelle,B.-A., Andrews,A.W., Calman,B.G., Sellers,J.R., Greenberg,R.M. and Smith,W.C. Direct Submission Submitted (29-APR-1998) Whitney Laboratory, University of Florida, 9505 Ocean Shore Blvd., St. Augustine, FL 32086, USA 3 (bases 1 to 3808) Battelle,B.-A., Andrews,A.W., Calman,B.G., Sellers,J.R., Greenberg,R.M. and Smith,W.C. Direct Submission Submitted (02-MAR-2000) Whitney Laboratory, University of Florida, 9505 Ocean Shore Blvd., St. Augustine, FL 32086, USA Sequence update by submitter On Mar 2, 2000 this sequence version replaced gi: ACCESSION VERSION REFERENCE AUTHORS TITLE JOURNAL MEDLINE PUBMED REFERENCE AUTHORS TITLE JOURNAL REFERENCE AUTHORS TITLE JOURNAL REMARK COMMENT AF AF GI: Egyedi azonosító (fix) GB azonosító (változhat!)
30 GeneBankaszekvenciaeredete(Atlantitőrfarkú) LOCUS DEFINITION ACCESSION VERSION KEYWORDS SOURCE ORGANISM AF bp mrna linear INV 23-OCT-2002 Limulus polyphemus myosin III mrna, complete cds. AF AF GI: Limulus polyphemus (Atlantic horseshoe crab) Limulus polyphemus Eukaryota; Metazoa; Arthropoda; Chelicerata; Merostomata; Xiphosura; Limulidae; Limulus. SOURCE Limulus polyphemus (Atlantic horseshoe crab) REFERENCE 1 (bases 1 to 3808) ORGANISM Limulus polyphemus AUTHORS Battelle,B.-A., Andrews,A.W., Calman,B.G., Sellers,J.R., Eukaryota; Greenberg,R.M. and Metazoa; Smith,W.C.Arthropoda; Chelicerata; Merostomata; Xiphosura; Limulidae; Limulus. TITLE A myosin III from Limulus eyes is a clock-regulated phosphoprotein JOURNAL J. Neurosci. 18 (12), (1998) MEDLINE PUBMED REFERENCE 2 (bases 1 to 3808) AUTHORS Battelle,B.-A., Andrews,A.W., Calman,B.G., Sellers,J.R., Greenberg,R.M. and Smith,W.C. TITLE Direct Submission JOURNAL Submitted (29-APR-1998) Whitney Laboratory, University of Florida, 9505 Ocean Shore Blvd., St. Augustine, FL 32086, USA REFERENCE 3 (bases 1 to 3808) AUTHORS Battelle,B.-A., Andrews,A.W., Calman,B.G., Sellers,J.R., Greenberg,R.M. and Smith,W.C. TITLE Direct Submission JOURNAL Submitted (02-MAR-2000) Whitney Laboratory, University of Florida, 9505 Ocean Shore Blvd., St. Augustine, FL 32086, USA REMARK Sequence update by submitter COMMENT On Mar 2, 2000 this sequence version replaced gi: NCBI Taxonómia
31 GeneBankreferenciák LOCUS DEFINITION ACCESSION VERSION KEYWORDS SOURCE ORGANISM AF bp mrna linear INV 23-OCT-2002 Limulus polyphemus myosin III mrna, complete cds. AF AF GI: Limulus polyphemus (Atlantic horseshoe crab) Limulus polyphemus Eukaryota; Metazoa; Arthropoda; Chelicerata; Merostomata; Xiphosura; Limulidae; Limulus. REFERENCE 1 (bases 1 to 3808) AUTHORS Battelle,B.-A., Andrews,A.W., Calman,B.G., Sellers,J.R., Greenberg,R.M. and Smith,W.C. TITLE A myosin III from Limulus eyes is a clock-regulated phosphoprotein REFERENCE 1 (bases 1 to 3808) JOURNAL J. Neurosci. 18 (12), (1998) AUTHORS Battelle,B.-A., Andrews,A.W., Calman,B.G., Sellers,J.R., MEDLINE PUBMED Greenberg,R.M. and Smith,W.C. A myosin III from Limulus eyes is a clock-regulated REFERENCETITLE 2 (bases 1 to 3808) phosphoprotein AUTHORS Battelle,B.-A., Andrews,A.W., Calman,B.G., Sellers,J.R., JOURNAL J. Neurosci. 18 (12), (1998) Greenberg,R.M. and Smith,W.C. TITLE MEDLINE Direct Submission PUBMED JOURNAL Submitted (29-APR-1998) Whitney Laboratory, University of Florida, 9505 Ocean Shore Blvd., St. Augustine, FL 32086, USA REFERENCE 3 (bases 1 to 3808) AUTHORS Battelle,B.-A., Andrews,A.W., Calman,B.G., Sellers,J.R., Greenberg,R.M. and Smith,W.C. TITLE Direct Submission JOURNAL Submitted (02-MAR-2000) Whitney Laboratory, University of Florida, 9505 Ocean Shore Blvd., St. Augustine, FL 32086, USA REMARK Sequence update by submitter COMMENT On Mar 2, 2000 this sequence version replaced gi: szakirodalom kereszthivatkozás
32 AGeneBanktulajdonságtábla FEATURES source CDS Location/Qualifiers /organism="limulus polyphemus" /db_xref="taxon:6850" /tissue_type="lateral eye" /note="n-terminal protein kinase domain; C-terminal myosin heavy chain head; substrate for PKA" /codon_start=1 /product="myosin III" /protein_id="aac " /db_xref="gi: " /translation="meykcisehlpfetlpdpgdrfevqelvgtgtyatvysaidkqa NKKVALKIIGHIAENLLDIETEYRIYKAVNGIQFFPEFRGAFFKRGERESDNEVWLGI EFLEEGTAADLLATHRRFGIHLKEDLIALIIKEVVRAVQYLHENSIIHRDIRAANIMF SKEGYVKLIDFGLSASVKNTNGKAQSSVGSPYWMAPEVISCDCLQEPYNYTCDVWSIG ITAIELADTVPSLSDIHALRAMFRINRNPPPSVKRETRWSETLKDFISECLVKNPEYR PCIQEIPQHPFLAQVEGKEDQLRSELVDILKKNPGEKLRNKPYNVTFKNGHLKTISGQ 1201 a 689 c 782 g 1136 t /protein_id="aac " /db_xref="gi: " fehérje adatbank kereszthivatkozás BASE COUNT ORIGIN 1 tcgacatctg tggtcgcttt ttttagtaat aaaaaattgt attatgacgt cctatctgtt 3781 aagatacagt aactagggaa aaaaaaaa //
33 AGeneBank(GenePept)bejegyzésFASTAformátumban >gi gb AF Limulus polyphemus myosin III mrna, complete cds TCGACATCTGTGGTCGCTTTTTTTAGTAATAAAAAATTGTATTATGACGTCCTATC TGTTGTTGTGTTACACAGGTACATATTAATAACAGGTAGCTAACGTACTTATATAT ACATATATATAATTGGTCTGTTACTTTCAGTTACTCCCTGACTTGTGATCCTACTTG TTGCTGTGTTATACAGGTATATATCACTAAAACAGACTGCTAACGTGCATATATTT ATATATGTGTAGCTTTGTTAATGCTTTAACATGGAGTATAAGTGTATCAGTGAACA TTTACCATTTGAGACTCTGCCTGATCCAGGTGATCGGTTTGAAGTACAAGAACTCG TTGGAACAGGAACTTATGCTACCGTATACTCAGCGATTGATAAGCAAGCAAACAA GAAGGTAGCGCTGAAGATTATAGGACACATTGCGGAAAATCTACTTGATATCGAA ACTGAATATCGTATTTATAAAGCTGTCAATGGAATCAGTTTTTCCCCGAATTCCGT GGTGCTTTCTTCAAGCGTGGGGAACGAGAATCTGACAATGAGTATGGCTGGGAAT TGAGTTTCTGGAAGAAGGGACAGCAGCTGACTTGCTTGCAACACACAGAAGGTTT GGAATTCACTTGAAGAAGACTTGATTGCTTTAATAATCAAGGAGGTTGTACGAGC TGTGCAGTACTTACATGAAAACAGCATTATCCACAGAGATATTCGTGCTGCCAAT ATAATGTTTTCTAAAGAGGGATATGTCAAATTAATTGACTTTGGTCTTTCTGCTTC AGTAAAGAACACGAACGGCAAAGCACAGTCTTCTGTGGGCTCCCCCTATTGGATG GCTCCTGAGGTGATATCCTGTGACTGTCTTCAAGAACCTTATAACTACACATGTGA CGTTTGGTCTATGGAATAACTGCTATAGAATTAGCAGACACAGTGCCCTCACTTA GCGATATTCATGCTTTAGCGCCATGTTTCGGATTAACAGAAATCCTCCCCCTAGTG TTAAGAGGGAAACACGCTGGTCAGAAACATTGAAAGATTTTATCAGCGAATGTTT GGTGAAAAATCCCGAATATCGACCGTGTATCCAAGAAATTCCCCAACACCCATTTT T...
34 Továbbiszekvenciaformátumok ID SQ EMBL // nameless standard; DNA; UNC; 457 BP. Sequence 457 BP; GGCGAAGATT CGGCCAGGCA AAGAAGAGCG CGACGAATGG GAGCATGTAG ATTCCTCCGT ACCCCATGCC CCAACCATGC GAGTCAAACC TTCGTGAAAG GCAGCCCGAA GACGATGGCC ATCCACACGA CGTGAATGAA CCGCGCCCTC GCGGTTTTTC \\\ AATGATCGAA AGGGAATCGG CAACTTGAGT GGAGTCAGTC nameless_1 TTCGTTGCCT CGGACCTGCA TCCCTTTGGG ENTRY AGATGCTTCA GAGAGCAAGG nameless_1 TTGATTGCGC 475 bases ATACAGATGC TITLE TCGGTTCGCG ACGGCCTGCG TGAGGATACC SEQUENCE CCGTGCGACG ACCATTAATA AGGCGCTCCA CAGTTCCCGC GGGACACTAG AGTGTCCACT GGGCAGCGGG 1 M S R CAGGGTCTCC T V T I E P VCCCGGGA T R I E G H A R I T L Q nameless_1 PIR GCG 31 D A K F H L T Q F R G F E K F C E G R P 61 T C G I C P V S H V L A S N K A C D H L 91 K L R R I I N L A Q L T Q S H A L S F F 121 W D S D P V S R N I F G V M R Q D P A L 151 G Q T I I E T L G G K K I H P T W V V P 181 K R D A M L K L I P E G L E I A K R T Y 211 K D E A N H F G S Q P T M F L S L V S P Length: L R L K D A QNov G R I15, L E 2004 D M V P 10:24 P H E Y 271 F S Y M K F P Y Y K P H G Y P N G I Y R CGGCCAGGCA AAGAAGAGCG CGACGAATGG 301 D A C G T P Y A D V A L A E F H M L Q E 331 H Y A R L V E I I Y A L ECCAACCATGC M M E R L L K ATTCCTCCGT ACCCCATGCC 361 A R A R S N R Y E G I G V A E A P R G I ATCACCACGA GCAGCCCGAA 391 G L I T W V N L I I A T GGACGATGGCC H N N L A M N 421 V D G N N L Q E G M L N R V E A V I R C CCGCGCCCTC CATGCGGTCC GCGGTTTTTC 451 A F G E M P L A I E L K D A T G R V V D nameless_1 1 GGCGAAGATT 51 CCATGGCTGT 101 TTCGTGAAAG 151 CGTGAATGAA 201 AGGGAATCGG /// CAACTTGAGT 251 AGATGCTTCA GAGAGCAAGG 301 ATACAGATGC TCGGTTCGCG 351 CGGTTCCAGT CCGTGCGACG 401 GGGACACTAG CCAACCGGGC 451 CCCGGGA GGAGTCAGTC TTCGTTGCCT ACGGCCTGCG ACCATTAATA AGTGTCCACT ATATCAGGAG CGGACCTGCA TTGATTGCGC AGGCGCTCCA GGGCAGCGGG CCATGGCTGT ATCACCACGA CATGCGGTCC ATATCAGGAG TCACCCAACC CGGTTCCAGT CCAACCGGGC L G D A G E V E Y R E M P A L T A R L S V S I P P T G E H L S S P D L L L G A K D G I R L R Q I G G V S E P L T Q E A F F K T L V P K F K G H L E H Y D G F E Check: R L I G E7178 A V E D.. V G P L A R L N N V GAGCATGTAG S G P I A S S F H Y D P T I L D A R V R GAGTCAAACC L M H H Y R I D D E ATCCACACGA Q S I R Q V A D A Y F D P C L S C A S H AATGATCGAA T L R R G TCCCTTTGGG TCACCCAACC TGAGGATACC CAGTTCCCGC CAGGGTCTCC
35 Konverziókülönféleszekvenciaformátumokközött Akülönféleszekvenciaformátumokkönnyenátkonvertálhatóakegymásba Seqret programazembosscsomagból helyilegtelepítettváltozattal (Bio)perlscriptsegítségével EBIReadSeqportálján
36 Másodikrész
37 Szekvenciaevolúció AlegtöbbDNSpolimeráznagyonhűenmásol. (AzE.coliDNSpolimerázanagyjábólegyhibátvéttízmilliónukleotidonként) Elegendőhosszúidőalattszámospontmutáció Kromoszómaátrendeződésselhirtelen,nagyobbváltozások(inszerció,deléció) történhetnek ADNS(vagyfehérje)szekvenciaösszehasonlításávalevolúciósrokonságifokis kimutatható: rdnsszekvenciaelemzésalapjánfelállítottuniverzálistörzsfa
38 Változásokaszekvenciákban A T C C T A T T C A C A G A T A A T C C A C A G A T A A T C C G A T T A A C A G A T A A T C C G A T T A A C A G A T A pontmutációk inszerció/deléció A T C C C C A A T A C A G A T A A T C C T A T T G GC A G A T A inverzió
39 Szekvenciaevolúció Homológszekvenciák: hasonlóak közösősrevezethetőekvissza Analógszekvenciák: hasonlóságközösevolúciósősnélkül leszármazott#2 leszármazott#1 (rombusz) (téglalap) közösős (paralelogramma) ortológ:ahomológfehérjékkétkülönfajbantalálhatók,afunkcióáltalábanazonos Pl.szarvasmarhainzulin emberiinzulin paralóg:ahomológfehérjékugyanazonfajbantalálhatók(általábannemteljesen azonosfunkció) Pl.emberihemoglobinAéshemoglobinBláncok
40 Homológiakeresésahőskorban:Dotplot zaj AkétszekvenciaazXilletveYtengelyrekerül MindenXpozíciótmindenYpozícióvalösszehasonlítunk Aholegyezésvan,odaegypontotteszünk Azközösrégiókátlósvonalkéntjelennekmeg
41 Homológiakeresés:szekvenciaillesztés Nukleinsavvagyfehérjeszekvenciákegymáshozrendezése Nagyonsokillesztéslehetséges Melyikalegjobb?Valóshasonlóságotmutat?Tényleghomológakétszekvencia? Azillesztésekkiértékeléséhezpontozásirendszerszükséges Szekvencia1 Szekvencia2 actaccagttcatttgatacttctcaaa taccattaccgtgttaactgaaaggacttaaagact actaccagttcatttgatacttctcaaa taccattaccgtgttaactgaaaggacttaaagact actaccagttcatttgatacttctcaaa taccattaccgtgttaactgaaaggacttaaagact actaccagttcatttgatacttctcaaa taccattaccgtgttaactgaaaggacttaaagact actaccagttcatttgatacttctcaaa taccattaccgtgttaactgaaaggacttaaagact actaccagttcatttgatacttctcaaa actaccagttcatttgatacttctcaaa taccattaccgtgttaactgaaaggacttaaagact taccattaccgtgttaactgaaaggacttaaagact actaccagttcatttgatacttctcaaa taccattaccgtgttaactgaaaggacttaaagact
42 Homológiakeresés:pontozás actaccagttcatttgatacttctcaaa Szekvencia1 taccattaccgtgttaactgaaaggacttaaagact Szekvencia2 Negatívértékbüntetiazeltéréseket: A T C G A T C -4-4 G Illeszkedik:5 Nemilleszkedik:19 Score:5x5+19x( 4)= 51
43 ADNSpontozásirendszerhibája CCTCCTTTGT Pont=50 CCTCCTTTGT Pro A A T C T G 4-4 Leu G 5-4 C CCTCCTTTGG Pont=32 CCTCCCTTAG Pro Leu Nemveszifigyelembe,hogyegyaminosavattöbbkodoniskódolhat(némamutációk)
44 Fehérjepontozásirendszer aháttér Azaminosavaknakkülönbözőfizikai kémiaitulajdonságaikvannak, ezekbefolyásoljákakicserélhetőségüket Példáulavalin(V)ésazizoleucin(I)kicserélhetőegymásra alifás L hidrofób P C S+S I M V pici A kicsi G CSH S K E D T F Y W H R aromás N Q pozitív poláris töltött A:alanin,R:arginin,N:aszparagin,D:aszparaginsav,C:cisztein,Q:glutamin,E:glutaminsav,G:glicerin, H:hisztidin,I:izoleucin,L:leucin,K:lizin,M:metionin,F:fenilalanin,P:prolin,S:serin,T:treonin, W:triptofán,Y:tirozin
45 Fehérjepontozásirendszerek(mátrixok) pontszámotrendelazösszeslehetségesaminosav aminosavcseréhez fehérjeszekvenciáktöbbszörösillesztésénekvizsgálatábólszármazóadatok Blossum62 esmátrix
46 Szekvenciaillesztés:globálisvagylokális Globálisillesztés:ateljesszekvenciátigyekszikoptimálisanelrendezni Σ 50pont Lokális:alegnagyobbjólilleszkedőközösszakasztkeresimeg Σ 55pont Természetesenakétmódszereltérőillesztéstad
47 BLAST:BasicLocalAlignmentTool BLAST:egyszerűlokálisszekvenciaillesztőeszköz azncbiportálonhozzáférhető: igengyors,igenelterjedt alkalmasnagyméretűszekvenciaadatbázisokbantörténőhomológiakeresésre programvariációk: szekvencia adatbázis program nukleotid nukleotid blastn fehérje fehérje blastp transzlált nukleotid fehérje blastx fehérje transzlált nukleotid tblastn transzlált nukleotid transzlált nukleotid tblastn
48 BLAST:BasicLocalAlignmentTool keresett szekvencia (query) Választhatófehérjeblastadatbankok: melyik adatbankban keressen nr ismétlődéstőlmentes,~genepept refseq jóljellemzett,felülvizsgáltadatok(ncbi) swissprot jóljellemzett,felülvizsgáltadatok(swissinstitute) pat szabadalmakhozkacspolódószekvenciák pdb ismert3d smodellelrendelkezőszekvenciák env környezetiszekvenálásokeredményei
49 Keresésfolyamatban... Becsülthátralévőidő(minimum)
50 Eredmények... Fajokszerintrendezve találatok Pontszám színkód
51 Eredmények... találatneve+hivatkozás találatleírása Eérték megengedettcsere azonosaminosavak deléció(gap) nemmegengedettcsere
52 Milyeninformációkatkaphatunkfehérjeszekvenciák vizsgálatával Afőkérdés:miazadottfehérjepontosszerepe,funkciója? Segítt easzekvenciaismereteafunkciómeghatározásában?
53 Fehérjeaminosavsorrendmeghatározzaatérszerkezetet Anfinsen,1961 UreahatásáraazRNázkicsapódik,(harmadlagostérszerkezeteelvész) AzureaeltávolításautánazRNázkülsősegítségnélkülvisszanyerteatérszerkezetét, ésazaktivitását! katalitikuszseb diszulfidhidak hidrofiloldalláncok hidrofóbmag
54 Fehérjeszekvenciaanalízisrévénfunkciójóslás Hasonlószekvenciakereséseadatbázisban,ismertfunkcióval Hasonlószekvenciakereséseadatbázisban,ismerttérszerkezettel Ismertfunkcióvalbíródomain ekazonosításaazismeretlenszekvencián Funkciójóslás Pusztánszekvenciaanalízisselafehérjefunkciójátnemlehetmegállapítani Abioinformatikaivizsgálatokötleteket,kiindulópontotadnakakísérletes munkához
55 ProteinDataBank Kísérletesenmeghatározottháromdimenziósfehérjeszerkezetimodellek
56 KeresésaPDBadatbázisban
57 CDDkonzerváltdomainadatbank Domain:afehérjénbelülirészegység,amelyjóldefiniáltstrukturálisvagyfunkcióbeli szerepettöltbe.egyfehérjénbelülgyakrantöbbdomainttalálunk,amelyek együttesenjárulnakhozzáafehérjeműködéséhez
58 CDDkonzerváltdomainadatbanktalálat azonosítottaktívközpont,szubsztrátkötőhelyek azonosítottdomain ek
59 NCBIportál azinformációözöne
60 Entrez:azNCBIintegráltkeresőmotorja OMIM PubMed PubMedCentral 3DDomains Journals Structure Books CDD/CDART Entrez Protein Taxonomy Genome GEO/GDS UniSTS UniGene Nucleotide PopSet SNP
61 Szakirodalmiadatbázis:Pubmed közel5300tudományosfolyóiratcikkeinekösszefoglalóibankereshetünk aszabadonletölthetőteljescikkekrehivatkozás
62 Szakirodalmiadatbázis:Pubmed amegtaláltösszefoglalómunkákat(reviewarticle)különiskilistázhatjuk
63 Szakirodalmiadatbázis:Pubmed különféleikonokjelzik,hogyamegtaláltteljescikkhozzáférhető e ingyenes hozzáférés
64 PubmedCentral 500szabadon,elektronikusanelérhetőfolyóirat
65 Mapviewer interaktívgenetikaitérképekazelkészültésafolyamatbanlévőgenomprojektekhez
66 Mapviewer Kulcsszavaskeresés találatokakromoszómákon
67 HumángenetikaiadatbázisOMIM Örökletesbetegségekkelkapcsolatosinformációk
68 Rendszertaniadatbázis(taxonómia)
69 Szabadonolvashatókönyvek:NCBIBooks
70 Szabadonolvashatókönyvek:NCBIBooks Berg,JeremyM.;Tymoczko,JohnL.;andStryer,Lubert. NewYork:W.H.FreemanandCo.;c2002 Biochemistry Cooper,GeoffreyM. Sunderland(MA):SinauerAssociates,Inc.;c2000 TheCell AMolecularApproach Gilbert,ScottF. Sunderland(MA):SinauerAssociates,Inc.;c2000 DevelopmentalBiology Janeway,CharlesA.;Travers,Paul;Walport,Mark;Shlomchik,Mark NewYorkandLondon:GarlandScience;c2001 Immunobiology Lodish,Harvey;Berk,Arnold;Zipursky,S.Lawrence; Matsudaira,Paul;Baltimore,David;Darnell,JamesE. NewYork:W.H.Freeman&Co.;c1999 MolecularCellBiology Coffin,JohnM.;Hughes,StephenH.;Varmus,HaroldE. Plainview(NY):ColdSpringHarborLaboratoryPress;c1997 Retroviruses
71 Egyébhasznosadatbankok:BRENDAenzimadatbázis Átfogóadatgyűjteményenzimekről azenzimhelyeametabolikushálózatban azenzimáltalkatalizáltreakciókleírása előforduláskülönféleélőlényekben,irodalmihivatkozások aktivitásadatok,enzimkinetikaiadatok optimálishőmérséklet,phadatok gátlószerekhatása enzymes.org/
72 KEGGanyagcsereútvonaladatbázis
Bioinformatika és genomanalízis az orvostudományban. Biológiai adatbázisok. Cserző Miklós 2018
 Bioinformatika és genomanalízis az orvostudományban Biológiai adatbázisok Cserző Miklós 2018 A mai előadás Mi az adatbázis A biológia kapcsolata az adatbázisokkal Az adatbázisok típusai Adatbázis formátumok,
Bioinformatika és genomanalízis az orvostudományban Biológiai adatbázisok Cserző Miklós 2018 A mai előadás Mi az adatbázis A biológia kapcsolata az adatbázisokkal Az adatbázisok típusai Adatbázis formátumok,
Juhász Angéla MTA ATK MI Alkalmazott Genomikai Osztály SZEKVENCIA ADATBÁZISOK
 Juhász Angéla MTA ATK MI Alkalmazott Genomikai Osztály SZEKVENCIA ADATBÁZISOK Fehérjét kódol? Tulajdonságai? -Hol lokalizálódik? -Oldható? -3D szerkezete? -Accession #? -Annotációja elérhető? Már benne
Juhász Angéla MTA ATK MI Alkalmazott Genomikai Osztály SZEKVENCIA ADATBÁZISOK Fehérjét kódol? Tulajdonságai? -Hol lokalizálódik? -Oldható? -3D szerkezete? -Accession #? -Annotációja elérhető? Már benne
Molekuláris biológiai adatbázisok és adatbázis keresések. Barta Endre Tóth Gábor MBK Bioinformatikai Csoport
 Molekuláris biológiai adatbázisok és adatbázis keresések Barta Endre Tóth Gábor MBK Bioinformatikai Csoport Adatbázisok: megvalósítás Szöveges adatbázis általában szekvenciális, néha indexelt megfelelő
Molekuláris biológiai adatbázisok és adatbázis keresések Barta Endre Tóth Gábor MBK Bioinformatikai Csoport Adatbázisok: megvalósítás Szöveges adatbázis általában szekvenciális, néha indexelt megfelelő
Appendix. A. DNA sequences of the UZ3/4 TCR chains. Appendix 116. TCR alpha 7 chain
 Appendix 116 Appendix A. DNA sequences of the UZ3/4 TCR chains TCR alpha 7 chain LOCUS TCR alpha 822 bp DNA ACCESSION TCR alpha 7 KEYWORDS L + recombined V alpha 7.2 J alpha 18 + C alpha SOURCE CD8+ T
Appendix 116 Appendix A. DNA sequences of the UZ3/4 TCR chains TCR alpha 7 chain LOCUS TCR alpha 822 bp DNA ACCESSION TCR alpha 7 KEYWORDS L + recombined V alpha 7.2 J alpha 18 + C alpha SOURCE CD8+ T
NÖVÉNYI GENOMIKA JÓRI BALÁZS
 NÖVÉNYI GENOMIKA JÓRI BALÁZS Eötvös Loránd Tudományegyetem, Növényélettani és Molekuláris Növénybiológia Tanszék, 1117 Budapest, Pázmány P. sétány 1/c. Elfogadva: 2004. december 29. Bot. Közlem. 91(1 2):
NÖVÉNYI GENOMIKA JÓRI BALÁZS Eötvös Loránd Tudományegyetem, Növényélettani és Molekuláris Növénybiológia Tanszék, 1117 Budapest, Pázmány P. sétány 1/c. Elfogadva: 2004. december 29. Bot. Közlem. 91(1 2):
A tárgy címe: Bioinformatika
 A tárgy címe: Bioinformatika Kötelezően választható tárgy IV. és V. évfolyamos biológus hallgatók számára; heti 2+3 óra Előkövetelmény: Biokémia főkollégium; genetika főkollégium; alapszintű számítógépes
A tárgy címe: Bioinformatika Kötelezően választható tárgy IV. és V. évfolyamos biológus hallgatók számára; heti 2+3 óra Előkövetelmény: Biokémia főkollégium; genetika főkollégium; alapszintű számítógépes
Human genome project
 Human genome project Pataki Bálint Ármin 2017.03.14. Pataki Bálint Ármin Human genome project 2017.03.14. 1 / 14 Agenda 1 Biológiai bevezető 2 A human genome project lefolyása 3 Alkalmazások, kitekintés
Human genome project Pataki Bálint Ármin 2017.03.14. Pataki Bálint Ármin Human genome project 2017.03.14. 1 / 14 Agenda 1 Biológiai bevezető 2 A human genome project lefolyása 3 Alkalmazások, kitekintés
Bakteriális identifikáció 16S rrns gén szekvencia alapján
 Bakteriális identifikáció 16S rrns gén szekvencia alapján MOHR ANITA SIPOS RITA, SZÁNTÓ-EGÉSZ RÉKA, MICSINAI ADRIENN 2100 Gödöllő, Szent-Györgyi Albert út 4. info@biomi.hu, www.biomi.hu TÖRZS AZONOSÍTÁS
Bakteriális identifikáció 16S rrns gén szekvencia alapján MOHR ANITA SIPOS RITA, SZÁNTÓ-EGÉSZ RÉKA, MICSINAI ADRIENN 2100 Gödöllő, Szent-Györgyi Albert út 4. info@biomi.hu, www.biomi.hu TÖRZS AZONOSÍTÁS
Manuscript Title: Identification of a thermostable fungal lytic polysaccharide monooxygenase and
 1 2 3 4 5 Journal name: Applied Microbiology and Biotechnology Manuscript Title: Identification of a thermostable fungal lytic polysaccharide monooxygenase and evaluation of its effect on lignocellulosic
1 2 3 4 5 Journal name: Applied Microbiology and Biotechnology Manuscript Title: Identification of a thermostable fungal lytic polysaccharide monooxygenase and evaluation of its effect on lignocellulosic
A bioinformatika gyökerei
 A bioinformatika gyökerei 1944: Avery a transforming principle a DNS 1952: Hershey és Chase perdöntő bizonyíték: a bakteriofágok szaporodásakor csak a DNS jut be a sejtbe 1953: Watson és Crick a DNS szerkezete
A bioinformatika gyökerei 1944: Avery a transforming principle a DNS 1952: Hershey és Chase perdöntő bizonyíték: a bakteriofágok szaporodásakor csak a DNS jut be a sejtbe 1953: Watson és Crick a DNS szerkezete
ÚJ GENERÁCIÓS SZEKVENÁLÁS
 VÍZMIKROBIOLÓGUSOK XI. ORSZÁGOS KONFERENCIÁJA - 2012 ÚJ GENERÁCIÓS SZEKVENÁLÁS LEHETŐSÉG, VAGY NEHÉZSÉG? MÁRIALIGETI KÁROLY EÖTVÖS LORÁND TUDOMÁNYEGYETEM, BUDAPEST 1953 JAMES D. WATSON, FRANCIS CRICK:
VÍZMIKROBIOLÓGUSOK XI. ORSZÁGOS KONFERENCIÁJA - 2012 ÚJ GENERÁCIÓS SZEKVENÁLÁS LEHETŐSÉG, VAGY NEHÉZSÉG? MÁRIALIGETI KÁROLY EÖTVÖS LORÁND TUDOMÁNYEGYETEM, BUDAPEST 1953 JAMES D. WATSON, FRANCIS CRICK:
DNS-szekvencia meghatározás
 DNS-szekvencia meghatározás Gilbert 1980 (1958) Sanger 3-1 A DNS-polimerázok jellemzői 5'-3' polimeráz aktivitás 5'-3' exonukleáz 3'-5' exonukleáz aktivitás Az új szál szintéziséhez kell: templát DNS primer
DNS-szekvencia meghatározás Gilbert 1980 (1958) Sanger 3-1 A DNS-polimerázok jellemzői 5'-3' polimeráz aktivitás 5'-3' exonukleáz 3'-5' exonukleáz aktivitás Az új szál szintéziséhez kell: templát DNS primer
Bioinformatika 2 4. előadás
 4. előadás Prof. Poppe László BME Szerves Kémia és Technológia Tsz. Bioinformatika proteomika Előadás és gyakorlat 2018.09.24. Biológiai adatbázisok Felhasználó Keresõprogram BLAST Biológiai adatbázisok
4. előadás Prof. Poppe László BME Szerves Kémia és Technológia Tsz. Bioinformatika proteomika Előadás és gyakorlat 2018.09.24. Biológiai adatbázisok Felhasználó Keresõprogram BLAST Biológiai adatbázisok
Orvosi Genomtudomány 2014 Medical Genomics 2014. Április 8 Május 22 8th April 22nd May
 Orvosi Genomtudomány 2014 Medical Genomics 2014 Április 8 Május 22 8th April 22nd May Hét / 1st week (9. kalendariumi het) Takács László / Fehér Zsigmond Magyar kurzus Datum/ido Ápr. 8 Apr. 9 10:00 10:45
Orvosi Genomtudomány 2014 Medical Genomics 2014 Április 8 Május 22 8th April 22nd May Hét / 1st week (9. kalendariumi het) Takács László / Fehér Zsigmond Magyar kurzus Datum/ido Ápr. 8 Apr. 9 10:00 10:45
 ő í í ü í ö ú í ö ú ö í ú ő í Ó ő ü í Í ö ö Í í í í í Í í ű ő ö í ő ö ö íá í íí í ő ö ő Í ö Ó ö ö ü ö ö ö ő É í í Í ő ő ő ő ő ő ő ő ö ú ő ú ú ő ö ö ú ú ö ú í ő Ó ö ő Í í ü í ö ú ő ö ő ú ő í ő ö ü Í í ö
ő í í ü í ö ú í ö ú ö í ú ő í Ó ő ü í Í ö ö Í í í í í Í í ű ő ö í ő ö ö íá í íí í ő ö ő Í ö Ó ö ö ü ö ö ö ő É í í Í ő ő ő ő ő ő ő ő ö ú ő ú ú ő ö ö ú ú ö ú í ő Ó ö ő Í í ü í ö ú ő ö ő ú ő í ő ö ü Í í ö
A HUMÁN GENOM PROJEKT Sasvári-Székely Mária* Semmelweis Egyetem, Orvosi Vegytani, Molekuláris Biológiai és Pathobiokémiai Intézet
 A HUMÁN GENOM PROJEKT Sasvári-Székely Mária* Semmelweis Egyetem, Orvosi Vegytani, Molekuláris Biológiai és Pathobiokémiai Intézet *Levelezési cím: Dr. Sasvári-Székely Mária, Semmelweis Egyetem, Orvosi
A HUMÁN GENOM PROJEKT Sasvári-Székely Mária* Semmelweis Egyetem, Orvosi Vegytani, Molekuláris Biológiai és Pathobiokémiai Intézet *Levelezési cím: Dr. Sasvári-Székely Mária, Semmelweis Egyetem, Orvosi
GENOMIKA TÖBBFÉLE MAKROMOLEKULA VIZSGÁLATA EGYIDŐBEN
 GENOMIKA TÖBBFÉLE MAKROMOLEKULA VIZSGÁLATA EGYIDŐBEN Strukturális genomika Genomkönyvtárak DNS szekvenálás Genom programok Polimorfizmusok RFLP DNS könyvtár készítés humán genom 1. Emésztés RE-kal Emberi
GENOMIKA TÖBBFÉLE MAKROMOLEKULA VIZSGÁLATA EGYIDŐBEN Strukturális genomika Genomkönyvtárak DNS szekvenálás Genom programok Polimorfizmusok RFLP DNS könyvtár készítés humán genom 1. Emésztés RE-kal Emberi
Forensic SNP Genotyping using Nanopore MinION Sequencing
 Forensic SNP Genotyping using Nanopore MinION Sequencing AUTHORS Senne Cornelis 1, Yannick Gansemans 1, Lieselot Deleye 1, Dieter Deforce 1,#,Filip Van Nieuwerburgh 1,#,* 1 Laboratory of Pharmaceutical
Forensic SNP Genotyping using Nanopore MinION Sequencing AUTHORS Senne Cornelis 1, Yannick Gansemans 1, Lieselot Deleye 1, Dieter Deforce 1,#,Filip Van Nieuwerburgh 1,#,* 1 Laboratory of Pharmaceutical
Makromolekulák. Fehérjetekeredé. rjetekeredés. Biopolimer. Polimerek
 Biopolimerek Makromolekulá Makromolekulák. Fehé Fehérjetekeredé rjetekeredés. Osztódó sejt magorsófonala 2011. November 16. Huber Tamá Tamás Dohány levél epidermális sejtjének aktin hálózata Bakteriofágból
Biopolimerek Makromolekulá Makromolekulák. Fehé Fehérjetekeredé rjetekeredés. Osztódó sejt magorsófonala 2011. November 16. Huber Tamá Tamás Dohány levél epidermális sejtjének aktin hálózata Bakteriofágból
Biológus MSc. Molekuláris biológiai alapismeretek
 Biológus MSc Molekuláris biológiai alapismeretek A nukleotidok építőkövei A nukleotidok szerkezete Nukleotid = N-tartalmú szerves bázis + pentóz + foszfát N-glikozidos kötés 5 1 4 2 3 (Foszfát)észter-kötés
Biológus MSc Molekuláris biológiai alapismeretek A nukleotidok építőkövei A nukleotidok szerkezete Nukleotid = N-tartalmú szerves bázis + pentóz + foszfát N-glikozidos kötés 5 1 4 2 3 (Foszfát)észter-kötés
Genetika. Tartárgyi adatlap: tantárgy adatai
 Genetika Előadás a I. éves Génsebészet szakos hallgatók számára Tartárgyi adatlap: tantárgy adatai 2.1. Tantárgy címe Genetika 2.2. Előadás felelőse Dr. Mara Gyöngyvér, docens 2.3. Egyéb oktatási tevékenységek
Genetika Előadás a I. éves Génsebészet szakos hallgatók számára Tartárgyi adatlap: tantárgy adatai 2.1. Tantárgy címe Genetika 2.2. Előadás felelőse Dr. Mara Gyöngyvér, docens 2.3. Egyéb oktatási tevékenységek
Genetikai panel kialakítása a hazai tejhasznú szarvasmarha állományok hasznos élettartamának növelésére
 Genetikai panel kialakítása a hazai tejhasznú szarvasmarha állományok hasznos élettartamának növelésére Dr. Czeglédi Levente Dr. Béri Béla Kutatás-fejlesztés támogatása a megújuló energiaforrások és agrár
Genetikai panel kialakítása a hazai tejhasznú szarvasmarha állományok hasznos élettartamának növelésére Dr. Czeglédi Levente Dr. Béri Béla Kutatás-fejlesztés támogatása a megújuló energiaforrások és agrár
Bioinformatika előadás
 Bioinformatika 2 11. előadás Prof. Poppe László BME Szerves Kémia és Technológia Tsz. Bioinformatika proteomika Előadás és gyakorlat 2016.11.28. Bioinformatics Szerkezeti genomika, proteomika, biológia
Bioinformatika 2 11. előadás Prof. Poppe László BME Szerves Kémia és Technológia Tsz. Bioinformatika proteomika Előadás és gyakorlat 2016.11.28. Bioinformatics Szerkezeti genomika, proteomika, biológia
10. CSI. A molekuláris biológiai technikák alkalmazásai
 10. CSI. A molekuláris biológiai technikák alkalmazásai A DNS mint azonosító 3 milliárd bázispár az emberi DNS-ben (99.9%-ban azonos) 0.1%-nyi különbség elegendő az egyedek megkülönböztetéséhez Genetikai
10. CSI. A molekuláris biológiai technikák alkalmazásai A DNS mint azonosító 3 milliárd bázispár az emberi DNS-ben (99.9%-ban azonos) 0.1%-nyi különbség elegendő az egyedek megkülönböztetéséhez Genetikai
Bevezetés a bioinformatikába. Harangi János DE, TEK, TTK Biokémiai Tanszék
 Bevezetés a bioinformatikába Harangi János DE, TEK, TTK Biokémiai Tanszék Bioinformatika Interdiszciplináris tudomány, amely magába foglalja a biológiai adatok gyűjtésének,feldolgozásának, tárolásának,
Bevezetés a bioinformatikába Harangi János DE, TEK, TTK Biokémiai Tanszék Bioinformatika Interdiszciplináris tudomány, amely magába foglalja a biológiai adatok gyűjtésének,feldolgozásának, tárolásának,
Biokémiai kutatások ma
 Nyitray László Biokémiai Tanszék Hb Biokémiai kutatások ma Makromolekulák szerkezet-funkció kutatása Molekuláris biológia minden szinten Redukcionista molekuláris biológia vs. holisztikus rendszerbiológia
Nyitray László Biokémiai Tanszék Hb Biokémiai kutatások ma Makromolekulák szerkezet-funkció kutatása Molekuláris biológia minden szinten Redukcionista molekuláris biológia vs. holisztikus rendszerbiológia
TDK lehetőségek az MTA TTK Enzimológiai Intézetben
 TDK lehetőségek az MTA TTK Enzimológiai Intézetben Vértessy G. Beáta egyetemi tanár TDK mind 1-3 helyezettek OTDK Pro Scientia különdíj 1 második díj Diákjaink Eredményei Zsűri különdíj 2 első díj OTDK
TDK lehetőségek az MTA TTK Enzimológiai Intézetben Vértessy G. Beáta egyetemi tanár TDK mind 1-3 helyezettek OTDK Pro Scientia különdíj 1 második díj Diákjaink Eredményei Zsűri különdíj 2 első díj OTDK
A Multi Locus Sequence Typing (MLST) alkalmazhatósága az élelmiszermikrobiológiában
 A Multi Locus Sequence Typing (MLST) alkalmazhatósága az élelmiszermikrobiológiában Sipos Rita, Lukács Alena, Simon Janka, Szántó-Egész Réka, Micsinai Adrienn 2100 Gödöllő, Szent-Györgyi Albert út 4. info@biomi.hu,
A Multi Locus Sequence Typing (MLST) alkalmazhatósága az élelmiszermikrobiológiában Sipos Rita, Lukács Alena, Simon Janka, Szántó-Egész Réka, Micsinai Adrienn 2100 Gödöllő, Szent-Györgyi Albert út 4. info@biomi.hu,
MIT TEHET A FIZIKUS A RÁKKUTATÁSÉRT? Pipek Orsolya ELTE TTK Komplex rendszerek fizikája tanszék. Atomoktól a csillagokig, Budapest, február 23.
 MIT TEHET A FIZIKUS A RÁKKUTATÁSÉRT? Pipek Orsolya ELTE TTK Komplex rendszerek fizikája tanszék Atomoktól a csillagokig, Budapest, 2017. február 23. Pipek Orsolya, ELTE TTK Komplex rendszerek fizikája
MIT TEHET A FIZIKUS A RÁKKUTATÁSÉRT? Pipek Orsolya ELTE TTK Komplex rendszerek fizikája tanszék Atomoktól a csillagokig, Budapest, 2017. február 23. Pipek Orsolya, ELTE TTK Komplex rendszerek fizikája
TEMATIKA Biokémia és molekuláris biológia IB kurzus (bb5t1301)
 Biokémia és molekuláris biológia I. kurzus (bb5t1301) Tematika 1 TEMATIKA Biokémia és molekuláris biológia IB kurzus (bb5t1301) 0. Bevezető A (a biokémiáról) (~40 perc: 1. heti előadás) A BIOkémia tárgya
Biokémia és molekuláris biológia I. kurzus (bb5t1301) Tematika 1 TEMATIKA Biokémia és molekuláris biológia IB kurzus (bb5t1301) 0. Bevezető A (a biokémiáról) (~40 perc: 1. heti előadás) A BIOkémia tárgya
Molekuláris evolúció második gyakorlat
 Molekuláris evolúció második gyakorlat Szekvenciák illesztése (alignment készítés) Szekvenciák szerkesztése Programok: ClustalX (http://evolution.genetics.washington.edu/phylip/software.html) GeneDoc (http://www.psc.edu/biomed/genedoc/)
Molekuláris evolúció második gyakorlat Szekvenciák illesztése (alignment készítés) Szekvenciák szerkesztése Programok: ClustalX (http://evolution.genetics.washington.edu/phylip/software.html) GeneDoc (http://www.psc.edu/biomed/genedoc/)
Genomadatbázisok Ld. Entrez Genome: Összes ismert genom, hierarchikus szervezésben (kromoszóma, térképek, gének, stb.)
 Genomika Új korszak, paradigmaváltás, forradalom: a teljes genomok ismeretében a biológia adatokban gazdag tudománnyá válik. Új kutatási módszerek, új szemlélet. Hajtóerõk: Genomszekvenálási projektek
Genomika Új korszak, paradigmaváltás, forradalom: a teljes genomok ismeretében a biológia adatokban gazdag tudománnyá válik. Új kutatási módszerek, új szemlélet. Hajtóerõk: Genomszekvenálási projektek
I. Strukturális Genomika II. Funkcionális Genomika III. Integratív Genomika
 I. Strukturális Genomika II. Funkcionális Genomika III. Integratív Genomika A genomika főbb területei Strukturális (szerkezeti ) genomika Funkcionális (működési) genomika Transzkriptomika Proteomika Integratív
I. Strukturális Genomika II. Funkcionális Genomika III. Integratív Genomika A genomika főbb területei Strukturális (szerkezeti ) genomika Funkcionális (működési) genomika Transzkriptomika Proteomika Integratív
 ö Á ö É É ü ü É É Ő ö É ö Á ó ü É Ó Ö Á ú é ü ö é Ö é ü é é ü ü é é Ü é ö ö Ö ö é Á é é é é é ó é é é é ü é ö ö ö í é ü ú é é é ü ü é é é ü é é ö é ö é é ó ö ü é é é é ó ó ö í ó é ó é é é ó é é é ű ö é
ö Á ö É É ü ü É É Ő ö É ö Á ó ü É Ó Ö Á ú é ü ö é Ö é ü é é ü ü é é Ü é ö ö Ö ö é Á é é é é é ó é é é é ü é ö ö ö í é ü ú é é é ü ü é é é ü é é ö é ö é é ó ö ü é é é é ó ó ö í ó é ó é é é ó é é é ű ö é
 Á Ó Á Ü ő ű Ú ö í ő Ó ú ö Á ú Ű Ó ű Ó í ű ö í ö ő ö ö í ö ö ő É ö Á ű Ó ö Á Ó ö í Á í í ö ű ö ú ö ö ú ö Ú ö ű Ó Ú ö Á í Ó í í Í í í Í ö Ú ö Á ú í Ó ő í ú ö Á ú Á í ú ö Á ú í ö Á ú í Ó ö ű Ó Ú Ú ű ő ö ü
Á Ó Á Ü ő ű Ú ö í ő Ó ú ö Á ú Ű Ó ű Ó í ű ö í ö ő ö ö í ö ö ő É ö Á ű Ó ö Á Ó ö í Á í í ö ű ö ú ö ö ú ö Ú ö ű Ó Ú ö Á í Ó í í Í í í Í ö Ú ö Á ú í Ó ő í ú ö Á ú Á í ú ö Á ú í ö Á ú í Ó ö ű Ó Ú Ú ű ő ö ü
 Á Á É Á Ü ö ű ű ő í ő ö ő í ő ö í É ő í ű ö ő ő í ö ü ő ő ü ő ü í ö ö ü ö ü ő ő ü ü ő ü ö ő ő ő ő íő ö ö ö ü ő ő ő ő í ú ő ő í ü ö ő í ű ü ö ő ő ő ő í ú ö ö ő ö ö ö ö ü ő ő ö ő ő í í ő ö ü ö í ö ö ö ö
Á Á É Á Ü ö ű ű ő í ő ö ő í ő ö í É ő í ű ö ő ő í ö ü ő ő ü ő ü í ö ö ü ö ü ő ő ü ü ő ü ö ő ő ő ő íő ö ö ö ü ő ő ő ő í ú ő ő í ü ö ő í ű ü ö ő ő ő ő í ú ö ö ő ö ö ö ö ü ő ő ö ő ő í í ő ö ü ö í ö ö ö ö
 ó Í ó ó Ü ó ő Ú ő É ó É Í ő Ö ő ő ó Íó ó Ú ó É Ö ó ő ő Ú Íő ő ő ő ő ő Ú ő ó ó ő ő ő ő ó ő ő ő ő ő ő Í ő ő ó ő ő ó ő Í ő ó ő ő ő ő ő ó ó ó ő ő ó ő ő ő ő ő ő ó ő ő ő ó ő ő Á ű ő ő ő ő ő ő Í ó ő ő ő ő ó ó
ó Í ó ó Ü ó ő Ú ő É ó É Í ő Ö ő ő ó Íó ó Ú ó É Ö ó ő ő Ú Íő ő ő ő ő ő Ú ő ó ó ő ő ő ő ó ő ő ő ő ő ő Í ő ő ó ő ő ó ő Í ő ó ő ő ő ő ő ó ó ó ő ő ó ő ő ő ő ő ő ó ő ő ő ó ő ő Á ű ő ő ő ő ő ő Í ó ő ő ő ő ó ó
 Á Á Í ó ó ó ö ó Ü ö ú Í ó ö ö ó ú ö ó ö ö Ü ö ú ó ó ó ó ö ü ó ö ö ü Ü ö ö ú ó ó ö ú ö ó ó ó ó ö ó ö ó ö ó ö ű ö ö ö ű ö ö ű ö ö ö ű ö ö ó ö ö ó ó ü ö ö ű ö ö ö ó ö ű ö Ü ö ö ú ó ö ó ü ü ö ü ü ö Í ö ü ö
Á Á Í ó ó ó ö ó Ü ö ú Í ó ö ö ó ú ö ó ö ö Ü ö ú ó ó ó ó ö ü ó ö ö ü Ü ö ö ú ó ó ö ú ö ó ó ó ó ö ó ö ó ö ó ö ű ö ö ö ű ö ö ű ö ö ö ű ö ö ó ö ö ó ó ü ö ö ű ö ö ö ó ö ű ö Ü ö ö ú ó ö ó ü ü ö ü ü ö Í ö ü ö
 ó ő ó ó ö ö ú Á Í ö ó ő ö ú Í ó ü ó ő ö ú ö ó ő ó ő ü ő ű ö ö ü ő ü ó Ó ö ó ó ő ő ő ö Í ó ö ö ö ó ő ö ő Í ü ö ö ö ö ö ö ő ö ö ö ö ú ú ű ö ű ó ó ö ö ő ű ö ú ö ö ö ö ö ó Á ö ö ö ő ő ó ő ő Ö ő ú ó ö ú ú ű
ó ő ó ó ö ö ú Á Í ö ó ő ö ú Í ó ü ó ő ö ú ö ó ő ó ő ü ő ű ö ö ü ő ü ó Ó ö ó ó ő ő ő ö Í ó ö ö ö ó ő ö ő Í ü ö ö ö ö ö ö ő ö ö ö ö ú ú ű ö ű ó ó ö ö ő ű ö ú ö ö ö ö ö ó Á ö ö ö ő ő ó ő ő Ö ő ú ó ö ú ú ű
 É ő ő íí í ú í ő Ő ő ü ü ü ü ü Ü Ü ő ő ő ő í ő ő ő í íí í ő ű í Ó Ó Ó í Ö Ö í Á Ö Ü Ö É í Ö í ő Ö Ö Ö Á í Á ő ő ő ő É Í Í ő ú Ú ú Ö í ő Á Ö ő Í Í ő ű í ő ú ü íí í Ö ő ő ő ő Í ő ő ő ő í ő ő ő ő í É É í
É ő ő íí í ú í ő Ő ő ü ü ü ü ü Ü Ü ő ő ő ő í ő ő ő í íí í ő ű í Ó Ó Ó í Ö Ö í Á Ö Ü Ö É í Ö í ő Ö Ö Ö Á í Á ő ő ő ő É Í Í ő ú Ú ú Ö í ő Á Ö ő Í Í ő ű í ő ú ü íí í Ö ő ő ő ő Í ő ő ő ő í ő ő ő ő í É É í
 ö Ö ö Ö ö ö ö ö ö ö ö Ö ö Ö ö ö ö ö ö ű ö ö ö ö Ö ö Ő Ü ö ö Ö Ö ö ö ö ö ö ö ö ö Ü ö ö ö ű ö ö ö ö ű ö ű ö Ö Ü Ü ö ö ú Ű ÍŐ Ö Ő ÍŐ ö ö ö ö ű ö Ö Ö Ó ö ö Ö ö ö Ö ö ö Ö ö ű ö ö É ö ö Í Á Á Ő ű ö ű ú Ö Ü Á
ö Ö ö Ö ö ö ö ö ö ö ö Ö ö Ö ö ö ö ö ö ű ö ö ö ö Ö ö Ő Ü ö ö Ö Ö ö ö ö ö ö ö ö ö Ü ö ö ö ű ö ö ö ö ű ö ű ö Ö Ü Ü ö ö ú Ű ÍŐ Ö Ő ÍŐ ö ö ö ö ű ö Ö Ö Ó ö ö Ö ö ö Ö ö ö Ö ö ű ö ö É ö ö Í Á Á Ő ű ö ű ú Ö Ü Á
 í ö Ö Á í ö í í ö í ö ö í í ö ö ö ö í í ö í ö í ö í ü í í ö í í í í í ö ö í í í ú ö í í ö Á Á Á ü ú í ö Á í í í ö í í ü ö ö ö ö í ö í í í ú í í ű ú í í í í ö í ű í ö ö ü ö ű ö ö í í í í í ö ü í ö í ö ű
í ö Ö Á í ö í í ö í ö ö í í ö ö ö ö í í ö í ö í ö í ü í í ö í í í í í ö ö í í í ú ö í í ö Á Á Á ü ú í ö Á í í í ö í í ü ö ö ö ö í ö í í í ú í í ű ú í í í í ö í ű í ö ö ü ö ű ö ö í í í í í ö ü í ö í ö ű
 ű í ö ö Á ü ü ö ö ö í í É ú ú ö ö ű í ö ü ö ú ü ű ú ö í í ú ö ú í ö ü í í ö í Á Ó É í ű ö ü ö ü ú ü ö ü ú ű ö ü ű ü í ü ű ü ü ö ű í ü í ö ü í í í í ö í ö ö ö Á ű ú ű ö ö ű í ö ö í ú í í ű í ö ú ö ö í Á
ű í ö ö Á ü ü ö ö ö í í É ú ú ö ö ű í ö ü ö ú ü ű ú ö í í ú ö ú í ö ü í í ö í Á Ó É í ű ö ü ö ü ú ü ö ü ú ű ö ü ű ü í ü ű ü ü ö ű í ü í ö ü í í í í ö í ö ö ö Á ű ú ű ö ö ű í ö ö í ú í í ű í ö ú ö ö í Á
 ö é Ö é ü ö é ü ö é Ö é ü í ü ü ü é é ü é é Ö ö é é é é ö ü ö ü ö é é ö é é ö é é ö ö é í é ü é é é í é ö é é ö é ö é ü é ü ú é é é é é í é é é é ö ö é é ö ö é é í í é í é ü ö ü Á é ö Á í ö í é ö ü ö é
ö é Ö é ü ö é ü ö é Ö é ü í ü ü ü é é ü é é Ö ö é é é é ö ü ö ü ö é é ö é é ö é é ö ö é í é ü é é é í é ö é é ö é ö é ü é ü ú é é é é é í é é é é ö ö é é ö ö é é í í é í é ü ö ü Á é ö Á í ö í é ö ü ö é
 É ö ö Í Í Í Ó Í Í Á Ó Á Ü Ú Í Á Á ű Á Ó Í Í É Á Ó Á Á ö ö Á Í Á Á ö ö ű ö ö Í Í ű Ö ű ö ö ű Í Í Ü ö ö Ó ű Í ö ö Í ö ö Ó ö Ö Í ö ö Ö ö ű ö ö Ó Í ű Ó ö ö ű ö ű Ö Ü Ö ű ű ö ö ö ö ö ö Íö ö Í Ö Ó ű ö ű ö ö
É ö ö Í Í Í Ó Í Í Á Ó Á Ü Ú Í Á Á ű Á Ó Í Í É Á Ó Á Á ö ö Á Í Á Á ö ö ű ö ö Í Í ű Ö ű ö ö ű Í Í Ü ö ö Ó ű Í ö ö Í ö ö Ó ö Ö Í ö ö Ö ö ű ö ö Ó Í ű Ó ö ö ű ö ű Ö Ü Ö ű ű ö ö ö ö ö ö Íö ö Í Ö Ó ű ö ű ö ö
 Ő Ö Ü Ö Ö ő ü ó í ü ü ő ü ó Ö ó ő ó ó ő ó ő í ő í ü ő ö ö ö ü í ü ö ö ö ö Ö ő ő Ö ő í ó ő ó ő Ö í ő ő ő ő ü ő ő ö ó ű ö ó ö ú ő ő ó ü ö í ü ö ö ó í ú ő ó ő í ö ö ö í ő ö ő ő ó ü ö ú ü ő ó ó ő ó ő ó í í
Ő Ö Ü Ö Ö ő ü ó í ü ü ő ü ó Ö ó ő ó ó ő ó ő í ő í ü ő ö ö ö ü í ü ö ö ö ö Ö ő ő Ö ő í ó ő ó ő Ö í ő ő ő ő ü ő ő ö ó ű ö ó ö ú ő ő ó ü ö í ü ö ö ó í ú ő ó ő í ö ö ö í ő ö ő ő ó ü ö ú ü ő ó ó ő ó ő ó í í
 É É É Ó Ö É í Ö ő ü ó ő ó ű Á ű ó ő ó ü ó ő ű ő Ö ü É É É ó É ó ü ű í Ö ü ó ű í ó ő ó ő ü ó ü ő ó É Í ő ő ő Ú ó ő ő ő ó ű ó ő ó ü ő ő ő í ü ő ü ő ó Ü ő ó ő ő ó ő Ú ő ő ó ő í ó ő ü ó Í ő ő ü ő É í ő ü ó
É É É Ó Ö É í Ö ő ü ó ő ó ű Á ű ó ő ó ü ó ő ű ő Ö ü É É É ó É ó ü ű í Ö ü ó ű í ó ő ó ő ü ó ü ő ó É Í ő ő ő Ú ó ő ő ő ó ű ó ő ó ü ő ő ő í ü ő ü ő ó Ü ő ó ő ő ó ő Ú ő ő ó ő í ó ő ü ó Í ő ő ü ő É í ő ü ó
 ú Ö ü ő ő ú ú ű ő í ó ó í ó ú ő ü ú ű ő í ó ó í ó ű í ó ő Í ő ü ú ő ő í ó ú Ö ő Ü ó ő ő É ó ó ó ó ő ő ú ű ő í ó ú ű ő ú ú ő ű ő í ő ó í ű ő ü ú ó ő ő ó ű ő ő í í í í ó ű ú ő Á ó ő Á ú ó ó ő ó í ó ű í í
ú Ö ü ő ő ú ú ű ő í ó ó í ó ú ő ü ú ű ő í ó ó í ó ű í ó ő Í ő ü ú ő ő í ó ú Ö ő Ü ó ő ő É ó ó ó ó ő ő ú ű ő í ó ú ű ő ú ú ő ű ő í ő ó í ű ő ü ú ó ő ő ó ű ő ő í í í í ó ű ú ő Á ó ő Á ú ó ó ő ó í ó ű í í
 ú ő ó ú ö ő ü ú ö ő ó ó ó ü ő í ö í ó ú ő ó ó ó ú ó ú ó ő ő ö ö ő ó ú ó ő ó ő í Á Á ö ö ó ő ú ö ő ú ó í ő ü ü ü í ú ü ü ü ó ú í ü í ó ő ó ő í ú ü ú ó ü ü ö ó ü ó í ü ó ő ö ö í ü ú ó ő ó í ó ő ó í ó ó í
ú ő ó ú ö ő ü ú ö ő ó ó ó ü ő í ö í ó ú ő ó ó ó ú ó ú ó ő ő ö ö ő ó ú ó ő ó ő í Á Á ö ö ó ő ú ö ő ú ó í ő ü ü ü í ú ü ü ü ó ú í ü í ó ő ó ő í ú ü ú ó ü ü ö ó ü ó í ü ó ő ö ö í ü ú ó ő ó í ó ő ó í ó ó í
 Á ó ü ő Ö Á ü ó ü ő Í ü Í Ó ü ő ő ó ó ó Í ó ü ó ő ő ó ó ü ú Í ő ő ó Ó ő ó ü ó Á ü ó ő ó Í Á Í ő ó ó ó ő ő Á ó ó ú ő Í ő ű ó Ó ü ó ó ú ó ő ú ü ő ó ó ó ő ó ó Ö ó ó ő ó ő ó ő ü ű ő ó ó ő ú ő ú ü Í ü ő ó ó
Á ó ü ő Ö Á ü ó ü ő Í ü Í Ó ü ő ő ó ó ó Í ó ü ó ő ő ó ó ü ú Í ő ő ó Ó ő ó ü ó Á ü ó ő ó Í Á Í ő ó ó ó ő ő Á ó ó ú ő Í ő ű ó Ó ü ó ó ú ó ő ú ü ő ó ó ó ő ó ó Ö ó ó ő ó ő ó ő ü ű ő ó ó ő ú ő ú ü Í ü ő ó ó
 ü ö Ö ü ó ü ó ó ó Á Ő É ö Ö ü ó ü ú ó ó ó ö ó í í ö ú Ó É ö Ö ü ó ü ü ó ó ó ö ó í ü ö Ö ó ü ü ü ó ó ó ö ó ü í í í ó í ú ű ű ü ű ú í ü ö ö í ö ú ü ó ú ú ű í ü ö ö ó ú ó í ü ú ó ü ó ó ű ó í ü ű ü í ű í
ü ö Ö ü ó ü ó ó ó Á Ő É ö Ö ü ó ü ú ó ó ó ö ó í í ö ú Ó É ö Ö ü ó ü ü ó ó ó ö ó í ü ö Ö ó ü ü ü ó ó ó ö ó ü í í í ó í ú ű ű ü ű ú í ü ö ö í ö ú ü ó ú ú ű í ü ö ö ó ú ó í ü ú ó ü ó ó ű ó í ü ű ü í ű í
 Í ú ó ú ó ú ó ó Á ó ó ö ű ú Á ú ó ó ó Í ó ö ö ö Í ö ó ó ö ó ó ó ö ó ö ö ö ö ó ö ó ö ó ü ó ó ü ó ü ö ö ö ö Ő ó ó Íó ó ó ü ó ű ó ó ű ű ó ö ü ö ú ö ü ű ö ö ö ö ó ú ö ö ö ü Í Í Í Á ó ó ú ü ú Á ü ö Á ó ü ó
Í ú ó ú ó ú ó ó Á ó ó ö ű ú Á ú ó ó ó Í ó ö ö ö Í ö ó ó ö ó ó ó ö ó ö ö ö ö ó ö ó ö ó ü ó ó ü ó ü ö ö ö ö Ő ó ó Íó ó ó ü ó ű ó ó ű ű ó ö ü ö ú ö ü ű ö ö ö ö ó ú ö ö ö ü Í Í Í Á ó ó ú ü ú Á ü ö Á ó ü ó
 í ö ő í ú ö ö í íí ü Ú Í Á ú ü í ö í ő í ö ő ű Í í ö ü ü ő ő ú í ő í ő ü ü ő Í ő Í í ü ö ö ö ö í ű ő ö ö ö í ü í Ó ö í ő ő í í ő Ó Ú Ő Íő Ő Ó ő ö ő ü ű í í ü ú Ő Í ő ő ő í ü ő É í Ő í ü ü ö ő í ü ö ö ü
í ö ő í ú ö ö í íí ü Ú Í Á ú ü í ö í ő í ö ő ű Í í ö ü ü ő ő ú í ő í ő ü ü ő Í ő Í í ü ö ö ö ö í ű ő ö ö ö í ü í Ó ö í ő ő í í ő Ó Ú Ő Íő Ő Ó ő ö ő ü ű í í ü ú Ő Í ő ő ő í ü ő É í Ő í ü ü ö ő í ü ö ö ü
 Í ö Í ű ú ö ö ú ö É í í ö Ó ű í ö ö í ö ö ö í í ö í í ö ö í ö ö ö ű í ö ö ö ö ö ö ö ú ö í ö ö í ö ö ö ö ö ú ű ű ú ö ö í ö É í ö ö í ö ö ö ú ű ö ö í ö ú ű ö ö í í ú ö ö í ö í í ö ö ö ú ö ö ö ö Í ö ú ö ú
Í ö Í ű ú ö ö ú ö É í í ö Ó ű í ö ö í ö ö ö í í ö í í ö ö í ö ö ö ű í ö ö ö ö ö ö ö ú ö í ö ö í ö ö ö ö ö ú ű ű ú ö ö í ö É í ö ö í ö ö ö ú ű ö ö í ö ú ű ö ö í í ú ö ö í ö í í ö ö ö ú ö ö ö ö Í ö ú ö ú
 Ő Ö ö Ö É Á Ü É ó É ó ü É É Ö Ö Á É Ő ú É Á ú Ő Ö Ü Ö Ö ü ó ó ü Ü ű ö ú ó Á í ó ö ö ö ö ó ü í í Á í Ó í ó ü Ö ö ú ó ó ö ü ó ó ö í í ű ö ó í ü í ö í í ű ö ü Ő ü ú Ö ö ó ö ó ö ö ö ü ó ö í ó Ö ö Ő ü Ö Ö ü
Ő Ö ö Ö É Á Ü É ó É ó ü É É Ö Ö Á É Ő ú É Á ú Ő Ö Ü Ö Ö ü ó ó ü Ü ű ö ú ó Á í ó ö ö ö ö ó ü í í Á í Ó í ó ü Ö ö ú ó ó ö ü ó ó ö í í ű ö ó í ü í ö í í ű ö ü Ő ü ú Ö ö ó ö ó ö ö ö ü ó ö í ó Ö ö Ő ü Ö Ö ü
 ö ú í í í ő ű Ü Ű Í í Ő Á Á Ö Ő Ű Í ö ú í í í ú ő ö ű í í í ö Ó ő í í í ö ú í ö ö ö ö Ü ő ö ö ö ú ű ő ú ű ö ö ú ö ö ő Ü ö ö í í ő ö í í í í í í ö ö í ö ö í í ő í ő ö ő í ú í ö í ö í í ö ű ö ö Ó Ü ö ő ő
ö ú í í í ő ű Ü Ű Í í Ő Á Á Ö Ő Ű Í ö ú í í í ú ő ö ű í í í ö Ó ő í í í ö ú í ö ö ö ö Ü ő ö ö ö ú ű ő ú ű ö ö ú ö ö ő Ü ö ö í í ő ö í í í í í í ö ö í ö ö í í ő í ő ö ő í ú í ö í ö í í ö ű ö ö Ó Ü ö ő ő
 ú ű ö ö ü ü Í ö ö ö ö É Í É ú ú É ú ú ö É ö Í Ü ú Í ö ö Í ú ö ö ö ö ü ö ö ú ü Ü ö ü Í ö ö ű ö ö Í ű ú ö ö ö ö Í ö ö ű ö ö Í ü Í ü ú Í É ö ö ü ö ö Ü ö ö Í ü Í ö ü Í Í ö Í ö Í ü ö ú Í ú Í ö É ú Í ö ö Í É
ú ű ö ö ü ü Í ö ö ö ö É Í É ú ú É ú ú ö É ö Í Ü ú Í ö ö Í ú ö ö ö ö ü ö ö ú ü Ü ö ü Í ö ö ű ö ö Í ű ú ö ö ö ö Í ö ö ű ö ö Í ü Í ü ú Í É ö ö ü ö ö Ü ö ö Í ü Í ö ü Í Í ö Í ö Í ü ö ú Í ú Í ö É ú Í ö ö Í É
 ő ö é ü ö é Ö é ő ü é í ü é é ő ö é ő ö Á ó ü ö é í é ö é Ö é ő ü ü é í é é ó é é í í é é ő ü í ő Ö í é ő é é ő é ő éü ú ü ö ő í Ú Ú ö É í í ü ó ó ó ü ő ö é í ó ö é í ö é é í ö é ó ű ő ö é ő ű ő í é í
ő ö é ü ö é Ö é ő ü é í ü é é ő ö é ő ö Á ó ü ö é í é ö é Ö é ő ü ü é í é é ó é é í í é é ő ü í ő Ö í é ő é é ő é ő éü ú ü ö ő í Ú Ú ö É í í ü ó ó ó ü ő ö é í ó ö é í ö é é í ö é ó ű ő ö é ő ű ő í é í
 ü ó Ö ü í ü ü ü ö É ó ó í ó ó ö ó ö ö ö í í ű ü ü ü Í í ü ü ü ö í ó í ó ó í ó í É ü ö í Í É í ö ú í ó í ö ö ó í ö ó ó ó ö ó ö í í ó ó í ó ó Ö í ö ö ó ö ó ú ó ö ó í ó ó í í ü ó í ö ó ó ü ü ó ö ó ú í ó í
ü ó Ö ü í ü ü ü ö É ó ó í ó ó ö ó ö ö ö í í ű ü ü ü Í í ü ü ü ö í ó í ó ó í ó í É ü ö í Í É í ö ú í ó í ö ö ó í ö ó ó ó ö ó ö í í ó ó í ó ó Ö í ö ö ó ö ó ú ó ö ó í ó ó í í ü ó í ö ó ó ü ü ó ö ó ú í ó í
 ü Ü ö ö ú Í ó í í ó ó ó ü ó ű ó í ó ó í ö ó ö ú ü ö Í í í ó ó ó ó Í ó ü ű ó í ó ó í ó Í í ó ü ö ú ó ó ó í í ó í í ű í ü ö í ó í ö í ú ó í ú ü ú Í í ü Í í í ó ü ö í ó í ó ü ö ó Í í í ó Í É ó ó ó Í í ö ö
ü Ü ö ö ú Í ó í í ó ó ó ü ó ű ó í ó ó í ö ó ö ú ü ö Í í í ó ó ó ó Í ó ü ű ó í ó ó í ó Í í ó ü ö ú ó ó ó í í ó í í ű í ü ö í ó í ö í ú ó í ú ü ú Í í ü Í í í ó ü ö í ó í ó ü ö ó Í í í ó Í É ó ó ó Í í ö ö
Humán genom variációk single nucleotide polymorphism (SNP)
 Humán genom variációk single nucleotide polymorphism (SNP) A genom ~ 97 %-a két különböző egyedben teljesen azonos ~ 1% különbség: SNP miatt ~2% különbség: kópiaszámbeli eltérés, deléciók miatt 11-12 millió
Humán genom variációk single nucleotide polymorphism (SNP) A genom ~ 97 %-a két különböző egyedben teljesen azonos ~ 1% különbség: SNP miatt ~2% különbség: kópiaszámbeli eltérés, deléciók miatt 11-12 millió
transzláció DNS RNS Fehérje A fehérjék jelenléte nélkülözhetetlen minden sejt számára: enzimek, szerkezeti fehérjék, transzportfehérjék
 Transzláció A molekuláris biológia centrális dogmája transzkripció transzláció DNS RNS Fehérje replikáció Reverz transzkriptáz A fehérjék jelenléte nélkülözhetetlen minden sejt számára: enzimek, szerkezeti
Transzláció A molekuláris biológia centrális dogmája transzkripció transzláció DNS RNS Fehérje replikáció Reverz transzkriptáz A fehérjék jelenléte nélkülözhetetlen minden sejt számára: enzimek, szerkezeti
SOLiD Technology. library preparation & Sequencing Chemistry (sequencing by ligation!) Imaging and analysis. Application specific sample preparation
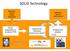 SOLiD Technology Application specific sample preparation Application specific data analysis library preparation & emulsion PCR Sequencing Chemistry (sequencing by ligation!) Imaging and analysis SOLiD
SOLiD Technology Application specific sample preparation Application specific data analysis library preparation & emulsion PCR Sequencing Chemistry (sequencing by ligation!) Imaging and analysis SOLiD
12/4/2014. Genetika 7-8 ea. DNS szerkezete, replikáció és a rekombináció. 1952 Hershey & Chase 1953!!!
 Genetika 7-8 ea. DNS szerkezete, replikáció és a rekombináció 1859 1865 1869 1952 Hershey & Chase 1953!!! 1879 1903 1951 1950 1944 1928 1911 1 1. DNS szerkezete Mi az örökítő anyag? Friedrich Miescher
Genetika 7-8 ea. DNS szerkezete, replikáció és a rekombináció 1859 1865 1869 1952 Hershey & Chase 1953!!! 1879 1903 1951 1950 1944 1928 1911 1 1. DNS szerkezete Mi az örökítő anyag? Friedrich Miescher
 ö ö ü ü ű ö Í ö ö ö ű Í ü ű ö ö ö ü ű ö ö ö ö ö Í ű ű ü ü Ó ű ö ö É ü ö ö ö ü ü É ö ü ö Á ü Á ű ü ű ű ű ű Í ÍÁ ü ö ö ö ü ü ü É ü ü Á ö ü ü ö ö ű ü ö ü ü ü ö ü ü ü ö ü ü ü ö ö ü ű ö ű ü ö ü ü ö ű ü Í ü
ö ö ü ü ű ö Í ö ö ö ű Í ü ű ö ö ö ü ű ö ö ö ö ö Í ű ű ü ü Ó ű ö ö É ü ö ö ö ü ü É ö ü ö Á ü Á ű ü ű ű ű ű Í ÍÁ ü ö ö ö ü ü ü É ü ü Á ö ü ü ö ö ű ü ö ü ü ü ö ü ü ü ö ü ü ü ö ö ü ű ö ű ü ö ü ü ö ű ü Í ü
 Í ű Á Á ű ü ü ü ű Í ü ü ü ü Í ű ű ü ü ű ü ü ű ü Í Í É Á Á Á É Á Ö Á Á Á ü É Ó Á Á Á Á É É Á ű É É Á ű ű Á Í Á Í É Á Á Á Á Á Á Ó Á ű ű ü ű ű ű ű ű ü ű Ó ü ű ü ü ű ü ű Í Í ü ű ü ü ü ü ü ű ü ű ü ü ü ü ü ű
Í ű Á Á ű ü ü ü ű Í ü ü ü ü Í ű ű ü ü ű ü ü ű ü Í Í É Á Á Á É Á Ö Á Á Á ü É Ó Á Á Á Á É É Á ű É É Á ű ű Á Í Á Í É Á Á Á Á Á Á Ó Á ű ű ü ű ű ű ű ű ü ű Ó ü ű ü ü ű ü ű Í Í ü ű ü ü ü ü ü ű ü ű ü ü ü ü ü ű
 ó ö ó Í Í Ó Í Á Í Í Í Ó Ú ó Í Ó ó Ó ó Í Ó Ó Ó Ó Ó Ó Ó ó Á Ó Ó ó ö ó Ú Í Í Ó Ó Ó Í Ó Ú É Í Í Í Ú Ó ő Í Í Ó Ó Ú Ó Ó ó Í ó Á Ó Ó Ó ó ó Í Ó Ó Ó Ó Ó Í Ú Í Í É ö Ó Ó Í Ó Ú Ó Ú Ó Ö Í Í Ú Ó Ó ó Ű Ó Ó Ó Ó Ó Ó Ó
ó ö ó Í Í Ó Í Á Í Í Í Ó Ú ó Í Ó ó Ó ó Í Ó Ó Ó Ó Ó Ó Ó ó Á Ó Ó ó ö ó Ú Í Í Ó Ó Ó Í Ó Ú É Í Í Í Ú Ó ő Í Í Ó Ó Ú Ó Ó ó Í ó Á Ó Ó Ó ó ó Í Ó Ó Ó Ó Ó Í Ú Í Í É ö Ó Ó Í Ó Ú Ó Ú Ó Ö Í Í Ú Ó Ó ó Ű Ó Ó Ó Ó Ó Ó Ó
 ó ö ÍÁ í ó í Á Á Á Á Á ü ö í í ó Í ő í ó ö ő ó ó ő ö ő í ö ú í í ő ó í í ö ö í ő í í ü ó ü ö ö ó ó ö ó ö ö ő ü ö ö ő ó ő ö í ü ö ö ó í ö ó ó ó ö ö ö ü ö ó ó ö í ó ó ó ö í í ú ö í í í í ü ő ö ö ó ű í ő
ó ö ÍÁ í ó í Á Á Á Á Á ü ö í í ó Í ő í ó ö ő ó ó ő ö ő í ö ú í í ő ó í í ö ö í ő í í ü ó ü ö ö ó ó ö ó ö ö ő ü ö ö ő ó ő ö í ü ö ö ó í ö ó ó ó ö ö ö ü ö ó ó ö í ó ó ó ö í í ú ö í í í í ü ő ö ö ó ű í ő
Bioinformatika előadás
 Bioinformatika 2 11. előadás Prof. Poppe László BME Szerves Kémia és Technológia Tsz. Bioinformatika proteomika Előadás és gyakorlat 2018.11.22. Szerkezeti genomika, proteomika, biológia A biológia forradalma
Bioinformatika 2 11. előadás Prof. Poppe László BME Szerves Kémia és Technológia Tsz. Bioinformatika proteomika Előadás és gyakorlat 2018.11.22. Szerkezeti genomika, proteomika, biológia A biológia forradalma
Az örökítőanyag. Az élőlények örökítőanyaga minden esetben nukleinsav (DNS,RNS) (1)Griffith, (2)Avery, MacLeod and McCarty (3)Hershey and Chase
 SZTE, Orv. Biol. Int., Mol- és Sejtbiol. Gyak., VIII. Az örökítőanyag Az élőlények örökítőanyaga minden esetben nukleinsav (DNS,RNS) (1)Griffith, (2)Avery, MacLeod and McCarty (3)Hershey and Chase Ez az
SZTE, Orv. Biol. Int., Mol- és Sejtbiol. Gyak., VIII. Az örökítőanyag Az élőlények örökítőanyaga minden esetben nukleinsav (DNS,RNS) (1)Griffith, (2)Avery, MacLeod and McCarty (3)Hershey and Chase Ez az
Poligénes v. kantitatív öröklődés
 1. Öröklődés komplexebb sajátosságai 2. Öröklődés molekuláris alapja Poligénes v. kantitatív öröklődés Azok a tulajdonságokat amelyek mértékegységgel nem, vagy csak nehezen mérhetők, kialakulásuk kevéssé
1. Öröklődés komplexebb sajátosságai 2. Öröklődés molekuláris alapja Poligénes v. kantitatív öröklődés Azok a tulajdonságokat amelyek mértékegységgel nem, vagy csak nehezen mérhetők, kialakulásuk kevéssé
A genetikai vizsgálatok jelene, jövője a Ritka Betegségek vonatkozásában
 Budapest, 2014. február 22. Ritka Betegségek Világnapja A genetikai vizsgálatok jelene, jövője a Ritka Betegségek vonatkozásában dr. Kósa János PentaCore Laboratórium, Budapest Semmelweis Egyetem I. sz.
Budapest, 2014. február 22. Ritka Betegségek Világnapja A genetikai vizsgálatok jelene, jövője a Ritka Betegségek vonatkozásában dr. Kósa János PentaCore Laboratórium, Budapest Semmelweis Egyetem I. sz.
Proteomika alapfogalmak, módszerek, példák a proteomika alkalmazására
 Proteomika alapfogalmak, módszerek, példák a proteomika alkalmazására Alapfogalmak és omikák : Genomika Teljes humán genom szekvenciájának meghatározása: 2001. február Genom: Winkler, 1920; GENes and chromosomes
Proteomika alapfogalmak, módszerek, példák a proteomika alkalmazására Alapfogalmak és omikák : Genomika Teljes humán genom szekvenciájának meghatározása: 2001. február Genom: Winkler, 1920; GENes and chromosomes
Fehérje expressziós rendszerek. Gyógyszerészi Biotechnológia
 Fehérje expressziós rendszerek Gyógyszerészi Biotechnológia Expressziós rendszerek Cél: rekombináns fehérjék előállítása nagy tisztaságban és nagy mennyiségben kísérleti ill. gyakorlati (therapia) felhasználásokra
Fehérje expressziós rendszerek Gyógyszerészi Biotechnológia Expressziós rendszerek Cél: rekombináns fehérjék előállítása nagy tisztaságban és nagy mennyiségben kísérleti ill. gyakorlati (therapia) felhasználásokra
Könyvtári informatika
 Tanmenet 2015/2016 tavaszi félév Doktori Iskola Könyvtári informatika Tantárgyfelelős: Ortutayné dr. Léces Melinda Oktató: Farkas Boglárka Ortutayné dr. Léces Melinda Pándi Dóra Sötét Krisztina Cél: A
Tanmenet 2015/2016 tavaszi félév Doktori Iskola Könyvtári informatika Tantárgyfelelős: Ortutayné dr. Léces Melinda Oktató: Farkas Boglárka Ortutayné dr. Léces Melinda Pándi Dóra Sötét Krisztina Cél: A
Mapping Sequencing Reads to a Reference Genome
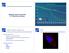 Mapping Sequencing Reads to a Reference Genome High Throughput Sequencing RN Example applications: Sequencing a genome (DN) Sequencing a transcriptome and gene expression studies (RN) ChIP (chromatin immunoprecipitation)
Mapping Sequencing Reads to a Reference Genome High Throughput Sequencing RN Example applications: Sequencing a genome (DN) Sequencing a transcriptome and gene expression studies (RN) ChIP (chromatin immunoprecipitation)
pjnc-pgluc Codon-optimized Gaussia luciferase (pgluc) Vector backbone: JM83 pjnc-pgluc is resistant to ampicillin and neomycin High or low copy:
 pjnc-pgluc Gene/Insert name: Codon-optimized Gaussia luciferase (pgluc) Vector backbone: pcmv-jnc Vector type: Mammalian cells Backbone size w/o insert (bp): 5375 Bacterial resistance: Ampicillin and neomycin
pjnc-pgluc Gene/Insert name: Codon-optimized Gaussia luciferase (pgluc) Vector backbone: pcmv-jnc Vector type: Mammalian cells Backbone size w/o insert (bp): 5375 Bacterial resistance: Ampicillin and neomycin
Fehérjék rövid bevezetés
 Receptorfehérj rjék szerkezetének felderítése Homológia modellezés Fehérjék rövid bevezetés makromolekulák számos biológiai funkció hordozói: enzimatikus katalízis, molekula transzport, immunválaszok,
Receptorfehérj rjék szerkezetének felderítése Homológia modellezés Fehérjék rövid bevezetés makromolekulák számos biológiai funkció hordozói: enzimatikus katalízis, molekula transzport, immunválaszok,
A DNS szerkezete. Genom kromoszóma gén DNS genotípus - allél. Pontos méretek Watson genomja. J. D. Watson F. H. C. Crick. 2 nm C G.
 1955: 46 emberi kromoszóma van 1961: mrns 1975: DNS szekvenálás 1982: gén-bank adatbázisok 1983: R (polymerase chain reaction) Mérföldkövek 1 J. D. Watson F. H.. rick 2008 1953 2003 Watson genomja DNS
1955: 46 emberi kromoszóma van 1961: mrns 1975: DNS szekvenálás 1982: gén-bank adatbázisok 1983: R (polymerase chain reaction) Mérföldkövek 1 J. D. Watson F. H.. rick 2008 1953 2003 Watson genomja DNS
Gyakorlati bioinformatika
 Gyakorlati bioinformatika Szekvenciaillesztés PhD kurzus 2. Szekvenciaillesztés Bagossi Péter Fajtái: - egyszer ill. többszörös illesztés - globális ill. lokális illesztés Alkalmazása: - adatbázisokban
Gyakorlati bioinformatika Szekvenciaillesztés PhD kurzus 2. Szekvenciaillesztés Bagossi Péter Fajtái: - egyszer ill. többszörös illesztés - globális ill. lokális illesztés Alkalmazása: - adatbázisokban
Iridovírus izolálása törpeharcsából Magyarországon
 Iridovírus izolálása törpeharcsából Magyarországon Juhász Tamás, Woynárovichné Láng Mária, Csaba György, Dán Ádám NÉBIH Állategészségügyi Diagnosztikai Igazgatóság 2013. január 29. Iridovírus CSALÁD: IRIDOVIRIDAE
Iridovírus izolálása törpeharcsából Magyarországon Juhász Tamás, Woynárovichné Láng Mária, Csaba György, Dán Ádám NÉBIH Állategészségügyi Diagnosztikai Igazgatóság 2013. január 29. Iridovírus CSALÁD: IRIDOVIRIDAE
TÉMAKÖRÖK. Ősi RNS világ BEVEZETÉS. RNS-ek tradicionális szerepben
 esirna mirtron BEVEZETÉS TÉMAKÖRÖK Ősi RNS világ RNS-ek tradicionális szerepben bevezetés BIOLÓGIAI MOLEKULÁK FEHÉRJÉK NUKLEINSAVAK DNS-ek RNS-ek BIOLÓGIAI MOLEKULÁK FEHÉRJÉK NUKLEINSAVAK DNS-ek RNS-ek
esirna mirtron BEVEZETÉS TÉMAKÖRÖK Ősi RNS világ RNS-ek tradicionális szerepben bevezetés BIOLÓGIAI MOLEKULÁK FEHÉRJÉK NUKLEINSAVAK DNS-ek RNS-ek BIOLÓGIAI MOLEKULÁK FEHÉRJÉK NUKLEINSAVAK DNS-ek RNS-ek
10. Genomika 2. Microarrayek és típusaik
 10. Genomika 2. 1. Microarray technikák és bioinformatikai vonatkozásaik Microarrayek és típusaik Korrelált génexpresszió mint a funkcionális genomika eszköze 2. Kombinált megközelítés a funkcionális genomikában
10. Genomika 2. 1. Microarray technikák és bioinformatikai vonatkozásaik Microarrayek és típusaik Korrelált génexpresszió mint a funkcionális genomika eszköze 2. Kombinált megközelítés a funkcionális genomikában
A genomikai oktatás helyzete a Debreceni Egyetemen
 A genomikai oktatás helyzete a Debreceni Egyetemen Bálint Bálint L. GNTP Oktatás és Tudásmenedzsment Munkabizottság, 2009. június 10. Tények Debreceni Egyetemről 21000 nappali és 33000 összes hallgató
A genomikai oktatás helyzete a Debreceni Egyetemen Bálint Bálint L. GNTP Oktatás és Tudásmenedzsment Munkabizottság, 2009. június 10. Tények Debreceni Egyetemről 21000 nappali és 33000 összes hallgató
BIOINFORMATIKA Ungvári Ildikó
 1 BIOINFORMATIKA Ungvári Ildikó Az elmúlt évtizedekben a molekuláris biológiai, genomikai technológiák robbanásszerű fejlődése a biológiai adatok mennyiségének exponenciális növekedéséhez vezetett. Ebben
1 BIOINFORMATIKA Ungvári Ildikó Az elmúlt évtizedekben a molekuláris biológiai, genomikai technológiák robbanásszerű fejlődése a biológiai adatok mennyiségének exponenciális növekedéséhez vezetett. Ebben
3. Sejtalkotó molekulák III. Fehérjék, enzimműködés, fehérjeszintézis (transzkripció, transzláció, poszt szintetikus módosítások)
 3. Sejtalkotó molekulák III. Fehérjék, enzimműködés, fehérjeszintézis (transzkripció, transzláció, poszt szintetikus módosítások) 3.1 Fehérjék, enzimek A genetikai információ egyik fő manifesztálódása
3. Sejtalkotó molekulák III. Fehérjék, enzimműködés, fehérjeszintézis (transzkripció, transzláció, poszt szintetikus módosítások) 3.1 Fehérjék, enzimek A genetikai információ egyik fő manifesztálódása
Fehérjeszerkezet, és tekeredés. Futó Kinga
 Fehérjeszerkezet, és tekeredés Futó Kinga Polimerek Polimer: hasonló alegységekből (monomer) felépülő makromolekulák Alegységek száma: tipikusan 10 2-10 4 Titin: 3,435*10 4 aminosav C 132983 H 211861 N
Fehérjeszerkezet, és tekeredés Futó Kinga Polimerek Polimer: hasonló alegységekből (monomer) felépülő makromolekulák Alegységek száma: tipikusan 10 2-10 4 Titin: 3,435*10 4 aminosav C 132983 H 211861 N
 Á Í Á Ó É ö á í á ő á á Á ő ő á ő á í á ő á á á á í ő ö í á á í á á ö ő á í ő áí á á ő á í í á ú ü ö á ú ö á í á á á ö á á ő á á á ő á ő á ú ü á ő á í ő ő ő áí á á ö ő á ő á á ő ő á í á ő á ő á á á ü ő
Á Í Á Ó É ö á í á ő á á Á ő ő á ő á í á ő á á á á í ő ö í á á í á á ö ő á í ő áí á á ő á í í á ú ü ö á ú ö á í á á á ö á á ő á á á ő á ő á ú ü á ő á í ő ő ő áí á á ö ő á ő á á ő ő á í á ő á ő á á á ü ő
A humán mitokondriális genom: Evolúció, mutációk, polimorfizmusok, populációs vonatkozások. Egyed Balázs ELTE Genetikai Tanszék
 A humán mitokondriális genom: Evolúció, mutációk, polimorfizmusok, populációs vonatkozások Egyed Balázs ELTE Genetikai Tanszék Endoszimbiotikus gén-transzfer (Timmis et al., 2004, Nat Rev Gen) Endoszimbiotikus
A humán mitokondriális genom: Evolúció, mutációk, polimorfizmusok, populációs vonatkozások Egyed Balázs ELTE Genetikai Tanszék Endoszimbiotikus gén-transzfer (Timmis et al., 2004, Nat Rev Gen) Endoszimbiotikus
DNS, RNS, Fehérjék. makromolekulák biofizikája. Biológiai makromolekulák. A makromolekulák TÖMEG szerinti mennyisége a sejtben NAGY
 makromolekulák biofizikája DNS, RNS, Fehérjék Kellermayer Miklós Tér Méret, alak, lokális és globális szerkezet Idő Fluktuációk, szerkezetváltozások, gombolyodás Kölcsönhatások Belső és külső kölcsöhatások,
makromolekulák biofizikája DNS, RNS, Fehérjék Kellermayer Miklós Tér Méret, alak, lokális és globális szerkezet Idő Fluktuációk, szerkezetváltozások, gombolyodás Kölcsönhatások Belső és külső kölcsöhatások,
Adatgy jtés, bibliográfia, hivatkozások. Az adatgy jtés folyamata
 Adatgy jtés, bibliográfia, hivatkozások Az adatgy jtés folyamata A tananyag nem tárgyalja a tudományos kutatás módszereit. Az egyes tudományágak sajátos kutatásmódszertannal rendelkeznek. A szakterületek
Adatgy jtés, bibliográfia, hivatkozások Az adatgy jtés folyamata A tananyag nem tárgyalja a tudományos kutatás módszereit. Az egyes tudományágak sajátos kutatásmódszertannal rendelkeznek. A szakterületek
Semmelweis Egyetem / Élettani Intézet / Budapest. Bioinformatika és genomanalízis az orvostudományban. Bevezetés. Cserző Miklós 2018
 Bioinformatika és genomanalízis az orvostudományban Bevezetés Cserző Miklós 2018 A mai előadás A kurzus menete Hol találkozunk bioinformatikával Mi a bioinformatika Miért van bioinformatika A számítógépekről
Bioinformatika és genomanalízis az orvostudományban Bevezetés Cserző Miklós 2018 A mai előadás A kurzus menete Hol találkozunk bioinformatikával Mi a bioinformatika Miért van bioinformatika A számítógépekről
Hamar Péter. RNS világ. Lánczos Kornél Gimnázium, Székesfehérvár, 2014. október 21. www.meetthescientist.hu 1 26
 Hamar Péter RNS világ Lánczos Kornél Gimnázium, Székesfehérvár, 2014. október 21. 1 26 Főszereplők: DNS -> RNS -> fehérje A kód lefordítása Dezoxy-ribo-Nuklein-Sav: DNS az élet kódja megkettőződés (replikáció)
Hamar Péter RNS világ Lánczos Kornél Gimnázium, Székesfehérvár, 2014. október 21. 1 26 Főszereplők: DNS -> RNS -> fehérje A kód lefordítása Dezoxy-ribo-Nuklein-Sav: DNS az élet kódja megkettőződés (replikáció)
 Ó Ö ő ü ü Ö ő ü ó í ó ő í ő ó ü ő ő ő Ő É Ü Ö ő ü ü Ö ő ü ó í ó ő ü Ö ő ü ő ü ó ó ó ő ő ő Ö Ö ó í í ü ő ó ó í ü ü Ö ű ő í í í í í í ő Ó ó Ó ó Ó ő Ö ű í ő ó Ó ó í ő ő ő ő ü í í ű í í í ő ü ő ú ű í ű ő ó
Ó Ö ő ü ü Ö ő ü ó í ó ő í ő ó ü ő ő ő Ő É Ü Ö ő ü ü Ö ő ü ó í ó ő ü Ö ő ü ő ü ó ó ó ő ő ő Ö Ö ó í í ü ő ó ó í ü ü Ö ű ő í í í í í í ő Ó ó Ó ó Ó ő Ö ű í ő ó Ó ó í ő ő ő ő ü í í ű í í í ő ü ő ú ű í ű ő ó
Nan Wang, Qingming Dong, Jingjing Li, Rohit K. Jangra, Meiyun Fan, Allan R. Brasier, Stanley M. Lemon, Lawrence M. Pfeffer, Kui Li
 Supplemental Material IRF3-dependent and NF- B-independent viral induction of the zinc-finger antiviral protein Nan Wang, Qingming Dong, Jingjing Li, Rohit K. Jangra, Meiyun Fan, Allan R. Brasier, Stanley
Supplemental Material IRF3-dependent and NF- B-independent viral induction of the zinc-finger antiviral protein Nan Wang, Qingming Dong, Jingjing Li, Rohit K. Jangra, Meiyun Fan, Allan R. Brasier, Stanley
Supplementary Table 1. Cystometric parameters in sham-operated wild type and Trpv4 -/- rats during saline infusion and
 WT sham Trpv4 -/- sham Saline 10µM GSK1016709A P value Saline 10µM GSK1016709A P value Number 10 10 8 8 Intercontractile interval (sec) 143 (102 155) 98.4 (71.4 148) 0.01 96 (92 121) 109 (95 123) 0.3 Voided
WT sham Trpv4 -/- sham Saline 10µM GSK1016709A P value Saline 10µM GSK1016709A P value Number 10 10 8 8 Intercontractile interval (sec) 143 (102 155) 98.4 (71.4 148) 0.01 96 (92 121) 109 (95 123) 0.3 Voided
Az orvosi biotechnológiai mesterképzés megfeleltetése az Európai Unió új társadalmi kihívásainak a Pécsi Tudományegyetemen és a Debreceni Egyetemen
 Az orvosi biotechnológiai mesterképzés megfeleltetése az Európai Unió új társadalmi kihívásainak a Pécsi Tudományegyetemen és a Debreceni Egyetemen Azonosító szám: TÁMOP-4.1.2-08/1/A-2009-0011 Az orvosi
Az orvosi biotechnológiai mesterképzés megfeleltetése az Európai Unió új társadalmi kihívásainak a Pécsi Tudományegyetemen és a Debreceni Egyetemen Azonosító szám: TÁMOP-4.1.2-08/1/A-2009-0011 Az orvosi
Supporting Information
 Supporting Information Paired design of dcas9 as a systematic platform for the detection of featured nucleic acid sequences in pathogenic strains Yihao Zhang 1,2,8, Long Qian 4,8, Weijia Wei 1,3,7,8, Yu
Supporting Information Paired design of dcas9 as a systematic platform for the detection of featured nucleic acid sequences in pathogenic strains Yihao Zhang 1,2,8, Long Qian 4,8, Weijia Wei 1,3,7,8, Yu
Bioinformatics: Blending. Biology and Computer Science
 Bioinformatics: Blending Biology and Computer Science MDNMSITNTPTSNDACLSIVHSLMCHRQ GGESETFAKRAIESLVKKLKEKKDELDSL ITAITTNGAHPSKCVTIQRTLDGRLQVAG RKGFPHVIYARLWRWPDLHKNELKHVK YCQYAFDLKCDSVCVNPYHYERVVSPGI DLSGLTLQSNAPSSMMVKDEYVHDFEG
Bioinformatics: Blending Biology and Computer Science MDNMSITNTPTSNDACLSIVHSLMCHRQ GGESETFAKRAIESLVKKLKEKKDELDSL ITAITTNGAHPSKCVTIQRTLDGRLQVAG RKGFPHVIYARLWRWPDLHKNELKHVK YCQYAFDLKCDSVCVNPYHYERVVSPGI DLSGLTLQSNAPSSMMVKDEYVHDFEG

