Glyma10g15850 aagggatccattctggaaccatatcttgctgtg ttgggtacccttggatgcaggatgacacg AtMKK6, AT5g56580
|
|
|
- Barnabás Zsolt Gál
- 5 évvel ezelőtt
- Látták:
Átírás
1 Supplemental table 1. Soybean MAPK, MAPKK, and MAPKKK genes and primers for VIGS cloning Gene_ID Forward Primer Reverse Primer Arabidopsis Ortholog Glyma04g03210 aagggatcctgcagcaagaaccagttcat ttgggtaccctaatggatgcgcattaggataaag AtMPK1,At1g10210 Glyma12g07770 aagggatccttcttgtaccagattcttcgtgg ttgggtacccctctgtcaat AGGCGCATCT G AtMPK3, At3g45640 Glyma16g03670 aagggatccctgtatcaattgttacgagggctc ttgggtacccctctgtgataagtctcagctgatga AtMPK4a, At4g01370 Glyma02g15690 aagggatccccagagcttctgttgaactcttc ttgggtacccatgttgttcaaagtcaaagttgaagg AtMPK6, At2g43790 Glyma08g05700 aagggatccatgggtggtggaggaacact ttgggtacccctttttgtgatgatccatggaag AtMPK9, AT3G18040 Glyma09g39190 aagggatccctgtatcagttgttacgaggactcaa ttgggtaccccagcctgagc TGATGAACAT AAT AtMPK4c Glyma11g15590 aagggatccctcttgcgaggattgaagtacatc ttgggtacccctcagttataagggccaactgttg AtMPK13, At1g07880 Glyma12g07850 aagggatcccaactcttgcgagggttgaa ttgggtacccctcagttataagggccaactgct AtMPK13, At1g07880 Glyma13g28120 aagggatcccagattcttcgtgggcttaagt ttgggtacccctctgtcaataggcgcatctg MPK16/17, At5g19010 Glyma17g02220 aagggatccggaagctatggtgttgtttgctc ttgggtaccctcggtgtatatacttcaagcctcg MPK16, At5g19010 Glyma13g05700 aagggatccggcgaagttgcttctccagt ttgggtacccgagaacacgtagctgtgaaagga SNF1 kinase homolog 10, At3G01090 Glyma13g33860 aagggatccacgcttatgaccaaagggtc ttgggtaccctgacaatccatactgaaaggcc MPK19, At3g14720 Glyma08g42240 aagggatcctcatatgatcctaggtcattcatcag ttgggtacccaggaccaacttttctatgagcagt MPK20, At2G42880 Glyma15g18860 aagggatccgaaatccaaatgcctattgagga ttgggtacccgttaggcatgcagcgaaaag AtMKK1, AT4G29810 or At4g26070 Glyma17g06020 aagggatccgtgctgaagggtctagtttatctcc ttgggtaccctgcagagataaatgagcaaaattct AtMKK2, AT4G29810, Glyma13g16650 aagggatccgtgctgaagggtttagtttatctcc ttgggtacccatgctgagataaatgagcaaaattct AtMKK2, AT4G29810, Glyma19g00220 aagggatcc TCTGGGAAACAACATCGTGG ttgggtaccc GCTAGTTGGT TCGCATCCAA TA AtMKK3, At5g40440 Glyma07g00520 aagggatcc AATCAGACGATGGATCCGTG ttgggtacccgagtagctga TGGAGGTTGG GA AtMKK4/5, At1G51660, AT3G21220 Glyma08g23900 aagggatcc AATCAGACGATGGATCCGTG ttgggtaccc GAGTAGCTGA TGGAGGTTTG GA AtMKK5, AT3G21220 Glyma10g15850 aagggatccattctggaaccatatcttgctgtg ttgggtacccttggatgcaggatgacacg AtMKK6, AT5g56580 Glyma02g41340 aagggatcctatgctttgggagttgttctattg ttgggtacccacactctgga ACTCCTAGCA TAGCT Protein kinase superfamily protein, AT5G Glyma14g39690 aagggatccgctaaacctttgctgagaaaagg ttgggtacccatgaccatagagaaaatcatcatcc Protein kinase superfamily protein, AT5G Glyma15g05390 aagggatccatgggtaataaaattggtagggc ttgggtacccggtgaagccttcatagacggt AtMEKK1a, AT4G08500 Glyma15g05400 aagggatccatggaagctgagcggaag ttgggtacccgtggtcatcagcaaaaccag AtMEKK1b, AT4G08500 Glyma08g08300 aagggatccaccactcctcctcctcacac ttgggtacccattgaagccttcatagacagttcc AtMEKK1c, AT4G08500 Glyma05g25290 aagggatccatggactggaggcgcag ttgggtacccggaggaagaagaaaccgca AtMEKK1d, AT4G08500 Glyma19g42340 aagggatccaacgatgatgatatgtgtgtgattg ttgggtaccctggtgacctgcttttaggagg AtNPK1, AT1g09000 Glyma06g15870 aagggatccagcaagagcaaggaccaacaa ttgggtacccgtgacaggtagggtgatcatcgta AtMKKK3, AT1G53570 Glyma06g03970 aagggatcctcgtgtaaagatcacagagaactctct ttgggtaccctgcatgataaacagatccaaatgat AtMKKK5, Glyma11g02520 aagggatccgaagcccatacaccaaggaac ttgggtacccgaagcccatacaccaaggaac MEKK related, AT1G63700 Glyma17g19800 aagggatccgatatatgggctttgggatgtg ttgggtacccatcaaaaccccacttttcttgaaat MPKKK21, AT4G36950 Glyma13g28570 aagggatccctagagttgagtgcctctgcagaa ttgggtaccctgaatttttg AGTTCAGAAA CTATGACT MAPKK RELATED KINASE with ARM domain, AT5G18700
2 Supplemental table 2. False discovery rate (q value) and log2 fold change (FC) of ribosomal RNA probe sets Affymetrix Soybean Probe ID p value q value log2 FC soybean_rrna_114_at soybean_rrna_666_at soybean_rrna_692_at soybean_rrna_637_at soybean_rrna_918_at soybean_rrna_1352_at soybean_rrna_274_at soybean_rrna_218_at soybean_rrna_57_at soybean_rrna_176_at soybean_rrna_349_at soybean_rrna_176_rc_at soybean_rrna_114_rc_at soybean_rrna_1479_at soybean_rrna_718_at soybean_rrna_808_at soybean_rrna_1665_at soybean_rrna_485_at soybean_rrna_666_rc_at soybean_rrna_838_at soybean_rrna_57_rc_at soybean_rrna_1021_at soybean_rrna_918_rc_at soybean_rrna_776_at soybean_rrna_1531_at soybean_rrna_1057_at soybean_rrna_637_rc_at soybean_rrna_1391_at soybean_rrna_218_rc_at soybean_rrna_274_rc_at soybean_rrna_349_rc_at soybean_rrna_838_rc_at soybean_rrna_718_rc_at E 03 soybean_rrna_1721_at soybean_rrna_1057_rc_at soybean_rrna_1692_at soybean_rrna_776_rc_at soybean_rrna_808_rc_at soybean_rrna_485_rc_at soybean_rrna_692_rc_at soybean_rrna_1721_rc_at soybean_rrna_1391_rc_at soybean_rrna_1021_rc_at soybean_rrna_1479_rc_at soybean_rrna_1531_rc_at soybean_rrna_1665_rc_at soybean_rrna_1352_rc_at soybean_rrna_1692_rc_at 8.50E E
3 Supplemental table 3. False discovery rate (q value) and log2 fold change (FC) of defense related probe sets Affymetrix Soybean Probe ID Soybean gene locus p value q value log 2 FC Annotation GmaAffx S1_at Glyma13g E E Defensin, PDF2.4 Gma S1_at Glyma01g E E PR 5 protein GmaAffx S1_s_at Glyma01g E E PR 5 protein Gma S1_a_at Glyma11g E E PR 5 protein Gma S1_at Glyma11g E E PR 5 protein Gma S1_s_at Glyma05g E E PR5 Thaumatin family, response_to_abiotic_or_biotic_stimulus GmaAffx S1_at Glyma02g E E Syntaxin 121 GmaAffx S1_at Glyma14g E E NUDIX HYDROLASE RELATED GmaAffx S1_at Glyma15g E E CYSTEINE RICH SECRETORY PROTEIN (CRISP/SCP/TPX1) RELATED GmaAffx S1_at Glyma03g E E 07 5 Syringolide induced protein, Hairpin inducing protein GmaAffx A1_at Glyma02g E E 07 5 Peroxidase Gma S1_at Glyma09g E E Acidic chitinase GmaAffx S1_s_at Glyma15g E E CYSTEINE RICH SECRETORY PROTEIN (CRISP/SCP/TPX1) RELATED Gma S1_s_at Glyma02g E E Chitinase class I Gma S1_at Gma S1_at 1.98E E GLYCOLIPID TRANSFER PROTEIN RELATED GmaAffx S1_at Glyma15g E E CYSTEINE RICH SECRETORY PROTEIN (CRISP/SCP/TPX1) RELATED Gma S1_at Glyma10g E E negative_regulation_of_transcription regulation_of_systemic_acquired_resistance defense_response_to_bacterium GmaAffx S1_at No Soybean Match 5.33E E Glutamine synthetase PR 2 GmaAffx S1_s_at Glyma17g E E 06 4 Pathogenesis related protein Bet v I family GmaAffx S1_x_at Glyma16g E E Extensin GmaAffx S1_x_at Glyma07g E E Pathogenesis related protein Bet v I family Gma S1_at Glyma07g E E O METHYLTRANSFERASE GmaAffx S1_s_at Glyma15g E E Defense related protein containing SCP domain GmaAffx S1_at Glyma16g E E Avr9/Cf 9 rapidly elicited protein Gma A1_at Glyma19g E E pectin methylesterase GmaAffx S1_at Glyma13g E E GRP78/BiP/KAR2, HSP70 superfamily Gma S1_at Glyma01g E E CGI 141 RELATED/LIPASE CONTAINING PROTEIN; Lipase (class 3) GmaAffx S1_at Glyma17g E E Pathogenesis related protein 2 Gma S1_a_at Glyma07g E E O METHYLTRANSFERASE GmaAffx S1_at Glyma06g E E EDS1 like GmaAffx S1_x_at No Soybean Match Iden3.11E E Stress and pathogenesis related protein Gma S1_at Glyma15g E E Avr9/Cf 9 rapidly elicited protein GmaAffx S1_at Glyma17g E E 05 3 Pathogenesis related protein Bet v I family Gma S1_at Glyma16g E E Resistance protein LM12 GmaAffx S1_at Glyma13g E E GLUTAREDOXIN GmaAffx S1_at Glyma13g E E lead_ion_transport multidrug_transport response_to_biotic_stimulus Gma S1_s_at Glyma09g E E Pathogenesis related class 10 protein SPE 16 Gma S1_at Glyma16g E E Resistance protein MG13 GmaAffx S1_s_at Glyma01g E E lipase class 3 family protein GmaAffx S1_at Glyma11g E E Endo 1,3 beta glucanase GmaAffx S1_s_at Glyma09g E E Class 10 pathogenesis related protein GmaAffx S1_s_at No Soybean Match Iden1.65E Stress induced protein SAM22 Gma S1_at Glyma14g E E Vacuolar processing enzyme GmaAffx S1_at Glyma11g E E Glucan endo 1,3 beta d glucosidase Gma S1_at Glyma09g E E Calreticulin 3 Gma S1_at Glyma15g E E Pathogenesis related protein Bet v I family Gma S1_at Glyma20g E E DELLA protein Gma A1_at Glyma03g E E PDR like ABC transporter Gma S1_s_at Glyma07g E E Pathogenesis related protein Bet v I family Gma S1_s_at Glyma07g E E PR 10 like
4 Gma S1_s_at Glyma03g E E Oxalate oxidase GmaAffx A1_at No Soybean Match Iden 2.02E Thaumatin like protein Gma S1_at Glyma20g E E CHITINASE RELATED PR4 GmaAffx A1_s_at Glyma01g E E Thaumatin like protein Gma S1_s_at Glyma02g E E lipoxygenase_activity GmaAffx S1_s_at Glyma15g E E CYSTEINE PROTEASE FAMILY C1 RELATED Gma S1_at Glyma15g E E NPR1 like protein Gma S1_x_at Glyma15g E E Peroxidase GmaAffx S1_at AT2G E E Nematode resistance HS1pro1 protein Gma S1_x_at Glyma07g E E Intracellular Pathogenesis related protein or Stress induced protein SAM22 Gma S1_at Glyma14g E E Pathogenesis related genes transcriptional activator PTI6 Gma S1_at No Soybean Match Iden1.35E E Class 10 pathogenesis related protein 1 GmaAffx S1_s_at Glyma13g E E Pathogenesis related protein PR 1 Gma A1_s_at 8.30E E Salicylic acid binding protein 2 GmaAffx S1_at Glyma02g E E Syntaxin related protein 121 GmaAffx S1_at Glyma13g E E Pathogenesis related genes transcriptional activator PTI5 Gma S1_a_at Glyma02g E E TIR domain Gma A1_s_at Glyma02g E E Salicylic acid methyl transferase like protein Gma S1_at Glyma08g E E LSD1 GmaAffx S1_s_at Glyma05g E E LSD1 GmaAffx S1_x_at Glyma09g E E 05 1 SGT1 2 GmaAffx A1_at Glyma02g E E 04 1 Pathogenesis related genes transcriptional activator PTI6 GmaAffx S1_at Glyma02g E E Salicylic acid methyl transferase like protein Gma S1_at Glyma18g E E salicylic acid carboxyl methyltransferase like protein
5 Supplemental table 4. WRKY probe sets differentially expressed in MPK4 silenced plants Affymetrix Soybean Probe ID Soybean gene locus p value q value log2 FC Annotation No. W box GmaAffx S1_at AT2G E E WRKY33 Gma S1_at Glyma15g E E WRKY42 1 gmaaffx s1_at Glyma11g E E WRKY33 2 GmaAffx S1_at Glyma17g E E WRKY60 0 GmaAffx S1_at AT5G E E WRKY51 Gma S1_at Glyma14g E E WRKY40 0 GmaAffx S1_s_at Glyma11g E E WRKY33 2 gmaaffx s1_at Glyma17g E E WRKY40 0 GmaAffx S1_at Glyma08g E E WRKY51 0 GmaAffx S1_s_at Glyma06g E E WRKY51 0 Gma S1_at Glyma19g E E WRKY75 1 GmaAffx S1_at Glyma15g E E WRKY31 0 gmaaffx s1_at Glyma06g E E WRKY51 0 GmaAffx S1_at Glyma09g E E WRKY31 1 Gma S1_at Glyma18g E E WRKY70 1 GmaAffx A1_at Glyma17g E E WRKY40 0 GmaAffx S1_at Glyma13g E E WRKY31 0 gmaaffx s1_at Glyma13g E E WRKY6 0 Gma S1_at Glyma06g E E WRKY53 4 Gma S1_at Glyma01g E E WRKY53 4 GmaAffx S1_at AT1G E E WRKY6 GmaAffx S1_at Glyma06g E E WRKY40 0 Gma S1_s_at Glyma05g E E WRKY65 0 gmaaffx s1_x_at Glyma09g E E WRKY31 1 GmaAffx S1_at Glyma07g E E WRKY6 1 gmaaffx s1_at Glyma07g E E WRKY6 1 Gma S1_a_at Glyma04g E E WRKY70 2 GmaAffx S1_x_at Glyma02g E E WRKY26 4 gma s1_x_at Glyma09g E E WRKY31 1 gma s1_at Glyma09g E E WRKY31 1 gma s1_s_at Glyma09g E E WRKY31 1 gmaaffx s1_at Glyma02g E E WRKY26 4 gma s1_at Glyma19g E E WRKY23 0 GmaAffx S1_at Glyma01g E E WRKY33 4 Gma S1_at Glyma16g E E WRKY22/29 2 gmaaffx s1_s_at Glyma07g E E WRKY6 1 gma s1_at Glyma13g E E WRKY6 0 gmaaffx s1_s_at Glyma02g E E WRKY33 4 GmaAffx S1_at Glyma05g E E WRKY53/55 1 gma s1_a_at AT2G E E WRKY33 GmaAffx S1_at Glyma02g E E WRKY57 0 GmaAffx S1_at Glyma09g E E WRKY70 0 gmaaffx s1_at Glyma02g E E WRKY57 0 gmaaffx s1_x_at Glyma02g E E WRKY33 4 gmaaffx s1_at Glyma19g E E WRKY41 3 Gma S1_s_at Glyma03g E E WRKY23 1 gmaaffx s1_at AT4G E E WRKY53 Gma A1_at Glyma01g E E WRKY4 0 GmaAffx S1_at Glyma08g E E WRKY4 0 Gma S1_a_at Glyma13g E E WRKY11 2 gma s1_at Glyma13g E E WRKY11 2 GmaAffx S1_at Glyma17g E E WRKY48 1 GmaAffx S1_at Glyma13g E E WRKY47 2 gma s1_at Glyma03g E E WRKY23 1 Gma S1_at Glyma17g E E WRKY11 1 GmaAffx S1 Glyma08g E E WRKY53 3 GmaAffx S1_s_at Glyma13g E E WRKY69 0 Gma S1_at Glyma17g E E WRKY15 4 GmaAffx S1_at Glyma17g E E WRKY31 0 GmaAffx S1_x_at AT1G E E WRKY4 gmaaffx s1_at Glyma07g E E WRKY53 2 gmaaffx s1_at No Soybean Match 1.40E E WRKY11 GmaAffx S1_at Glyma06g E E WRKY11 0 GmaAffx S1_at Glyma08g E E WRKY75 3 Gma S1_s_at Glyma12g E E WRKY69 0 GmaAffx S1_at Glyma01g E E WRKY15 4 gmaaffx s1_s_at Glyma06g E E WRKY17 0 gmaaffx s1_at Glyma19g E E WRKY42 1 gmaaffx s1_at No Soybean Match 6.51E E WRKY33 GmaAffx S1_at Glyma02g E E WRKY1 1 GmaAffx S1_at Glyma16g E E WRKY46 2 Gma A1_at Glyma03g E E WRKY35 0 Gma A1_at Glyma03g E E WRKY21 2 GmaAffx S1_at Glyma09g E E WRKY13 1 GmaAffx S1_at Glyma03g E E WRKY2/TTG2 2
6 Supplemental table 5. Differentially expressed probe sets corresponding to auxin regulated genes in MPK4 silenced plants Affymetrix Soybean Probe ID Soybean gene locus p value q value log2 FC Annotation Gma A1_s_at Glyma10g E E 06 6 Auxin down regulated protein Gma S1_at Glyma08g E E Auxin induced protein AUX22 GmaAffx S1_at Glyma11g E E Auxin transporter like protein 5 GmaAffx S1_at Glyma09g E E Auxin efflux carrier GmaAffx S1_at Glyma13g E E Response_to_auxin_stimulus Gma A1_at Glyma08g E E GH1 protein GmaAffx S1_at Glyma07g E E Auxin response factor 18 GmaAffx S1_at Glyma06g E E Auxin induced protein 15 Gma S1_at Glyma12g E E Auxin responsive protein; SAUR protein Gma S1_at Glyma10g E E Auxin regulated protein GmaAffx S1_at Glyma02g E E 04 3 Auxin induced protein IAA4 GmaAffx S1_at Glyma06g E E 04 3 Auxin responsive protein GmaAffx S1_at Glyma08g E E 05 3 Indole 3 acetic acid induced protein ARG7 Gma S1_at AT4G E Auxin induced protein GmaAffx S1_at Glyma11g E E Auxin response factor; Aux/IAA_ARF Gma S1_at Glyma17g E E Auxin binding protein ABP19 GmaAffx S1_at Glyma08g E E response_to_auxin_stimulus root_development Gma S1_at Glyma14g E E Proteolysis response_to_auxin_stimulus lateral_root_morphogenesis Gma S1_at Glyma10g E E Auxin induced protein AUX28 GmaAffx S1_at Glyma12g E E Auxin response factor; Aux/IAA_ARF_ GmaAffx S1_at Glyma08g E E Auxin conjugate hydrolase Gma S1_at Glyma10g E E Putative indole 3 acetic acid amido synthetase GH3.9 Gma S1_at Glyma13g E E Auxin induced protein 22 (AUX22) GmaAffx S1_at Glyma09g E E Auxin responsive protein Gma S1_at Glyma08g E E AUX/IAA family GmaAffx S1_at Glyma16g E E Auxin response factor 18 GmaAffx S1_at Glyma08g E E Auxin responsive protein Gma S1_s_at Glyma08g E E Response_to_auxin_stimulus root_development GO: Gma S1_at Glyma07g E E Response_to_auxin_stimulus Gma S1_at Glyma18g E E AUX1 like auxin influx carrier protein Gma S1_a_at Glyma08g E E Auxin_polar_transport establishment_or_maintenance_of_transmembrane_electrochemical_grad GmaAffx S1_s_at AT5G E E Auxin response factor 8 GmaAffx S1_s_at Glyma02g E E Response_to_auxin_stimulus auxin_mediated_signaling_pathway GO: GmaAffx S1_at Glyma17g E E unidimensional_cell_growth auxin_polar_transport phloem_or_xylem_histogenesis GO: GmaAffx S1_s_at Glyma10g E E Regulation_of_transcription,_DNA dependent response_to_auxin_stimulus regulation_of_stomata Gma S1_a_at Glyma16g E E Auxin binding protein ABP19 GmaAffx S1_at Glyma13g E E Auxin response factor; Aux/IAA_ARF_ GmaAffx S1_at Glyma20g E E Unidimensional cell growth auxin polar_transport phloem or xylem histogenesis GO: Gma S1_at Glyma13g E E _DNA dependent auxin_metabolic_process response_to_auxin_stimulus Gma S1_at Glyma07g E E auxin polar transport establishment Gma S1_at Glyma05g E E Auxin responsive protein IAA27 GmaAffx A1_at Glyma16g E E auxin polar transport GmaAffx S1_at Glyma18g E E Putative auxin independent growth promoter protein (At3g30300) GmaAffx A1_at Glyma16g E auxin polar transport
7 Gma S1_at Glyma12g E Putative auxin response factor ARF16 Gma S1_at Glyma15g E E 05 2 Auxin responsive protein IAA26 Gma S1_at Glyma20g E E 05 2 AUX/IAA family, Auxin regulated protein GmaAffx S1_at AT2G E E 06 2 PIN1 like auxin transport protein GmaAffx A1_at Glyma15g E E 06 2 DNA dependent auxin_metabolic_process response_to_auxin_stimulus Gma S1_at Glyma10g E E 03 2 Auxin induced protein IAA4; IAA31? GmaAffx S1_s_at AT3G E E Putative uncharacterized protein; response_to_auxin_stimulus; transcription_factor_activity Gma A1_at Glyma04g E E Auxin responsive protein GmaAffx A1_at Glyma12g E E Auxin response factor 4; Aux/IAA_ARF_ GmaAffx S1_at Glyma07g E E Putative uncharacterized protein GmaAffx A1_at Glyma09g E E Auxin induced protein X10A GmaAffx S1_at NO BLASTX HIT (E< E E Auxin response factor GmaAffx S1_x_at Glyma20g E E Aux/IAA protein, AXR3 Gma S1_at Glyma06g E E Auxin responsive protein IAA29 Gma A1_at Glyma07g E E Auxin response factor 9 GmaAffx S1_at Glyma13g E E Auxin response factor GmaAffx A1_at Glyma17g E E Auxin response factor 5 GmaAffx S1_at Glyma09g E E Auxin efflux carrier protein; PIN1 like auxin transport protein; PIN7 GmaAffx S1_at Glyma14g E E Auxin response factor 8 Gma S1_at Glyma19g E E Auxin induced protein 22 GmaAffx S1_at AT2G E Auxin response factor Gma A1_at Glyma10g E E Auxin induced protein PCNT115 Gma S1_at AT4G E E Auxin induced protein AUX28 Gma S1_at AT5G E E GH1 protein GmaAffx S1_s_at Glyma02g E E Auxin induced protein 22 GmaAffx A1_at Glyma19g E E Putative auxin response factor 75 GmaAffx A1_at Glyma09g E E Auxin induced protein 10 GmaAffx S1_at Glyma08g E E Auxin efflux carrier GmaAffx S1_at Glyma19g E E Multidrug resistance protein 1 Gma A1_at Glyma11g E E Auxin transporter like protein 1 Gma A1_at Glyma11g E E Auxin transporter like protein LAX1 Gma S1_a_at Glyma15g E E Auxin binding protein ABP20 GmaAffx Glyma13g E E Auxin responsive family protein GmaAffx S1_at Glyma10g E E Putative indole 3 acetic acid amido synthetase GH3.9 GmaAffx S1_at Glyma02g E E Auxin induced protein Gma S1_at Glyma09g E E Auxin induced protein Gma A1_s_at AT2G E E Aux/IAA protein GmaAffx S1_x_at Glyma10g E E Auxin responsive protein IAA7 GmaAffx S1_at Glyma01g E E Auxin regulated protein; AUX/IAA family GmaAffx A1_at Glyma12g E E Indole 3 acetic acid amido synthetase GH3.6 GmaAffx S1_at Glyma13g E E Putative auxin efflux carrier protein GmaAffx S1_s_at Glyma15g E E regulation_of_translation response_to_auxin_stimulus GmaAffx S1_at Glyma18g E E Auxin response factor 8 Gma S1_a_at Glyma19g E E 04 1 ubiquitin dependent_protein_catabolic_process response_to_auxin_stimulus pollen_maturation s GmaAffx S1_at Glyma07g E E 05 1 Auxin induced related / indole 3 acetic acid induced related like Gma S1_at Glyma09g E E 03 1 GH1 protein; Aux/IAA protein
Nan Wang, Qingming Dong, Jingjing Li, Rohit K. Jangra, Meiyun Fan, Allan R. Brasier, Stanley M. Lemon, Lawrence M. Pfeffer, Kui Li
 Supplemental Material IRF3-dependent and NF- B-independent viral induction of the zinc-finger antiviral protein Nan Wang, Qingming Dong, Jingjing Li, Rohit K. Jangra, Meiyun Fan, Allan R. Brasier, Stanley
Supplemental Material IRF3-dependent and NF- B-independent viral induction of the zinc-finger antiviral protein Nan Wang, Qingming Dong, Jingjing Li, Rohit K. Jangra, Meiyun Fan, Allan R. Brasier, Stanley
Architecture of the Trypanosome RNA Editing Accessory Complex, MRB1
 Architecture of the Trypanosome RNA Editing Accessory Complex, MRB1 Michelle L. Ammerman, Kurtis M. Downey, Hassan Hashimi, John C. Fisk, Danielle L. Tomasello, Drahomíra Faktorová, Lucie Hanzálková, Tony
Architecture of the Trypanosome RNA Editing Accessory Complex, MRB1 Michelle L. Ammerman, Kurtis M. Downey, Hassan Hashimi, John C. Fisk, Danielle L. Tomasello, Drahomíra Faktorová, Lucie Hanzálková, Tony
Supplementary Table 2. Gossypol-induced Decrease of Levels of Reduced Cysteine-containing Peptides
 Supplementary Table 2. Gossypol-induced Decrease of Levels of Reduced Cysteine-containing Peptides Accession Description Sequences of Cys-containing peptides IPI00021263 14-3-3 protein zeta/delta DICNDVLSLLEK
Supplementary Table 2. Gossypol-induced Decrease of Levels of Reduced Cysteine-containing Peptides Accession Description Sequences of Cys-containing peptides IPI00021263 14-3-3 protein zeta/delta DICNDVLSLLEK
Supplemental Table S1. Overview of MYB transcription factor genes analyzed for expression in red and pink tomato fruit.
 Supplemental Table S1. Overview of MYB transcription factor genes analyzed for expression in red and pink tomato fruit. MYB Primer pairs TC AtMYB Forward Reverse TC199266 MYB12 AGGCTCTTGGAGGTCGTTACC CAACTCTTTCCGCATCTCAATAATC
Supplemental Table S1. Overview of MYB transcription factor genes analyzed for expression in red and pink tomato fruit. MYB Primer pairs TC AtMYB Forward Reverse TC199266 MYB12 AGGCTCTTGGAGGTCGTTACC CAACTCTTTCCGCATCTCAATAATC
Akt1 Akt kinase activity Creb signaling CCTTACAGCCCTCAAGTACTCATTC GGCGTACTCCATGACAAAGCA Arc Actin binding
 Additional File 1 (Table S1): Description and primer sequences of 80 candidate genes tested as potential synaptic plasticity formation genes in qrt-pcr array. Abbreviations: LTP: long-term potentiation,
Additional File 1 (Table S1): Description and primer sequences of 80 candidate genes tested as potential synaptic plasticity formation genes in qrt-pcr array. Abbreviations: LTP: long-term potentiation,
AZ ALACSONY HŐMÉRSÉKLET HATÁSÁRA BEKÖVETKEZŐ REDOX ÉS GÉNEXPRESSZIÓS VÁLTOZÁSOK GABONAFÉLÉKBEN
 A martonvásári agrárkutatások hatodik évtizede AZ ALAONY HŐMÉRSÉKLET HATÁSÁRA BEKÖVETKEZŐ REDOX ÉS GÉNEXPRESSZIÓS VÁLTOZÁSOK GABONAFÉLÉKBEN KOY GÁBOR, VÁGÚJFALVI ATTILA, TÓTH BALÁZS, SZALAI GABRIELLA,
A martonvásári agrárkutatások hatodik évtizede AZ ALAONY HŐMÉRSÉKLET HATÁSÁRA BEKÖVETKEZŐ REDOX ÉS GÉNEXPRESSZIÓS VÁLTOZÁSOK GABONAFÉLÉKBEN KOY GÁBOR, VÁGÚJFALVI ATTILA, TÓTH BALÁZS, SZALAI GABRIELLA,
Expression analysis of PIN genes in root tips and nodules of Lotus japonicus
 Article Expression analysis of PIN genes in root tips and nodules of Lotus japonicus Izabela Sańko-Sawczenko 1, Dominika Dmitruk 1, Barbara Łotocka 1, Elżbieta Różańska 1 and Weronika Czarnocka 1, * 1
Article Expression analysis of PIN genes in root tips and nodules of Lotus japonicus Izabela Sańko-Sawczenko 1, Dominika Dmitruk 1, Barbara Łotocka 1, Elżbieta Różańska 1 and Weronika Czarnocka 1, * 1
T-helper Type 2 driven Inflammation Defines Major Subphenotypes of Asthma
 T-helper Type 2 driven Inflammation Defines Major Subphenotypes of Asthma Prescott G. Woodruff, M.D., M.P.H., Barmak Modrek, Ph.D., David F. Choy, B.S., Guiquan Jia, M.D., Alexander R. Abbas, Ph.D., Almut
T-helper Type 2 driven Inflammation Defines Major Subphenotypes of Asthma Prescott G. Woodruff, M.D., M.P.H., Barmak Modrek, Ph.D., David F. Choy, B.S., Guiquan Jia, M.D., Alexander R. Abbas, Ph.D., Almut
Supplementary Material. Promoter deletion analysis reveals root-specific expression of the alkenal reductase gene (OS-AER1) in Oryza sativa
 10.1071/FP18237_AC CSIRO 2019 Supplementary Material: Functional Plant Biology, 2019, 46(4), 376 391. Supplementary Material Promoter deletion analysis reveals root-specific expression of the alkenal reductase
10.1071/FP18237_AC CSIRO 2019 Supplementary Material: Functional Plant Biology, 2019, 46(4), 376 391. Supplementary Material Promoter deletion analysis reveals root-specific expression of the alkenal reductase
Környezetvédelmi szakemberképzés, elméleti kutatások és gyakorlati alkalmazások a Szegedi Tudományegyetemen
 Dr. Rákhely Gábor Intézetvezető SZTE TTIK Környezettudományi Intézet Környezetvédelmi szakemberképzés, elméleti kutatások és gyakorlati alkalmazások a Szegedi Tudományegyetemen Sikeres és tanulságos kármentesítési
Dr. Rákhely Gábor Intézetvezető SZTE TTIK Környezettudományi Intézet Környezetvédelmi szakemberképzés, elméleti kutatások és gyakorlati alkalmazások a Szegedi Tudományegyetemen Sikeres és tanulságos kármentesítési
kpis(ppk20) kpis(ppk46)
 Table S1. C. elegans strains used in this study Strains carrying transgenes Strain Transgene Genotype Reference IT213 tcer-1 prom:tcer-1orf:gfp:tcer-1 3'utr unc-119(ed3) III; kpis(ppk6) This study IT283
Table S1. C. elegans strains used in this study Strains carrying transgenes Strain Transgene Genotype Reference IT213 tcer-1 prom:tcer-1orf:gfp:tcer-1 3'utr unc-119(ed3) III; kpis(ppk6) This study IT283
Modular Optimization of Hemicellulose-utilizing Pathway in. Corynebacterium glutamicum for Consolidated Bioprocessing of
 [Supplementary materials] Modular Optimization of Hemicellulose-utilizing Pathway in Corynebacterium glutamicum for Consolidated Bioprocessing of Hemicellulosic Biomass Sung Sun Yim 1, Jae Woong Choi 1,
[Supplementary materials] Modular Optimization of Hemicellulose-utilizing Pathway in Corynebacterium glutamicum for Consolidated Bioprocessing of Hemicellulosic Biomass Sung Sun Yim 1, Jae Woong Choi 1,
Átlagtermés és rekordtermés 8 növénykultúrában
 Átlagtermés és rekordtermés 8 növénykultúrában Növény Rekord termés * Átlag termés * Veszteség típusa Biotikus Abiotikus Abiotikus stressz okozta kiesés (% a rekord terméshez képest) Kukorica 19300 4600
Átlagtermés és rekordtermés 8 növénykultúrában Növény Rekord termés * Átlag termés * Veszteség típusa Biotikus Abiotikus Abiotikus stressz okozta kiesés (% a rekord terméshez képest) Kukorica 19300 4600
Supporting Information
 Supporting Information Paired design of dcas9 as a systematic platform for the detection of featured nucleic acid sequences in pathogenic strains Yihao Zhang 1,2,8, Long Qian 4,8, Weijia Wei 1,3,7,8, Yu
Supporting Information Paired design of dcas9 as a systematic platform for the detection of featured nucleic acid sequences in pathogenic strains Yihao Zhang 1,2,8, Long Qian 4,8, Weijia Wei 1,3,7,8, Yu
Apoptózis. 1. Bevezetés 2. Külső jelút 3. Belső jelút
 Jelutak Apoptózis 1. Bevezetés 2. Külső jelút 3. Belső jelút Apoptózis Sejtmag 1. Kondenzálódó sejtmag apoptózis autofágia nekrózis Lefűződések Összezsugorodás Fragmentálódó sejtmag Apoptotikus test Fagocita
Jelutak Apoptózis 1. Bevezetés 2. Külső jelút 3. Belső jelút Apoptózis Sejtmag 1. Kondenzálódó sejtmag apoptózis autofágia nekrózis Lefűződések Összezsugorodás Fragmentálódó sejtmag Apoptotikus test Fagocita
Jelutak. Apoptózis. Apoptózis Bevezetés 2. Külső jelút 3. Belső jelút. apoptózis autofágia nekrózis. Sejtmag. Kondenzálódó sejtmag
 Jelutak Apoptózis 1. Bevezetés 2. Külső jelút 3. Belső jelút Apoptózis Sejtmag Kondenzálódó sejtmag 1. autofágia nekrózis Lefűződések Összezsugorodás Fragmentálódó sejtmag Apoptotikus test Fagocita bekebelezi
Jelutak Apoptózis 1. Bevezetés 2. Külső jelút 3. Belső jelút Apoptózis Sejtmag Kondenzálódó sejtmag 1. autofágia nekrózis Lefűződések Összezsugorodás Fragmentálódó sejtmag Apoptotikus test Fagocita bekebelezi
Supplemental Information. Investigation of Penicillin Binding Protein. (PBP)-like Peptide Cyclase and Hydrolase
 Cell Chemical Biology, Volume upplemental Information Investigation of Penicillin Binding Protein (PBP)-like Peptide Cyclase and ydrolase in urugamide on-ribosomal Peptide Biosynthesis Yongjun Zhou, Xiao
Cell Chemical Biology, Volume upplemental Information Investigation of Penicillin Binding Protein (PBP)-like Peptide Cyclase and ydrolase in urugamide on-ribosomal Peptide Biosynthesis Yongjun Zhou, Xiao
Flowering time. Col C24 Cvi C24xCol C24xCvi ColxCvi
 Flowering time Rosette leaf number 50 45 40 35 30 25 20 15 10 5 0 Col C24 Cvi C24xCol C24xCvi ColxCvi Figure S1. Flowering time in three F 1 hybrids and their parental lines as measured by leaf number
Flowering time Rosette leaf number 50 45 40 35 30 25 20 15 10 5 0 Col C24 Cvi C24xCol C24xCvi ColxCvi Figure S1. Flowering time in three F 1 hybrids and their parental lines as measured by leaf number
Horizontal gene transfer drives adaptive colonization of apple trees by the fungal pathogen Valsa mali. Zhiyuan Yin, Baitao Zhu, Hao Feng, Lili Huang*
 Supplementary Information Horizontal gene transfer drives adaptive colonization of apple trees by the fungal pathogen Valsa mali Zhiyuan Yin, Baitao Zhu, Hao Feng, Lili Huang* State Key Laboratory of Crop
Supplementary Information Horizontal gene transfer drives adaptive colonization of apple trees by the fungal pathogen Valsa mali Zhiyuan Yin, Baitao Zhu, Hao Feng, Lili Huang* State Key Laboratory of Crop
Supplemental Information. RNase H1-Dependent Antisense Oligonucleotides. Are Robustly Active in Directing RNA Cleavage
 YMTHE, Volume 25 Supplemental Information RNase H1-Dependent Antisense Oligonucleotides Are Robustly Active in Directing RNA Cleavage in Both the Cytoplasm and the Nucleus Xue-Hai Liang, Hong Sun, Joshua
YMTHE, Volume 25 Supplemental Information RNase H1-Dependent Antisense Oligonucleotides Are Robustly Active in Directing RNA Cleavage in Both the Cytoplasm and the Nucleus Xue-Hai Liang, Hong Sun, Joshua
A LISZTHARMAT ÉS A FOGÉKONY SZŐLŐ KÖZÖTTI MOLEKULÁRIS KAPCSOLAT. Tóth Zsófia 1 -Kiss Erzsébet 2
 A LISZTHARMAT ÉS A FOGÉKONY SZŐLŐ KÖZÖTTI MOLEKULÁRIS KAPCSOLAT Tóth Zsófia 1 -Kiss Erzsébet 2 1 Szent István Egyetem, Genetika és Biotechnológiai Intézet, Gödöllő 2100 Páter Károly utca 1., 0628/522069,
A LISZTHARMAT ÉS A FOGÉKONY SZŐLŐ KÖZÖTTI MOLEKULÁRIS KAPCSOLAT Tóth Zsófia 1 -Kiss Erzsébet 2 1 Szent István Egyetem, Genetika és Biotechnológiai Intézet, Gödöllő 2100 Páter Károly utca 1., 0628/522069,
A BAKTÉRIUMOK ÁLTAL KIVÁLTOTT NÖVÉNYI ÁLTALÁNOS VÉDEKEZÉS (BR) HATÁSA VÍRUSFERTŐZÉSRE, VALAMINT BR SORÁN MEGVÁLTOZOTT AKTIVITÁSÚ GÉNEK VIZSGÁLATA
 DOKTORI ÉRTEKEZÉS A BAKTÉRIUMOK ÁLTAL KIVÁLTOTT NÖVÉNYI ÁLTALÁNOS VÉDEKEZÉS (BR) HATÁSA VÍRUSFERTŐZÉSRE, VALAMINT BR SORÁN MEGVÁLTOZOTT AKTIVITÁSÚ GÉNEK VIZSGÁLATA ZSIROS LÁSZLÓ TÉMAVEZETŐK: PALKOVICS
DOKTORI ÉRTEKEZÉS A BAKTÉRIUMOK ÁLTAL KIVÁLTOTT NÖVÉNYI ÁLTALÁNOS VÉDEKEZÉS (BR) HATÁSA VÍRUSFERTŐZÉSRE, VALAMINT BR SORÁN MEGVÁLTOZOTT AKTIVITÁSÚ GÉNEK VIZSGÁLATA ZSIROS LÁSZLÓ TÉMAVEZETŐK: PALKOVICS
pleics-06 Amp pleics-13 Amp/Zeo N-His 10/ 3 x
 Protease Sequence primers Vector Name AR Tag/Flag cleavage Tag and linker pleics-01 Amp N-His 6 TEV Extra SM His6-SSGVDLGT-TEV site-1 T7 Promoter pleics-01-seq-r pleics-02 Amp N-GST TEV Extra SM GST-SS-TEV
Protease Sequence primers Vector Name AR Tag/Flag cleavage Tag and linker pleics-01 Amp N-His 6 TEV Extra SM His6-SSGVDLGT-TEV site-1 T7 Promoter pleics-01-seq-r pleics-02 Amp N-GST TEV Extra SM GST-SS-TEV
Forensic SNP Genotyping using Nanopore MinION Sequencing
 Forensic SNP Genotyping using Nanopore MinION Sequencing AUTHORS Senne Cornelis 1, Yannick Gansemans 1, Lieselot Deleye 1, Dieter Deforce 1,#,Filip Van Nieuwerburgh 1,#,* 1 Laboratory of Pharmaceutical
Forensic SNP Genotyping using Nanopore MinION Sequencing AUTHORS Senne Cornelis 1, Yannick Gansemans 1, Lieselot Deleye 1, Dieter Deforce 1,#,Filip Van Nieuwerburgh 1,#,* 1 Laboratory of Pharmaceutical
DOKTORI ÉRTEKEZÉS TÉZISEI AZ OPPORTUNISTA HUMÁNPATOGÉN CANDIDA PARAPSILOSIS ÉLESZTŐGOMBA ELLENI TERMÉSZETES ÉS ADAPTÍV IMMUNVÁLASZ VIZSGÁLATA
 DOKTORI ÉRTEKEZÉS TÉZISEI AZ OPPORTUNISTA HUMÁNPATOGÉN CANDIDA PARAPSILOSIS ÉLESZTŐGOMBA ELLENI TERMÉSZETES ÉS ADAPTÍV IMMUNVÁLASZ VIZSGÁLATA TÓTH ADÉL TÉMAVEZETŐ: DR. GÁCSER ATTILA TUDOMÁNYOS FŐMUNKATÁRS
DOKTORI ÉRTEKEZÉS TÉZISEI AZ OPPORTUNISTA HUMÁNPATOGÉN CANDIDA PARAPSILOSIS ÉLESZTŐGOMBA ELLENI TERMÉSZETES ÉS ADAPTÍV IMMUNVÁLASZ VIZSGÁLATA TÓTH ADÉL TÉMAVEZETŐ: DR. GÁCSER ATTILA TUDOMÁNYOS FŐMUNKATÁRS
Genetikai panel kialakítása a hazai tejhasznú szarvasmarha állományok hasznos élettartamának növelésére
 Genetikai panel kialakítása a hazai tejhasznú szarvasmarha állományok hasznos élettartamának növelésére Dr. Czeglédi Levente Dr. Béri Béla Kutatás-fejlesztés támogatása a megújuló energiaforrások és agrár
Genetikai panel kialakítása a hazai tejhasznú szarvasmarha állományok hasznos élettartamának növelésére Dr. Czeglédi Levente Dr. Béri Béla Kutatás-fejlesztés támogatása a megújuló energiaforrások és agrár
Involvement of ER Stress in Dysmyelination of Pelizaeus-Merzbacher Disease with PLP1 Missense Mutations Shown by ipsc-derived Oligodendrocytes
 Stem Cell Reports, Volume 2 Supplemental Information Involvement of ER Stress in Dysmyelination of Pelizaeus-Merzbacher Disease with PLP1 Missense Mutations Shown by ipsc-derived Oligodendrocytes Yuko
Stem Cell Reports, Volume 2 Supplemental Information Involvement of ER Stress in Dysmyelination of Pelizaeus-Merzbacher Disease with PLP1 Missense Mutations Shown by ipsc-derived Oligodendrocytes Yuko
13. RNS szintézis és splicing
 13. RNS szintézis és splicing 1 Visszatekintés: Az RNS típusai és szerkezete Hírvivő RNS = mrns (messenger RNA = mrna) : fehérjeszintézis pre-mrns érett mrns (intronok kivágódnak = splicing) Transzfer
13. RNS szintézis és splicing 1 Visszatekintés: Az RNS típusai és szerkezete Hírvivő RNS = mrns (messenger RNA = mrna) : fehérjeszintézis pre-mrns érett mrns (intronok kivágódnak = splicing) Transzfer
Supplementary materials to: Whole-mount single molecule FISH method for zebrafish embryo
 Supplementary materials to: Whole-mount single molecule FISH method for zebrafish embryo Yuma Oka and Thomas N. Sato Supplementary Figure S1. Whole-mount smfish with and without the methanol pretreatment.
Supplementary materials to: Whole-mount single molecule FISH method for zebrafish embryo Yuma Oka and Thomas N. Sato Supplementary Figure S1. Whole-mount smfish with and without the methanol pretreatment.
Receptor Tyrosine-Kinases
 Receptor Tyrosine-Kinases MAPkinase pathway PI3Kinase Protein Kinase B pathway PI3K/PK-B pathway Phosphatidyl-inositol-bisphosphate...(PI(4,5)P 2...) Phosphatidyl-inositol-3-kinase (PI3K) Protein kinase
Receptor Tyrosine-Kinases MAPkinase pathway PI3Kinase Protein Kinase B pathway PI3K/PK-B pathway Phosphatidyl-inositol-bisphosphate...(PI(4,5)P 2...) Phosphatidyl-inositol-3-kinase (PI3K) Protein kinase
Supplementary Table 1. Cystometric parameters in sham-operated wild type and Trpv4 -/- rats during saline infusion and
 WT sham Trpv4 -/- sham Saline 10µM GSK1016709A P value Saline 10µM GSK1016709A P value Number 10 10 8 8 Intercontractile interval (sec) 143 (102 155) 98.4 (71.4 148) 0.01 96 (92 121) 109 (95 123) 0.3 Voided
WT sham Trpv4 -/- sham Saline 10µM GSK1016709A P value Saline 10µM GSK1016709A P value Number 10 10 8 8 Intercontractile interval (sec) 143 (102 155) 98.4 (71.4 148) 0.01 96 (92 121) 109 (95 123) 0.3 Voided
SOLiD Technology. library preparation & Sequencing Chemistry (sequencing by ligation!) Imaging and analysis. Application specific sample preparation
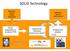 SOLiD Technology Application specific sample preparation Application specific data analysis library preparation & emulsion PCR Sequencing Chemistry (sequencing by ligation!) Imaging and analysis SOLiD
SOLiD Technology Application specific sample preparation Application specific data analysis library preparation & emulsion PCR Sequencing Chemistry (sequencing by ligation!) Imaging and analysis SOLiD
TRANSZPORTEREK Szakács Gergely
 TRANSZPORTEREK Szakács Gergely Összefoglalás A biológiai membránokon keresztüli anyagáramlást számos membránfehérje szabályozza. E fehérjék változatos funkciója és megjelenésük mintázata biztosítja a sejtek
TRANSZPORTEREK Szakács Gergely Összefoglalás A biológiai membránokon keresztüli anyagáramlást számos membránfehérje szabályozza. E fehérjék változatos funkciója és megjelenésük mintázata biztosítja a sejtek
Bevezetés Áttekintés
 Bevezetés Áttekintés Fogalmak Egy lymphocita életének áttekintése Human Ig-izotípusok morfológiája Human Ig-izotípusok funkciói Miért? Válasz: más Ig effektor funkciók C fixáció FcR Polymerizáció Izotípusváltás
Bevezetés Áttekintés Fogalmak Egy lymphocita életének áttekintése Human Ig-izotípusok morfológiája Human Ig-izotípusok funkciói Miért? Válasz: más Ig effektor funkciók C fixáció FcR Polymerizáció Izotípusváltás
AZ ÁRPA SZÁRAZSÁGTŰRÉSÉNEK VIZSGÁLATA: QTL- ÉS ASSZOCIÁCIÓS ANALÍZIS, MARKER ALAPÚ SZELEKCIÓ, TILLING
 Hagyomány és haladás a növénynemesítésben AZ ÁRPA SZÁRAZSÁGTŰRÉSÉNEK VIZSGÁLATA: QTL- ÉS ASSZOCIÁCIÓS ANALÍZIS, MARKER ALAPÚ SZELEKCIÓ, BÁLINT ANDRÁS FERENC 1, SZIRA FRUZSINA 1, ANDREAS BÖRNER 2, KERSTIN
Hagyomány és haladás a növénynemesítésben AZ ÁRPA SZÁRAZSÁGTŰRÉSÉNEK VIZSGÁLATA: QTL- ÉS ASSZOCIÁCIÓS ANALÍZIS, MARKER ALAPÚ SZELEKCIÓ, BÁLINT ANDRÁS FERENC 1, SZIRA FRUZSINA 1, ANDREAS BÖRNER 2, KERSTIN
KUTATÁSI TÉMAJAVASLAT ITC hallgatónak jelentkezők számára ROP GTPÁZOK ÁLTAL KÖZVETÍTETT JELÁTVITEL NÖVÉNYEKBEN
 KUTATÁSI TÉMAJAVASLAT ITC hallgatónak jelentkezők számára témavezető: MÉNESI Dalma, M.Sc. intézet: Növénybiológiai Intézet e-mail cím: menesi.dalma@brc.mta.hu CV: http://www.szbk.hu/file/cv/plant_menesi_dalma_hu.pdf
KUTATÁSI TÉMAJAVASLAT ITC hallgatónak jelentkezők számára témavezető: MÉNESI Dalma, M.Sc. intézet: Növénybiológiai Intézet e-mail cím: menesi.dalma@brc.mta.hu CV: http://www.szbk.hu/file/cv/plant_menesi_dalma_hu.pdf
Manuscript Title: Identification of a thermostable fungal lytic polysaccharide monooxygenase and
 1 2 3 4 5 Journal name: Applied Microbiology and Biotechnology Manuscript Title: Identification of a thermostable fungal lytic polysaccharide monooxygenase and evaluation of its effect on lignocellulosic
1 2 3 4 5 Journal name: Applied Microbiology and Biotechnology Manuscript Title: Identification of a thermostable fungal lytic polysaccharide monooxygenase and evaluation of its effect on lignocellulosic
Dr. Ottó Szabolcs Országos Onkológiai Intézet
 Nemzeti Kutatási és Fejlesztési Program 1. Főirány: Életminőség javítása Nemzeti Onkológiai Kutatás-Fejlesztési Konzorcium a daganatos halálozás csökkentésére 1/48/2001. Részjelentés: 200. November 0.-2004.
Nemzeti Kutatási és Fejlesztési Program 1. Főirány: Életminőség javítása Nemzeti Onkológiai Kutatás-Fejlesztési Konzorcium a daganatos halálozás csökkentésére 1/48/2001. Részjelentés: 200. November 0.-2004.
Gene Expression Profiling of Vascular Endothelial Cells Exposed to Fluid Mechanical Forces: Relevance for Focal Susceptibility to Atherosclerosis
 Endothelium, 11:45 57, 2004 Copyright c Taylor & Francis Inc. ISSN: 1062-3329 print / 1029-2373 online DOI: 10.1080/10623320490432470 Gene Expression Profiling of Vascular Endothelial Cells Exposed to
Endothelium, 11:45 57, 2004 Copyright c Taylor & Francis Inc. ISSN: 1062-3329 print / 1029-2373 online DOI: 10.1080/10623320490432470 Gene Expression Profiling of Vascular Endothelial Cells Exposed to
Markerless Escherichia coli rrn Deletion Strains for Genetic Determination of Ribosomal Binding Sites
 Markerless Escherichia coli rrn Deletion Strains for Genetic Determination of Ribosomal Binding Sites Selwyn Quan *, Ole Skovgaard, Robert E. McLaughlin, Ed T. Buurman,1, and Catherine L. Squires * Department
Markerless Escherichia coli rrn Deletion Strains for Genetic Determination of Ribosomal Binding Sites Selwyn Quan *, Ole Skovgaard, Robert E. McLaughlin, Ed T. Buurman,1, and Catherine L. Squires * Department
Supporting Information
 Supporting Information Cell-free GFP simulations Cell-free simulations of degfp production were consistent with experimental measurements (Fig. S1). Dual emmission GFP was produced under a P70a promoter
Supporting Information Cell-free GFP simulations Cell-free simulations of degfp production were consistent with experimental measurements (Fig. S1). Dual emmission GFP was produced under a P70a promoter
pjnc-pgluc Codon-optimized Gaussia luciferase (pgluc) Vector backbone: JM83 pjnc-pgluc is resistant to ampicillin and neomycin High or low copy:
 pjnc-pgluc Gene/Insert name: Codon-optimized Gaussia luciferase (pgluc) Vector backbone: pcmv-jnc Vector type: Mammalian cells Backbone size w/o insert (bp): 5375 Bacterial resistance: Ampicillin and neomycin
pjnc-pgluc Gene/Insert name: Codon-optimized Gaussia luciferase (pgluc) Vector backbone: pcmv-jnc Vector type: Mammalian cells Backbone size w/o insert (bp): 5375 Bacterial resistance: Ampicillin and neomycin
The beet R locus encodes a new cytochrome P450 required for red. betalain production.
 The beet R locus encodes a new cytochrome P450 required for red betalain production. Gregory J. Hatlestad, Rasika M. Sunnadeniya, Neda A. Akhavan, Antonio Gonzalez, Irwin L. Goldman, J. Mitchell McGrath,
The beet R locus encodes a new cytochrome P450 required for red betalain production. Gregory J. Hatlestad, Rasika M. Sunnadeniya, Neda A. Akhavan, Antonio Gonzalez, Irwin L. Goldman, J. Mitchell McGrath,
University of Bristol - Explore Bristol Research
 Al-Salihi, S. A. A., Scott, T. A., Bailey, A. M., & Foster, G. D. (2017). Improved vectors for Agrobacterium mediated genetic manipulation of Hypholoma spp. and other homobasidiomycetes. Journal of Microbiological
Al-Salihi, S. A. A., Scott, T. A., Bailey, A. M., & Foster, G. D. (2017). Improved vectors for Agrobacterium mediated genetic manipulation of Hypholoma spp. and other homobasidiomycetes. Journal of Microbiological
Fiatal Biotechnológusok Országos Konferenciája SZERDA
 Fiatal Biotechnológusok Országos Konferenciája 2018 2018.03.28. - SZERDA 9:00-10:00 REGISZTRÁCIÓ, POSZTEREK ELHELYEZÉSE 10:00-10:15 MEGNYITÓ Fehér Attila, Tamás László 10:15-10:45 PLENÁRIS ELŐADÁS - PLE1
Fiatal Biotechnológusok Országos Konferenciája 2018 2018.03.28. - SZERDA 9:00-10:00 REGISZTRÁCIÓ, POSZTEREK ELHELYEZÉSE 10:00-10:15 MEGNYITÓ Fehér Attila, Tamás László 10:15-10:45 PLENÁRIS ELŐADÁS - PLE1
PIAL1 (At1g08910) ORF sequence
 PIAL1 (At1g08910) ORF sequence ATGGTTATTCCGGCGACTTCTAGGTTTGGGTTTCGTGCTGAATTCAACACCAAGGAGTTTCAAGCTTCCTGCATCTCTCT! CGCTAATGAAATCGATGCGGCTATTGGGAGAAATGAAGTTCCAGGGAATATTCAAGAGCTCGCTTTAATCCTCAATAATG! TGTGCCGACGTAAATGTGATGATTATCAAACCAGGGCGGTGGTAATGGCGCTGATGATCTCAGTCAAGAGTGCTTGTCAG!
PIAL1 (At1g08910) ORF sequence ATGGTTATTCCGGCGACTTCTAGGTTTGGGTTTCGTGCTGAATTCAACACCAAGGAGTTTCAAGCTTCCTGCATCTCTCT! CGCTAATGAAATCGATGCGGCTATTGGGAGAAATGAAGTTCCAGGGAATATTCAAGAGCTCGCTTTAATCCTCAATAATG! TGTGCCGACGTAAATGTGATGATTATCAAACCAGGGCGGTGGTAATGGCGCTGATGATCTCAGTCAAGAGTGCTTGTCAG!
DI-, TETRA- ÉS HEXAPLOID TRITICUM FAJOK GENOMJAINAK ELEMZÉSE ÉS AZOK ÖSSZEHASONLÍTÁSA FLUORESZCENS IN SITU HIBRIDIZÁCIÓVAL
 Hagyomány és haladás a növénynemesítésben DI-, TETRA- ÉS HEXAPLOID TRITICUM FAJOK GENOMJAINAK ELEMZÉSE ÉS AZOK ÖSSZEHASONLÍTÁSA FLUORESZCENS IN SITU HIBRIDIZÁCIÓVAL VARGA MÓNIKA, MOLNÁR ISTVÁN ÉS KOVÁCS
Hagyomány és haladás a növénynemesítésben DI-, TETRA- ÉS HEXAPLOID TRITICUM FAJOK GENOMJAINAK ELEMZÉSE ÉS AZOK ÖSSZEHASONLÍTÁSA FLUORESZCENS IN SITU HIBRIDIZÁCIÓVAL VARGA MÓNIKA, MOLNÁR ISTVÁN ÉS KOVÁCS
Szövettípusokra jellemző folyamatok az UV RESISTANCE LOCUS 8 (UVR8) UV-B fotoreceptor jelátviteli rendszerében
 Szövettípusokra jellemző folyamatok az UV RESISTANCE LOCUS 8 (UVR8) UV-B fotoreceptor jelátviteli rendszerében Doktori (Ph.D.) értekezés Készítette: Bernula Péter Témavezetők: Prof. Dr. Nagy Ferenc Dr.
Szövettípusokra jellemző folyamatok az UV RESISTANCE LOCUS 8 (UVR8) UV-B fotoreceptor jelátviteli rendszerében Doktori (Ph.D.) értekezés Készítette: Bernula Péter Témavezetők: Prof. Dr. Nagy Ferenc Dr.
Az izomváltozások szabályozása a smoothelin-like 1 fehérje által. Tudományos hétfő előadássorozat. Lontay Beáta Orvosi Vegytani Intézet
 Az izomváltozások szabályozása a smoothelin-like 1 fehérje által Tudományos hétfő előadássorozat Lontay Beáta Orvosi Vegytani Intézet Kutatási témák: 1. A protein foszfatázok és kinázok szerepe a neurotranszmitter
Az izomváltozások szabályozása a smoothelin-like 1 fehérje által Tudományos hétfő előadássorozat Lontay Beáta Orvosi Vegytani Intézet Kutatási témák: 1. A protein foszfatázok és kinázok szerepe a neurotranszmitter
Transzgénikus növények alkalmazása a funkcionális genomikai kutatásokban
 Transzgénikus növények alkalmazása a funkcionális genomikai kutatásokban MTA Agrártudományi Kutatóközpont Növényi Sejtbiológia Osztály Gyakorlatban alkalmazható transzgénikus növények létrehozásának alapfeltétele:
Transzgénikus növények alkalmazása a funkcionális genomikai kutatásokban MTA Agrártudományi Kutatóközpont Növényi Sejtbiológia Osztály Gyakorlatban alkalmazható transzgénikus növények létrehozásának alapfeltétele:
Léptetőmotorok. Előnyök: Hátrányok:
 Léptetőmotorok A léptetőmotorok lényeges tulajdonsága, hogy egy körülforduláshoz hány lépés szükséges. Ezt megadhatják fokban, ekkor az egy lépésre eső szögelfordulást adják meg. Illetve megadhatják az
Léptetőmotorok A léptetőmotorok lényeges tulajdonsága, hogy egy körülforduláshoz hány lépés szükséges. Ezt megadhatják fokban, ekkor az egy lépésre eső szögelfordulást adják meg. Illetve megadhatják az
A doktori értekezés tézisei. A növényi NRP fehérjék lehetséges szerepe a hiszton defoszforiláció szabályozásában, és a hőstressz válaszban.
 A doktori értekezés tézisei A növényi NRP fehérjék lehetséges szerepe a hiszton defoszforiláció szabályozásában, és a hőstressz válaszban. Bíró Judit Témavezető: Dr. Fehér Attila Magyar Tudományos Akadémia
A doktori értekezés tézisei A növényi NRP fehérjék lehetséges szerepe a hiszton defoszforiláció szabályozásában, és a hőstressz válaszban. Bíró Judit Témavezető: Dr. Fehér Attila Magyar Tudományos Akadémia
Extracelluláris vezikulák szerepe autoimmun betegségekben. Pállinger Éva
 Extracelluláris vezikulák szerepe autoimmun betegségekben Pállinger Éva Az extracelluláris vezikulák osztályozása György-Szabó et al. Cell Mol Life Sci 2011. Exoszómák képződése Mikrovezikulák képződése
Extracelluláris vezikulák szerepe autoimmun betegségekben Pállinger Éva Az extracelluláris vezikulák osztályozása György-Szabó et al. Cell Mol Life Sci 2011. Exoszómák képződése Mikrovezikulák képződése
Eredeti gyógyszerkutatás. ELTE TTK vegyészhallgatók számára Dr Arányi Péter 2009 március, 4.ea. Szerkezet optimalizálás (I.)
 Eredeti gyógyszerkutatás ELTE TTK vegyészhallgatók számára Dr Arányi Péter 2009 március, 4.ea. Szerkezet optimalizálás (I.) 1 How do we proceed? New target Internal Literature Validation Selected Target
Eredeti gyógyszerkutatás ELTE TTK vegyészhallgatók számára Dr Arányi Péter 2009 március, 4.ea. Szerkezet optimalizálás (I.) 1 How do we proceed? New target Internal Literature Validation Selected Target
Az Oxidatív stressz hatása a PIBF receptor alegységek összeszerelődésére.
 Újabban világossá vált, hogy a Progesterone-induced blocking factor (PIBF) amely a progesteron számos immunológiai hatását közvetíti, nem csupán a lymphocytákban és terhességgel asszociált szövetekben,
Újabban világossá vált, hogy a Progesterone-induced blocking factor (PIBF) amely a progesteron számos immunológiai hatását közvetíti, nem csupán a lymphocytákban és terhességgel asszociált szövetekben,
 www.szentagothailab.sote.hu Glükokortikoid receptorok felépítése, műküdése Szignalizációs kapcsolatok szívizom, bél immunrendszer Idegrendszer (nem szinaptikus kapcsolatok) Korai embrionális fejlődés Szignalizációs
www.szentagothailab.sote.hu Glükokortikoid receptorok felépítése, műküdése Szignalizációs kapcsolatok szívizom, bél immunrendszer Idegrendszer (nem szinaptikus kapcsolatok) Korai embrionális fejlődés Szignalizációs
Suppl. Materials. Polyhydroxyalkanoate (PHA) Granules Have no Phospholipids. Germany
 Suppl. Materials Polyhydroxyalkanoate (PHA) Granules Have no Phospholipids Stephanie Bresan 1, Anna Sznajder 1, Waldemar Hauf 2, Karl Forchhammer 2, Daniel Pfeiffer 3 and Dieter Jendrossek 1 1 Institute
Suppl. Materials Polyhydroxyalkanoate (PHA) Granules Have no Phospholipids Stephanie Bresan 1, Anna Sznajder 1, Waldemar Hauf 2, Karl Forchhammer 2, Daniel Pfeiffer 3 and Dieter Jendrossek 1 1 Institute
Paralóg jelátviteli útvonalak finom szabályozásának szerkezeti alapú vizsgálata
 Paralóg jelátviteli útvonalak finom szabályozásának szerkezeti alapú vizsgálata Tézisfüzet Glatz Gábor okleveles biológus ELTE Biológia Doktori Iskola, Szerkezeti Biokémia Program Iskolavezető: Dr. Erdei
Paralóg jelátviteli útvonalak finom szabályozásának szerkezeti alapú vizsgálata Tézisfüzet Glatz Gábor okleveles biológus ELTE Biológia Doktori Iskola, Szerkezeti Biokémia Program Iskolavezető: Dr. Erdei
Expressziós microarray. Dr. Győrffy Balázs
 Expressziós microarray Dr. Győrffy Balázs Áttekintő 1. A technológia 2. Feldolgozás 2.1. Minőség-ellenőrzés 2.2. Jellemző kiválasztás 2.3. Vizualizálás 2.4. Kereszt-elemzés 2.5. Online diagnosztika 3.
Expressziós microarray Dr. Győrffy Balázs Áttekintő 1. A technológia 2. Feldolgozás 2.1. Minőség-ellenőrzés 2.2. Jellemző kiválasztás 2.3. Vizualizálás 2.4. Kereszt-elemzés 2.5. Online diagnosztika 3.
Mozgás - Gyógyszer TÁMOP-4.2.2/08/1-2008-0006
 TÁMOP-4.2.2/08/1-2008-0006 Mozgás - Gyógyszer Varga Csaba, Puskás László G, Balogh László, Kovács Gyula, Kiss Gábor, Seres Erika, Pósa Anikó, Magyariné Berkó Anikó, Molnár Andor, Szablics Péter, Orbán
TÁMOP-4.2.2/08/1-2008-0006 Mozgás - Gyógyszer Varga Csaba, Puskás László G, Balogh László, Kovács Gyula, Kiss Gábor, Seres Erika, Pósa Anikó, Magyariné Berkó Anikó, Molnár Andor, Szablics Péter, Orbán
Megérkeztek a legújabb DELL Latitude E modellek. DELL NB Latitude E5500
 Megérkeztek a legújabb DELL Latitude E modellek DELL NB Latitude E5500 Core 2 Duo P8400 2,26GHz 160GB 7200rpm Hard drive (Free Fall Sensor) 2GB 800MHz DDR2 memory (2 x 1GB) DVD+/-RW 15.4in Widescreen WXGA+
Megérkeztek a legújabb DELL Latitude E modellek DELL NB Latitude E5500 Core 2 Duo P8400 2,26GHz 160GB 7200rpm Hard drive (Free Fall Sensor) 2GB 800MHz DDR2 memory (2 x 1GB) DVD+/-RW 15.4in Widescreen WXGA+
Trinucleotide Repeat Diseases: CRISPR Cas9 PacBio no PCR Sequencing MFMER slide-1
 Trinucleotide Repeat Diseases: CRISPR Cas9 PacBio no PCR Sequencing 2015 MFMER slide-1 Fuch s Eye Disease TCF 4 gene Fuchs occurs in about 4% of the US population. Leads to deteriorating vision without
Trinucleotide Repeat Diseases: CRISPR Cas9 PacBio no PCR Sequencing 2015 MFMER slide-1 Fuch s Eye Disease TCF 4 gene Fuchs occurs in about 4% of the US population. Leads to deteriorating vision without
Supporting Information
 Supporting Information Plasmid Construction DNA cloning was carried out according to standard protocols. The sequence of all plasmids was confirmed by DNA sequencing and amplified using the E. coli strain
Supporting Information Plasmid Construction DNA cloning was carried out according to standard protocols. The sequence of all plasmids was confirmed by DNA sequencing and amplified using the E. coli strain
Biodízel előállítás alapanyagainak és melléktermékeinek vizsgálata állatkísérletekben. DOKTORI (PhD) ÉRTEKEZÉS TÉZISEI. Dr.
 Biodízel előállítás alapanyagainak és melléktermékeinek vizsgálata állatkísérletekben DOKTORI (PhD) ÉRTEKEZÉS TÉZISEI Dr. Szele Eszter B-149 Daganatok molekuláris epidemiológiája Doktori Iskola vezetője:
Biodízel előállítás alapanyagainak és melléktermékeinek vizsgálata állatkísérletekben DOKTORI (PhD) ÉRTEKEZÉS TÉZISEI Dr. Szele Eszter B-149 Daganatok molekuláris epidemiológiája Doktori Iskola vezetője:
Proper gibberellin localization in vascular tissue is required to regulate adventitious root development in tobacco
 Journal of Experimental Botany, Vol. 64, No. 11, pp. 3411 3424, 2013 doi:10.1093/jxb/ert186 This paper is available online free of all access charges (see http://jxb.oxfordjournals.org/open_access.html
Journal of Experimental Botany, Vol. 64, No. 11, pp. 3411 3424, 2013 doi:10.1093/jxb/ert186 This paper is available online free of all access charges (see http://jxb.oxfordjournals.org/open_access.html
Biotranszformáció. Csala Miklós. Semmelweis Egyetem Orvosi Vegytani, Molekuláris Biológiai és Patobiokémiai Intézet
 Biotranszformáció Csala Miklós Semmelweis Egyetem rvosi Vegytani, Molekuláris Biológiai és Patobiokémiai Intézet direkt bilirubin hem (porfirin) X koleszterin X epesavak piruvát acil-koa citoplazma piruvát
Biotranszformáció Csala Miklós Semmelweis Egyetem rvosi Vegytani, Molekuláris Biológiai és Patobiokémiai Intézet direkt bilirubin hem (porfirin) X koleszterin X epesavak piruvát acil-koa citoplazma piruvát
Vezikuláris transzport
 Molekuláris Sejtbiológia Vezikuláris transzport Dr. habil KŐHIDAI László Semmelweis Egyetem, Genetikai, Sejt- és Immunbiológiai Intézet 2005. november 3. Intracelluláris vezikul uláris transzport Kommunikáció
Molekuláris Sejtbiológia Vezikuláris transzport Dr. habil KŐHIDAI László Semmelweis Egyetem, Genetikai, Sejt- és Immunbiológiai Intézet 2005. november 3. Intracelluláris vezikul uláris transzport Kommunikáció
Egy új genetikai módszerrel azonosított Arabidopsis A4A hősokk faktor funkcionális jellemzése
 Egy új genetikai módszerrel azonosított Arabidopsis A4A hősokk faktor funkcionális jellemzése Tézisfüzet Immaculada Pérez Salamó Témavezető: Dr. Szabados László MTA Szegedi Biológiai Központ Növénybiológia
Egy új genetikai módszerrel azonosított Arabidopsis A4A hősokk faktor funkcionális jellemzése Tézisfüzet Immaculada Pérez Salamó Témavezető: Dr. Szabados László MTA Szegedi Biológiai Központ Növénybiológia
CzB 2010. Élettan: a sejt
 CzB 2010. Élettan: a sejt Sejt - az élet alapvető egysége Prokaryota -egysejtű -nincs sejtmag -nincsenek sejtszervecskék -DNS = egy gyűrű - pl., bactériumok Eukaryota -egy-/többsejtű -sejmag membránnal
CzB 2010. Élettan: a sejt Sejt - az élet alapvető egysége Prokaryota -egysejtű -nincs sejtmag -nincsenek sejtszervecskék -DNS = egy gyűrű - pl., bactériumok Eukaryota -egy-/többsejtű -sejmag membránnal
MikroRNS-ek szerepe az autoimmun és reumatológiai kórképek kialakulásában
 MikroRNS-ek szerepe az autoimmun és reumatológiai kórképek kialakulásában Butz Henriett SE Laboratóriumi Medicina Intézet MTA-SE Molekuláris Medicina Kutatócsoport 2017. április 20-21 Amiről szó lesz 1.
MikroRNS-ek szerepe az autoimmun és reumatológiai kórképek kialakulásában Butz Henriett SE Laboratóriumi Medicina Intézet MTA-SE Molekuláris Medicina Kutatócsoport 2017. április 20-21 Amiről szó lesz 1.
Logisztikus regresszió október 27.
 Logisztikus regresszió 2017. október 27. Néhány példa Mi a valószínűsége egy adott betegségnek a páciens bizonyos megfigyelt jellemzői (pl. nem, életkor, laboreredmények, BMI stb.) alapján? Mely genetikai
Logisztikus regresszió 2017. október 27. Néhány példa Mi a valószínűsége egy adott betegségnek a páciens bizonyos megfigyelt jellemzői (pl. nem, életkor, laboreredmények, BMI stb.) alapján? Mely genetikai
Szakmai Zárójelentés
 48765 számú OTKA pályázat ''Egy fork-head típusú transzkripciós faktor sejtosztódásban játszott szerepének elemzése'' Szakmai Zárójelentés A projekt fő célkitűzései: 1. A sep1 protein szabályozásában szerepet
48765 számú OTKA pályázat ''Egy fork-head típusú transzkripciós faktor sejtosztódásban játszott szerepének elemzése'' Szakmai Zárójelentés A projekt fő célkitűzései: 1. A sep1 protein szabályozásában szerepet
Pro- és antioxidáns hatások szerepe az endoplazmás retikulum eredetű stresszben és apoptózisban
 Pro- és antioxidáns hatások szerepe az endoplazmás retikulum eredetű stresszben és apoptózisban Az endoplazmás retikulum (ER) számos környezeti és metabolikus hatás szenzora. Mindazon tényezők, melyek
Pro- és antioxidáns hatások szerepe az endoplazmás retikulum eredetű stresszben és apoptózisban Az endoplazmás retikulum (ER) számos környezeti és metabolikus hatás szenzora. Mindazon tényezők, melyek
Autoan'testek vizsgáló módszerei, HLA 'pizálás. Immunológiai és Biotechnológiai Intézet PTE- ÁOK
 Autoan'testek vizsgáló módszerei, HLA 'pizálás Immunológiai és Biotechnológiai Intézet PTE- ÁOK Természetes autoan'testek Pathológiás autoan'testek Természetes autoan'testek IgM Alacsony affinitás Alacsony
Autoan'testek vizsgáló módszerei, HLA 'pizálás Immunológiai és Biotechnológiai Intézet PTE- ÁOK Természetes autoan'testek Pathológiás autoan'testek Természetes autoan'testek IgM Alacsony affinitás Alacsony
Transzgénikus növények alkalmazása a funkcionális genomikai kutatásokban
 Transzgénikus növények alkalmazása a funkcionális genomikai kutatásokban MTA Agrártudományi Kutatóközpont Növényi Sejtbiológia Osztály Gyakorlatban alkalmazható transzgénikus növények létrehozásának alapfeltétele:
Transzgénikus növények alkalmazása a funkcionális genomikai kutatásokban MTA Agrártudományi Kutatóközpont Növényi Sejtbiológia Osztály Gyakorlatban alkalmazható transzgénikus növények létrehozásának alapfeltétele:
Tűzfal építés az alapoktól. Kadlecsik József KFKI RMKI kadlec@sunserv.kfki.hu
 Tűzfal építés az alapoktól Kadlecsik József KFKI RMKI kadlec@sunserv.kfki.hu Tartalom Szoftver-telepítés: kernel, iptables Routing Stateless és stateful szűrési példák NAT Szabály-finomítás iptables-save,
Tűzfal építés az alapoktól Kadlecsik József KFKI RMKI kadlec@sunserv.kfki.hu Tartalom Szoftver-telepítés: kernel, iptables Routing Stateless és stateful szűrési példák NAT Szabály-finomítás iptables-save,
Az F# nyelv erőforrásanalízise
 Az F# nyelv erőforrásanalízise Góbi Attila Eötvös Loránd Tudományegyetem Támogatta a KMOP-1.1.2-08/1-2008-0002 és az Európai Regionális Fejlesztési Alap. 2012. Június 19. Góbi Attila (ELTE) Az F# nyelv
Az F# nyelv erőforrásanalízise Góbi Attila Eötvös Loránd Tudományegyetem Támogatta a KMOP-1.1.2-08/1-2008-0002 és az Európai Regionális Fejlesztési Alap. 2012. Június 19. Góbi Attila (ELTE) Az F# nyelv
Plug-on transmitter - can power 48V phantom powered microphones Series SK 250-UHF-A RF Miniature Transmitter Ft Ft
 3000 Series 009974 SKP 3000 Plug-on transmitter - can power 48V phantom powered microphones 5000 Series 275 512 Ft 349 900 Ft 005525 SK 250-UHF-A RF Miniature Transmitter 1 016 457 Ft 1 290 900 Ft 500621
3000 Series 009974 SKP 3000 Plug-on transmitter - can power 48V phantom powered microphones 5000 Series 275 512 Ft 349 900 Ft 005525 SK 250-UHF-A RF Miniature Transmitter 1 016 457 Ft 1 290 900 Ft 500621
Fehérje expressziós rendszerek. Gyógyszerészi Biotechnológia
 Fehérje expressziós rendszerek Gyógyszerészi Biotechnológia Expressziós rendszerek Cél: rekombináns fehérjék előállítása nagy tisztaságban és nagy mennyiségben kísérleti ill. gyakorlati (therapia) felhasználásokra
Fehérje expressziós rendszerek Gyógyszerészi Biotechnológia Expressziós rendszerek Cél: rekombináns fehérjék előállítása nagy tisztaságban és nagy mennyiségben kísérleti ill. gyakorlati (therapia) felhasználásokra
A mutáns fenotípushoz szorosan kapcsolt markerek (1N1R és U212D) segítségével BAC (Bacterial Artifical Chromosome) klónokat azonosítottunk egy másik
 A szimbiotikus gümő kialakulásában résztvevő két gén azonosítása Medicago truncatulaból térképezésen alapuló génizolálással c. OTKA pályázat (T046645) zárójelentése A pályázat célja a Sinorhizobium meliloti
A szimbiotikus gümő kialakulásában résztvevő két gén azonosítása Medicago truncatulaból térképezésen alapuló génizolálással c. OTKA pályázat (T046645) zárójelentése A pályázat célja a Sinorhizobium meliloti
(A F) H&E staining of the duodenum in the indicated genotypes at E16.5 (A, D), E18.5
 Fig. S1. Brg1 ablation leads to abnormal villous structure in the duodenum in the perinatal period (A F) H&E staining of the duodenum in the indicated genotypes at E16.5 (A, D), E18.5 (B, E), and P0.5
Fig. S1. Brg1 ablation leads to abnormal villous structure in the duodenum in the perinatal period (A F) H&E staining of the duodenum in the indicated genotypes at E16.5 (A, D), E18.5 (B, E), and P0.5
Értékcsökkenési leírás
 Társasági adó Gyakorlat Értékcsökkenési leírás A Futár 2000 Kft. által birtokolt gépjárművek adatait az alábbi táblázat tartalmazza. A vállalkozás számviteli politikája szerint az értékcsökkenést a táblázatban
Társasági adó Gyakorlat Értékcsökkenési leírás A Futár 2000 Kft. által birtokolt gépjárművek adatait az alábbi táblázat tartalmazza. A vállalkozás számviteli politikája szerint az értékcsökkenést a táblázatban
TAKARMÁNYOZÁSTAN. Az Agrármérnöki MSc szak tananyagfejlesztése TÁMOP-4.1.2-08/1/A-2009-0010
 TAKARMÁNYOZÁSTAN Az Agrármérnöki MSc szak tananyagfejlesztése TÁMOP-4.1.2-08/1/A-2009-0010 Takarmányok fehérjetartalma Az állati szervezet létfontosságú vegyületei fehérje természetűek Az állati termékek
TAKARMÁNYOZÁSTAN Az Agrármérnöki MSc szak tananyagfejlesztése TÁMOP-4.1.2-08/1/A-2009-0010 Takarmányok fehérjetartalma Az állati szervezet létfontosságú vegyületei fehérje természetűek Az állati termékek
Tézis tárgyköréhez kapcsolódó tudományos közlemények
 Tézis tárgyköréhez kapcsolódó tudományos közlemények ABC transzporterek és lipidkörnyezetük kölcsönhatásának vizsgálata membrán koleszterin tartalom hatása az ABCG2 (BCRP/MXR) fehérje működésére Ph.D.
Tézis tárgyköréhez kapcsolódó tudományos közlemények ABC transzporterek és lipidkörnyezetük kölcsönhatásának vizsgálata membrán koleszterin tartalom hatása az ABCG2 (BCRP/MXR) fehérje működésére Ph.D.
A cell-based screening system for RNA Polymerase I inhibitors
 Electronic Supplementary Material (ESI) for MedChemComm. This journal is The Royal Society of Chemistry 2019 Supporting Information A cell-based screening system for RNA Polymerase I inhibitors Xiao Tan,
Electronic Supplementary Material (ESI) for MedChemComm. This journal is The Royal Society of Chemistry 2019 Supporting Information A cell-based screening system for RNA Polymerase I inhibitors Xiao Tan,
Table S1. Top and Bottom 10 most up- and down-regulated statistically significant transcripts (H 2 O 2 vs. untreated zebrafish larvae)
 Table S1. Top and Bottom 10 most up- and down-regulated statistically significant transcripts (H 2 O 2 vs. untreated zebrafish larvae) Rank Symbol logfc P- value Description 1 hsp70l 2.6 0.030 heat shock
Table S1. Top and Bottom 10 most up- and down-regulated statistically significant transcripts (H 2 O 2 vs. untreated zebrafish larvae) Rank Symbol logfc P- value Description 1 hsp70l 2.6 0.030 heat shock
Új generációs sporttudományi képzés és tartalomfejlesztés, hazai és nemzetközi hálózatfejlesztés és társadalmasítás a Szegedi Tudományegyetemen
 Új generációs sporttudományi képzés és tartalomfejlesztés, hazai Pályázati azonosító: TÁMOP-4.1.2.E-15/1/Konv-2015-0002 3. alprojekt Sportélettani képzés és tartalomfejlesztés Pilot 1: Dinamikus változások
Új generációs sporttudományi képzés és tartalomfejlesztés, hazai Pályázati azonosító: TÁMOP-4.1.2.E-15/1/Konv-2015-0002 3. alprojekt Sportélettani képzés és tartalomfejlesztés Pilot 1: Dinamikus változások
(AD) β (A ) (2) ACDP ACDP ACDP APP. APP-C99 γ (3) A 40 A 42 A A A A 42/A 40. in vivo A A A A
 (AD) β (A) A AAPP APP-C99 γ A A40 A42 A AA42/A40 γ A A γ AD A A A A APP APP APP A A A A A A (1)AD A42 A AD AD (2) ACDP AACDP ACDP A A ACDP (3) (1)A in vivo A APP Gray and Whittaker 1962; Perez-Otano et
(AD) β (A) A AAPP APP-C99 γ A A40 A42 A AA42/A40 γ A A γ AD A A A A APP APP APP A A A A A A (1)AD A42 A AD AD (2) ACDP AACDP ACDP A A ACDP (3) (1)A in vivo A APP Gray and Whittaker 1962; Perez-Otano et
Minőségjavító kísérlettervezés
 . példa J.J. Pignatiello, J.S. Ramberg: J. Quality Technology, 17 198-06 (1985) kézbentartható -1 1 A: high heat temperature ( 0 F) 1840 1880 B: heating time (s) 3 5 C: transfer time (s) 10 1 D: hold down
. példa J.J. Pignatiello, J.S. Ramberg: J. Quality Technology, 17 198-06 (1985) kézbentartható -1 1 A: high heat temperature ( 0 F) 1840 1880 B: heating time (s) 3 5 C: transfer time (s) 10 1 D: hold down
Fehérjeglikoziláció az endoplazmás retikulumban mint lehetséges daganatellenes támadáspont
 Fehérjeglikoziláció az endoplazmás retikulumban mint lehetséges daganatellenes támadáspont Doktori tézisek Dr. Konta Laura Éva Semmelweis Egyetem Molekuláris Orvostudományok Tudományági Doktori Iskola
Fehérjeglikoziláció az endoplazmás retikulumban mint lehetséges daganatellenes támadáspont Doktori tézisek Dr. Konta Laura Éva Semmelweis Egyetem Molekuláris Orvostudományok Tudományági Doktori Iskola
Szakmai zárójelentés
 Szakmai zárójelentés Kései szárazságtűrésben szerepet játszó génjelöltek asszociációs vizsgálata árpában EcoTILLING módszerrel című OTKA pályázat (OTKA azonosító: 72366) Kutatásaink elsődleges célja a
Szakmai zárójelentés Kései szárazságtűrésben szerepet játszó génjelöltek asszociációs vizsgálata árpában EcoTILLING módszerrel című OTKA pályázat (OTKA azonosító: 72366) Kutatásaink elsődleges célja a
A CSONTPÓTLÓ MŰTÉTEK BIOLÓGIAI ALAPJAI, A JÖVŐ LEHETŐSÉGEI
 Semmelweis Egyetem Arc- Állcsont- Szájsebészeti- és Fogászati Klinika Igazgató: Prof. Németh Zsolt A CSONTPÓTLÓ MŰTÉTEK BIOLÓGIAI ALAPJAI, A JÖVŐ LEHETŐSÉGEI Dr. Barabás Péter, Dr. Huszár Tamás SE Szak-
Semmelweis Egyetem Arc- Állcsont- Szájsebészeti- és Fogászati Klinika Igazgató: Prof. Németh Zsolt A CSONTPÓTLÓ MŰTÉTEK BIOLÓGIAI ALAPJAI, A JÖVŐ LEHETŐSÉGEI Dr. Barabás Péter, Dr. Huszár Tamás SE Szak-
klorid ioncsatorna az ABC (ATP Binding Casette) fehérjecsaládba tartozik, amelyek általánosságban részt vesznek a gyógyszerek olyan alapvetı
 Szegedi Tudományegyetem Farmakológiai és Farmakoterápiai Intézet Igazgató: Prof. Dr. Varró András 6720 Szeged, Dóm tér 12., Tel.: (62) 545-682 Fax: (62) 545-680 e-mail: varro.andras@med.u-szeged.hu Opponensi
Szegedi Tudományegyetem Farmakológiai és Farmakoterápiai Intézet Igazgató: Prof. Dr. Varró András 6720 Szeged, Dóm tér 12., Tel.: (62) 545-682 Fax: (62) 545-680 e-mail: varro.andras@med.u-szeged.hu Opponensi
A humán tripszinogén 4 expressziója és eloszlási mintázata az emberi agyban
 A humán tripszinogén 4 expressziója és eloszlási mintázata az emberi agyban Doktori (PhD) értekezés Siklódi Erika Rozália Biológia Doktori Iskola Iskolavezető: Prof. Erdei Anna, tanszékvezető egyetemi
A humán tripszinogén 4 expressziója és eloszlási mintázata az emberi agyban Doktori (PhD) értekezés Siklódi Erika Rozália Biológia Doktori Iskola Iskolavezető: Prof. Erdei Anna, tanszékvezető egyetemi
tccattaattcgacagaccagagttaaataatccttgtatgccattgtgatcacatctacagttcagattttgtatttca
 Figure Legends for Supplementary Materials: Fig. 1. Nucleotide sequence of the zebrafish lumican (zlum) gene. Exons are indicated by uppercase letters, and introns are indicated by lowercase letters. The
Figure Legends for Supplementary Materials: Fig. 1. Nucleotide sequence of the zebrafish lumican (zlum) gene. Exons are indicated by uppercase letters, and introns are indicated by lowercase letters. The
Biológus MSc. Molekuláris biológiai alapismeretek
 Biológus MSc Molekuláris biológiai alapismeretek A nukleotidok építőkövei A nukleotidok szerkezete Nukleotid = N-tartalmú szerves bázis + pentóz + foszfát N-glikozidos kötés 5 1 4 2 3 (Foszfát)észter-kötés
Biológus MSc Molekuláris biológiai alapismeretek A nukleotidok építőkövei A nukleotidok szerkezete Nukleotid = N-tartalmú szerves bázis + pentóz + foszfát N-glikozidos kötés 5 1 4 2 3 (Foszfát)észter-kötés
Mely humán génvariációk és környezeti faktorok járulnak hozzá az allergiás megbetegedések kialakulásához?
 Mely humán génvariációk és környezeti faktorok járulnak hozzá az allergiás megbetegedések kialakulásához? Falus András Genetikai, Sejt- és Immunbiológiai Intézet Semmelweis Egyetem Antal Péter Hadadi Éva
Mely humán génvariációk és környezeti faktorok járulnak hozzá az allergiás megbetegedések kialakulásához? Falus András Genetikai, Sejt- és Immunbiológiai Intézet Semmelweis Egyetem Antal Péter Hadadi Éva
A bakteriális efflux pumpák szerepe a multidrog rezisztenciában
 A bakteriális efflux pumpák szerepe a multidrog rezisztenciában MIKROBIOLÓGIA jelentősége a mindennapokban Otthon: konyhai műveletek (például kelt tészták, megfelelő hőkezelés, tartósítás) kozmetikumok
A bakteriális efflux pumpák szerepe a multidrog rezisztenciában MIKROBIOLÓGIA jelentősége a mindennapokban Otthon: konyhai műveletek (például kelt tészták, megfelelő hőkezelés, tartósítás) kozmetikumok
A koleszterin-anyagcsere szabályozása (Csala Miklós)
 A koleszterin-anyagcsere szabályozása (Csala Miklós) A koleszterin fontos építőeleme az emberi sejteknek, fontos szerepe van a biológiai membránok fluiditásának szabályozásában. E mellett hormonok és epesavak
A koleszterin-anyagcsere szabályozása (Csala Miklós) A koleszterin fontos építőeleme az emberi sejteknek, fontos szerepe van a biológiai membránok fluiditásának szabályozásában. E mellett hormonok és epesavak
Savak bázisok. Csonka Gábor Általános Kémia: 7. Savak és bázisok Dia 1 /43
 Savak bázisok 121 Az Arrhenius elmélet röviden 122 BrønstedLowry elmélet 123 A víz ionizációja és a p skála 124 Erős savak és bázisok 125 Gyenge savak és bázisok 126 Több bázisú savak 127 Ionok mint savak
Savak bázisok 121 Az Arrhenius elmélet röviden 122 BrønstedLowry elmélet 123 A víz ionizációja és a p skála 124 Erős savak és bázisok 125 Gyenge savak és bázisok 126 Több bázisú savak 127 Ionok mint savak
