Horizontal gene transfer drives adaptive colonization of apple trees by the fungal pathogen Valsa mali. Zhiyuan Yin, Baitao Zhu, Hao Feng, Lili Huang*
|
|
|
- Boglárka Kisné
- 6 évvel ezelőtt
- Látták:
Átírás
1 Supplementary Information Horizontal gene transfer drives adaptive colonization of apple trees by the fungal pathogen Valsa mali Zhiyuan Yin, Baitao Zhu, Hao Feng, Lili Huang* State Key Laboratory of Crop Stress Biology for Arid Areas and College of Plant Protection, Northwest A&F University, Yangling 712, Shaanxi, China *Corresponding author Lili Huang,
2 Supplementary Figures Supplementary Figure S1. Dead tissues of apple bark caused by Valsa mali in the fields.
3 Supplementary Figure S2. Bleomycin resistance assays of three fungi under different levels of bleomycin stress. were grown on the potato dextrose agar (PDA) media at 25 C in the dark for two days.
4 Supplementary Figure S3. Generation of gene deletion mutants. (a) Gene replacement strategy of hygromycin-resistant cassette. (b) Gene replacement strategy of G418-resistant cassette.
5 Supplementary Tables Supplementary Table S1. The genes involved in lysine biosynthesis in V. mali Gene Description Ortholog/homolog in V.mali a Genes involved in α-aminoadipate (AAA) pathway b LYS20/LYS21 Homocitrate synthase VM1G_07016 LYS4 Homoaconitase VM1G_10252 LYS12 Homoisocitrate VM1G_06170 dehydrogenase LYS2 α-aminoadipate reductase VM1G_02319, VM1G_105, VM1G_06405, VM1G_03154, VM1G_05645 LYS9 Saccharopine reductase VM1G_06828 LYS1 Saccahropine dehydrogenase VM1G_01424 Genes involved in succinylase (DAP) pathway c Ask Aspartate kinase VM1G_06620 Asd Aspartate semialdehyde VM1G_082 dehydrogenase DapA Dihydrodipicolinate VM1G_02804 synthase DapB Dihydrodipicolinate NA reductase DapD THDPA succinylase NA DapC succinyl-dap VM1G_011 aminotransferase DapE succinyl-dap VM1G_00056 desuccinylase DapF DAP epimerase NA LysA DAP decarboxylase VM1G_069 a Orthologs to genes involved in AAA pathway of Saccharomyces cerevisiae were identified by orthagogue (Ekseth O K, Kuiper M, Mironov V. orthagogue: an agile tool for the rapid prediction of orthology relations. Bioinformatics, 2013: btt582.); homologs to genes involved in DAP pathway were identified by blastp searches using known bacterial genes. b Refer to "Xu H, Andi B, Qian J, et al. The α-aminoadipate pathway for lysine biosynthesis in fungi. Cell biochemistry and biophysics, 2006, 46(1): " c Refer to "Liu Y, White R H, Whitman W B. Methanococci use the diaminopimelate aminotransferase (DapL) pathway for lysine biosynthesis. Journal of bacteriology, 2010, 192(13): "
6 Supplementary Table S2. Primers used for gene deletion and qrt-pcr. Names Sequences (5'-3') HGT32-1F CCATTGCGGAGAAGGTTAG HGT32-2R CAGATACGGCAGAGAAATCGCAACCTCCAAATGCTGCGTGGGTAA HGT32-3F GTTTAGATTCCAAGTGTCTACTGCTGGCTGGGTAGCGTTGTAGTTGT ATG HGT32-4R TGAGAAGGGAGCCAGCAT HGT32-5F ACGAATAACTCGCCTGTGG HGT32-6R AGCCGGGTCCTGAAATG HGT32-7F GCCAGAGCCAAGTCAACC HGT32-8R GCCCGTCCAGTATGTAATCA HGT32-CF GCAACAACCGAAGACGAAA HGT32-CR CTGACAAGGCAGGAGGAGA G852-F TCGGCTATGACTGGGCACAACA G850-R GAGCGGCGATACCGTAAAGCAC G856-F GAATGGTCAAATCAAACTGCTAGATAT G855-F TGTTGGGTTTGAGCTAGGTGGG HGT32-qPCR-F TGGACGATGTAGACGAGGTAT HGT32-qPCR-R TCCTGAAATGTCACAGTCCTAAC HGT14-1F GGCTCCAAATCCCCTCG HGT14-2R TTGACCTCCACTAGCTCCAGCCAAGCCAATACCACTGCTAATACAA TGCTG HGT14-3F GAATAGAGTAGATGCCGACCGCGGGTTGGGAAAGACTACGCCGAA TG HGT14-4R GCGTCCATAGAGCGGAAGAT HGT14-5F CGTCACTGCCCGTCTCA HGT14-6R TCCTCAACAGTCAAGTCCCTC HGT14-CF TCTAGCGAGCCGATTACAGT HGT14-CR CCCACATTGACCACCCAC H852 ATGTTGGCGACCTCGTATTGG H850 TTCCTCCCTTTATTTCAGATTCAA H855-R GCTGATCTGACCAGTTGC H856-F GTCGATGCGACGCAATCGT HGT14-qPCR-F GTGGGAGATATCGAGACGAAAC HGT14-qPCR-R ATCTGCCAGCCCTTGATTAC G6PDH-F TCAGAACAAGTTCGAGGGCGACAA G6PDH-R TGAGGGCAATAGAGGGCTTGTTCA
7 WP_ _Sinorhizobium_meliloti XP_ _Aspergillus_terreus WP_ _Bradyrhizobium_sp WP_ _Bradyrhizobium_sp WP_ _Methylobacterium_sp WP_ _Rhizobium_grahamii XP_ _Rhinocladiella_mackenziei WP_ _Bosea_thiooxidans KQR77015_Burkholderia_sp KGO54378_Penicillium_expansum XP_ _Pestalotiopsis_fici AIC2_Rhizobium_etli KIH762_Sporothrix_brasiliensis KEQ566_Aureobasidium_melanogenum XP_ _Exophiala_aquamarina WP_ _Bosea_thiooxidans XP_ _Aureobasidium_namibiae KFX361_Pseudogymnoascus_pannorum WP_ _Rhizobium_etli XP_ _Exophiala_xenobiotica KJR86105_Sporothrix_schenckii KKY16813_Phaeomoniella_chlamydospora WP_ _Bosea_sp KIW133_Exophiala_spinifera XP_ _Pseudocercospora_fijiensis WP_ _Elioraea_tepidiphila XP_ _Capronia_epimyces CRL21828_Penicillium_camemberti CEJ10414_bacterium_YEK KEQ90_Aureobasidium_pullulans VM1G_00056_Valsa_mali KIW34027_Cladophialophora_immunda XP_ _Talaromyces_stipitatus CDX52783_Mesorhizobium_plurifarium XP_ _Aureobasidium_subglaciale KQU83927_Mesorhizobium_sp CRG84517_Talaromyces_islandicus WP_ _Elioraea_tepidiphila GAM33785_Talaromyces_cellulolyticus XP_ _Fonsecaea_pedrosoi XP_ _Cyphellophora_europaea KRE00578_Bosea_sp EPE08060_Ophiostoma_piceae ERS919_Sporothrix_schenckii WP_ _Paenibacillus_odorifer GAD627_Byssochlamys_spectabilis KRE06713_Bosea_sp WP_ _Mesorhizobium_sp XP_ _Baudoinia_panamericana Bordetella_spp. Fusarium_spp. VM1G_00056
8 WP _Sphingobium_yanoikuyae EYE663_Aspergillus_ruber WP _Kutzneria_albida XP _Colletotrichum_fioriniae KKO503_Trichoderma_harzianum WP _Terracidiphilus_gabretensis KMS54549_Sphingobium_czechense WP _Sphingobium_sp WP _Streptomyces_sp WP _Caulobacter_henricii WP _Sphingomonas_sp CAK41643_Aspergillus_niger WP _Sphingomonas_sp WP _Novosphingobium_malaysiense WP018364_Streptomyces_sp XP _Bipolaris_sorokiniana XP _Zymoseptoria_tritici KOC10800_Aspergillus_flavus WP _Sphingobium_chungbukense WP _Novosphingobium_sp XP _Setosphaeria_turcica WP _Caulobacter_henricii WP _Caulobacter_sp WP _Sphingobium_sp EWM18849_Kutzneria_sp GAA86910_Aspergillus_kawachii EAU69263_Stigmatella_aurantiaca XP _Aspergillus_niger KKY241_Diaporthe_ampelina GAM05656_Novosphingobium_sp WP _Sphingomonas_sp KFY42778_Pseudogymnoascus_sp WP _Granulicella_mallensis WP _Sphingobium_japonicum WP _Asticcacaulis_biprosthecium XP _Bipolaris_zeicola WP _Shewanella_sp CCT70624_Fusarium_fujikuroi XP _Pantholops_hodgsonii GAP82757_Rosellinia_necatrix WP _Sphingobium_yanoikuyae CEJ83726_Torrubiella_hemipterigena WP _Rhodanobacter_sp XP _Fusarium_pseudograminearum XP00859_Beauveria_bassiana WP _Rhodanobacter_sp WP _Stigmatella_aurantiaca WP _Actinoplanes_friuliensis XP _Colletotrichum_gloeosporioides KLO763_Fusarium_fujikuroi EZP83146_Novosphingobium_resinovorum WP _Caulobacter_sp EHA27409_Aspergillus_niger WP _Rhizobacter_sp KNG444_Stemphylium_lycopersici KJK67468_Aspergillus_parasiticus WP _Kutzneria_albida WP _Novosphingobium_sp KMS50525_Novosphingobium_barchaimii EMF10247_Sphaerulina_musiva ABZ728_Caulobacter_sp WP _Pedobacter_sp XP681701_Aspergillus_nidulans WP _Rhodanobacter_fulvus WP _Kutzneria_sp VM1G_00616_Valsa_mali XP _Bipolaris_oryzae KEQ82843_Aureobasidium_pullulans XP _Pestalotiopsis_fici KGQ057_Beauveria_bassiana AEU37208_Granulicella_mallensis WP _Novosphingobium_sp KJX524_Zymoseptoria_brevis WP _Sphingobium_yanoikuyae WP _Microbispora_rosea WP _Sphingobium_sp WP _Novosphingobium_lindaniclasticum WP _Methylibium_sp WP _Amycolatopsis_balhimycina XP _Pyrenophora_tritici WP _Sphingomonas_sp WP _Caulobacter_sp WP _Caulobacter_sp WP _Novosphingobium_resinovorum XP _Aspergillus_flavus XP _Aureobasidium_subglaciale WP _Sphingomonas-like KEQ67483_Aureobasidium_melanogenum WP _Novosphingobium_sp WP _Sphingobium_yanoikuyae EQB52649_Colletotrichum_gloeosporioides WP _alpha_proteobacterium XP _Marssonina_brunnea KIS30550_Novosphingobium_sp XP _Pyrenophora_teres WP _Caulobacter_sp XP _Parastagonospora_nodorum XP _Aureobasidium_namibiae XP _Bipolaris_victoriae WP _Hassallia_byssoidea XP _Fusarium_graminearum VM1G_00616
9 KKY35092_Diaporthe_ampelina WP_01111_Rhodopirellula_baltica WP_ _Paenibacillus_sp WP_ _Verrucomicrobium_sp WP_ _Rhodopirellula_baltica WP_ _Rubinisphaera_brasiliensis KLU07046_Rhodopirellula_sp WP_ _Gimesia_maris WP_00308_Rhodopirellula_baltica WP_ _Haloferula_sp XP_ _Naegleria_gruberi WP_00228_Paenibacillus_sp WP_ _Rhodopirellula_sp WP_ _Verrucomicrobium_sp WP_ _Pirellula_staleyi EMI25847_Rhodopirellula_europaea WP_ _Pedosphaera_parvula WP_00913_Verrucomicrobium_spinosum EPX57826_Cystobacter_fuscus WP_ _Rhodopirellula_sp WP_ _Paenibacillus_lactis WP_ _Chthoniobacter_flavus WP_ _Zavarzinella_formosa WP_ _Cystobacter_fuscus WP_ _Rhodopirellula_maiorica WP_ _Verrucomicrobium_sp ADG68792_Planctopirus_limnophila WP_ _Rhodopirellula_baltica ADB15537_Pirellula_staleyi WP_ _Paenibacillus_sp WP_ _Singulisphaera_acidiphila WP_ _Paenibacillus_sp VM1G_00776_Valsa_mali WP_ _Planctopirus_limnophila WP_ _Rhodopirellula_europaea WP_ _Paenibacillus_sp WP_ _Verrucomicrobium_sp WP_ _Blastopirellula_marina WP_ _Rhodopirellula_europaea M1G_00776
10 EKX68546_Streptomyces_ipomoeae XP_ _Eutypa_lata VM1G_01172_Valsa_mali AAA67510_Streptomyces_glaucescens AGP609_Streptomyces_rapamycinicus KIM364_Oidiodendron_maius WP_ _Streptomyces_sp KIM9_Oidiodendron_maius WP_ _Streptomyces_caatingaensis WP_ _Lechevalieria_aerocolonigenes KJK50809_Lechevalieria_aerocolonigenes WP_ _Streptomyces_iranensis CEL20688_Kibdelosporangium_sp WP_ _Streptomyces_caatingaensis CDM28614_Penicillium_roqueforti WP_ _Herbidospora_cretacea WP_ _Pseudonocardia_acaciae WP_ _Allokutzneria_albata WP_00403_Saccharopolyspora_erythraea AFU90406_Streptomyces_sp WP_ _Streptomyces_bottropensis WP_ _Streptomyces_ipomoeae WP_ _Streptomyces_sp WP_ _Allokutzneria_albata WP_01870_Streptomyces_purpureus WP_ _Streptomyces_violaceusniger WP_ _Streptomyces_sp XP_ _Pestalotiopsis_fici WP_ _Streptomyces_himastatinicus WP_ _Mycobacterium_llatzerense WP_ _Streptomyces_mobaraensis WP_ _Streptomyces_sp WP_ _Amycolatopsis_rifamycinica AFV25479_Streptomyces_sp WP_01436_Streptomyces_sulphureus WP_ _Kibdelosporangium_phytohabitans WP_ _Streptomyces_scabiei WP_ _Archangium_gephyra WP_ _Streptomyces_hygroscopicus Mycobacterium_spp Mycobacterium_spp VM1G_01172
11 WP_ _Novosphingobium_malaysiense WP_ _Sphingomonas_sp KOP40348_Madurella_mycetomatis XP_ _Eutypa_lata WP_ _Sphingomonas_phyllosphaerae WP_01681_Sphingomonas_sp XP_ _Coniosporium_apollinis WP_ _Sphingomonas-like WP_ _Sphingomonas_sp KMS52368_Sphingobium_czechense WP_ _Novosphingobium_sp WP_ _Sphingomonas_sp KKY366_Diaporthe_ampelina WP_ _Sphingopyxis_alaskensis WP_ _Novosphingobium_resinovorum WP_ _Sphingomonas_paucimobilis WP_ _Novosphingobium_sp XP_ _Togninia_minima WP_ _Sphingomonas_sp KFA55220_Stachybotrys_chartarum WP_ _Sphingomonadaceae XP_ _Microsporum_gypseum WP_ _Sphingomonas_sp XP_ _Aureobasidium_subglaciale KMS54367_Novosphingobium_barchaimii WP_ _Sphingomonas_paucimobilis WP_ _Sphingomonas_sp WP_ _Sphingopyxis_sp WP_ _Sphingobium_sp WP_ _Sphingomonas_sp WP_04385_Novosphingobium_sp WP_ _Novosphingobium_sp EKG044_Macrophomina_phaseolina WP_ _Novosphingobium_sp WP_ _Novosphingobium_sp WP_ _Sphingomonadaceae WP_ _Novosphingobium_sp WP_ _Novosphingobium_lindaniclasticum WP_ _Novosphingobium_sp WP_ _Sphingomonas_sp KEQ619_Aureobasidium_melanogenum WP_ _Tolypothrix_campylonemoides VM1G_01377_Valsa_mali XP_ _Capronia_epimyces Sphingomonas_spp. Fusarium_spp. Pseudogymnoascus_spp. VM1G_01377
12 WP_ _Micromonospora_sp WP_ _Candidatus_Solibacter KRB77082_Nocardioides_sp WP_ _Granulicella_mallensis WP_ _Nocardiopsis_alba WP_ _bacterium_UASB WP_ _Acidobacteriaceae_bacterium KKY217_Diaporthe_ampelina WP_ _Nocardiopsis_baichengensis WP_ _Terriglobus_sp WP_ _Acidobacteriaceae_bacterium WP_ _Nocardiopsis_alba WP_ _Nocardiopsis_potens WP_ _Micromonospora_parva WP_ _Candidatus_Koribacter XP_ _Nectria_haematococca WP_ _Acidobacterium_sp KQP78010_Aeromicrobium_sp WP_ _Acidobacteria_bacterium WP_ _Edaphobacter_aggregans WP_ _Kribbella_flavida WP_ _Acidobacteriaceae_bacterium ACO33744_Acidobacterium_capsulatum WP_ _Acidobacteriaceae_bacterium WP_ _Granulicella_tundricola KQP26891_Aeromicrobium_sp WP_ _Acidobacteriaceae_bacterium VM1G_01892_Valsa_mali WP_ _Nocardiopsis_halophila WP_ _Acidobacteriaceae_bacterium XP_ _Pestalotiopsis_fici WP_ _Acidobacterium_capsulatum WP_ _Terriglobus_roseus WP_ _Nocardiopsis_sp WP_ _Nocardiopsis_chromatogenes Streptomyces_spp VM1G_01892
13 KJJ21279_Penicillium_solitum XP_ _Agaricus_bisporus VM1G_02284_Valsa_mali KIY62258_Cylindrobasidium_torrendii XP_ _Rhinocladiella_mackenziei CAI43283_Malassezia_sympodialis XP_ _Auricularia_subglabra KGO75825_Penicillium_italicum KIJ090_Laccaria_amethystina CEH18770_Ceraceosorus_bombacis EEB884_Moniliophthora_perniciosa KIW63858_Capronia_semiimmersa CEJ58114_Penicillium_brasilianum EKV115_Penicillium_digitatum XP_ _Moniliophthora_roreri XP_00260_Agaricus_bisporus CRG84226_Talaromyces_islandicus XP_ _Auricularia_subglabra XP_00272_Agaricus_bisporus KOS16334_Malassezia_pachydermatis XP_ _Anthracocystis_flocculosa KOS16335_Malassezia_pachydermatis KPV76585_Rhodotorula_graminis XP_ _Tilletiaria_anomala XP_ _Auricularia_subglabra XP_ _Capronia_coronata XP_ _Moniliophthora_roreri XP_ _Malassezia_globosa KIW34086_Cladophialophora_immunda CDI54338_Melanopsichium_pennsylvanicum KGO420_Penicillium_expansum KIW42617_Exophiala_oligosperma CDI554_Melanopsichium_pennsylvanicum XP_ _Schizophyllum_commune XP_ _Ustilago_maydis EMS260_Rhodosporidium_toruloides ETS65339_Pseudozyma_aphidis GAK65507_Pseudozyma_antarctica CBQ6_Sporisorium_reilianum GAK684_Pseudozyma_antarctica XP_ _Exophiala_xenobiotica KIY44082_Fistulina_hepatica CDM35827_Penicillium_roqueforti XP_ _Malassezia_globosa XP_ _Schizophyllum_commune CCF50110_Ustilago_hordei GAC024_Pseudozyma_antarctica KKY36776_Diaporthe_ampelina ETS61786_Pseudozyma_aphidis GAD92055_Byssochlamys_spectabilis KIY70183_Cylindrobasidium_torrendii KIW17623_Exophiala_spinifera XP_ _Pseudozyma_hubeiensis XP_ _Malassezia_globosa XP_ _Pseudozyma_hubeiensis EST09215_Pseudozyma_brasiliensis GAC71446_Pseudozyma_antarctica XP_ _Anthracocystis_flocculosa KIK66615_Gymnopus_luxurians EGU12718_Rhodotorula_glutinis XP_ _Talaromyces_marneffei KDQ31492_Pleurotus_ostreatus GAM40240_Talaromyces_cellulolyticus XP_ _Penicillium_rubens KIY01127_Fonsecaea_multimorphosa KOS37086_Penicillium_nordicum XP_ _Exophiala_dermatitidis CRL26481_Penicillium_camemberti KIV78046_Exophiala_sideris XP_ _Capronia_epimyces XP_ _Talaromyces_stipitatus Basidiomycota Basidiomycota Ascomycota VM1G_02284
14 WP_ _Streptomyces_sp GAM366_Talaromyces_cellulolyticus KOS44646_Penicillium_nordicum XP_ _Rhinocladiella_mackenziei XP_ _Cladophialophora_carrionii CCD565_Botrytis_cinerea KGO68501_Penicillium_italicum KIW350_Cladophialophora_bantiana XP_ _Eutypa_lata WP_ _Vibrio_nigripulchritudo GAM832_fungal_sp WP_ _Saccharospirillum_impatiens KMO772_Mycobacterium_obuense XP_ _Colletotrichum_fioriniae XP_ _Cladophialophora_yegresii EPE030_Ophiostoma_piceae XP_ _Marssonina_brunnea KIW024_Capronia_semiimmersa CEJ516_Penicillium_brasilianum XP_ _Botrytis_cinerea KIW16780_Exophiala_spinifera KQY06207_Mycobacterium_sp CDK28010_Kuraishia_capsulata KKY27433_Diplodia_seriata KEY74755_Stachybotrys_chartarum KKP037_Trichoderma_harzianum WP_ _Streptomyces_griseoflavus KIX3_Fonsecaea_multimorphosa KQR7_Williamsia_sp XP_ _Capronia_coronata KIW34586_Cladophialophora_immunda KIW00318_Verruconis_gallopava ETW9_Candidatus_Entotheonella XP_ _Sclerotinia_sclerotiorum KIV82153_Exophiala_sideris KFY76565_Pseudogymnoascus_pannorum CRG85410_Talaromyces_islandicus CDM27871_Penicillium_roqueforti WP_ _Streptomyces_sp KGO40120_Penicillium_expansum KFZ104_Pseudogymnoascus_pannorum VM1G_02549_Valsa_mali XP_ _Pestalotiopsis_fici WP_ _Limnohabitans_sp CRL18751_Penicillium_camemberti XP_ _Grosmannia_clavigera XP_ _Coniosporium_apollinis WP_ _Mycobacterium_sp XP_ _Rasamsonia_emersonii KJJ10128_Penicillium_solitum WP_ _Chelativorans_sp KPI35356_Phialophora_attae KKY18201_Phaeomoniella_chlamydospora XP_ _Talaromyces_marneffei KIW880_Cladophialophora_bantiana KFX42121_Talaromyces_marneffei WP_ _Mycobacterium_obuense XP_ _Cladophialophora_psammophila ETR7_Trichoderma_reesei XP_ _Penicillium_rubens XP_ _Fonsecaea_pedrosoi WP_ _Mycobacterium_obuense XP_ _Exophiala_xenobiotica XP_ _Fonsecaea_pedrosoi EMR87546_Botrytis_cinerea WP_ _Mycobacterium_vulneris ESZ586_Sclerotinia_borealis XP_ _Togninia_minima XP_ _Eutypa_lata KPI424_Phialophora_attae KFA45846_Stachybotrys_chartarum WP_ _Streptomyces_toyocaensis Bordetella_spp. Acinetobacter_spp. VM1G_02549
15 KIW16782_Exophiala_spinifera KEC78234_Sinorhizobium_americanum WP_ _Bordetella_bronchiseptica WP_03723_Rhodococcus_wratislaviensis WP_ _Streptomyces_sp KIW00312_Verruconis_gallopava WP_ _Bordetella_bronchiseptica XP_ _Neofusicoccum_parvum XP_00702_Pseudocercospora_fijiensis WP_ _Burkholderia_mimosarum WP_ _Bacillus_sp KFX42120_Talaromyces_marneffei WP_ _Sodalis_praecaptivus XP_ _Rhinocladiella_mackenziei WP_ _Acinetobacter_sp WP_ _Novosphingobium_malaysiense WP_ _Streptomyces_scabiei WP_ _Vibrio_sp WP_ _Streptomyces_sp WP_ _Rhodococcus_sp WP_ _Mycobacterium_vaccae WP_ _Bacillus_bataviensis AAW28146_Rhizobium_sp XP_ _Togninia_minima WP_ _Streptomyces_sp WP_ _Streptomyces_griseus WP_ _Acinetobacter_brisouii WP_ _Novosphingobium_resinovorum GAM368_Talaromyces_cellulolyticus EPE023_Ophiostoma_piceae WP_ _Streptomyces_xylophagus WP_ _Streptomyces_ipomoeae WP_ _Sodalis_glossinidius WP_ _Erwinia_typographi WP_ _Arthrobacter_sp WP_ _Sinorhizobium_americanum WP_ _Nocardia_higoensis WP_ _Klebsiella_pneumoniae WP_ _Bordetella_bronchiseptica WP_ _Bradyrhizobium_sp XP_ _Pestalotiopsis_fici WP_ _Pimelobacter_simplex KKO72846_Kerstersia_gyiorum KEY74761_Stachybotrys_chartarum XP_ _Talaromyces_marneffei WP_ _Mycobacterium_sp WP_ _Ensifer_sp WP_ _Hyphomicrobium_sp WP_ _Streptomyces_phaeochromogenes XP_ _Capronia_coronata WP_ _Streptomyces_acidiscabies VM1G_02555_Valsa_mali KFA81538_Stachybotrys_chartarum WP_ _Raoultella_terrigena EGD06287_Burkholderia_sp WP_ _Bordetella_bronchiseptica WP_ _Pusillimonas_noertemannii KKY16861_Phaeomoniella_chlamydospora WP_ _Streptomyces_sp XP_ _Eutypa_lata WP_ _Bordetella_parapertussis WP_ _Bacillus_sp WP_ _Streptomyces_acidiscabies WP_ _Bacillus_sp WP_ _Nocardioides_alkalitolerans WP_ _Amycolatopsis_taiwanensis KPI35307_Phialophora_attae XP_ _Grosmannia_clavigera WP_ _Rhodococcus_sp WP_ _Pusillimonas_noertemannii Achromobacter_spp Photorhabdus_spp VM1G_02555
16 KDQ13290_Botryobasidium_botryosum CEL57465_Rhizoctonia_solani KDQ58426_Jaapia_argillacea EUC53763_Rhizoctonia_solani KIO242_Tulasnella_calospora XP_ _Punctularia_strigosozonata KIO24292_Tulasnella_calospora XP_ _Gloeophyllum_trabeum VM1G_02910_Valsa_mali CCO26677_Rhizoctonia_solani EJU02432_Dacryopinax_sp XP_ _Auricularia_subglabra CUA77546_Rhizoctonia_solani KIO24291_Tulasnella_calospora XP_ _Fomitiporia_mediterranea KII87568_Plicaturopsis_crispa KLO122_Schizopora_paradoxa KIO24307_Tulasnella_calospora KLO13341_Schizopora_paradoxa XP_ _Pseudocercospora_fijiensis XP_ _Auricularia_subglabra KIO216_Tulasnella_calospora KIO26062_Tulasnella_calospora KIO24280_Tulasnella_calospora CED84805_Xanthophyllomyces_dendrorhous KIM123_Serendipita_vermifera CCA74032_Piriformospora_indica Cryptococcus_spp Basidiomycota Basidiomycota Ascomycota VM1G_02910
17 EYE023_Aspergillus_ruber KGO304_Penicillium_expansum KIV762_Exophiala_sideris EUN26561_Bipolaris_victoriae XP_ _Melampsora_larici XP_ _Pestalotiopsis_fici XP_ _Colletotrichum_gloeosporioides KGO75074_Penicillium_italicum KOS478_Penicillium_nordicum KDN66752_Colletotrichum_sublineola AF181866_synthetic_construct XP_456462_Debaryomyces_hansenii XP_00829_Punctularia_strigosozonata KIV86820_Exophiala_sideris XP_ _Aureobasidium_subglaciale XP_ _Talaromyces_stipitatus XP_ _Bipolaris_sorokiniana XP_ _Setosphaeria_turcica 4RSL_A_4RSL_A XP_ _Nectria_haematococca CEJ56755_Penicillium_brasilianum KGO47403_Penicillium_expansum XP_ _Talaromyces_stipitatus XP_ _Stereum_hirsutum KKY34247_Diaporthe_ampelina EQB58906_Colletotrichum_gloeosporioides BAD00185_Penicillium_terrenum XP_ _Aureobasidium_namibiae XP_ _Pyrenophora_teres CRL24868_Penicillium_camemberti EPE02569_Ophiostoma_piceae XP_ _Exophiala_xenobiotica KIW10542_Exophiala_spinifera XP_ _Nectria_haematococca XP_ _Grosmannia_clavigera XP_ _Exophiala_dermatitidis XP_ _Neofusicoccum_parvum BAE140_Ulocladium_sp XP_ _Bipolaris_maydis XP_ _Exophiala_xenobiotica KII88422_Plicaturopsis_crispa KIY458_Fistulina_hepatica XP_ _Bipolaris_zeicola ENH83077_Colletotrichum_orbiculare XP_681392_Aspergillus_nidulans XP_ _Eutypa_lata XP_ _Puccinia_graminis CDK252_Kuraishia_capsulata CDZ665_Xanthophyllomyces_dendrorhous CDM34716_Penicillium_roqueforti XP_ _Bipolaris_oryzae XP_ _Pyrenophora_tritici XP_777019_Cryptococcus_neoformans KNZ529_Puccinia_sorghi VM1G_020_Valsa_mali GAM35130_Talaromyces_cellulolyticus KNE892_Puccinia_striiformis XP_ _Parastagonospora_nodorum KEZ43556_Scedosporium_apiospermum XP_ _Heterobasidion_irregulare KDQ51628_Jaapia_argillacea XP_00255_Penicillium_rubens KEQ614_Aureobasidium_melanogenum XP_ _Capronia_coronata KJJ23348_Penicillium_solitum CEP22126_Cyberlindnera_jadinii XP_568763_Cryptococcus_neoformans KEQ80229_Aureobasidium_pullulans BAD00186_Coniochaeta_sp XP_ _Colletotrichum_graminicola XP_ _Cryptococcus_neoformans XP_ _Schizophyllum_commune XP_ _Gloeophyllum_trabeum XP_ _Wickerhamomyces_ciferrii Fusarium_spp Cryptococcus_spp Basidiomycota Ascomycota VM1G_020
18 WP_ _Burkholderia_sp AJC22888_Pandoraea_pulmonicola WP_ _Burkholderia_sp WP_ _Xenophilus_azovorans WP_ _Rhodococcus_sp KIM700_Oidiodendron_maius WP_ _Azohydromonas_australica WP_ _Burkholderia_xenovorans WP_ _Comamonas_testosteroni WP_ _Nocardia_jiangxiensis WP_ _Rhodococcus_sp WP_ _Streptomyces_europaeiscabiei KPI23374_Actinobacteria_bacterium KRB187_Hydrogenophaga_sp WP_ _Sciscionella_sp WP_ _Sphingobium_baderi WP_ _Bosea_sp KMS61908_Sphingobium_baderi WP_ _Amorphus_coralli KEY74811_Stachybotrys_chartarum KFA4_Stachybotrys_chartarum GAO20820_Alicycliphilus_sp WP_ _Ottowia_thiooxydans EIF318_Burkholderia_sp WP_ _Pandoraea_pulmonicola KRB85535_Sphingomonas_sp WP_ _Burkholderia_sp WP_ _Sphingopyxis_sp WP_ _Sciscionella_marina EYS85102_Cupriavidus_sp AJW201_Sphingomonas_sp XP_008823_Colletotrichum_graminicola XP_ _Setosphaeria_turcica WP_ _Massilia_sp WP_ _Burkholderia_sp WP_ _Burkholderia_multivorans KGO63929_Penicillium_italicum WP_ _Burkholderia_multivorans EIF32032_Burkholderia_sp WP_ _Cupriavidus_sp XP_ _Rhinocladiella_mackenziei WP_ _Rhodococcus_jostii VM1G_03090_Valsa_mali AKP92220_Achromobacter_xylosoxidans VM1G_03090
19 0.3 KQV30626_Rhizobium_sp WP_ _Arenibacter_algicola KFZ12530_Pseudogymnoascus_pannorum EWG42788_Fusarium_verticillioides KPA45902_Fusarium_langsethiae WP_ _Candidatus_Solibacter WP_01623_Eudoraea_adriatica WP_ _Acidobacteriaceae_bacterium KFY43621_Pseudogymnoascus_pannorum CRK16278_Verticillium_longisporum KFY710_Pseudogymnoascus_pannorum XP_ _Arthroderma_otae KKY31257_Diaporthe_ampelina WP_02615_Rhizobium_leguminosarum KIL912_Fusarium_avenaceum KFY30424_Pseudogymnoascus_pannorum WP_ _Flavobacterium_filum XP_ _Colletotrichum_fioriniae XP_ _Verticillium_dahliae EXM07238_Fusarium_oxysporum VM1G_03108_Valsa_mali WP_ _Lacinutrix_himadriensis WP_ _Arenitalea_lutea WP_ _Candidatus_Phaeomarinobacter WP_ _Xanthomonas_sp KFX86644_Pseudogymnoascus_pannorum XP_ _Fusarium_pseudograminearum XP_ _Verticillium_alfalfae ENH68826_Fusarium_oxysporum XP_ _Anthracocystis_flocculosa CRK14657_Verticillium_longisporum WP_ _Acidobacteriaceae_bacterium WP_ _Niabella_soli XP_ _Cyphellophora_europaea CRL17750_Penicillium_camemberti KFH68184_Mortierella_verticillata GAO88553_Neosartorya_udagawae WP_ _Corynebacterium_genitalium WP_ _Rhizobium_grahamii ALM086_Sediminicola_sp CCT66908_Fusarium_fujikuroi EMT68080_Fusarium_oxysporum XP_ _Pseudocercospora_fijiensis XP_ _Melampsora_larici EWZ727_Fusarium_oxysporum WP_ _Terriglobus_sp XP_ _Microsporum_gypseum WP_02903_Rhizobium_leguminosarum KFX2_Pseudogymnoascus_pannorum KLO632_Fusarium_fujikuroi XP_ _Fusarium_graminearum WP_ _Sphingobacterium_sp Chryseobacterium_spp VM1G_03108
20 XP _Parastagonospora_nodorum XP _Neosartorya_fischeri WP _Chryseobacterium_sp GAM319_Talaromyces_cellulolyticus GAD648_Byssochlamys_spectabilis CEJ62862_Penicillium_brasilianum WP _Lactobacillus_plantarum KIY01641_Fonsecaea_multimorphosa KIV86245_Exophiala_sideris KIV87176_Exophiala_sideris KJK64811_Aspergillus_parasiticus XP _Phaeoacremonium_minimum XP _Dichomitus_squalens WP _Frankia_sp WP _Nocardia_niigatensis GAQ11743_Aspergillus_lentulus CAJ62281_Frankia_alni XP00752_Eutypa_lata CRL172_Penicillium_camemberti XP _Colletotrichum_graminicola GAO858_Neosartorya_udagawae KFY420_Pseudogymnoascus_sp XP _Glarea_lozoyensis XP _Capronia_epimyces WP _Kutzneria_albida GAM35249_Talaromyces_cellulolyticus WP _Algoriphagus_sp EMR911_Botrytis_cinerea XP7449_Aspergillus_fumigatus XP _Bipolaris_maydis WP _Lactobacillus_silagei KJK63712_Aspergillus_parasiticus WP _WP_ WP _Nocardia_vinacea KLP06860_Fusarium_fujikuroi XP _Capronia_coronata KIV78870_Exophiala_sideris KKZ655_Emmonsia_crescens VM1G_03556_Valsa_mali KJJ25285_Penicillium_solitum WP04176_Frankia_alni XP _Rhinocladiella_mackenziei WP _Paenibacillus_sp XP _Capronia_coronata KIW012_Exophiala_spinifera CRL24591_Penicillium_camemberti XP _Botrytis_cinerea XP _Moniliophthora_roreri XP _Exophiala_xenobiotica KDN71869_Colletotrichum_sublineola XP _Talaromyces_stipitatus XP _Cyphellophora_europaea XP _Thielavia_terrestris KOS216_Escovopsis_weberi GAM43929_Talaromyces_cellulolyticus XP _Pestalotiopsis_fici KOS482_Penicillium_nordicum WP _Rhodococcus_rhodochrous XP _Cladophialophora_carrionii XP _Colletotrichum_fioriniae XP _Exophiala_dermatitidis XP _Aspergillus_flavus KJA23206_Hypholoma_sublateritium CRG83420_Talaromyces_islandicus WP _Sphingomonas_sp XP _Colletotrichum_gloeosporioides Ascomycota Ascomycota Basidiomycota VM1G_03556
21 WP_ _Gordonia_sputi WP_ _Microbispora_rosea VM1G_04133_Valsa_mali WP_ _Microbacterium_testaceum WP_ _Shinella_sp WP_02001_Mycobacterium_sp WP_04701_Nonomuraea_sp WP_02681_Mycobacterium_sp KQM34063_Rhizobium_sp WP_ _Micromonospora_chokoriensis WP_ _Microbacterium_hominis WP_ _Azorhizobium_doebereinerae WP_ _Gordonia_otitidis KQQ534_Rhizobium_sp WP_ _Nonomuraea_sp WP_ _Mycobacterium_sp ESY88146_Mesorhizobium_sp WP_ _Terriglobus_roseus WP_ _Mycobacterium_sp WP_ _Shinella_sp WP_ _Mesorhizobium_sp WP_ _Mycobacterium_llatzerense WP_ _Nocardia_higoensis WP_ _Nonomuraea_sp WP_ _Pedosphaera_parvula WP_ _Streptomyces_scabrisporus WP_ _Mycobacterium_sp WP_ _Mesorhizobium_sp XP_ _Baudoinia_panamericana VM1G_04133
22 WP_ _Rhizobium_leguminosarum XP_ _Nicotiana_sylvestris WP_ _Streptomyces_cellulosae WP_ _Paenibacillus_sp WP_ _Arthrobacter_sp XP_ _Nicotiana_tomentosiformis XP_ _Ogataea_parapolymorpha ABR16229_Picea_sitchensis XP_ _Erythranthe_guttatus AFZ84925_Physcomitrella_patens KCW88013_Eucalyptus_grandis WP_ _Runella_limosa KCW88014_Eucalyptus_grandis WP_ _Paenibacillus_peoriae CDK279_Kuraishia_capsulata WP_ _Photobacterium_sanctipauli EFL20921_Streptomyces_himastatinicus WP_ _Paenibacillus_senegalensis EIF45186_Brettanomyces_bruxellensis WP_ _Cylindrospermum_stagnale WP_ _Clostridiales_bacterium WP_ _Paenibacillus_sp XP_ _Solanum_lycopersicum WP_ _Corynebacterium-like WP_ _Bacillus_sp XP_ _Brachypodium_distachyon AFZ84926_Physcomitrella_patens WP_ _Devosia_sp XP_ _Togninia_minima BAJ480_Candidatus_Caldiarchaeum XP_ _Elaeis_guineensis WP_ _Streptomyces_violaceoruber KON30581_miscellaneous_Crenarchaeota WP_ _Eubacterium_sp WP_ _Halomonas_salina BAJ471_Candidatus_Caldiarchaeum WP_ _Clostridium_sp AFZ84924_Physcomitrella_patens WP_ _Paenibacillus_sp WP_ _Mesorhizobium_sp AFZ84927_Physcomitrella_patens XP_ _Physcomitrella_patens WP_ _Streptomyces_sp WP_ _Clostridiales_bacterium AFZ84928_Physcomitrella_patens KPN14136_Bacillus_sp VM1G_04190_Valsa_mali WP_ _Scytonema_hofmanni WP_ _Bacillus_sp XP_ _Candida_tenuis Viridiplantae Viridiplantae VM1G_04190
23 0.09 XP_ _Colletotrichum_graminicola XP_ _Pestalotiopsis_fici XP_ _Phytophthora_parasitica KKY13676_Diplodia_seriata ETM415_Phytophthora_parasitica XP_ _Phytophthora_parasitica ETL31091_Phytophthora_parasitica CEJ56904_Penicillium_brasilianum EKV05820_Penicillium_digitatum XP_664276_Aspergillus_nidulans XP_ _Phytophthora_infestans XP_ _Colletotrichum_gloeosporioides ETO70679_Phytophthora_parasitica KGO77832_Penicillium_italicum ETI48538_Phytophthora_parasitica KPM40080_Neonectria_ditissima CCX34009_Pyronema_omphalodes EME43478_Dothistroma_septosporum EKG17_Macrophomina_phaseolina KJX177_Zymoseptoria_brevis ENH83916_Colletotrichum_orbiculare XP_00140_Phytophthora_sojae EQB47601_Colletotrichum_gloeosporioides EMF12245_Sphaerulina_musiva XP_ _Neofusicoccum_parvum XP_ _Phytophthora_sojae XP_ _Penicillium_rubens ETL88722_Phytophthora_parasitica CRL22911_Penicillium_camemberti XP_ _Nectria_haematococca XP_ _Phytophthora_parasitica XP_ _Phytophthora_infestans XP_ _Melampsora_larici XP_ _Phytophthora_sojae XP_ _Phytophthora_infestans XP_ _Colletotrichum_fioriniae CCF36503_Colletotrichum_higginsianum KGO46541_Penicillium_expansum ETO306_Phytophthora_parasitica XP_ _Phytophthora_sojae XP_ _Pseudocercospora_fijiensis XP_ _Phytophthora_infestans KDN63372_Colletotrichum_sublineola VM1G_04692_Valsa_mali XP_ _Puccinia_graminis KJJ039_Penicillium_solitum ETI44692_Phytophthora_parasitica KOS396_Penicillium_nordicum Fusarium_spp Ascomycota Ascomycota Basidiomycota Oomycetes VM1G_04692
24 WP_ _Streptomyces_europaeiscabiei ELQ35678_Magnaporthe_oryzae WP_ _Streptomyces_galbus KPI324_Actinobacteria_bacterium XP_ _Togninia_minima WP_ _Clostridium_scatologenes WP_ _Rhodococcus_rhodnii XP_ _Eutypa_lata VM1G_04714_Valsa_mali WP_ _Streptomyces_sp WP_ _Micromonospora_carbonacea WP_ _Streptomyces_scabiei ELP70762_Streptomyces_turgidiscabies WP_ _Streptomyces_bottropensis WP_ _Verrucosispora_maris KLU857_Magnaporthiopsis_poae WP_ _Kibdelosporangium_aridum WP_ _Jiangella_alkaliphila GAP83078_Rosellinia_necatrix XP_ _Leptosphaeria_maculans XP_ _Magnaporthe_oryzae WP_ _Streptomyces_phaeochromogenes KRD202_Streptomyces_sp EKX634_Streptomyces_ipomoeae WP_ _Streptomyces_griseus WP_ _Streptomyces_turgidiscabies KKY35501_Diaporthe_ampelina WP_ _Streptomyces_bingchenggensis WP_ _Jiangella_alkaliphila WP_ _Jiangella_gansuensis WP_ _Streptomyces_sp WP_ _Herbidospora_cretacea WP_ _Actinoplanes_globisporus WP_ _Streptomyces_afghaniensis XP_ _Gaeumannomyces_graminis ELQ63839_Magnaporthe_oryzae WP_ _Jiangella_muralis ADI039_Streptomyces_bingchenggensis WP_ _Streptomyces_ipomoeae KPM46055_Neonectria_ditissima KFF211_Streptomyces_scabiei WP_ _Jiangella_muralis WP_ _Jiangella_muralis WP_ _Actinospica_robiniae WP_ _Clostridium_drakei WP_ _Streptomyces_sp WP_ _Streptomyces_sp EPJ42815_Streptomyces_afghaniensis WP_03331_Streptomyces_yerevanensis WP_ _Streptomyces_griseoruber VM1G_04714
25 KFY72129_Pseudogymnoascus_pannorum WP_ _Pseudomonas_sp WP_ _Pseudomonas_sp WP_ _Agrobacterium_rhizogenes GAD0_Byssochlamys_spectabilis EQB50603_Colletotrichum_gloeosporioides EXK762_Fusarium_oxysporum XP_ _Nectria_haematococca VM1G_05155_Valsa_mali XP_ _Nectria_haematococca XP_ _Colletotrichum_gloeosporioides KFY12076_Pseudogymnoascus_pannorum WP_ _Caulobacter_segnis WP_ _Acinetobacter_baumannii KGO69138_Penicillium_expansum WP_ _Paenibacillus_sp KFY05889_Pseudogymnoascus_pannorum XP_ _Neofusicoccum_parvum WP_ _Agrobacterium_vitis KIL86485_Fusarium_avenaceum WP_ _Pseudomonas_fluorescens XP_ _Rasamsonia_emersonii WP_ _Serratia_sp WP_ _Rhodococcus_sp WP_ _Rhizobium_leguminosarum KFZ05818_Pseudogymnoascus_pannorum WP_ _Pseudomonas_brassicacearum EXL915_Fusarium_oxysporum XP_ _Colletotrichum_fioriniae WP_ _Paenibacillus_sp KGO47902_Penicillium_expansum XP_ _Nectria_haematococca KGO617_Penicillium_expansum Ascomycota Ascomycota VM1G_05155
26 0.3 WP_ _Bacillus_thuringiensis VM1G_05405_Valsa_mali EGV00186_Mycoplasma_columbinum KIR032_Lachnospiraceae_bacterium EMR90169_Botrytis_cinerea XP_ _Sclerotinia_sclerotiorum ESZ91503_Sclerotinia_borealis EWC47146_Drechslerella_stenobrocha WP_04660_Bacillus_cereus XP_ _Tuber_melanosporum AET42542_Emiliania_huxleyi WP_ _Mycoplasma_hyopneumoniae WP_048722_Bacillus_cytotoxicus CNS05861_Salmonella_enterica AET610_Emiliania_huxleyi CCX04588_Pyronema_omphalodes WP_ _Mycoplasma_bovis XP_ _Micromonas_pusilla WP_ _Bacillus_cereus WP_ _Mycoplasma_anatis WP_ _Ruminococcus_albus WP_ _Mycoplasma_agalactiae WP_ _Bacillus_cereus AKU788_Spiroplasma_turonicum KKY246_Diaporthe_ampelina WP_ _Bacillus_cereus KPU55275_Bacillus_cereus CDC49071_Clostridium_sp WP_ _Spiroplasma_apis CCA68150_Piriformospora_indica WP_01782_Ruminococcus_flavefaciens EGS282_Mycoplasma_anatis WP_ _WP_ WP_ _Bacillus_cereus WP_ _Mycoplasma_columbinum WP_ _Mycoplasma_columbinum WP_ _Mycoplasma_agalactiae WP_02338_Mycoplasma_glycophilum WP_ _Ruminococcus_albus WP_ _Mycoplasma_hyopneumoniae WP_ _Bacillus_thuringiensis WP_ _Bacillus_cereus XP_ _Botrytis_cinerea XP_ _Dactylellina_haptotyla CCD50810_Botrytis_cinerea WP_ _Mycoplasma_hyopneumoniae CCD50809_Botrytis_cinerea WP_ _Spiroplasma_syrphidicola WP_ _Bacillus_cereus YP_ _Cafeteria_roenbergensis AKA50185_Mycoplasma_gallinaceum WP_ _Mycoplasma_flocculare XP_ _Arthrobotrys_oligospora WP_ _Spiroplasma_apis KPI390_Phialophora_attae WP_ _Bacillus_cereus Ascomycota VM1G_05405
27 VM1G_05720 WP_ _Streptomycetaceae_bacterium 85 WP_ _Streptomyces_vitaminophilus WP_ _Streptomyces_peruviensis 83 WP_ _Streptomyces_viridochromogenes 74 WP_ _Streptomyces_viridochromogenes 67 WP_ _Streptomyces_aureofaciens WP_ _Streptomyces_scabrisporus 80 WP_ _Nonomuraea_coxensis KRF34641_Nocardioides_sp KRE443_Nocardioides_sp 84 KGO636_Penicillium_italicum KJJ26316_Penicillium_solitum 42 KGO43380_Penicillium_expansum KGO5_Penicillium_expansum 64 KIN08901_Oidiodendron_maius 75 VM1G_05720_Valsa_mali XP_ _Aureobasidium_namibiae GAM913_fungal_sp WP_01291_Kribbella_flavida WP_ _Kribbella_catacumbae WP_ _WP_
28 WP_ _Marinobacter_nanhaiticus KPV61707_Bathyarchaeota_archaeon ESN65425_Serratia_sp EKG16230_Macrophomina_phaseolina WP_ _Thermoanaerobacter_italicus WP_ _Clostridium_sp WP_ _Bifidobacterium_reuteri ADI02052_Syntrophothermus_lipocalidus WP_ _Clostridium_sp WP_ _Stigmatella_aurantiaca WP_ _Syntrophothermus_lipocalidus WP_ _Clostridium_sp WP_ _Clostridium_sp XP_ _Pestalotiopsis_fici EAU64700_Stigmatella_aurantiaca WP_ _Thermoanaerobacter_sp WP_ _Thermoanaerobacter_ethanolicus WP_ _Brenneria_goodwinii WP_ _Thermoanaerobacter_sp WP_ _Frankia_sp WP_ _Marinobacterium_litorale KFA45684_Stachybotrys_chartarum WP_ _Thermoanaerobacter_wiegelii WP_ _Clostridium_sp WP_ _Serratia_sp WP_ _Butyrivibrio_crossotus XP_ _Grosmannia_clavigera KEY71523_Stachybotrys_chartarum XP_ _Togninia_minima WP_ _Clostridium_sp WP_ _Clostridiaceae_bacterium KFI83206_Bifidobacterium_reuteri WP_ _Frankia_sp XP_ _Neofusicoccum_parvum KFA6_Stachybotrys_chlorohalonata WP_ _Pseudomonas_sp KFA71331_Stachybotrys_chartarum WP_ _Desulfotomaculum_thermocisternum VM1G_05918_Valsa_mali WP_ _Thermoanaerobacter_sp WP_ _Clostridium_argentinense WP_ _Caldanaerobacter_subterraneus CCY766_Butyrivibrio_crossotus KKY26283_Phaeomoniella_chlamydospora XP_00713_Capronia_epimyces WP_ _Acetivibrio_cellulolyticus WP_ _Desulfotomaculum_alkaliphilum WP_ _Ruminiclostridium_thermocellum WP_ _Clostridiales_bacterium VM1G_05918
29 KPI44658_Phialophora_attae XP _Cyphellophora_europaea VM1G_07081_Valsa_mali KIW13928_Exophiala_spinifera CCF657_Nocardia_cyriacigeorgica ADI11806_Streptomyces_bingchenggensis WP _Amycolatopsis_jejuensis WP03192_Nocardia_otitidiscaviarum XP _Talaromyces_stipitatus WP _Nocardia_farcinica WP _Mycobacterium_mageritense WP _Nocardia_cyriacigeorgica XP _Trichoderma_atroviride XP _Coccidioides_immitis EJZ13104_Mycobacterium_fortuitum CRZ15879_Mycobacterium_neworleansense WP _Mycobacterium_sp KMU87049_Coccidioides_immitis CRG873_Talaromyces_islandicus WP _Nocardia_cyriacigeorgica XP _Exophiala_aquamarina XP _Coccidioides_posadasii WP _Rhodococcus_erythropolis CEO59207_Penicillium_brasilianum WP _Streptomyces_bingchenggensis Fusarium_spp Mycobacterium_spp Mycobacterium_spp VM1G_07081
30 WP_ _Prevotella_veroralis WP_ _Methyloversatilis_universalis WP_ _Mesorhizobium_sp WP_ _Sphingopyxis_sp WP_ _Prevotella_melaninogenica WP_ _Pelomonas_sp WP_ _Anaerolinea_thermophila WP_ _Sphingomonas_sp WP_ _Nocardioides_sp WP_ _Prevotella_sp WP_04854_Tetrasphaera_japonica ABQ71016_Sphingomonas_wittichii WP_ _Mycobacterium_vaccae WP_ _Prevotella_multiformis CDA82828_Bacteroides_sp WP_ _Frigoribacterium_sp WP_ _Pseudomonas_fluorescens WP_ _Nocardioides_sp WP_ _Bacteroides_sp WP_ _Nocardioides_sp WP_ _Prevotella_nanceiensis WP_ _Georgenia_sp CRK15713_Verticillium_longisporum WP_02782_Anaerobiospirillum_succiniciproducens WP_ _Prevotella_sp EFC729_Prevotella_melaninogenica WP_ _Pseudomonas_sp WP_ _Methylocystis_sp WP_ _Sphingomonas_sp VM1G_088_Valsa_mali WP_ _Mycobacterium_goodii WP_ _Methyloversatilis_universalis WP_ _Frigoribacterium_sp WP_ _Sphingopyxis_macrogoltabida WP_ _Hyphomicrobium_zavarzinii WP_ _Curtobacterium_sp WP_ _Prevotella_sp WP_ _Prevotella_veroralis WP_ _Alkanindiges_illinoisensis WP_ _Phycicoccus_jejuensis WP_ _Bacteroides_sp WP_ _Mobilicoccus_sp KOR33108_Achromatium_sp CUO758_Bacteroides_xylanisolvens WP_ _Bacteroides_xylanisolvens WP_ _Pseudomonas_tuomuerensis WP_ _Mobilicoccus_pelagius WP_ _Pseudomonas_fluorescens WP_ _Candidatus_Achromatium AIV40612_Curtobacterium_sp WP_ _Pseudomonas_chlororaphis WP_ _Roseibacterium_elongatum WP_05641_Sphingomonas_sp VM1G_088
31 WP_ _Rubinisphaera_brasiliensis WP_ _Turicibacter_sanguinis WP_01134_Hahella_chejuensis KGZ109_Raoultella_planticola KPL13614_Bacteroides_sp WP_ _Gimesia_maris WP_ _Blastopirellula_marina WP_ _Methylosarcina_fibrata WP_ _Turicibacter_sanguinis KIW16080_Exophiala_spinifera WP_ _Helicobacter_ailurogastricus WP_ _Streptomyces_neyagawaensis AIT56388_uncultured_bacterium XP_ _Rhinocladiella_mackenziei WP_ _Streptomyces_mirabilis ADB40010_Spirosoma_linguale WP_ _Raoultella_planticola WP_ _Frankia_sp WP_ _Frankia_sp WP_ _Actinoplanes_globisporus CRF52922_Helicobacter_ailurogastricus XP_ _Capronia_coronata WP_ _Frankia_sp WP_ _Amycolatopsis_halophila XP_ _Exophiala_dermatitidis WP_ _Frankia_sp WP_ _Nocardiopsis_potens KIV80839_Exophiala_sideris WP_ _Turicibacter_sp WP_ _Streptomyces_europaeiscabiei KJE22112_Frankia_sp ADO74692_Stigmatella_aurantiaca WP_ _Streptomyces_bottropensis WP_ _Raoultella_sp KFD06387_Raoultella_planticola VM1G_09114_Valsa_mali WP_ _Stigmatella_aurantiaca KKY36417_Diaporthe_ampelina WP_ _Porphyrobacter_sp KQM04280_Frankia_sp WP_ _Caloramator_sp WP_ _Turicibacter_sanguinis VM1G_09114
32 0.1 WP_ _Rhodopseudomonas_sp WP_ _Pandoraea_sp WP_ _Burkholderia_contaminans WP_ _Rhodopseudomonas_sp WP_ _Burkholderiaceae_bacterium WP_ _Pandoraea_sp WP_ _Burkholderia_bannensis VM1G_10126_Valsa_mali WP_ _Cupriavidus_basilensis WP_ _Rhodopseudomonas_palustris WP_ _Cupriavidus_sp WP_040_Rhodopseudomonas_palustris AHB77920_Pandoraea_sp WP_ _Burkholderia_mimosarum WP_ _Burkholderia_phymatum WP_01129_Cupriavidus_pinatubonensis WP_ _Rhodopseudomonas_palustris WP_ _Burkholderia_sp WP_ _Rhodopseudomonas_palustris WP_02539_Pandoraea_pnomenusa WP_ _Pseudomonas_sp AHN788_Pandoraea_pnomenusa VM1G_10126
33 WP_ _Lampropedia_sp KKY15566_Phaeomoniella_chlamydospora XP_ _Bipolaris_oryzae ESW512_Rhodobacter_sp WP_ _Ochrobactrum_sp CDE50821_Faecalibacterium_sp KKY13883_Diplodia_seriata WP_ _unclassified_Clostridiales XP_ _Exophiala_xenobiotica WP_ _Ochrobactrum_intermedium CCD47517_Botrytis_cinerea WP_ _Lampropedia_sp XP_ _Bipolaris_zeicola ABP50426_Pyrobaculum_arsenaticum ABO08726_Pyrobaculum_calidifontis WP_047510_Rhizobium_sp XP_ _Notothenia_coriiceps WP_ _Rhizobium_sp BAN8_Aeropyrum_camini AFA320_Pyrobaculum_oguniense XP_ _Notothenia_coriiceps WP_ _Nitrospina_gracilis WP_ _WP_ XP_ _Capronia_epimyces WP_ _Caldivirga_maquilingensis XP_ _Cladophialophora_psammophila WP_ _Desulfurococcus_mucosus ESZ587_Sclerotinia_borealis WP_ _Rhizobium_sp XP_ _Maylandia_zebra XP_ _Fonsecaea_pedrosoi CDX58527_Mesorhizobium_plurifarium WP_ _Ochrobactrum_anthropi WP_ _Thermoproteus_tenax WP_ _Rhizobium_alamii KIW71727_Capronia_semiimmersa XP_ _Salmo_salar XP_00761_Bipolaris_sorokiniana BAA79068_Aeropyrum_pernix WP_ _Pyrobaculum_islandicum XP_ _Selaginella_moellendorffii WP_ _Pyrodictium_delaneyi KIV85086_Exophiala_sideris ACB407_Pyrobaculum_neutrophilum WP_ _Rhizobium_alamii KIX314_Fonsecaea_multimorphosa EEQ130_Ochrobactrum_intermedium VM1G_10361_Valsa_mali WP_ _Ochrobactrum_anthropi XP_ _Branchiostoma_floridae KNG44746_Stemphylium_lycopersici WP_ _Martelella_endophytica XP_ _Capronia_coronata AAL64263_Pyrobaculum_aerophilum WP_ _Pyrolobus_fumarii WP_ _Ochrobactrum_sp KJS17081_Hoeflea_sp WP_ _Ochrobactrum_intermedium XP_ _Cladophialophora_carrionii WP_ _Rhizobium_leguminosarum XP_ _Papilio_polytes CDK24869_Kuraishia_capsulata ERM02390_Ochrobactrum_intermedium XP_ _Aplysia_californica XP_ _Ogataea_parapolymorpha WP_ _Ochrobactrum_intermedium WP_ _Alphaproteobacteria WP_ _Ochrobactrum_intermedium WP_ _Ochrobactrum_sp WP_ _Desulfurococcus_mobilis WP_ _Arthrobacter_sp AKT35671_Pyrobaculum_sp KIW464_Exophiala_oligosperma EUN231_Bipolaris_victoriae KIW368_Cladophialophora_bantiana XP_ _Rhinocladiella_mackenziei KJS17077_Hoeflea_sp KIY45135_Fistulina_hepatica ADM28010_Ignisphaera_aggregans WP_ _Martelella_endophytica ADZ72558_Polymorphum_gilvum XP_ _Pyrenophora_teres AJY45479_Martelella_endophytica KIW25045_Cladophialophora_immunda WP_ _Ochrobactrum_sp XP_ _Bipolaris_maydis XP_ _Pestalotiopsis_fici KPQ17907_Rhodobacteraceae_bacterium WP_ _Rhizobium_sp WP_ _Thiothrix_flexilis WP_ _Hungatella_hathewayi KIW15468_Exophiala_spinifera XP_ _Sclerotinia_sclerotiorum XP_ _Leptosphaeria_maculans XP_ _Grosmannia_clavigera XP_ _Setosphaeria_turcica XP_ _Cladophialophora_yegresii KGK40729_Pichia_kudriavzevii WP_ _Celeribacter_indicus AJE45757_Celeribacter_indicus WP_ _Ochrobactrum_anthropi /Archaea VM1G_10361
34 0.3 WP_02663_Nocardia_brasiliensis KJR86022_Sporothrix_schenckii ERS929_Sporothrix_schenckii WP_ _Actinopolyspora_erythraea WP_ _Gordonia_effusa WP_ _Nocardia_brasiliensis WP_ _Mycobacterium_abscessus WP_ _Streptacidiphilus_anmyonensis WP_ _Mycobacterium_iranicum WP_ _Streptomyces_xylophagus WP_ _Nocardia_brasiliensis WP_ _Mycobacterium_gilvum WP_ _Mycobacterium_aromaticivorans XP_ _Togninia_minima WP_ _Mycobacterium_aurum WP_ _Nocardia_nova WP_ _Mycobacterium_vaccae WP_ _Sandarakinorhabdus_sp KIH915_Sporothrix_brasiliensis WP_ _Nocardia_vulneris KDE0_Mycobacterium_aromaticivorans WP_ _Streptomyces_monomycini VM1G_10685_Valsa_mali VM1G_10685
35 WP_ _Clostridiales_bacterium WP_ _Clostridiales_bacterium KON206_miscellaneous_Crenarchaeota WP_ _Moorella_thermoacetica WP_ _Anaerosalibacter_sp WP_ _Thermococcus_litoralis WP_ _Acetonema_longum WP_ _Caldivirga_maquilingensis WP_ _Pyrococcus_horikoshii CBK79287_Coprococcus_catus WP_ _Lysinibacillus_fusiformis WP_ _Blautia_producta WP_ _Lysinibacillus_sphaericus VM1G_109_Valsa_mali KON29082_miscellaneous_Crenarchaeota WP_ _bacterium_OL WP_ _Lysinibacillus_sphaericus WP_ _Pyramidobacter_piscolens WP_ _Blautia_producta WP_ _bacterium_UASB WP_00526_Blautia_hydrogenotrophica WP_ _Photobacterium_sanctipauli WP_ _Clostridiales_bacterium WP_ _Spirochaeta_bajacaliforniensis WP_ _Pyrococcus_sp WP_ _Anaerobaculum_hydrogeniformans Outgroup Outgroup /Archaea VM1G_109
Manuscript Title: Identification of a thermostable fungal lytic polysaccharide monooxygenase and
 1 2 3 4 5 Journal name: Applied Microbiology and Biotechnology Manuscript Title: Identification of a thermostable fungal lytic polysaccharide monooxygenase and evaluation of its effect on lignocellulosic
1 2 3 4 5 Journal name: Applied Microbiology and Biotechnology Manuscript Title: Identification of a thermostable fungal lytic polysaccharide monooxygenase and evaluation of its effect on lignocellulosic
Forensic SNP Genotyping using Nanopore MinION Sequencing
 Forensic SNP Genotyping using Nanopore MinION Sequencing AUTHORS Senne Cornelis 1, Yannick Gansemans 1, Lieselot Deleye 1, Dieter Deforce 1,#,Filip Van Nieuwerburgh 1,#,* 1 Laboratory of Pharmaceutical
Forensic SNP Genotyping using Nanopore MinION Sequencing AUTHORS Senne Cornelis 1, Yannick Gansemans 1, Lieselot Deleye 1, Dieter Deforce 1,#,Filip Van Nieuwerburgh 1,#,* 1 Laboratory of Pharmaceutical
Supporting Information
 Supporting Information Paired design of dcas9 as a systematic platform for the detection of featured nucleic acid sequences in pathogenic strains Yihao Zhang 1,2,8, Long Qian 4,8, Weijia Wei 1,3,7,8, Yu
Supporting Information Paired design of dcas9 as a systematic platform for the detection of featured nucleic acid sequences in pathogenic strains Yihao Zhang 1,2,8, Long Qian 4,8, Weijia Wei 1,3,7,8, Yu
University of Bristol - Explore Bristol Research
 Al-Salihi, S. A. A., Scott, T. A., Bailey, A. M., & Foster, G. D. (2017). Improved vectors for Agrobacterium mediated genetic manipulation of Hypholoma spp. and other homobasidiomycetes. Journal of Microbiological
Al-Salihi, S. A. A., Scott, T. A., Bailey, A. M., & Foster, G. D. (2017). Improved vectors for Agrobacterium mediated genetic manipulation of Hypholoma spp. and other homobasidiomycetes. Journal of Microbiological
4 vana, vanb, vanc1, vanc2
 77 1) 2) 2) 3) 4) 5) 1) 2) 3) 4) 5) 16 12 7 17 4 8 2003 1 2004 7 3 Enterococcus 5 Enterococcus faecalis 1 Enterococcus avium 1 Enterococcus faecium 2 5 PCR E. faecium 1 E-test MIC 256 mg/ml vana MRSA MRSA
77 1) 2) 2) 3) 4) 5) 1) 2) 3) 4) 5) 16 12 7 17 4 8 2003 1 2004 7 3 Enterococcus 5 Enterococcus faecalis 1 Enterococcus avium 1 Enterococcus faecium 2 5 PCR E. faecium 1 E-test MIC 256 mg/ml vana MRSA MRSA
Modular Optimization of Hemicellulose-utilizing Pathway in. Corynebacterium glutamicum for Consolidated Bioprocessing of
 [Supplementary materials] Modular Optimization of Hemicellulose-utilizing Pathway in Corynebacterium glutamicum for Consolidated Bioprocessing of Hemicellulosic Biomass Sung Sun Yim 1, Jae Woong Choi 1,
[Supplementary materials] Modular Optimization of Hemicellulose-utilizing Pathway in Corynebacterium glutamicum for Consolidated Bioprocessing of Hemicellulosic Biomass Sung Sun Yim 1, Jae Woong Choi 1,
T-helper Type 2 driven Inflammation Defines Major Subphenotypes of Asthma
 T-helper Type 2 driven Inflammation Defines Major Subphenotypes of Asthma Prescott G. Woodruff, M.D., M.P.H., Barmak Modrek, Ph.D., David F. Choy, B.S., Guiquan Jia, M.D., Alexander R. Abbas, Ph.D., Almut
T-helper Type 2 driven Inflammation Defines Major Subphenotypes of Asthma Prescott G. Woodruff, M.D., M.P.H., Barmak Modrek, Ph.D., David F. Choy, B.S., Guiquan Jia, M.D., Alexander R. Abbas, Ph.D., Almut
Supplemental Information. Investigation of Penicillin Binding Protein. (PBP)-like Peptide Cyclase and Hydrolase
 Cell Chemical Biology, Volume upplemental Information Investigation of Penicillin Binding Protein (PBP)-like Peptide Cyclase and ydrolase in urugamide on-ribosomal Peptide Biosynthesis Yongjun Zhou, Xiao
Cell Chemical Biology, Volume upplemental Information Investigation of Penicillin Binding Protein (PBP)-like Peptide Cyclase and ydrolase in urugamide on-ribosomal Peptide Biosynthesis Yongjun Zhou, Xiao
Seven gene deletions in seven days: Fast generation of Escherichia coli strains tolerant to acetate and osmotic stress Supplementary Figure S1.
 Seven gene deletions in seven days: Fast generation of Escherichia coli strains tolerant to acetate and osmotic stress Sheila I. Jensen 1*, Rebecca M. Lennen 1, Markus J. Herrgård 1, Alex T. Nielsen 1*
Seven gene deletions in seven days: Fast generation of Escherichia coli strains tolerant to acetate and osmotic stress Sheila I. Jensen 1*, Rebecca M. Lennen 1, Markus J. Herrgård 1, Alex T. Nielsen 1*
Involvement of ER Stress in Dysmyelination of Pelizaeus-Merzbacher Disease with PLP1 Missense Mutations Shown by ipsc-derived Oligodendrocytes
 Stem Cell Reports, Volume 2 Supplemental Information Involvement of ER Stress in Dysmyelination of Pelizaeus-Merzbacher Disease with PLP1 Missense Mutations Shown by ipsc-derived Oligodendrocytes Yuko
Stem Cell Reports, Volume 2 Supplemental Information Involvement of ER Stress in Dysmyelination of Pelizaeus-Merzbacher Disease with PLP1 Missense Mutations Shown by ipsc-derived Oligodendrocytes Yuko
A cell-based screening system for RNA Polymerase I inhibitors
 Electronic Supplementary Material (ESI) for MedChemComm. This journal is The Royal Society of Chemistry 2019 Supporting Information A cell-based screening system for RNA Polymerase I inhibitors Xiao Tan,
Electronic Supplementary Material (ESI) for MedChemComm. This journal is The Royal Society of Chemistry 2019 Supporting Information A cell-based screening system for RNA Polymerase I inhibitors Xiao Tan,
Nan Wang, Qingming Dong, Jingjing Li, Rohit K. Jangra, Meiyun Fan, Allan R. Brasier, Stanley M. Lemon, Lawrence M. Pfeffer, Kui Li
 Supplemental Material IRF3-dependent and NF- B-independent viral induction of the zinc-finger antiviral protein Nan Wang, Qingming Dong, Jingjing Li, Rohit K. Jangra, Meiyun Fan, Allan R. Brasier, Stanley
Supplemental Material IRF3-dependent and NF- B-independent viral induction of the zinc-finger antiviral protein Nan Wang, Qingming Dong, Jingjing Li, Rohit K. Jangra, Meiyun Fan, Allan R. Brasier, Stanley
Supplementary materials to: Whole-mount single molecule FISH method for zebrafish embryo
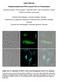
 Supplementary materials to: Whole-mount single molecule FISH method for zebrafish embryo Yuma Oka and Thomas N. Sato Supplementary Figure S1. Whole-mount smfish with and without the methanol pretreatment.
Supplementary materials to: Whole-mount single molecule FISH method for zebrafish embryo Yuma Oka and Thomas N. Sato Supplementary Figure S1. Whole-mount smfish with and without the methanol pretreatment.
Flowering time. Col C24 Cvi C24xCol C24xCvi ColxCvi
 Flowering time Rosette leaf number 50 45 40 35 30 25 20 15 10 5 0 Col C24 Cvi C24xCol C24xCvi ColxCvi Figure S1. Flowering time in three F 1 hybrids and their parental lines as measured by leaf number
Flowering time Rosette leaf number 50 45 40 35 30 25 20 15 10 5 0 Col C24 Cvi C24xCol C24xCvi ColxCvi Figure S1. Flowering time in three F 1 hybrids and their parental lines as measured by leaf number
AZ ALACSONY HŐMÉRSÉKLET HATÁSÁRA BEKÖVETKEZŐ REDOX ÉS GÉNEXPRESSZIÓS VÁLTOZÁSOK GABONAFÉLÉKBEN
 A martonvásári agrárkutatások hatodik évtizede AZ ALAONY HŐMÉRSÉKLET HATÁSÁRA BEKÖVETKEZŐ REDOX ÉS GÉNEXPRESSZIÓS VÁLTOZÁSOK GABONAFÉLÉKBEN KOY GÁBOR, VÁGÚJFALVI ATTILA, TÓTH BALÁZS, SZALAI GABRIELLA,
A martonvásári agrárkutatások hatodik évtizede AZ ALAONY HŐMÉRSÉKLET HATÁSÁRA BEKÖVETKEZŐ REDOX ÉS GÉNEXPRESSZIÓS VÁLTOZÁSOK GABONAFÉLÉKBEN KOY GÁBOR, VÁGÚJFALVI ATTILA, TÓTH BALÁZS, SZALAI GABRIELLA,
Expression analysis of PIN genes in root tips and nodules of Lotus japonicus
 Article Expression analysis of PIN genes in root tips and nodules of Lotus japonicus Izabela Sańko-Sawczenko 1, Dominika Dmitruk 1, Barbara Łotocka 1, Elżbieta Różańska 1 and Weronika Czarnocka 1, * 1
Article Expression analysis of PIN genes in root tips and nodules of Lotus japonicus Izabela Sańko-Sawczenko 1, Dominika Dmitruk 1, Barbara Łotocka 1, Elżbieta Różańska 1 and Weronika Czarnocka 1, * 1
(A F) H&E staining of the duodenum in the indicated genotypes at E16.5 (A, D), E18.5
 Fig. S1. Brg1 ablation leads to abnormal villous structure in the duodenum in the perinatal period (A F) H&E staining of the duodenum in the indicated genotypes at E16.5 (A, D), E18.5 (B, E), and P0.5
Fig. S1. Brg1 ablation leads to abnormal villous structure in the duodenum in the perinatal period (A F) H&E staining of the duodenum in the indicated genotypes at E16.5 (A, D), E18.5 (B, E), and P0.5
Supplementary Table 1. Cystometric parameters in sham-operated wild type and Trpv4 -/- rats during saline infusion and
 WT sham Trpv4 -/- sham Saline 10µM GSK1016709A P value Saline 10µM GSK1016709A P value Number 10 10 8 8 Intercontractile interval (sec) 143 (102 155) 98.4 (71.4 148) 0.01 96 (92 121) 109 (95 123) 0.3 Voided
WT sham Trpv4 -/- sham Saline 10µM GSK1016709A P value Saline 10µM GSK1016709A P value Number 10 10 8 8 Intercontractile interval (sec) 143 (102 155) 98.4 (71.4 148) 0.01 96 (92 121) 109 (95 123) 0.3 Voided
Markerless Escherichia coli rrn Deletion Strains for Genetic Determination of Ribosomal Binding Sites
 Markerless Escherichia coli rrn Deletion Strains for Genetic Determination of Ribosomal Binding Sites Selwyn Quan *, Ole Skovgaard, Robert E. McLaughlin, Ed T. Buurman,1, and Catherine L. Squires * Department
Markerless Escherichia coli rrn Deletion Strains for Genetic Determination of Ribosomal Binding Sites Selwyn Quan *, Ole Skovgaard, Robert E. McLaughlin, Ed T. Buurman,1, and Catherine L. Squires * Department
Glyma10g15850 aagggatccattctggaaccatatcttgctgtg ttgggtacccttggatgcaggatgacacg AtMKK6, AT5g56580
 Supplemental table 1. Soybean MAPK, MAPKK, and MAPKKK genes and primers for VIGS cloning Gene_ID Forward Primer Reverse Primer Arabidopsis Ortholog Glyma04g03210 aagggatcctgcagcaagaaccagttcat ttgggtaccctaatggatgcgcattaggataaag
Supplemental table 1. Soybean MAPK, MAPKK, and MAPKKK genes and primers for VIGS cloning Gene_ID Forward Primer Reverse Primer Arabidopsis Ortholog Glyma04g03210 aagggatcctgcagcaagaaccagttcat ttgggtaccctaatggatgcgcattaggataaag
(11) Lajstromszám: E 005 557 (13) T2 EURÓPAI SZABADALOM SZÖVEGÉNEK FORDÍTÁSA
 !HU000007T2! (19) HU (11) Lajstromszám: E 00 7 (13) T2 MAGYAR KÖZTÁRSASÁG Magyar Szabadalmi Hivatal EURÓPAI SZABADALOM SZÖVEGÉNEK FORDÍTÁSA (21) Magyar ügyszám: E 03 026690 (22) A bejelentés napja: 03.
!HU000007T2! (19) HU (11) Lajstromszám: E 00 7 (13) T2 MAGYAR KÖZTÁRSASÁG Magyar Szabadalmi Hivatal EURÓPAI SZABADALOM SZÖVEGÉNEK FORDÍTÁSA (21) Magyar ügyszám: E 03 026690 (22) A bejelentés napja: 03.
Phenotype. Genotype. It is like any other experiment! What is a bioinformatics experiment? Remember the Goal. Infectious Disease Paradigm
 It is like any other experiment! What is a bioinformatics experiment? You need to know your data/input sources You need to understand your methods and their assumptions You need a plan to get from point
It is like any other experiment! What is a bioinformatics experiment? You need to know your data/input sources You need to understand your methods and their assumptions You need a plan to get from point
Architecture of the Trypanosome RNA Editing Accessory Complex, MRB1
 Architecture of the Trypanosome RNA Editing Accessory Complex, MRB1 Michelle L. Ammerman, Kurtis M. Downey, Hassan Hashimi, John C. Fisk, Danielle L. Tomasello, Drahomíra Faktorová, Lucie Hanzálková, Tony
Architecture of the Trypanosome RNA Editing Accessory Complex, MRB1 Michelle L. Ammerman, Kurtis M. Downey, Hassan Hashimi, John C. Fisk, Danielle L. Tomasello, Drahomíra Faktorová, Lucie Hanzálková, Tony
Akt1 Akt kinase activity Creb signaling CCTTACAGCCCTCAAGTACTCATTC GGCGTACTCCATGACAAAGCA Arc Actin binding
 Additional File 1 (Table S1): Description and primer sequences of 80 candidate genes tested as potential synaptic plasticity formation genes in qrt-pcr array. Abbreviations: LTP: long-term potentiation,
Additional File 1 (Table S1): Description and primer sequences of 80 candidate genes tested as potential synaptic plasticity formation genes in qrt-pcr array. Abbreviations: LTP: long-term potentiation,
Hibridspecifikus tápanyag-és vízhasznosítás kukoricánál csernozjom talajon
 Hibridspecifikus tápanyag-és vízhasznosítás kukoricánál csernozjom talajon Karancsi Lajos Gábor Debreceni Egyetem Agrár és Gazdálkodástudományok Centruma Mezőgazdaság-, Élelmiszertudományi és Környezetgazdálkodási
Hibridspecifikus tápanyag-és vízhasznosítás kukoricánál csernozjom talajon Karancsi Lajos Gábor Debreceni Egyetem Agrár és Gazdálkodástudományok Centruma Mezőgazdaság-, Élelmiszertudományi és Környezetgazdálkodási
Crash Course in Omics Terminology, Concepts & Data Types
 Crash Course in Omics Terminology, Concepts & Data Types 12 th Annual EuPathDB Workshop: 2017 Jessie Kissinger June 18, 2017 FungiDB MicrosporidiaDB AmoebaDB PlasmoDB ToxoDB CryptoDB PiroplasmaDB TrichDB
Crash Course in Omics Terminology, Concepts & Data Types 12 th Annual EuPathDB Workshop: 2017 Jessie Kissinger June 18, 2017 FungiDB MicrosporidiaDB AmoebaDB PlasmoDB ToxoDB CryptoDB PiroplasmaDB TrichDB
Suppl. Materials. Polyhydroxyalkanoate (PHA) Granules Have no Phospholipids. Germany
 Suppl. Materials Polyhydroxyalkanoate (PHA) Granules Have no Phospholipids Stephanie Bresan 1, Anna Sznajder 1, Waldemar Hauf 2, Karl Forchhammer 2, Daniel Pfeiffer 3 and Dieter Jendrossek 1 1 Institute
Suppl. Materials Polyhydroxyalkanoate (PHA) Granules Have no Phospholipids Stephanie Bresan 1, Anna Sznajder 1, Waldemar Hauf 2, Karl Forchhammer 2, Daniel Pfeiffer 3 and Dieter Jendrossek 1 1 Institute
- Supplementary Data
 1 Open Access Asian Australas. J. Anim. Sci. Vol. 29, No. 3 : 321-326 March 2016 http://dx.doi.org/10.5713/ajas.15.0331 www.ajas.info pissn 1011-2367 eissn 1976-5517 Analysis of Swine Leukocyte Antigen
1 Open Access Asian Australas. J. Anim. Sci. Vol. 29, No. 3 : 321-326 March 2016 http://dx.doi.org/10.5713/ajas.15.0331 www.ajas.info pissn 1011-2367 eissn 1976-5517 Analysis of Swine Leukocyte Antigen
Supplemental Table S1. Overview of MYB transcription factor genes analyzed for expression in red and pink tomato fruit.
 Supplemental Table S1. Overview of MYB transcription factor genes analyzed for expression in red and pink tomato fruit. MYB Primer pairs TC AtMYB Forward Reverse TC199266 MYB12 AGGCTCTTGGAGGTCGTTACC CAACTCTTTCCGCATCTCAATAATC
Supplemental Table S1. Overview of MYB transcription factor genes analyzed for expression in red and pink tomato fruit. MYB Primer pairs TC AtMYB Forward Reverse TC199266 MYB12 AGGCTCTTGGAGGTCGTTACC CAACTCTTTCCGCATCTCAATAATC
Oxacillin MIC µ g ml borderline oxacillin-resistant Staphylococcus aureus. oxacillin Staphylococcus aureus MRSA
 oxacillin Staphylococcus aureus MRSA 8 17 11 1 Oxacillin MIC 1248 µ gml borderline oxacillin-resistant Staphylococcus aureus BORSA79 NCCLS CLSIM100-S142004 cefoxitin disk oxacillin cefoxitin methicillin-resistant
oxacillin Staphylococcus aureus MRSA 8 17 11 1 Oxacillin MIC 1248 µ gml borderline oxacillin-resistant Staphylococcus aureus BORSA79 NCCLS CLSIM100-S142004 cefoxitin disk oxacillin cefoxitin methicillin-resistant
Supplementary Figure 1
 Supplementary Figure 1 Plot of delta-afe of sequence variants detected between resistant and susceptible pool over the genome sequence of the WB42 line of B. vulgaris ssp. maritima. The delta-afe values
Supplementary Figure 1 Plot of delta-afe of sequence variants detected between resistant and susceptible pool over the genome sequence of the WB42 line of B. vulgaris ssp. maritima. The delta-afe values
 ö ö Ü Á Á Ő É ü ú ü ö Á É ö ú ü ö ö ö ü ö ö ö ü ö ü ö ö ö ö ö ö Í ú ö ö ö ö ü ü ű ö ö ö ö ű ú ü ö ö ű Á É Í Ő É É Á Í É Á Í Í Á ü ö ö ü ö ö ü ú ű ú ú ü ö ö ö ű ú ö ú ö ü ú ö ö ú ö ü ü ú ú ü ú ú ö ö ö
ö ö Ü Á Á Ő É ü ú ü ö Á É ö ú ü ö ö ö ü ö ö ö ü ö ü ö ö ö ö ö ö Í ú ö ö ö ö ü ü ű ö ö ö ö ű ú ü ö ö ű Á É Í Ő É É Á Í É Á Í Í Á ü ö ö ü ö ö ü ú ű ú ú ü ö ö ö ű ú ö ú ö ü ú ö ö ú ö ü ü ú ú ü ú ú ö ö ö
AZ ÁRPA SZÁRAZSÁGTŰRÉSÉNEK VIZSGÁLATA: QTL- ÉS ASSZOCIÁCIÓS ANALÍZIS, MARKER ALAPÚ SZELEKCIÓ, TILLING
 Hagyomány és haladás a növénynemesítésben AZ ÁRPA SZÁRAZSÁGTŰRÉSÉNEK VIZSGÁLATA: QTL- ÉS ASSZOCIÁCIÓS ANALÍZIS, MARKER ALAPÚ SZELEKCIÓ, BÁLINT ANDRÁS FERENC 1, SZIRA FRUZSINA 1, ANDREAS BÖRNER 2, KERSTIN
Hagyomány és haladás a növénynemesítésben AZ ÁRPA SZÁRAZSÁGTŰRÉSÉNEK VIZSGÁLATA: QTL- ÉS ASSZOCIÁCIÓS ANALÍZIS, MARKER ALAPÚ SZELEKCIÓ, BÁLINT ANDRÁS FERENC 1, SZIRA FRUZSINA 1, ANDREAS BÖRNER 2, KERSTIN
University of Bristol - Explore Bristol Research
 Tan, B. G., Wellesley, F. C., Savery, N. J., & Szczelkun, M. D. (2016). Length heterogeneity at conserved sequence block 2 in human mitochondrial DNA acts as a rheostat for RNA polymerase POLRMT activity.
Tan, B. G., Wellesley, F. C., Savery, N. J., & Szczelkun, M. D. (2016). Length heterogeneity at conserved sequence block 2 in human mitochondrial DNA acts as a rheostat for RNA polymerase POLRMT activity.
A LISZTHARMAT ÉS A FOGÉKONY SZŐLŐ KÖZÖTTI MOLEKULÁRIS KAPCSOLAT. Tóth Zsófia 1 -Kiss Erzsébet 2
 A LISZTHARMAT ÉS A FOGÉKONY SZŐLŐ KÖZÖTTI MOLEKULÁRIS KAPCSOLAT Tóth Zsófia 1 -Kiss Erzsébet 2 1 Szent István Egyetem, Genetika és Biotechnológiai Intézet, Gödöllő 2100 Páter Károly utca 1., 0628/522069,
A LISZTHARMAT ÉS A FOGÉKONY SZŐLŐ KÖZÖTTI MOLEKULÁRIS KAPCSOLAT Tóth Zsófia 1 -Kiss Erzsébet 2 1 Szent István Egyetem, Genetika és Biotechnológiai Intézet, Gödöllő 2100 Páter Károly utca 1., 0628/522069,
PIAL1 (At1g08910) ORF sequence
 PIAL1 (At1g08910) ORF sequence ATGGTTATTCCGGCGACTTCTAGGTTTGGGTTTCGTGCTGAATTCAACACCAAGGAGTTTCAAGCTTCCTGCATCTCTCT! CGCTAATGAAATCGATGCGGCTATTGGGAGAAATGAAGTTCCAGGGAATATTCAAGAGCTCGCTTTAATCCTCAATAATG! TGTGCCGACGTAAATGTGATGATTATCAAACCAGGGCGGTGGTAATGGCGCTGATGATCTCAGTCAAGAGTGCTTGTCAG!
PIAL1 (At1g08910) ORF sequence ATGGTTATTCCGGCGACTTCTAGGTTTGGGTTTCGTGCTGAATTCAACACCAAGGAGTTTCAAGCTTCCTGCATCTCTCT! CGCTAATGAAATCGATGCGGCTATTGGGAGAAATGAAGTTCCAGGGAATATTCAAGAGCTCGCTTTAATCCTCAATAATG! TGTGCCGACGTAAATGTGATGATTATCAAACCAGGGCGGTGGTAATGGCGCTGATGATCTCAGTCAAGAGTGCTTGTCAG!
There are no translations available. CURRENT APPOINTMENT(S):
 There are no translations available. CURRENT APPOINTMENT(S): Research fellow of Biochemistry and Molecular Biology, Department of Biochemistry and Molecular Biology, Medical and Health Science Center (MHSC),
There are no translations available. CURRENT APPOINTMENT(S): Research fellow of Biochemistry and Molecular Biology, Department of Biochemistry and Molecular Biology, Medical and Health Science Center (MHSC),
INFO-CAPITALISM IN CENTRAL EUROPE: THE VISEGRAD STRATEGY. By Pál TAMÁS [Institute of Sociology, HAS, Budapest]
![INFO-CAPITALISM IN CENTRAL EUROPE: THE VISEGRAD STRATEGY. By Pál TAMÁS [Institute of Sociology, HAS, Budapest] INFO-CAPITALISM IN CENTRAL EUROPE: THE VISEGRAD STRATEGY. By Pál TAMÁS [Institute of Sociology, HAS, Budapest]](/thumbs/32/15680230.jpg) INFO-CAPITALISM IN CENTRAL EUROPE: THE VISEGRAD STRATEGY By Pál TAMÁS [Institute of Sociology, HAS, Budapest] A typology of FUAs, based on 5 functions: - population - transport - manufacturing GVA - no
INFO-CAPITALISM IN CENTRAL EUROPE: THE VISEGRAD STRATEGY By Pál TAMÁS [Institute of Sociology, HAS, Budapest] A typology of FUAs, based on 5 functions: - population - transport - manufacturing GVA - no
The beet R locus encodes a new cytochrome P450 required for red. betalain production.
 The beet R locus encodes a new cytochrome P450 required for red betalain production. Gregory J. Hatlestad, Rasika M. Sunnadeniya, Neda A. Akhavan, Antonio Gonzalez, Irwin L. Goldman, J. Mitchell McGrath,
The beet R locus encodes a new cytochrome P450 required for red betalain production. Gregory J. Hatlestad, Rasika M. Sunnadeniya, Neda A. Akhavan, Antonio Gonzalez, Irwin L. Goldman, J. Mitchell McGrath,
Magyar-kínai TéT együttműködés nyertesei a 2009-2010 évre
 Magyar-kínai TéT együttműködés nyertesei a 2009-2010 évre Az egyezmény alapján létrehozott kétoldalú TéT együttműködési Vegyes Bizottság legutóbbi ülésére 2008. november 18-án került sor. Magyar projektvezető
Magyar-kínai TéT együttműködés nyertesei a 2009-2010 évre Az egyezmény alapján létrehozott kétoldalú TéT együttműködési Vegyes Bizottság legutóbbi ülésére 2008. november 18-án került sor. Magyar projektvezető
kpis(ppk20) kpis(ppk46)
 Table S1. C. elegans strains used in this study Strains carrying transgenes Strain Transgene Genotype Reference IT213 tcer-1 prom:tcer-1orf:gfp:tcer-1 3'utr unc-119(ed3) III; kpis(ppk6) This study IT283
Table S1. C. elegans strains used in this study Strains carrying transgenes Strain Transgene Genotype Reference IT213 tcer-1 prom:tcer-1orf:gfp:tcer-1 3'utr unc-119(ed3) III; kpis(ppk6) This study IT283
Kína gazdasága, gazdasági fejlődése
 Kína gazdasága, gazdasági fejlődése Könyvek: Alden, Chris: Kína az afrikai kontinensen. Pécs : IDResearch Kft : Publikon K., 2010. 327 A 35 Bánhidi Ferenc: Kína gazdasági fejlődésének tendenciái a nyolcvanas
Kína gazdasága, gazdasági fejlődése Könyvek: Alden, Chris: Kína az afrikai kontinensen. Pécs : IDResearch Kft : Publikon K., 2010. 327 A 35 Bánhidi Ferenc: Kína gazdasági fejlődésének tendenciái a nyolcvanas
A KELET-BORSODI HELVÉTI BARNAKŐSZÉNTELEPEK TANI VIZSGÁLATA
 A KELET-BORSODI HELVÉTI BARNAKŐSZÉNTELEPEK TANI VIZSGÁLATA SZÉNKŐZET JUHÁSZ ANDRÁS* (3 ábrával) Összefoglalás: A szénkőzettani vizsgálatok céljául elsősorban a barnakőszéntelepek várható kiterjedésének
A KELET-BORSODI HELVÉTI BARNAKŐSZÉNTELEPEK TANI VIZSGÁLATA SZÉNKŐZET JUHÁSZ ANDRÁS* (3 ábrával) Összefoglalás: A szénkőzettani vizsgálatok céljául elsősorban a barnakőszéntelepek várható kiterjedésének
CELTIS FAJOK KOMPLEX ETIOLÓGIÁJÚ BETEGSÉGÉNEK DIAGNÓZISA (esettanulmány)
 NÖVÉNYVÉDELEM 46 (10), 2010 465 CELTIS FAJOK KOMPLEX ETIOLÓGIÁJÚ BETEGSÉGÉNEK DIAGNÓZISA (esettanulmány) Vajna László MTA Növényvédelmi Kutatóintézete, 1525 Budapest Pf. 102. Díszfák, erdei fafajok és
NÖVÉNYVÉDELEM 46 (10), 2010 465 CELTIS FAJOK KOMPLEX ETIOLÓGIÁJÚ BETEGSÉGÉNEK DIAGNÓZISA (esettanulmány) Vajna László MTA Növényvédelmi Kutatóintézete, 1525 Budapest Pf. 102. Díszfák, erdei fafajok és
A modern e-learning lehetőségei a tűzoltók oktatásának fejlesztésében. Dicse Jenő üzletfejlesztési igazgató
 A modern e-learning lehetőségei a tűzoltók oktatásának fejlesztésében Dicse Jenő üzletfejlesztési igazgató How to apply modern e-learning to improve the training of firefighters Jenő Dicse Director of
A modern e-learning lehetőségei a tűzoltók oktatásának fejlesztésében Dicse Jenő üzletfejlesztési igazgató How to apply modern e-learning to improve the training of firefighters Jenő Dicse Director of
Alkoholmentes italok 2002/3-4 51-53. Gyümölcslevek aktuális mikrobiológiai kérdései
 Gyümölcslevek aktuális mikrobiológiai kérdései Alkoholmentes italok 2002/3-4 51-53. Motto: A mikrobák alkalmazkodóképessége és változékonysága határtalan Tabajdiné dr. Pintér Veronika Összefoglaló A szerző
Gyümölcslevek aktuális mikrobiológiai kérdései Alkoholmentes italok 2002/3-4 51-53. Motto: A mikrobák alkalmazkodóképessége és változékonysága határtalan Tabajdiné dr. Pintér Veronika Összefoglaló A szerző
DI-, TETRA- ÉS HEXAPLOID TRITICUM FAJOK GENOMJAINAK ELEMZÉSE ÉS AZOK ÖSSZEHASONLÍTÁSA FLUORESZCENS IN SITU HIBRIDIZÁCIÓVAL
 Hagyomány és haladás a növénynemesítésben DI-, TETRA- ÉS HEXAPLOID TRITICUM FAJOK GENOMJAINAK ELEMZÉSE ÉS AZOK ÖSSZEHASONLÍTÁSA FLUORESZCENS IN SITU HIBRIDIZÁCIÓVAL VARGA MÓNIKA, MOLNÁR ISTVÁN ÉS KOVÁCS
Hagyomány és haladás a növénynemesítésben DI-, TETRA- ÉS HEXAPLOID TRITICUM FAJOK GENOMJAINAK ELEMZÉSE ÉS AZOK ÖSSZEHASONLÍTÁSA FLUORESZCENS IN SITU HIBRIDIZÁCIÓVAL VARGA MÓNIKA, MOLNÁR ISTVÁN ÉS KOVÁCS
Current Weed Control strategies in sorghum I
 Current Weed Control strategies in sorghum I In Pre-planting/pre-emergence chemichal fallow use of seed safeners residual herbicides efficacy uncertain high dependency on rains Current Weed Control strategies
Current Weed Control strategies in sorghum I In Pre-planting/pre-emergence chemichal fallow use of seed safeners residual herbicides efficacy uncertain high dependency on rains Current Weed Control strategies
Összefoglalás. Summary. Bevezetés
 A talaj kálium ellátottságának vizsgálata módosított Baker-Amacher és,1 M CaCl egyensúlyi kivonószerek alkalmazásával Berényi Sándor Szabó Emese Kremper Rita Loch Jakab Debreceni Egyetem Agrár és Műszaki
A talaj kálium ellátottságának vizsgálata módosított Baker-Amacher és,1 M CaCl egyensúlyi kivonószerek alkalmazásával Berényi Sándor Szabó Emese Kremper Rita Loch Jakab Debreceni Egyetem Agrár és Műszaki
Man-Made Plant diseases
 Fotó: Vajna László Mottó! CeCil Yarwood: Man-Made Plant diseases science, vol. 168. 218-200. 10 April 1970: (Cecil E. Yarwood, Department of Plant Pathology, University of California, Berkeley... ) 1907-1981
Fotó: Vajna László Mottó! CeCil Yarwood: Man-Made Plant diseases science, vol. 168. 218-200. 10 April 1970: (Cecil E. Yarwood, Department of Plant Pathology, University of California, Berkeley... ) 1907-1981
The role of aristolochene synthase in diphosphate activation
 Supporting information for The role of aristolochene synthase in diphosphate activation Juan A. Faraldos, Verónica González and Rudolf K. Allemann * School of Chemistry, Cardiff University, Main Building,
Supporting information for The role of aristolochene synthase in diphosphate activation Juan A. Faraldos, Verónica González and Rudolf K. Allemann * School of Chemistry, Cardiff University, Main Building,
Supplemental Information. RNase H1-Dependent Antisense Oligonucleotides. Are Robustly Active in Directing RNA Cleavage
 YMTHE, Volume 25 Supplemental Information RNase H1-Dependent Antisense Oligonucleotides Are Robustly Active in Directing RNA Cleavage in Both the Cytoplasm and the Nucleus Xue-Hai Liang, Hong Sun, Joshua
YMTHE, Volume 25 Supplemental Information RNase H1-Dependent Antisense Oligonucleotides Are Robustly Active in Directing RNA Cleavage in Both the Cytoplasm and the Nucleus Xue-Hai Liang, Hong Sun, Joshua
Bioinformatics: Blending. Biology and Computer Science
 Bioinformatics: Blending Biology and Computer Science MDNMSITNTPTSNDACLSIVHSLMCHRQ GGESETFAKRAIESLVKKLKEKKDELDSL ITAITTNGAHPSKCVTIQRTLDGRLQVAG RKGFPHVIYARLWRWPDLHKNELKHVK YCQYAFDLKCDSVCVNPYHYERVVSPGI DLSGLTLQSNAPSSMMVKDEYVHDFEG
Bioinformatics: Blending Biology and Computer Science MDNMSITNTPTSNDACLSIVHSLMCHRQ GGESETFAKRAIESLVKKLKEKKDELDSL ITAITTNGAHPSKCVTIQRTLDGRLQVAG RKGFPHVIYARLWRWPDLHKNELKHVK YCQYAFDLKCDSVCVNPYHYERVVSPGI DLSGLTLQSNAPSSMMVKDEYVHDFEG
A burgonya (Solanum tuberosum L.) sótűrő képességének és kalluszindukciójának vizsgálata in vitro módszerek alkalmazásával
 AGRÁRTUDOMÁNYI KÖZLEMÉNYEK, 25/18. A burgonya (Solanum tuberosum L.) sótűrő képességének és kalluszindukciójának vizsgálata in vitro módszerek alkalmazásával Pepó Pál Tóth Szilárd Debreceni Egyetem Agrártudományi
AGRÁRTUDOMÁNYI KÖZLEMÉNYEK, 25/18. A burgonya (Solanum tuberosum L.) sótűrő képességének és kalluszindukciójának vizsgálata in vitro módszerek alkalmazásával Pepó Pál Tóth Szilárd Debreceni Egyetem Agrártudományi
SZOFTVEREK A SORBANÁLLÁSI ELMÉLET OKTATÁSÁBAN
 SZOFTVEREK A SORBANÁLLÁSI ELMÉLET OKTATÁSÁBAN Almási Béla, almasi@math.klte.hu Sztrik János, jsztrik@math.klte.hu KLTE Matematikai és Informatikai Intézet Abstract This paper gives a short review on software
SZOFTVEREK A SORBANÁLLÁSI ELMÉLET OKTATÁSÁBAN Almási Béla, almasi@math.klte.hu Sztrik János, jsztrik@math.klte.hu KLTE Matematikai és Informatikai Intézet Abstract This paper gives a short review on software
A MUTATÓNÉVMÁSOK. A mutatónévmások az angolban is (mint a magyarban) betölthetik a mondatban
 A MUTATÓNÉVMÁSOK ez this /ðɪs/ az that /ðæt/ ezek these /ði:z/ azok those /ðəʊz / A mutatónévmások az angolban is (mint a magyarban) betölthetik a mondatban a) az ALANY szerepét - Ilyenkor (a már említett
A MUTATÓNÉVMÁSOK ez this /ðɪs/ az that /ðæt/ ezek these /ði:z/ azok those /ðəʊz / A mutatónévmások az angolban is (mint a magyarban) betölthetik a mondatban a) az ALANY szerepét - Ilyenkor (a már említett
A Fusarium solani-val szembeni ellenállóképesség vizsgálata különböző zöldborsó fajtákon és nemesítési kombinációkon
 A Fusarium solani-val szembeni ellenállóképesség vizsgálata különböző zöldborsó fajtákon és nemesítési kombinációkon Mendlerné Drienyovszki Nóra 1 Mándi Lajosné 2 Debreceni Egyetem Agrártudományi Centrum,
A Fusarium solani-val szembeni ellenállóképesség vizsgálata különböző zöldborsó fajtákon és nemesítési kombinációkon Mendlerné Drienyovszki Nóra 1 Mándi Lajosné 2 Debreceni Egyetem Agrártudományi Centrum,
Construction of a cube given with its centre and a sideline
 Transformation of a plane of projection Construction of a cube given with its centre and a sideline Exercise. Given the center O and a sideline e of a cube, where e is a vertical line. Construct the projections
Transformation of a plane of projection Construction of a cube given with its centre and a sideline Exercise. Given the center O and a sideline e of a cube, where e is a vertical line. Construct the projections
Planetary Nebulae, PN PNe PN
 2 2 e-mail: masaaki@oao.nao.ac.jp 719 0232 3037 5 Subaru Telescope, National Astronomical Observatory of Japan, 650 North A ohoku Place, Hilo, Hawaii 96720, U.S.A. e-mail: tajitsu@subaru.naoj.org 397 0101
2 2 e-mail: masaaki@oao.nao.ac.jp 719 0232 3037 5 Subaru Telescope, National Astronomical Observatory of Japan, 650 North A ohoku Place, Hilo, Hawaii 96720, U.S.A. e-mail: tajitsu@subaru.naoj.org 397 0101
A doktori értekezés tézisei. A növényi NRP fehérjék lehetséges szerepe a hiszton defoszforiláció szabályozásában, és a hőstressz válaszban.
 A doktori értekezés tézisei A növényi NRP fehérjék lehetséges szerepe a hiszton defoszforiláció szabályozásában, és a hőstressz válaszban. Bíró Judit Témavezető: Dr. Fehér Attila Magyar Tudományos Akadémia
A doktori értekezés tézisei A növényi NRP fehérjék lehetséges szerepe a hiszton defoszforiláció szabályozásában, és a hőstressz válaszban. Bíró Judit Témavezető: Dr. Fehér Attila Magyar Tudományos Akadémia
Stressz-élettani vizsgálatok aldo-keto reduktáz génekkel transzformált transzgenikus árpa vonalakon
 PhD értekezés tézisei Stressz-élettani vizsgálatok aldo-keto reduktáz génekkel transzformált transzgenikus árpa vonalakon Éva Csaba, okleveles biológus Témavezető: Tamás László PhD, habil. egyetemi docens
PhD értekezés tézisei Stressz-élettani vizsgálatok aldo-keto reduktáz génekkel transzformált transzgenikus árpa vonalakon Éva Csaba, okleveles biológus Témavezető: Tamás László PhD, habil. egyetemi docens
AGROTECHNIKAI TÉNYEZŐK HATÁSA A KULTÚRNÖVÉNYEKRE ÉS A GYOMOSODÁSRA
 PANNON EGYETEM GEORGIKON KAR NÖVÉNYVÉDELMI INTÉZET NÖVÉNYTERMESZTÉSI ÉS KERTÉSZETI TUDOMÁNYOK DOKTORI ISKOLA ISKOLA VEZETŐ DR. GÁBORJÁNYI RICHARD MTA DOKTORA AGROTECHNIKAI TÉNYEZŐK HATÁSA A KULTÚRNÖVÉNYEKRE
PANNON EGYETEM GEORGIKON KAR NÖVÉNYVÉDELMI INTÉZET NÖVÉNYTERMESZTÉSI ÉS KERTÉSZETI TUDOMÁNYOK DOKTORI ISKOLA ISKOLA VEZETŐ DR. GÁBORJÁNYI RICHARD MTA DOKTORA AGROTECHNIKAI TÉNYEZŐK HATÁSA A KULTÚRNÖVÉNYEKRE
Rotary District 1911 DISTRICT TÁMOGATÁS IGÉNYLŐ LAP District Grants Application Form
 1 A Future Vision pilot célja a Future Vision Plan (Jövőkép terv) egyszerűsített támogatási modelljének tesztelése, és a Rotaristák részvételének növelése a segélyezési folyamatokban. A teszt során a districteknek
1 A Future Vision pilot célja a Future Vision Plan (Jövőkép terv) egyszerűsített támogatási modelljének tesztelése, és a Rotaristák részvételének növelése a segélyezési folyamatokban. A teszt során a districteknek
Válasz Prof. Dr. Szabó János opponensi bírálatára
 Válasz Prof. Dr. Szabó János opponensi bírálatára Szeretném megköszönni Szabó Professzor Úr véleményét, építő jellegű kritikáját és a doktori értekezés elfogadására tett javaslatát. A különböző témakörökkel
Válasz Prof. Dr. Szabó János opponensi bírálatára Szeretném megköszönni Szabó Professzor Úr véleményét, építő jellegű kritikáját és a doktori értekezés elfogadására tett javaslatát. A különböző témakörökkel
Supporting Information
 Supporting Information Cell-free GFP simulations Cell-free simulations of degfp production were consistent with experimental measurements (Fig. S1). Dual emmission GFP was produced under a P70a promoter
Supporting Information Cell-free GFP simulations Cell-free simulations of degfp production were consistent with experimental measurements (Fig. S1). Dual emmission GFP was produced under a P70a promoter
Bird species status and trends reporting format for the period 2008-2012 (Annex 2)
 1. Species Information 1.1 Member State Hungary 1.2.2 Natura 2000 code A129 1.3 Species name Otis tarda 1.3.1 Sub-specific population 1.4 Alternative species name 1.5 Common name túzok 1.6 Season Breeding
1. Species Information 1.1 Member State Hungary 1.2.2 Natura 2000 code A129 1.3 Species name Otis tarda 1.3.1 Sub-specific population 1.4 Alternative species name 1.5 Common name túzok 1.6 Season Breeding
Trinucleotide Repeat Diseases: CRISPR Cas9 PacBio no PCR Sequencing MFMER slide-1
 Trinucleotide Repeat Diseases: CRISPR Cas9 PacBio no PCR Sequencing 2015 MFMER slide-1 Fuch s Eye Disease TCF 4 gene Fuchs occurs in about 4% of the US population. Leads to deteriorating vision without
Trinucleotide Repeat Diseases: CRISPR Cas9 PacBio no PCR Sequencing 2015 MFMER slide-1 Fuch s Eye Disease TCF 4 gene Fuchs occurs in about 4% of the US population. Leads to deteriorating vision without
Irodalomjegyzék. Emberi Jogok Egyetemes Nyilatkozata (angolul) elérhető: http://www.un.org/en/documents/udhr/ a letöltés napja: {2009.11.30.
 Irodalomjegyzék ENSZ (hivatalos) dokumentumok Emberi Jogok Egyetemes Nyilatkozata (angolul) elérhető: http://www.un.org/en/documents/udhr/ a letöltés napja: {2009.11.30.} Polgári és Politikai Jogok Egyezségokmánya
Irodalomjegyzék ENSZ (hivatalos) dokumentumok Emberi Jogok Egyetemes Nyilatkozata (angolul) elérhető: http://www.un.org/en/documents/udhr/ a letöltés napja: {2009.11.30.} Polgári és Politikai Jogok Egyezségokmánya
Road construction works
 Road construction works Info Version 2 Url http://com.mercell.com/permalink/45294693.aspx External tender id 246818-2014 Tender type Tender Document type Additional information Procurement procedure Open
Road construction works Info Version 2 Url http://com.mercell.com/permalink/45294693.aspx External tender id 246818-2014 Tender type Tender Document type Additional information Procurement procedure Open
PEST Hallgatói nemzetközi szakmai gyakorlat és mobilitás tapasztalatcseréjének projektje. Összefoglaló
 PEST Hallgatói nemzetközi szakmai gyakorlat és mobilitás tapasztalatcseréjének projektje Összefoglaló logo of the partner institutin Partnerek Lahti University of Applied Sciences Finnország MAC-Team aisbl
PEST Hallgatói nemzetközi szakmai gyakorlat és mobilitás tapasztalatcseréjének projektje Összefoglaló logo of the partner institutin Partnerek Lahti University of Applied Sciences Finnország MAC-Team aisbl
Crash Course in Omics Terminology, Concepts & Data Types
 Crash Course in Omics Terminology, Concepts & Data Types 13 th Annual EuPathDB Workshop Jessie Kissinger June 17th, 2018 FungiDB MicrosporidiaDB AmoebaDB PlasmoDB ToxoDB CryptoDB PiroplasmaDB TrichDB GiardiaDB
Crash Course in Omics Terminology, Concepts & Data Types 13 th Annual EuPathDB Workshop Jessie Kissinger June 17th, 2018 FungiDB MicrosporidiaDB AmoebaDB PlasmoDB ToxoDB CryptoDB PiroplasmaDB TrichDB GiardiaDB
Szalay Sándor a talaj-növény rendszerről Prof. Dr. Győri Zoltán intézetigazgató, az MTA doktora a DAB alelnöke
 Debreceni Egyetem Agrár- és Műszaki Tudományok Centruma Mezőgazdaságtudományi Kar Élelmiszertudományi, Minőségbiztosítási és Mikrobiológiai Intézet Szalay Sándor a talaj-növény rendszerről Prof. Dr. Győri
Debreceni Egyetem Agrár- és Műszaki Tudományok Centruma Mezőgazdaságtudományi Kar Élelmiszertudományi, Minőségbiztosítási és Mikrobiológiai Intézet Szalay Sándor a talaj-növény rendszerről Prof. Dr. Győri
DALI TERÁPIÁVAL SZERZETT KEZDETI TAPASZTALATAINK
 University of Debrecen Research University DALI TERÁPIÁVAL SZERZETT KEZDETI TAPASZTALATAINK Harangi Mariann, Paragh György, Balla József Debreceni Egyetem Orvos- és Egészségtudományi Centrum, Belgyógyászati
University of Debrecen Research University DALI TERÁPIÁVAL SZERZETT KEZDETI TAPASZTALATAINK Harangi Mariann, Paragh György, Balla József Debreceni Egyetem Orvos- és Egészségtudományi Centrum, Belgyógyászati
Pannon Egyetem Vegyészmérnöki- és Anyagtudományok Doktori Iskola
 Pannon Egyetem Vegyészmérnöki- és Anyagtudományok Doktori Iskola A KÉN-HIDROGÉN BIOKATALITIKUS ELTÁVOLÍTÁSA BIOGÁZBÓL SZUSZPENDÁLT SZAKASZOS ÉS RÖGZÍTETT FÁZISÚ FOLYAMATOS REAKTORBAN, AEROB ÉS MIKROAEROB
Pannon Egyetem Vegyészmérnöki- és Anyagtudományok Doktori Iskola A KÉN-HIDROGÉN BIOKATALITIKUS ELTÁVOLÍTÁSA BIOGÁZBÓL SZUSZPENDÁLT SZAKASZOS ÉS RÖGZÍTETT FÁZISÚ FOLYAMATOS REAKTORBAN, AEROB ÉS MIKROAEROB
Excel vagy Given-When-Then? Vagy mindkettő?
 TESZT & TEA BUDAPEST AGILE MEETUP Pénzügyi számítások automatizált agilis tesztelése: Excel vagy Given-When-Then? Vagy mindkettő? NAGY GÁSPÁR TechTalk developer coach Budapest, 2014 február 6. SpecFlow
TESZT & TEA BUDAPEST AGILE MEETUP Pénzügyi számítások automatizált agilis tesztelése: Excel vagy Given-When-Then? Vagy mindkettő? NAGY GÁSPÁR TechTalk developer coach Budapest, 2014 február 6. SpecFlow
(11) Lajstromszám: E 003 759 (13) T2 EURÓPAI SZABADALOM SZÖVEGÉNEK FORDÍTÁSA
 !HU00000379T2! (19) HU (11) Lajstromszám: E 003 79 (13) T2 MAGYAR KÖZTÁRSASÁG Magyar Szabadalmi Hivatal EURÓPAI SZABADALOM SZÖVEGÉNEK FORDÍTÁSA (21) Magyar ügyszám: E 0 728073 (22) A bejelentés napja:
!HU00000379T2! (19) HU (11) Lajstromszám: E 003 79 (13) T2 MAGYAR KÖZTÁRSASÁG Magyar Szabadalmi Hivatal EURÓPAI SZABADALOM SZÖVEGÉNEK FORDÍTÁSA (21) Magyar ügyszám: E 0 728073 (22) A bejelentés napja:
Néhány folyóiratkereső rendszer felsorolása és példa segítségével vázlatos bemutatása Sasvári Péter
 Néhány folyóiratkereső rendszer felsorolása és példa segítségével vázlatos bemutatása Sasvári Péter DOI: http://doi.org/10.13140/rg.2.2.28994.22721 A tudományos közlemények írása minden szakma művelésének
Néhány folyóiratkereső rendszer felsorolása és példa segítségével vázlatos bemutatása Sasvári Péter DOI: http://doi.org/10.13140/rg.2.2.28994.22721 A tudományos közlemények írása minden szakma művelésének
A forrás pontos megnevezésének elmulasztása valamennyi hivatkozásban szerzői jogsértés (plágium).
 A szakirodalmi idézések és hivatkozások rendszere és megadásuk szabályai A bibliográfia legfontosabb szabályai Fogalma: Bibliográfiai hivatkozáson azoknak a pontos és kellően részletezett adatoknak az
A szakirodalmi idézések és hivatkozások rendszere és megadásuk szabályai A bibliográfia legfontosabb szabályai Fogalma: Bibliográfiai hivatkozáson azoknak a pontos és kellően részletezett adatoknak az
FELADATMEGOLDÁSI SZOKÁSAINAK VIZSGÁLATA. Baranyai Tünde
 Volume 3, Number 1, 2013 3. kötet, 1. szám, 2013 A SZATMÁRNÉMETI TANÍTÓ- ÉS ÓVÓKÉPZŐS HALLGATÓK FELADATMEGOLDÁSI SZOKÁSAINAK VIZSGÁLATA THE EXAMINATION OF TEACHER TRAINING COLLEGE STUDENTS PROBLEM-SOLVING
Volume 3, Number 1, 2013 3. kötet, 1. szám, 2013 A SZATMÁRNÉMETI TANÍTÓ- ÉS ÓVÓKÉPZŐS HALLGATÓK FELADATMEGOLDÁSI SZOKÁSAINAK VIZSGÁLATA THE EXAMINATION OF TEACHER TRAINING COLLEGE STUDENTS PROBLEM-SOLVING
A teszt a következő diával indul! The test begins with the next slide!
 A teszt a következő diával indul! The test begins with the next slide! A KÖVETKEZŐKBEN SZÁMOZOTT KÉRDÉSEKET VAGY KÉPEKET LÁT SZÁMOZOTT KÉPLETEKKEL. ÍRJA A SZÁMOZOTT KÉRDÉSRE ADOTT VÁLASZT, VAGY A SZÁMOZOTT
A teszt a következő diával indul! The test begins with the next slide! A KÖVETKEZŐKBEN SZÁMOZOTT KÉRDÉSEKET VAGY KÉPEKET LÁT SZÁMOZOTT KÉPLETEKKEL. ÍRJA A SZÁMOZOTT KÉRDÉSRE ADOTT VÁLASZT, VAGY A SZÁMOZOTT
 ľ ó Ü ó ĺĺ ľ ľ í í í ó ó ó ó í Ü ö ĺ ó ó í í í ó ü ü Ü ö ü ü í í ö ó óó ĺ í ű ö Ü ö ö ű ó Ĺ ö Á Á ű í ű ó ü ú ű Ü ö ű ó ú ó ó ĺ ó í ö í ó ó ö ű ö í í ö ó ó ó Ú ĺ ó ó ó Ö ó ď í ö ű ó ę ó ű ö í í ó í Ü í
ľ ó Ü ó ĺĺ ľ ľ í í í ó ó ó ó í Ü ö ĺ ó ó í í í ó ü ü Ü ö ü ü í í ö ó óó ĺ í ű ö Ü ö ö ű ó Ĺ ö Á Á ű í ű ó ü ú ű Ü ö ű ó ú ó ó ĺ ó í ö í ó ó ö ű ö í í ö ó ó ó Ú ĺ ó ó ó Ö ó ď í ö ű ó ę ó ű ö í í ó í Ü í
A BURGONYA Y VÍRUS HC-Pro ÉS A BURGONYA StubGAL83 FEHÉRJÉJÉNEK KAPCSOLATA
 226 NÖVÉNYVÉDELEM 46 (5), 2010 A BURGONYA Y VÍRUS HC-Pro ÉS A BURGONYA StubGAL83 FEHÉRJÉJÉNEK KAPCSOLATA Beczner Farkas, Antal Ferenc és Bánfalvi Zsófia Mezôgazdasági Biotechnológiai Kutatóközpont, 2100
226 NÖVÉNYVÉDELEM 46 (5), 2010 A BURGONYA Y VÍRUS HC-Pro ÉS A BURGONYA StubGAL83 FEHÉRJÉJÉNEK KAPCSOLATA Beczner Farkas, Antal Ferenc és Bánfalvi Zsófia Mezôgazdasági Biotechnológiai Kutatóközpont, 2100
ERŐMŰI SZERKEZETI ELEMEK ÉLETTARTAM GAZ- DÁLKODÁSÁNAK TÁMOGATÁSA A TÖRÉSMECHANI- KA ALKALMAZÁSÁVAL
 Miskolci Egyetem, Multidiszciplináris tudományok, 1. kötet (2011) 1. szám, pp. 213-220. ERŐMŰI SZERKEZETI ELEMEK ÉLETTARTAM GAZ- DÁLKODÁSÁNAK TÁMOGATÁSA A TÖRÉSMECHANI- KA ALKALMAZÁSÁVAL Lukács János egyetemi
Miskolci Egyetem, Multidiszciplináris tudományok, 1. kötet (2011) 1. szám, pp. 213-220. ERŐMŰI SZERKEZETI ELEMEK ÉLETTARTAM GAZ- DÁLKODÁSÁNAK TÁMOGATÁSA A TÖRÉSMECHANI- KA ALKALMAZÁSÁVAL Lukács János egyetemi
EXKLUZÍV AJÁNDÉKANYAGOD EXTRA BEÉGETŐS MONDATOK - HOGY NE ÉGJ BE : -)
 EXTRA BEÉGETŐS MONDATOK - HOGY NE ÉGJ BE : -) Égesd be a mondatokat az agyadba! Ez a legjobb módja annak, hogy megragadjanak egyben a mondatok és amikor beszélned kell, akkor már csak át kell húznod más
EXTRA BEÉGETŐS MONDATOK - HOGY NE ÉGJ BE : -) Égesd be a mondatokat az agyadba! Ez a legjobb módja annak, hogy megragadjanak egyben a mondatok és amikor beszélned kell, akkor már csak át kell húznod más
(AD) β (A ) (2) ACDP ACDP ACDP APP. APP-C99 γ (3) A 40 A 42 A A A A 42/A 40. in vivo A A A A
 (AD) β (A) A AAPP APP-C99 γ A A40 A42 A AA42/A40 γ A A γ AD A A A A APP APP APP A A A A A A (1)AD A42 A AD AD (2) ACDP AACDP ACDP A A ACDP (3) (1)A in vivo A APP Gray and Whittaker 1962; Perez-Otano et
(AD) β (A) A AAPP APP-C99 γ A A40 A42 A AA42/A40 γ A A γ AD A A A A APP APP APP A A A A A A (1)AD A42 A AD AD (2) ACDP AACDP ACDP A A ACDP (3) (1)A in vivo A APP Gray and Whittaker 1962; Perez-Otano et
INRA, UMR 1388 Génétique, Physiologie et Systèmes d Elevage, F Castanet-Tolosan, France
 Supplementary information Non integrative strategy decreases chromosome instability and improves endogenous pluripotency genes reactivation in porcine induced pluripotent stem cells Annabelle Congras 1,
Supplementary information Non integrative strategy decreases chromosome instability and improves endogenous pluripotency genes reactivation in porcine induced pluripotent stem cells Annabelle Congras 1,
DOKTORI ÉRTEKEZÉS TÉZISEI AZ OPPORTUNISTA HUMÁNPATOGÉN CANDIDA PARAPSILOSIS ÉLESZTŐGOMBA ELLENI TERMÉSZETES ÉS ADAPTÍV IMMUNVÁLASZ VIZSGÁLATA
 DOKTORI ÉRTEKEZÉS TÉZISEI AZ OPPORTUNISTA HUMÁNPATOGÉN CANDIDA PARAPSILOSIS ÉLESZTŐGOMBA ELLENI TERMÉSZETES ÉS ADAPTÍV IMMUNVÁLASZ VIZSGÁLATA TÓTH ADÉL TÉMAVEZETŐ: DR. GÁCSER ATTILA TUDOMÁNYOS FŐMUNKATÁRS
DOKTORI ÉRTEKEZÉS TÉZISEI AZ OPPORTUNISTA HUMÁNPATOGÉN CANDIDA PARAPSILOSIS ÉLESZTŐGOMBA ELLENI TERMÉSZETES ÉS ADAPTÍV IMMUNVÁLASZ VIZSGÁLATA TÓTH ADÉL TÉMAVEZETŐ: DR. GÁCSER ATTILA TUDOMÁNYOS FŐMUNKATÁRS
CURRICULUM VITAE FODOR ISTVÁN
 CURRICULUM VITAE FODOR ISTVÁN Klebersberg K. St. 3 8237, Tihany Hungary Phone: +36 30 608 3553 e-mail: fodor.istvan@okologia.mta.hu ; prof1122332@gmail.com EDUCATION - 2007-2011 Czuczor Gergely High School
CURRICULUM VITAE FODOR ISTVÁN Klebersberg K. St. 3 8237, Tihany Hungary Phone: +36 30 608 3553 e-mail: fodor.istvan@okologia.mta.hu ; prof1122332@gmail.com EDUCATION - 2007-2011 Czuczor Gergely High School
GENERATÍV TEST (VIRÁGOS NÖVÉNYEK)
 GENERATÍV TEST (VIRÁGOS NÖVÉNYEK) MITÓZIS IVAROS szaporító szervek hím női (antheridium) (archegonium) IVARTALAN szaporító szervek Sporangium 2n n MEIÓZIS (2n 2n) MITÓZIS IVARTALAN SZAPORÍTÓSZERVEK (2n
GENERATÍV TEST (VIRÁGOS NÖVÉNYEK) MITÓZIS IVAROS szaporító szervek hím női (antheridium) (archegonium) IVARTALAN szaporító szervek Sporangium 2n n MEIÓZIS (2n 2n) MITÓZIS IVARTALAN SZAPORÍTÓSZERVEK (2n
Napraforgó fehérjék biofizikai tulajdonságainak jellemzése és emészthetőségének vizsgálata in vitro rendszerben
 PhD értekezés tézisei Napraforgó fehérjék biofizikai tulajdonságainak jellemzése és emészthetőségének vizsgálata in vitro rendszerben Berecz Bernadett Témavezető: Prof. emer. Láng Ferenc Konzulens: Dr.
PhD értekezés tézisei Napraforgó fehérjék biofizikai tulajdonságainak jellemzése és emészthetőségének vizsgálata in vitro rendszerben Berecz Bernadett Témavezető: Prof. emer. Láng Ferenc Konzulens: Dr.
Effect of the different parameters to the surface roughness in freeform surface milling
 19 November 0, Budapest Effect of the different parameters to the surface roughness in freeform surface milling Balázs MIKÓ Óbuda University 1 Abstract Effect of the different parameters to the surface
19 November 0, Budapest Effect of the different parameters to the surface roughness in freeform surface milling Balázs MIKÓ Óbuda University 1 Abstract Effect of the different parameters to the surface
Önkiszolgáló BI infrastruktúra az adatvezérelt teljesítménymenedzsmentben
 Önkiszolgáló BI infrastruktúra az adatvezérelt teljesítménymenedzsmentben Microsoft Future Decoded 2018.03.21. Krizsanovich Péter Ügyvezető igazgató, Stratégiai-, Tervezési és Controlling Igazgatóság Horváth
Önkiszolgáló BI infrastruktúra az adatvezérelt teljesítménymenedzsmentben Microsoft Future Decoded 2018.03.21. Krizsanovich Péter Ügyvezető igazgató, Stratégiai-, Tervezési és Controlling Igazgatóság Horváth
Lesson 1 On the train
 Let's Learn Hungarian! Lesson notes Lesson 1 On the train Dialogue for Lesson 1 (formal speech): Guard: Jó napot kívánok. Jó napot. Guard: Az útlevelét, kérem. Tessék. Guard: Köszönöm. Hmmmm, amerikai?
Let's Learn Hungarian! Lesson notes Lesson 1 On the train Dialogue for Lesson 1 (formal speech): Guard: Jó napot kívánok. Jó napot. Guard: Az útlevelét, kérem. Tessék. Guard: Köszönöm. Hmmmm, amerikai?
INHIBITION OF ETHYLENE BIOSYNTHESIS IN APPLE
 INHIBITION OF ETHYLENE BIOSYNTHESIS IN APPLE AND TAXONOMICAL COMPARISON OF DIFFERENT FESTUCA SPECIES USING MOLECULAR METHODS Ph.D. thesis Zsolt Galli Gödöllő 2004 Ph.D. School: Scientific Code: Ph.D. Program:
INHIBITION OF ETHYLENE BIOSYNTHESIS IN APPLE AND TAXONOMICAL COMPARISON OF DIFFERENT FESTUCA SPECIES USING MOLECULAR METHODS Ph.D. thesis Zsolt Galli Gödöllő 2004 Ph.D. School: Scientific Code: Ph.D. Program:
TÁMOPͲ4.2.2.AͲ11/1/KONVͲ2012Ͳ0029
 AUTOTECH Jármipari anyagfejlesztések: célzott alapkutatás az alakíthatóság, hkezelés és hegeszthetség témaköreiben TÁMOP4.2.2.A11/1/KONV20120029 www.autotech.unimiskolc.hu ANYAGSZERKEZETTANI ÉS ANYAGTECHNOLÓGIAI
AUTOTECH Jármipari anyagfejlesztések: célzott alapkutatás az alakíthatóság, hkezelés és hegeszthetség témaköreiben TÁMOP4.2.2.A11/1/KONV20120029 www.autotech.unimiskolc.hu ANYAGSZERKEZETTANI ÉS ANYAGTECHNOLÓGIAI
Receptor Tyrosine-Kinases
 Receptor Tyrosine-Kinases MAPkinase pathway PI3Kinase Protein Kinase B pathway PI3K/PK-B pathway Phosphatidyl-inositol-bisphosphate...(PI(4,5)P 2...) Phosphatidyl-inositol-3-kinase (PI3K) Protein kinase
Receptor Tyrosine-Kinases MAPkinase pathway PI3Kinase Protein Kinase B pathway PI3K/PK-B pathway Phosphatidyl-inositol-bisphosphate...(PI(4,5)P 2...) Phosphatidyl-inositol-3-kinase (PI3K) Protein kinase
H-1152 Budapest, Szentmihályi út 171. +36 (1) 414 7364 www.chinacham.hu
 KÍNAI MAGYAR KERESKEDELEM-FEJLESZTÉSI ÉS MARKETING KONFERENCIA A Minisztérium, Külkereskedelmi Osztálya, a Kínai Népköztársaság Kereskedelmi Minisztérium Külkereskedelem- fejlesztési Hivatala, a ChinaCham
KÍNAI MAGYAR KERESKEDELEM-FEJLESZTÉSI ÉS MARKETING KONFERENCIA A Minisztérium, Külkereskedelmi Osztálya, a Kínai Népköztársaság Kereskedelmi Minisztérium Külkereskedelem- fejlesztési Hivatala, a ChinaCham
A vadgazdálkodás minősítése a Dél-dunántúli régióban
 Acta Oeconomica Kaposváriensis (2007) Vol 1 No 1-2, 197-204 Kaposvári Egyetem, Gazdaságtudományi Kar, Kaposvár Kaposvár University, Faculty of Economic Science, Kaposvár A vadgazdálkodás minősítése a Dél-dunántúli
Acta Oeconomica Kaposváriensis (2007) Vol 1 No 1-2, 197-204 Kaposvári Egyetem, Gazdaságtudományi Kar, Kaposvár Kaposvár University, Faculty of Economic Science, Kaposvár A vadgazdálkodás minősítése a Dél-dunántúli
MELLÉKLET / ANNEX. EU MEGFELELŐSÉGI NYILATKOZAT-hoz for EU DECLARATION OF CONFORMITY
 TRACON BUDAPEST KFT. TRACON BUDAPEST LTD. 2120 DUNAKESZI, PALLAG U. 23. TEL.: (27) 540-000, FAX: (27) 540-005 WWW.TRACON.HU EU MEGFELELŐSÉGI NYILATKOZAT, a 79/1997. (XII. 31.) IKIM számú és a 374/2012.
TRACON BUDAPEST KFT. TRACON BUDAPEST LTD. 2120 DUNAKESZI, PALLAG U. 23. TEL.: (27) 540-000, FAX: (27) 540-005 WWW.TRACON.HU EU MEGFELELŐSÉGI NYILATKOZAT, a 79/1997. (XII. 31.) IKIM számú és a 374/2012.
BIZTONSÁGI ADATLAP. 1. Az anyag/keverék és a vállalat/vállalkozás azonosítása PV4421 FL-PGC1A PEPTIDE, 100 UM
 BIZTONSÁGI ADATLAP Az anyag/készítmény azonosítása 1. Az anyag/keverék és a vállalat/vállalkozás azonosítása Termék neve FL-PGC1A PEPTIDE, 100 UM A vállalat/vállalkozás azonosítása Life Technologies 5791
BIZTONSÁGI ADATLAP Az anyag/készítmény azonosítása 1. Az anyag/keverék és a vállalat/vállalkozás azonosítása Termék neve FL-PGC1A PEPTIDE, 100 UM A vállalat/vállalkozás azonosítása Life Technologies 5791
