Gén térképezés, génizolálás Dr. Karsai Ildikó, tudományos tanácsadó
|
|
|
- Máté Kelemen
- 9 évvel ezelőtt
- Látták:
Átírás
1 Gén térképezés, génizolálás Dr. Karsai Ildikó, tudományos tanácsadó Karsai Ildikó
2 Gén azonosítás tulajdonságok fenotípusos megnyilvánulásában meghatározó szerepet játszó genetikai komponensek, szabályozó mechanizmusok, biokémiai folyamatok megértése diagnosztikai markerek, módszerek kidolgozása természetes variabilitás tesztelése vad és termesztett fajtákon belül, valamint a rokon fajok csoportjában hasznos gének, gén allélek beépítése a nemesítési alapanyagokba marker szelekcióval egybekötött hagyományos keresztezések során gén transzformációs technikák alkalmazásával
3 Gén azonosítás főbb módszerei marker kapcsoltsági térképek és pozicionális klónozás Két szülős térképező populációk Széles genetikai bázist képviselő fajtakör jelölt gén megközelítés Genom pozíció függő stratégiák Összehasonlító genomikai stratégiák mesterséges genetikai variáció (indukált mutációs populációk) génfunkció és fenotípus közti kapcsolat Gén expressziós mintázatok elemzése
4 Gén térképezés főbb módszerei Két szülős térképező populációk marker kapcsoltsági térképek, QTL elemzés Széles genetikai bázist képviselő fajtakör LD térképezés, teljes genomot lefedő asszociációs elemzések
5 Két szülős populációk marker kapcsoltsági térképei Térképező populáció Molekuláris marker technikák Számítógépes térképező programok
6 Térképező populáció Keresztezési partnerek kiválasztása: távoli vagy speciális Populáció típusa: hasadó F2 populáció homozigóta populációk (DH, RIL, SSD) egyéb speciális populációk (NIL, egy kromoszómára rekombináns stb.) Populáció mérete Marker kapcsoltsági térképek DNS izolálás markeres vizsgálatok Genom szintű különbségek
7 Marker kapcsoltsági térképek Molekuláris marker technikák RFLP PCR alapú markerek ismeretlen kromoszómális elhelyezkedésű: RAPD, AFLP ismert kromoszómális elhelyezkedésű: SSR, STS funkcionális markerek: EST, SNP, Tillling HTP marker technikák (Illumina platform, DArT) Új generációs szekvenálás
8 Marker kapcsoltsági térképek Számítógépes térképező programok marker térkép készítés (MAPMAKER, GMENDEL, JOINMAP, GenStat) QTL elemző programok (MAPMAKER, WINQTL, JOINMAP, GenStat) Egyszerű intervallum térképezés (SIM) Összetett intervallum térképezés (CIM) Egy tulajdonság + egy környezet több tulajdonság + több környezet Tulajdonság komponens kölcsönhatás elemzés, QTL x környezet kölcsönhatás elemzés Fenotípusos vizsgálatok ismételhetőség, pontosság Fenomika
9 Marker kapcsoltsági térképek alkalmazási területei Genomot teljesen lefedő molekuláris marker térkép készítése Összehasonlító térképezés rokon fajok között, fajon belül különböző populációk között Genom szerveződés makro és mikro kolinearitás kromoszóma átrendeződések intergénikus és génklaszter szakaszok azonosítása
10 Marker kapcsoltsági térképek alkalmazási területei Gének azonosítása, fenotípusos hatásuk vizsgálata finom térképezés, pozicionális klónozás modell növény ismert génszekvenciája alapján Egyszerű és összetett tulajdonságok genetikai elemzése lókuszok számának és helyének azonosítása Adott lókusz szerepének elemzése; QTL dinamika, tulajdonság komponensek vizsgálata Allél gyakoriságok vizsgálata szélesebb genetikai bázison Marker szelekció
11 DNS markerek, mint ujjlenyomatok Markerek (M1 ) M1 M2 M3 N1 N2 N5 N10 N15 A szülő B szülő A x B keresztezés utódpopulációjának növényegyedei (N1 )
12 QTL elemzés marker kapcsoltsági térkép alkalmazásával Hasadó populáció Genotípusos jellemzés Fenotípusos jellemzés szülők AAABBAB ABBABABB BBBAA A B Adat mátrix *mst1011 ABAAABBBABBBABBBBBABBABBA *mst1012 ABBAABAABBAABBBBBBABBBBBB *mst102 AAABBBAABBABBBABBBBABBABB *wg4642 ABBAABAABBAABBBBBBABBBABB *bcd1752 ABBAAABBBA---ABBABBBABABAA *wg541 AA-BBBA---ABBABBABBABABAABA *v8h *uv16h *v16h *uv24h
13 QTL elemzés marker kapcsoltsági térkép alkalmazásával Marker kapcsoltsági térkép elkészítése 8> make chrom osom e chrom 2h chrom 4h chrom5h chromosomes 1 6 > adefined: s schrom2h i g n chrom framework: 2h chrom4h chrom5h H v M > - t a h n r c e h e o p r o l i o n c t u s o n c h r o m 2 h... c a n n o t r e - a s s i g n B m a g a n c h o r l o c u s o n c h r o m 2 h... c a n n o t r e - a s s i g n 9> seq hvm 36 bm ag125 B m a c Placing a n c h o r at l olog-likelihood c u s o n c h r o mthreshold 4 h... c a n n o t r e - a s s i g n sequence #1= hvm36 Markers H v M 6 7 bmag125 ' t r i p- l Start: ae nec rhr o r 9 l do 5 ce u1 t es 11 Distance c ot in 12 o n c ' h ri s o mo n 4. h... c a n n o t r e - a s s i g n Npt-2: (10) B m a g No a nabg358 unique c h o r placements l o c u s o20.6 n cfor h rcm o6 mremaining 5 h... c a nmarkers n o t r e - a s s i g n 10> anchor A Bchrom G h - a n c h o r l o c u s o n c h r o m 5 h... c a n n o t r e - a s s i g n HvM 36 A- Banchor G 8 locus - 5 Map: a s sopb162 on i g nchrom e d t o 2h c h r o m2.4 2 h cm a t L O D Markers Distance Bmag125 A - Banchor C locus 4 - a shvm36 s9 on i g Bmc222a nchrom2h e d t o c h r o m 2cM h a t L O D chromosome O P Hchrom h - aanchor(s): s s5 i g nopb162 e d t o HvM c h r 36 o m8.2 Bm 2 h ag125 a t L O D O P B a soph121 1 ABG8 s i g n e d t o c h r o m 2cM h a t L O D OPP102b 6.3 cm A B G a sabc311 s i gopa192 n e d t o c h r o m 2cM h a t L O D A B G a s12 s i gopq10 n e d t o c h r o m 2 h a t L O D A B G a s s i g n e d t o c h r o m 2 h a t L O D L i n k a g e G r o u p s a t m i n L O D , m a x D i s t a n c e T r i p l e t c r i t e r i a : L O D , M a x - D i s t , # L i n k a g e s 2 c o u n t i n g l i n k e d t r i p l e t s i n 2 l i n k a g e g r o u p s l o g - l i k e l i h o o d d i f f e r e n c e s c o u n t m a r k e r s a - b - c b - a - c a - c - b 1 : A B G 8 A B C O P H : A B G 8 A B C H v M : A B G 8 A B C O P B : A B G 8 A B C A B G : A B G 8 A B C A B G : A B G 8 A B C A B G : A B G 8 A B C B m c a markers 0 log-likelihood= ABG cm : A B G 8 A B C O P P b Markers placed relative to above map: 910 : AOPP102b B G 8 A9 B C O PcM 10 A : A B G 8 :-21-:--8-:-11-:--6-:-11-: A B C O P Q OPA *.:.**.:...:...:...:...: cm 7 2.*.:.**.:...:...:...:...: OPQ :.**.:..*.:...:...:...: :..*.:.**.:...:...:...: :...:.**.:..*.:...:...: cm 9 markers :...:.**.:..*.:...:...: Placing at log-likelihood threshold Start: log-likelihood= Npt-4: 9 5 (2) Npt-4: 9 5 (4) Npt-4: (3)
14 DK1DK2DK3DK4DK7DK8DK9 DK10 DK11 DK12 DK13 DK14 DK15 DK16 DK17 DK18 DK19 DK20 DK21 DK22 DK24 DK25 DK26 DK27 DK28 DK29 DK30 DK31 DK32 DK35 DK36 DK37 DK38 DK39 *mst102 A B A A A B B B A A B B A B B B A B B B A B B A B B A A B A A B A B *Bmac144d A B A A A B B B A B B A B B B B B B B B A B B A B B A A B A A B A B TCGA96 A B A A A B B B A B B A B B B A B B B B A B B A B B A A B A A B A B *MWG938 A B A A B B B B A B B A B B B A B B B B A B B A B B A A B A A B A B AGAT166/162 A B A A B B B B A B B A B B B A B B B B A B B A B B A A B A A B A B GCTC58 A B A A B B B B A B B A B B B A B A B B A B B A B B A A B A B B B B OPK16 A B A A B B B B A B B A B B B A B A B B A B B A B B A A B A B B B B *abc156.1 A B A A B B B B A B B A B B B A B A B B A B B A B B A A B A B B B B *BMAC213 A B A A B B B B A B B A B B B A B A B B A B B A B B A A B A B B B B GAAT76 A B A A B B B B A B B A B B B A B A B B A B B A B B A A B A B A B B *OPE171 A B A A B B B B A B B - B B B A B A B B A B B A B B B A B A B A B B GAAT414 A B A B B B B B A B B A B B B A B A B B A B B A B B B A B A B A B A *OPO18 A B A B B B B B A B B A B B B A B A B B A B B A B B B A B A B A B A *OPB16-1 A B A B B B B B A A B A B B B A B A A A A B B A B B B A B A B A B A *Bmag211 A B A B B B B B B A B A B B B A B A A A A B B A B B B A B A B A B A *OPS16 A B A B B B B B B A B A B B B A B A A A A B B A B B A A B A B A B A GCGA428 A B A B B B B B B A B A B B B A B A A A A B B A B B A A B A B A B A *Ebmac560a A B A B B B B B B A B A B B B A B A A A A B B A B B A A B A B A B A *OPB41 A B A B B B B B B A B A B B B A B A A A A B B A B B A A B A B A B A GAAT550 A B A B B B B B B A B A B B B A B A A A A B B A B B A A B A B A B A AGGT290 A B A B B B B B B A B A B B B B B A A A A B B A B B A A B A B A B A TCGA119 A B A B B B B B B A B A B B B B B A A A A B B A B B A A B A B A B A *OPT152 A B A B B B B B B A B A B B B B B A A A A B B A B B A A B A B A B A GAAT510 A B A B B B B B B A B A B B B B B A A B A B A A A A A B A A B B A A AGAT51 A B A B A A A B B A B A B B B B B B A B B B A A A A A B A A A B A B *Bmac144a A B A B A A A A B A B A B B B B B B A B B B A A A A A B A A A B A B GCGA269/270 A B A B A A A A B A B A B B B B B B A B B B A A A A A B A A A B A B *ABC152b A B A B A A A A B A B A B B B B B B A B B B A A A A A B A A A B A B *HvHVA1 A B B B A A A A B A B A B B B B B B A B B B B A A A A B A A A B B B *BCD265c A B B B A A A A B A B A B B B B B B A B B B B A A A A B A A A B B B GCGT132 A B B B A A A A B A B A B B B B B B B B B B B A A A A B A A A B B B *OPP142 A B B B A A A A B A B A B B B B B B B B B B B A A A A B A A A B B A
15 mst102 0 Bmac144d 4 TCGA96 6 MWG938 8 AGAT GCTC58 15 OPK16 17 abc Bmac GAAT76 24 OPE GAAT OPO18 31 OPB Bmag OPS16 50 GCGA Ebmac560a 54 OPB GAAT AGGT TCGA OPT GAAT AGAT51 99 Bmac144a 100 GCGA ABC152b 109 HvHVA1 125 BCD265c 126 GCGT OPP OPX H Bmac222a 0 ABG459 1 ABG HvM36 25 ABC GAGT ABG8 31 GCGA52 37 GAGA72 39 TCTC GAAT OPA AGAT AGAT GAGA GAGT62 58 GCGT abg459a 101 Bmac Bmac144b 108 bcd175b 109 Bmag GAGT TCGT TCAT TCTC Bmag003c 127 GCAT OPG H OPD07 0 OPS141 6 OPO GATC GCGA TCAT GAGA TCTC AGTC GAAT GCAT OPI AGAT OPS Bmag384b 75 TCTC GCGT OPK HvM60 99 AGAT AGAT GAAT AGGT GCGT OPH ABG4 152 TCAT Bmag GCAT TCTC OPS Hvm GAGA OPH Hvm GCGT TCAT GCAT GCGA TCGA ABC AGTC GAAT H HvPhyA 0 GCAT76 8 TCAT300 TCAT77 9 AGAT HvPhyB 21 CAB 22 Bmac30 24 AGAT Bmag Bmag173a 33 Bmac OPJ15 40 TCAT OPU AGAT46 59 ABG abg54 64 AGAT GCGT GCAT AGAT HvM67 85 OPS VRN-H2 HvSnf2 92 AGGT Hdamyb 100 4H OPT151 0 GCAT50 7 Bmag136b 8 GAGA556 9 Bmag GATC AGTC AGGA GAGA AGAT42 32 GAGA58 33 Bmag abc AGAT TCTC Bmag223 mr 41 GATC abg459b 51 OPH GCGT GCGA TCAT abc306d 90 OPK TCTC ABG AGTC Bmag381b 112 gms HvPhyC VRN-H1 117 abc TCGT GCAT ABC H BMAC316 0 AGGT256 2 TCTC386 9 AGAT ABC152c 23 Bmag Bmag21a 51 GAGA GCGT Bmag OPH Bmag HvCry1a HvCry2 GCGA48 71 AGGA62 72 OPI OPA abc306b 80 abc306c 81 abc AGGT Bmag003b 89 ebmac TCTC OPT14 95 GCTC GAGA AGGT TCAT GCAT AGAT wg464a GAGA GAGA TCTC TCAT GCGT H Hvm4 0 Bmac222b 9 GCAT TCAT TCTC abg TCTC abc152a 16 GCGT69 25 AGTC TCAT Bmag21c 32 GCAT Ebmac TCGT TCGA VRN-H3 50 KSuA1a 52 abc306a 59 TCGT TCGT64 62 AGGT64 65 OPE Bmac TCGT Bmag OPR OPS mst101b 88 Bmag AGAT AGAT GCGT OPQ TCTC OPB AGGT GCTC GAAT H B1 B3 B7 B9 B10 B12 B13 B5 B3 B3 B7 B7 B8 B15 B14 B16 B18 B19 B4 B5 B6 B10 B12 B14 B4 B5 B7 B11 B13 B18 B1 B3 B4 B8 B10 B12 B3 B6 B5 B9 B12 B10 Dicktoo Kompolti korai marker térkép (246 lókusz)
16
17 Egyedi térképek fajra jellemző konszenzus térkép Stein et al. TAG(2007)114:823
18 P92201D5-2/P91193D1-10 Ajana/WAWHT2074 Cadoux/Reeves EGA Blanco/Millewa wpt-5195* wpt-0643 wpt-6575 wpt-5587 wpt-3459 wpt-5934 wpt-6627* wpt-4916* wpt-0100* wpt-8235* gwm614b* wpt-8404* wpt-1041* wmc25* gwm319* wpt-3569* wpt-1650* wpt-1068* wpt-7937* wpt-0775* wpt-8294* wpt-0395* wpt-1494* wpt-2249* wpt-2907* wpt-0434* wpt-9131* barc183* wpt-0615* wpt-1127* wpt-5440* wpt-6144* wpt-0079* gwm630* wpt-9350* cfd56* AF112966* wpt-7625* gwm47* wmc477* gwm120* wpt-3109* wpt-9336* wpt-7350* wpt-4701 wpt-2266* gwm526c* barc1147* barc92 Marker kapcsoltsági térkép kromoszóma fizikai térképe gwm614 wpt-1663 wpt-5567 wpt-4527 cfd238 wmc25a barc318 wpt-9423 wpt-9402 wpt-1489 wpt-8072 wpt-4301 wpt-0462 wpt-3561 wpt-6932 wpt-3983 wpt-5707 wpt-2314 wpt-6199 wpt-8583 wpt-6477 wpt-4125 wpt-9644 wpt-7757 wpt-0408 wpt-0615 wpt-5672 wpt-8492 barc183b* gwm46 barc7 barc55 wmc474 cfa2278 wpt-0335 wpt-0709 wpt-7750 gwm374 gwm319 gwm630b gwm271 gdm14a gwm120a gwm191a* wpt-1140* wpt-7200 wpt-3132 wpt-0950 wpt-7404 wpt-5128* gwm120b wpt-4199 gdm61* wpt-4527 cfd238 wpt-8004 wpt-8326 wpt-9423 wpt-9402 wpt-1489 wpt-8072 wpt-5707 wpt-3983 wpt-4997 gwm429 barc13 wpt-6477 wpt-0615 wpt-7750 wpt-2430* wpt-8569* wpt-6575* wpt-5587* wpt-3459* wpt-5934* wpt-6970* wpt-6627* wpt-5195* wpt-0643* wpt-6805* wpt-0100* wpt-8760* wpt-2410* wpt-3565* gwm614* wpt-6223* barc35* barc297a* wpt-2106* wpt-8004* wpt-8326* wmc154 wpt-5374 wpt-3561 wpt-5707 wpt-7757 wpt-5672 wpt-5556 wpt-4125 barc55 wpt-6278 barc1114 cfa2043b gwm120 wpt-4199* wpt-2430 wpt-8569 wpt-0094 wpt-7200 wpt-3132 wpt-8460 wpt-5680 wpt-5242 wpt-0694 stm509acag wpt-4701 wpt-7004 wpt-4210 wpt-0047 barc1147 wpt-2135 wpt-3378 barc159 wpt-4559 wpt-6970 wpt-7123 wpt-9288 wpt-6311 wpt-8737 wpt-4916 wpt-9423 wpt-5587 wpt-5934 wpt-6575 wpt-9859 wpt-7995 wpt-9220 wpt-3459 wpt-3188 wpt-1549 wpt-1549 wpt-2249 wpt-2397 wpt-3807 wpt-5287 wpt-1650 wpt-8074 wpt-0408 wpt-0615 wpt-1489 wpt-1494 wpt-3188 wpt-3983 wpt-4701 wpt-4737 wpt-4997 wpt-5249 wpt-5556 wpt-5597 wpt-7348 wpt-7610 wpt-8072 wpt-8294 wpt-8460 wpt-8720 wpt-9402 wpt-6144 wpt-6053 wpt-6627 wpt-7408 wpt-7757 wpt-8070 wpt BS-3 2BS-1 2BL-6 Chromosome 2B
19 QTL elemzés marker kapcsoltsági térkép alkalmazásával QTL azonosítás a teljes marker kapcsoltsági térképre kiterjedve markerek közötti intervallumok vizsgálata Valószínűségi becslések LOD= lg(qtl csúcs jelenlétének valószínűsége/annak a valószínűsége, hogy nincs QTL hatás) LOD küszöbérték: M genom méret M marker sűrűség M populáció méret
20 cm Weight R 2 LOD OPJ15-ABG cm % % H25.0% H 5H % * % ** ============================================================= % ** % *** QTL-Map 14.0 for peak 3: 37.8% *** Confidence Interval: 38.2% Left Boundary= **** OPS31-Snf % **** 40 Right Boundary= Snf2-Hdamyb (off end) LOD values QTL elemzés marker kapcsoltsági térkép alkalmazásával % **** % **** % **** INTERVAL % LENGTH QTL-POS *** GENETICS WEIGHT 28.0 Snf2-Hdamyb % *** free ABG366-abg cm % *** % *** chi^2= (1 D.F.) log-likelihood= % ** mean= sigma^2= variance-explained= abg54-opr % cm % recombination distance (cm) ============================================================= % % HD16h uv HD16h vern HD24h uv HD24h vern
21 Kalászolási idő, mint vizsgált tulajdonság Populáció egyedei közti változékonyság DH vonalak száma h + hő ciklus X átl =77 nap Kompolti Dicktoo Kalászolási idő
22 Dicktoo x Kompolti korai DH vonalak HvSnf2 ZCCTH 4H árpa kromoszóma 0 HvPhyA 8 GCAT76 9 TCAT300 TCAT77 16 AGAT HvPhyB 22 CAB 24 Bmac30 29 AGAT Bmag Bmag173a 39 Bmac OPJ15 41 TCAT OPU AGAT46 62 ABG abg54 65 AGAT GCGT GCAT AGAT HvM67 86 OPS VRN-H2 HvSnf2 AGGT650 Hdamyb QTL elemzés vernalizálatlan vernalizált kezelés
23 Két szülős populációk QTL térképezés Hagyományos két:szülős térképező populációk hátrányai: Előállításuk idő és munka igényes Szűkebb mértékű polimorfizmus Lókuszonként csak két allél hatásának vizsgálatára van mód A két szülő közti hasonlóság korlátozza az egyes gének azonosítási lehetőségeit QTL térképezés pontossága változó Adott populációban azonosított QTL csúcshoz szorosan kapcsolt marker nem polimorf a nemesítési anyagban Gén (allél) kölcsönhatások vizsgálata limitált Alacsony hatékonyság
24 Gén térképezés főbb módszerei Két szülős térképező populációk marker kapcsoltsági térképek, QTL elemzés Széles genetikai bázist képviselő fajtakör LD térképezés, teljes genomot lefedő asszociációs elemzések
25 Széles genetikai bázisú fajtakör asszociációs genetika Nincs szükség a genetikai populáció kifejlesztésére A tesztelt populáció képviselheti a vadfajok, tájfajták és vagy a nemesítés számára releváns fajták széles körét Diverz és nagyméretű populációk vizsgálata a gének detektálásának valószínűségét fokozza Adott gén természetes allél variációi, és azok fenotípusos hatása könnyebben azonosítható Történelmi rekombinációk hasznosítása (evolúció adott környezetben folytatott nemesítés) nagyobb felbontású géntérképezést tesz lehetővé Régebbi fenotípusos vizsgálatok eredményei is felhasználhatók
26 Asszociációs térképezés a növényekben Széles genetikai bázisú fajtakör kiválasztása Széles körű fenotípusos jellemzés (különböző környezet, többszöri ismétlés) Genotipizálás molekuláris markerekkel (pl AFLP, SSR, DArT, SNP) LD jellemzése a marker adatbázisra alapozva Marker tulajdonság asszociáció (megfelelő megközelítés alkalmazásával) Tulajdonsággal kapcsolt markerek azonosítása Populáció szerkezet meghatározása (struktúra és rokonsági fokok) Azonosított lókuszok klónozása, biológiai funkciójuk jellemzése
27 Asszociációs térképezés Valós marker tulajdonság asszociáció azonosításának esélye függ Az alkalmazott molekuláris marker típustól A populációban lévő LD kialakulásának módjától és az LD mértékétől A populáció genomi sajátosságaitól A populáció mérettől és a szerkezetétől A vizsgált tulajdonság genetikai szabályozásának komplexitásától
28 Asszociációs térképezés Valós marker tulajdonság asszociáció azonosításának esélye függ Az alkalmazott molekuláris marker típustól A populációban lévő LD kialakulásának módjától és az LD mértékétől A populáció genomi sajátosságaitól A populáció mérettől és a szerkezetétől Alkalmazott statisztikai modelltől A vizsgált tulajdonság genetikai szabályozásának komplexitásától
29 Molekuláris marker típusok Kodomináns (SNP) domináns (AFLP, DArT) markerek Bialléles (SNP, DArT) : multialléles (SSR, RFLP, AFLP) markerek
30 Asszociációs térképezés Valós marker tulajdonság asszociáció azonosításának esélye függ Az alkalmazott molekuláris marker típustól A populációban lévő LD kialakulásának módjától és az LD mértékétől A populáció genomi sajátosságaitól A populáció mérettől és a szerkezetétől Alkalmazott statisztikai modelltől A vizsgált tulajdonság genetikai szabályozásának komplexitásától
31 Különböző lókuszok allél variánsai közötti kapcsoltsági viszonyok adott populáción belül Linkage disequilibrium (LD; kapcsoltsági egyensúlytalanság) Linkage equilibrium (LE; kapcsoltsági egyensúly)
32 Linkage disequilibrium (LD) LD: az allélok nem véletlenszerű szétválása (kromoszómán belül és kromoszómák között) Mértékének meghatározása D= P AB :P A P B ; Megfigyelt és a várt haplotípus gyakoriságok különbsége rekombinációs különbségek összegzése r 2 ; két marker lókusz közti korreláció négyzete rekombinációs és mutációs események összegzése
33 LD ábrázolása Páronkénti r 2 :ben kifejezett LD értékek grafikus ábrázolása KétMszülős marker térkép Fajta populáció marker mátrix Sok fajtás LD térkép
34 LD ábrázolása LD csökkenés (LD decay) a páronkénti marker távolság függvényében y = Ln(x) R 2 = két marker közti rekombinációs távolság (cm) két marker közti korreláció ( r 2 )
35 LD mértékét befolyásoló faktorok LD értékét növelő tényezők Öntermékenyülés Genetikai izoláció Strukturált populáció Rokonság Kis méretű kiinduló populáció Alacsony rekombinációs gyakoriság LD értékét csökkentő tényezők Idegen megtermékenyülés Rekombináció és mutáció nagy gyakorisága Ismétlődő mutációk
36 Az LD mértéke függ a populáció típusától, a rekombináció gyakoriságától (adott kromoszóma régió genetikai és fizikai távolsága közti arány), az LD blokkoktól LD mértéke árpában Cél gén Cél gén LD LD lassan szűnik meg Termesztett árpa ~5cM Rostoks et al., 2006 Kraakman et al., 2004 LD gyorsan szűnik meg Vadárpa Génen belül (~300 bp) Morrell et al., 2005 Árpa genom ~ 1250 cm
37 LD és az asszociációs vizsgálatok Nagy kiterjedésű LD Csökkenti az asszociációs térképezéshez szükséges marker sűrűséget Térképezés felbontóképessége csökkent (coarse mapping) Szűk LD Nagyszámú marker szükséges az asszociációs vizsgálatok kivitelezéséhez, egy adott gén markerezéséhez Térképezés felbontó képessége jelentősen megnő Független markerek közti LD Hamis marker tulajdonság kapcsolat azonosításának esélye megnő
38 1H Árpa genomot lefedő LD térkép 2H 3H Comadran et al. (2011) TAG 122: Mediterrán árpafajta 2132 SNP 4H LD cm:ban kifejezve 5H 6H 7H 1H 2H 3H 4H 5H 6H 7H
39 ps1443 ps1515 ps1725 ps1785 ps1839 ps1843 ps2032 ps2035 ps2036 ps2230 ps2322 ps2327 ps182 ps427 ps506 ps582 ps843 ps885 ps912 ps1093 ps1267 ps1348 ps1455 ps1489 ps1576 ps1596 ps1816 ps2008 ps182 ps427 ps506 ps582 ps843 ps885 ps912 ps1093 ps1267 ps1348 ps1455 ps1489 ps1576 ps1596 ps1816 ps2008 ps1443 ps1515 ps1725 ps1785 ps1839 ps1843 ps2032 ps2035 ps2036 ps2230 ps2322 ps >0.01 <0.01 <0.001 < lower r 2 upper Pvalue PpdH1 2H PpdH1 HvFT 7H HvCO1 7H HvFT HvCO1 ps1443 ps1515 ps1725 ps1785 ps1839 ps1843 ps2032 ps2035 ps2036 ps2230 ps2322 ps2327 ps182 ps427 ps506 ps582 ps843 ps885 ps912 ps1093 ps1267 ps1348 ps1455 ps1489 ps1576 ps1596 ps1816 ps2008 ps182 ps427 ps506 ps582 ps843 ps885 ps912 ps1093 ps1267 ps1348 ps1455 ps1489 ps1576 ps1596 ps1816 ps2008 ps1443 ps1515 ps1725 ps1785 ps1839 ps1843 ps2032 ps2035 ps2036 ps2230 ps2322 ps >0.01 <0.01 <0.001 < lower r 2 upper Pvalue PpdH1 2H PpdH1 HvFT1 7H HvCO1 7H HvFT1 HvCO1 B ps1443 ps1515 ps1725 ps1785 ps1839 ps1843 ps2032 ps2035 ps2036 ps2230 ps2322 ps2327 ps182 ps427 ps506 ps582 ps843 ps885 ps912 ps1093 ps1267 ps1348 ps1455 ps1489 ps1576 ps1596 ps1816 ps2008 ps182 ps427 ps506 ps582 ps843 ps885 ps912 ps1093 ps1267 ps1348 ps1455 ps1489 ps1576 ps1596 ps1816 ps2008 ps1443 ps1515 ps1725 ps1785 ps1839 ps1843 ps2032 ps2035 ps2036 ps2230 ps2322 ps >0.01 <0.01 <0.001 < lower r 2 upper Pvalue PpdH1 2H PpdH1 HvFT 7H HvCO1 7H HvFT HvCO1 ps1443 ps1515 ps1725 ps1785 ps1839 ps1843 ps2032 ps2035 ps2036 ps2230 ps2322 ps2327 ps182 ps427 ps506 ps582 ps843 ps885 ps912 ps1093 ps1267 ps1348 ps1455 ps1489 ps1576 ps1596 ps1816 ps2008 ps182 ps427 ps506 ps582 ps843 ps885 ps912 ps1093 ps1267 ps1348 ps1455 ps1489 ps1576 ps1596 ps1816 ps2008 ps1443 ps1515 ps1725 ps1785 ps1839 ps1843 ps2032 ps2035 ps2036 ps2230 ps2322 ps >0.01 <0.01 <0.001 < lower r 2 upper Pvalue PpdH1 2H PpdH1 HvFT1 7H HvCO1 7H HvFT1 HvCO >0.01 <0.01 <0.001 < lower r 2 upper Pvalue PpdH1 2H PpdH1 HvFT1 7H HvCO1 7H HvFT1 HvCO1 B Egyedi gének LD térképei Stracke et al. TAG(2009)118:259 LD kb:ban kifejezve
40 Széles genetikai bázisú fajtakör asszociációs genetika LD térképpel lehetővé válik a gén fenotípus asszociációk azonosítása a meglévő törzsgyűjteményekben Az evolúció során rögzített fenotípusos információkat, meiotikus és rekombinációs eseményeket hasznosítja Egy törzsgyűjteményben lehetőség nyílik számos fenotípusos tulajdonság párhuzamos vizsgálatára
41 Asszociációs térképezés Valós marker tulajdonság asszociáció azonosításának esélye függ Az alkalmazott molekuláris marker típustól A populációban lévő LD kialakulásának módjától és az LD mértékétől A populáció genomi sajátosságaitól A populáció mérettől és a szerkezetétől Alkalmazott statisztikai modelltől A vizsgált tulajdonság genetikai szabályozásának komplexitásától
42 Σ , H H H H H H H Őszi+ átmeneti őszi tavaszi 168 fajta Gén spec DArT Átlagos LD csökkenés (cm) Átl cm Össz cm Markerek LDMdecay kromoszómánkénti változásai 168 árpafajta körben
43 LDMdecay kromoszómánkénti változásai 168 árpafajta körben Átlagos LD csökkenés (cm) Egyedi kromoszómák és a teljes genomi markerek közti összefüggés Korreláció (r) alapján Párosított TMteszt (P érték) alapján 1H E:12 2H E:27 3H E:54 4H E:38 5H ns 6H EM239 7H E:10 Átlag 5.0 : :
44 Asszociációs térképezés Valós marker tulajdonság asszociáció azonosításának esélye függ Az alkalmazott molekuláris marker típustól A populációban lévő LD kialakulásának módjától és az LD mértékétől A populáció genomi sajátosságaitól A populáció mérettől és a szerkezetétől Alkalmazott statisztikai modelltől A vizsgált tulajdonság genetikai szabályozásának komplexitásától
45 Rawson(ND MW US CAP Core 102 Lines 1105 P-OPA2 markers; SM distance; UPGMA cluster Coefficient Barke Pasadena C-14 Farmington Scarlett Collins Franklin BISON5H BISON1H+5H BISON4H+5H Baronesse BISON1H BISON4H BISON7H BISON1H+4H BISON1H+4H+5H Geraldine Eslick Hays Radiant ACMetcalfe CDC-Kendall CDC-Stratus Newdale Merit Crest Klages Hockett Harrington Sublette Garnett CDCCopeland 2B TR306 B1202 B1215 2B Conrad Charles Rawson(ND Bowman Conlon ND21863 Craft Haxby Orca Flagship2 Arapiles Canela BCD47 BCD12 HarunaNijo CIho4196 Shenmai3 OWBRecessive Steptoe Sussex Wysor Nomini Price Doyce Strider Kold Thoroughbred Dicktoo Kompolti WA Hoody Belford Washford Morex 88Ab536 88Ab536-B Robust MNBrite Excel Lacey M123 6B Legacy 6B FEG90-31 FEG66-08 M122 6B FEG B Stander B1614 Stellar Foster 6B CDC-Sisler FEG59-09 B1602 Tradition Drummond Larker ND20448 ND20508 NDB112 OWBDominant
46 Strukturált populáció A populáción belül határozottan elkülönülő alcsoportok jelenléte LD kapcsolat egymástól távol, nem kapcsolt, vagy eltérő kromoszómákon levő lókuszok között Alcsoportonként változó LD blokkok
47 Rawson(ND MW US CAP Core 102 Lines 1105 P-OPA2 markers; SM distance; UPGMA cluster Coefficient Barke Pasadena C-14 Farmington Scarlett Collins Franklin BISON5H BISON1H+5H BISON4H+5H Baronesse BISON1H BISON4H BISON7H BISON1H+4H BISON1H+4H+5H Geraldine Eslick Hays Radiant ACMetcalfe CDC-Kendall CDC-Stratus Newdale Merit Crest Klages Hockett Harrington Sublette Garnett CDCCopeland 2B TR306 B1202 B1215 2B Conrad Charles Rawson(ND Bowman Conlon ND21863 Craft Haxby Orca Flagship2 Arapiles Canela BCD47 BCD12 HarunaNijo CIho4196 Shenmai3 OWBRecessive Steptoe Sussex Wysor Nomini Price Doyce Strider Kold Thoroughbred Dicktoo Kompolti WA Hoody Belford Washford Morex 88Ab536 88Ab536-B Robust MNBrite Excel Lacey M123 6B Legacy 6B FEG90-31 FEG66-08 M122 6B FEG B Stander B1614 Stellar Foster 6B CDC-Sisler FEG59-09 B1602 Tradition Drummond Larker ND20448 ND20508 NDB112 OWBDominant
48 Strukturált populációk Populáció szerkezetből származó fals asszociációk Populáció struktúra mint tényező beépítése az elemzésekbe K mátrix (pairwise Kinship values) (TASSEL, PCA, GenStat) Q mátrix (a határozottan elkülönülő alcsoportok azonosítása) (STRUCTURE) K + Q mátrix együttes használata
49 Alcsoportok számának meghatározása STRUCTURE programmal Goodness of fit k
50 Comadran et al. (2009) TAG 119: Mediterrán árpafajta 1130 DArT marker alapján
51 Alcsoportok és rokonsági kapcsolatok meghatározása főkomponens elemzéssel (PCA)
52 LD:decay populáció és alcsoport függő változásai 811 térképezett DArT marker alapján Comadran et al. (2009) TAG 119:175
53 Asszociációs térképezés Valós marker tulajdonság asszociáció azonosításának esélye függ Az alkalmazott molekuláris marker típustól A populációban lévő LD kialakulásának módjától és az LD mértékétől A populáció mérettől és a szerkezetétől Alkalmazott statisztikai modelltől A vizsgált tulajdonság genetikai szabályozásának komplexitásától
54 Asszociációs vizsgálatok alapstatisztikai eljárásai Egyszerű marker:tulajdonság asszociáció (naív modell) Általános lineáris modell (GLM) Kevert lineáris modell MLM+Kinship mátrix; MLMK Kevert lineáris modell + populáció struktúra MLMK + Q mátrix
55 MOR (naïve) 0.7 Cumulative % of p-value ITA (naïve) MOR (Q) ITA (Q) K Naïve Q 0.1 K Observed p-value Comadran et al. (2009) TAG 119:175
56 12 10 GLM naive GLM Q MLM K MLM Q_K 8 -log10p rekombinációs távolság (cm)
57 Asszociációs térképezés Valós marker tulajdonság asszociáció azonosításának esélye függ Az alkalmazott molekuláris marker típustól A populációban lévő LD kialakulásának módjától és az LD mértékétől A populáció mérettől és a szerkezetétől Alkalmazott statisztikai modelltől A vizsgált tulajdonság genetikai szabályozásának komplexitásától
58 Lisztharmat rezisztencia asszociációs vizsgálata árpában Comadran et al. (2009) TAG 119:175
59 Mennyiségi tulajdonságok Termőképesség genom széles asszociációs vizsgálata árpában (Comadran et al ) Mediterrán medence körül 12 termőhelyen két évjáratban (24 egyedi adatsor) Meta:analízis során fő QTL:ek meghatározása, egyedi termőhelyeken azonosítható QTL:ek
60 Mennyiségi tulajdonságok Termőképesség genom széles asszociációs vizsgálata árpában (Comadran et al )
61 Mennyiségi tulajdonságok Termőképesség genom széles asszociációs vizsgálata árpában (Comadran et al ) 2 7 QTL:t azonosítottak a termés különböző komponensei esetében A szignifikáns asszociációk egy része olyan SNP:k nél lokalizálódott, amelyek szorosan kapcsolódtak ismert génekhez QTL:ek együtt lokalizálódása a genomon termés meta:analíziseként 3 főhatású QTL:t azonosítottak, míg az egyedi termőhelyekre bontva ezek száma 27 volt (genotípus x környezet kölcsönhatás elemzése)
62 Strukturált populációk problematikája A populáció struktúra hatását számításba vevő módszerek nem minden esetben képesek a fals pozitív asszociációk kiszűrésére A populáció struktúrára szűrés megakadályozhatja az olyan biológiai jelentőséggel bíró QTL / génhatások azonosítását is, amelyek egybeesnek a populáció struktúrával A fenotípus szignifikáns korrelációja a populáció struktúrával megakadályozhatja a kisebb hatású allélok detektálását
63 Javasolt irodalom Abdurakhmonov et al. (2008) International Journal of Plant Genomics vol. 2008, article No: Comadran et al. (2009) Theor Appl Genet, 119:175 Comadran et al (2011) Theor Appl Genet 122:1363 Rostoks et al. (2006) PNAS 103:18656 Waugh et al. (2009) Curr Opin in Plant Biol 12:218
Genetikai térképezés, gén azonosítás Mészáros Klára, Karsai Ildikó meszarosk@mail.mgki.hu
 Genetikai térképezés, gén azonosítás, Karsai Ildikó meszarosk@mail.mgki.hu Növénynemesítés feladatai Termésbiztonság növelése, nagy szezonális szélsőségek csökkentése Biotikus és abiotikus stresszrezisztencia
Genetikai térképezés, gén azonosítás, Karsai Ildikó meszarosk@mail.mgki.hu Növénynemesítés feladatai Termésbiztonság növelése, nagy szezonális szélsőségek csökkentése Biotikus és abiotikus stresszrezisztencia
Genetikai térképezés, gén azonosítás
 Genetikai térképezés, gén azonosítás, Karsai Ildikó meszarosk@agrar.mta.hu Növénynemesítés feladatai Növénytermesztés hatékonyságának és a termésbiztonság növelése speciális termesztési rendszerek biztosítása
Genetikai térképezés, gén azonosítás, Karsai Ildikó meszarosk@agrar.mta.hu Növénynemesítés feladatai Növénytermesztés hatékonyságának és a termésbiztonság növelése speciális termesztési rendszerek biztosítása
Többgénes jellegek. 1. Klasszikus (poligénes) mennyiségi jellegek. 2.Szinte minden jelleg több gén irányítása alatt áll
 Többgénes jellegek Többgénes jellegek 1. 1. Klasszikus (poligénes) mennyiségi jellegek Multifaktoriális jellegek: több gén és a környezet által meghatározott jellegek 2.Szinte minden jelleg több gén irányítása
Többgénes jellegek Többgénes jellegek 1. 1. Klasszikus (poligénes) mennyiségi jellegek Multifaktoriális jellegek: több gén és a környezet által meghatározott jellegek 2.Szinte minden jelleg több gén irányítása
HAPMAP -2010 Nemzetközi HapMap Projekt. SNP GWA Haplotípus: egy kromoszóma szegmensen lévő SNP mintázat
 HAPMAP -2010 Nemzetközi HapMap Projekt A Nemzetközi HapMap Project célja az emberi genom haplotípus* térképének(hapmap; haplotype map) megszerkesztése, melynek segítségével katalogizálni tudjuk az ember
HAPMAP -2010 Nemzetközi HapMap Projekt A Nemzetközi HapMap Project célja az emberi genom haplotípus* térképének(hapmap; haplotype map) megszerkesztése, melynek segítségével katalogizálni tudjuk az ember
Hátterükben egyetlen gén áll, melynek általában számottevő a viselkedésre gyakorolt hatása, öröklési mintázata jellegzetes.
 Múlt órán: Lehetséges tesztfeladatok: Kitől származik a variáció-szelekció paradigma, mely szerint az egyéni, javarészt öröklött különbségek között a társadalmi harc válogat? Fromm-Reichmann Mill Gallton
Múlt órán: Lehetséges tesztfeladatok: Kitől származik a variáció-szelekció paradigma, mely szerint az egyéni, javarészt öröklött különbségek között a társadalmi harc válogat? Fromm-Reichmann Mill Gallton
A GABONAFÉLÉK EGYEDFEJLŐDÉSÉT ÉS KALÁSZOLÁSÁT MEGHATÁROZÓ GENETIKAI KOMPONENSEK TANULMÁNYOZÁSA
 A martonvásári agrárkutatások hatodik évtizede A GABONAFÉLÉK EGYEDFEJLŐDÉSÉT ÉS KALÁSZOLÁSÁT MEGHATÁROZÓ GENETIKAI KOMPONENSEK TANULMÁNYOZÁSA KARSAI ILDIKÓ 1, MÉSZÁROS KLÁRA 2, KŐSZEGI BÉLA 3, PATRICK
A martonvásári agrárkutatások hatodik évtizede A GABONAFÉLÉK EGYEDFEJLŐDÉSÉT ÉS KALÁSZOLÁSÁT MEGHATÁROZÓ GENETIKAI KOMPONENSEK TANULMÁNYOZÁSA KARSAI ILDIKÓ 1, MÉSZÁROS KLÁRA 2, KŐSZEGI BÉLA 3, PATRICK
Az Agrármérnöki MSc szak tananyagfejlesztése TÁMOP-4.1.2-08/1/A-2009-0010 projekt
 Az Agrármérnöki MSc szak tananyagfejlesztése TÁMOP-4.1.2-08/1/A-2009-0010 projekt ÁLLATGENETIKA Debreceni Egyetem Nyugat-magyarországi Egyetem Pannon Egyetem A projekt az Európai Unió támogatásával, az
Az Agrármérnöki MSc szak tananyagfejlesztése TÁMOP-4.1.2-08/1/A-2009-0010 projekt ÁLLATGENETIKA Debreceni Egyetem Nyugat-magyarországi Egyetem Pannon Egyetem A projekt az Európai Unió támogatásával, az
Populációgenetikai. alapok
 Populációgenetikai alapok Populáció = egyedek egy adott csoportja Az egyedek eltérnek egymástól morfológiailag, de viselkedésüket tekintve is = genetikai különbségek Fenotípus = külső jellegek morfológia,
Populációgenetikai alapok Populáció = egyedek egy adott csoportja Az egyedek eltérnek egymástól morfológiailag, de viselkedésüket tekintve is = genetikai különbségek Fenotípus = külső jellegek morfológia,
Intelligens Rendszerek Elmélete. Párhuzamos keresés genetikus algoritmusokkal
 Intelligens Rendszerek Elmélete Dr. Kutor László Párhuzamos keresés genetikus algoritmusokkal http://mobil.nik.bmf.hu/tantargyak/ire.html login: ire jelszó: IRE0 IRE / A természet általános kereső algoritmusa:
Intelligens Rendszerek Elmélete Dr. Kutor László Párhuzamos keresés genetikus algoritmusokkal http://mobil.nik.bmf.hu/tantargyak/ire.html login: ire jelszó: IRE0 IRE / A természet általános kereső algoritmusa:
Zárójelentés. OTKA NK72913 sz. pályázat
 Zárójelentés OTKA NK79 sz. pályázat A hőmérséklet szerepe a gabonafélék kalászolási idejének szabályozásában Témavezető: Dr. Karsai Ildikó 008 0 Martonvásár OTKA NK79 zárójelentés. Bevezetés Kontrollált
Zárójelentés OTKA NK79 sz. pályázat A hőmérséklet szerepe a gabonafélék kalászolási idejének szabályozásában Témavezető: Dr. Karsai Ildikó 008 0 Martonvásár OTKA NK79 zárójelentés. Bevezetés Kontrollált
Számítógépes döntéstámogatás. Genetikus algoritmusok
 BLSZM-10 p. 1/18 Számítógépes döntéstámogatás Genetikus algoritmusok Werner Ágnes Villamosmérnöki és Információs Rendszerek Tanszék e-mail: werner.agnes@virt.uni-pannon.hu BLSZM-10 p. 2/18 Bevezetés 1950-60-as
BLSZM-10 p. 1/18 Számítógépes döntéstámogatás Genetikus algoritmusok Werner Ágnes Villamosmérnöki és Információs Rendszerek Tanszék e-mail: werner.agnes@virt.uni-pannon.hu BLSZM-10 p. 2/18 Bevezetés 1950-60-as
ÁLLATTENYÉSZTÉSI GENETIKA
 TÁMOP-4.1.2-08/1/A-2009-0010 project ÁLLATTENYÉSZTÉSI GENETIKA University of Debrecen University of West Hungary University of Pannonia The project is supported by the European Union and co-financed by
TÁMOP-4.1.2-08/1/A-2009-0010 project ÁLLATTENYÉSZTÉSI GENETIKA University of Debrecen University of West Hungary University of Pannonia The project is supported by the European Union and co-financed by
Intelligens Rendszerek Elmélete. Párhuzamos keresés genetikus algoritmusokkal. A genetikus algoritmus működése. Az élet információ tárolói
 Intelligens Rendszerek Elmélete dr. Kutor László Párhuzamos keresés genetikus algoritmusokkal http://mobil.nik.bmf.hu/tantargyak/ire.html login: ire jelszó: IRE07 IRE 5/ Természetes és mesterséges genetikus
Intelligens Rendszerek Elmélete dr. Kutor László Párhuzamos keresés genetikus algoritmusokkal http://mobil.nik.bmf.hu/tantargyak/ire.html login: ire jelszó: IRE07 IRE 5/ Természetes és mesterséges genetikus
A Hardy-Weinberg egyensúly. 2. gyakorlat
 A Hardy-Weinberg egyensúly 2. gyakorlat A Hardy-Weinberg egyensúly feltételei: nincs szelekció nincs migráció nagy populációméret (nincs sodródás) nincs mutáció pánmixis van allélgyakoriság azonos hímekben
A Hardy-Weinberg egyensúly 2. gyakorlat A Hardy-Weinberg egyensúly feltételei: nincs szelekció nincs migráció nagy populációméret (nincs sodródás) nincs mutáció pánmixis van allélgyakoriság azonos hímekben
Algoritmusok Tervezése. 9. Előadás Genetikus Algoritmusok Dr. Bécsi Tamás
 Algoritmusok Tervezése 9. Előadás Genetikus Algoritmusok Dr. Bécsi Tamás Biológiai háttér (nagyvonalúan) A sejt genetikai információit hordozó DNS általában kromoszómának nevezett makromolekulákba van
Algoritmusok Tervezése 9. Előadás Genetikus Algoritmusok Dr. Bécsi Tamás Biológiai háttér (nagyvonalúan) A sejt genetikai információit hordozó DNS általában kromoszómának nevezett makromolekulákba van
Természetes szelekció és adaptáció
 Természetes szelekció és adaptáció Amiről szó lesz öröklődő és variábilis fenotípus természetes szelekció adaptáció evolúció 2. Természetes szelekció Miért fontos a természetes szelekció (TSZ)? 1. C.R.
Természetes szelekció és adaptáció Amiről szó lesz öröklődő és variábilis fenotípus természetes szelekció adaptáció evolúció 2. Természetes szelekció Miért fontos a természetes szelekció (TSZ)? 1. C.R.
Conserved ortholog set (COS) markerek térképezése Aegilops kromoszómákon
 Conserved ortholog set (COS) markerek térképezése Aegilops kromoszómákon Rövid tanulmányút 2011. 01.03-03. 30., John Inn Centre, Dept. of Crop Genetics, Norwich Research Park, Norwich NR4 7UH, UK Supervisor:
Conserved ortholog set (COS) markerek térképezése Aegilops kromoszómákon Rövid tanulmányút 2011. 01.03-03. 30., John Inn Centre, Dept. of Crop Genetics, Norwich Research Park, Norwich NR4 7UH, UK Supervisor:
Evolúció. Dr. Szemethy László egyetemi docens Szent István Egyetem VadVilág Megőrzési Intézet
 Evolúció Dr. Szemethy László egyetemi docens Szent István Egyetem VadVilág Megőrzési Intézet Mi az evolúció? Egy folyamat: az élőlények tulajdonságainak változása a környezethez való alkalmazkodásra Egy
Evolúció Dr. Szemethy László egyetemi docens Szent István Egyetem VadVilág Megőrzési Intézet Mi az evolúció? Egy folyamat: az élőlények tulajdonságainak változása a környezethez való alkalmazkodásra Egy
Tremmelné Tar Melinda
 SZENT ISTVÁN EGYETEM PCR alapú molekuláris markerek azonosítása és felhasználása a búza levélrozsda rezisztenciára való nemesítésben Doktori (PhD) értekezés tézisei Tremmelné Tar Melinda Gödöllı 2012.
SZENT ISTVÁN EGYETEM PCR alapú molekuláris markerek azonosítása és felhasználása a búza levélrozsda rezisztenciára való nemesítésben Doktori (PhD) értekezés tézisei Tremmelné Tar Melinda Gödöllı 2012.
Evolúció. Dr. Szemethy László egyetemi docens Szent István Egyetem VadVilág Megőrzési Intézet
 Evolúció Dr. Szemethy László egyetemi docens Szent István Egyetem VadVilág Megőrzési Intézet Mi az evolúció? Egy folyamat: az élőlények tulajdonságainak változása a környezethez való alkalmazkodásra Egy
Evolúció Dr. Szemethy László egyetemi docens Szent István Egyetem VadVilág Megőrzési Intézet Mi az evolúció? Egy folyamat: az élőlények tulajdonságainak változása a környezethez való alkalmazkodásra Egy
Orvosi Genomika. 1. Trendek a modern orvostudományban. 2. Genomika és modern orvostudomány
 1 1. ORVOSI GENOMIKA Orvosi Genomika 1. Trendek a modern orvostudományban DIA 1 Az emberi genom megfejtése óta eltelt 10 évben rendkívül gyors fejlődés ment végbe a genomikai technikákban, ill. a genomikai
1 1. ORVOSI GENOMIKA Orvosi Genomika 1. Trendek a modern orvostudományban DIA 1 Az emberi genom megfejtése óta eltelt 10 évben rendkívül gyors fejlődés ment végbe a genomikai technikákban, ill. a genomikai
Biomassza alapú bioalkohol előállítási technológia fejlesztése metagenomikai eljárással
 Biomassza alapú bioalkohol előállítási technológia fejlesztése metagenomikai eljárással Kovács Zoltán ügyvezető DEKUT Debreceni Kutatásfejlesztési Közhasznú Nonprofit Kft. Problémadefiníció Első generációs
Biomassza alapú bioalkohol előállítási technológia fejlesztése metagenomikai eljárással Kovács Zoltán ügyvezető DEKUT Debreceni Kutatásfejlesztési Közhasznú Nonprofit Kft. Problémadefiníció Első generációs
Szelekció. Szelekció. A szelekció típusai. Az allélgyakoriságok változása 3/4/2013
 Szelekció Ok: több egyed születik, mint amennyi túlél és szaporodni képes a sikeresség mérése: fitnesz Szelekció Ok: több egyed születik, mint amennyi túlél és szaporodni képes a sikeresség mérése: fitnesz
Szelekció Ok: több egyed születik, mint amennyi túlél és szaporodni képes a sikeresség mérése: fitnesz Szelekció Ok: több egyed születik, mint amennyi túlél és szaporodni képes a sikeresség mérése: fitnesz
2011. január április 10. IPK Gatersleben (Németország) május 17. Kruppa Klaudia
 2011. január 10. 2011. április 10. IPK Gatersleben (Németország) Gatersleben (G-life) Country State District Town Administration Germany Saxony-Anhalt Salzlandkreis Seeland Basic statistics Area 16.00
2011. január 10. 2011. április 10. IPK Gatersleben (Németország) Gatersleben (G-life) Country State District Town Administration Germany Saxony-Anhalt Salzlandkreis Seeland Basic statistics Area 16.00
Tudománytörténeti visszatekintés
 GENETIKA I. AZ ÖRÖKLŐDÉS TÖRVÉNYSZERŰSÉGEI Minek köszönhető a biológiai sokféleség? Hogyan történik a tulajdonságok átörökítése? Tudománytörténeti visszatekintés 1. Keveredés alapú öröklődés: (1761-1766,
GENETIKA I. AZ ÖRÖKLŐDÉS TÖRVÉNYSZERŰSÉGEI Minek köszönhető a biológiai sokféleség? Hogyan történik a tulajdonságok átörökítése? Tudománytörténeti visszatekintés 1. Keveredés alapú öröklődés: (1761-1766,
DOKTORI (PHD) ÉRTEKEZÉS CERNÁK ISTVÁN
 DOKTORI (PHD) ÉRTEKEZÉS CERNÁK ISTVÁN KESZTHELY 2008 2 PANNON EGYETEM GEORGIKON MEZŐGAZDASÁGTUDOMÁNYI KAR Növénytudományi és Biotechnológia Tanszék Növénytermesztés és Kertészeti Tudományok Doktori Iskola
DOKTORI (PHD) ÉRTEKEZÉS CERNÁK ISTVÁN KESZTHELY 2008 2 PANNON EGYETEM GEORGIKON MEZŐGAZDASÁGTUDOMÁNYI KAR Növénytudományi és Biotechnológia Tanszék Növénytermesztés és Kertészeti Tudományok Doktori Iskola
A metabolikus szindróma genetikai háttere. Kappelmayer János, Balogh István (www.kbmpi.hu)
 A metabolikus szindróma genetikai háttere Kappelmayer János, Balogh István (www.kbmpi.hu) Definíció WHO, 1999 EGIR, 1999 ATP III, 2001 Ha három vagy több komponens jelen van a betegben: Vérnyomás: > 135/85
A metabolikus szindróma genetikai háttere Kappelmayer János, Balogh István (www.kbmpi.hu) Definíció WHO, 1999 EGIR, 1999 ATP III, 2001 Ha három vagy több komponens jelen van a betegben: Vérnyomás: > 135/85
Evolúcióbiológia. Biológus B.Sc tavaszi félév
 Evolúcióbiológia Biológus B.Sc. 2011. tavaszi félév A biológiában minden csak az evolúció fényében válik érthetővé Theodosius Dobzhansky : Nothing in biology makes sense except in the light of evolution.
Evolúcióbiológia Biológus B.Sc. 2011. tavaszi félév A biológiában minden csak az evolúció fényében válik érthetővé Theodosius Dobzhansky : Nothing in biology makes sense except in the light of evolution.
Molekuláris genetikai vizsgáló. módszerek az immundefektusok. diagnosztikájában
 Molekuláris genetikai vizsgáló módszerek az immundefektusok diagnosztikájában Primer immundefektusok A primer immundeficiencia ritka, veleszületett, monogénes öröklődésű immunhiányos állapot. Családi halmozódást
Molekuláris genetikai vizsgáló módszerek az immundefektusok diagnosztikájában Primer immundefektusok A primer immundeficiencia ritka, veleszületett, monogénes öröklődésű immunhiányos állapot. Családi halmozódást
A genetikai lelet értelmezése monogénes betegségekben
 A genetikai lelet értelmezése monogénes betegségekben Tory Kálmán Semmelweis Egyetem, I. sz. Gyermekklinika A ~20 ezer fehérje-kódoló gén a 23 pár kromoszómán A kromoszómán található bázisok száma: 250M
A genetikai lelet értelmezése monogénes betegségekben Tory Kálmán Semmelweis Egyetem, I. sz. Gyermekklinika A ~20 ezer fehérje-kódoló gén a 23 pár kromoszómán A kromoszómán található bázisok száma: 250M
A HUMÁNGENETIKA LEGÚJABB EREDMÉNYEI Péterfy Miklós
 A HUMÁNGENETIKA LEGÚJABB EREDMÉNYEI Péterfy Miklós Összefoglalás A humángenetika korunk egyik legdinamikusabban fejlődő tudományága. Ennek a fejlődésnek legfőbb mozgatórugója az, hogy a humángenetika,
A HUMÁNGENETIKA LEGÚJABB EREDMÉNYEI Péterfy Miklós Összefoglalás A humángenetika korunk egyik legdinamikusabban fejlődő tudományága. Ennek a fejlődésnek legfőbb mozgatórugója az, hogy a humángenetika,
Genetika 3 ea. Bevezetés
 Genetika 3 ea. Mendel törvényeinek a kiegészítése: Egygénes öröklődés Többtényezős öröklődés Bevezetés Mendel által vizsgált tulajdonságok: diszkrétek, két különböző fenotípus Humán tulajdonságok nagy
Genetika 3 ea. Mendel törvényeinek a kiegészítése: Egygénes öröklődés Többtényezős öröklődés Bevezetés Mendel által vizsgált tulajdonságok: diszkrétek, két különböző fenotípus Humán tulajdonságok nagy
Informatikai Rendszerek Tervezése
 Sapientia - Erdélyi Magyar TudományEgyetem (EMTE) Csíkszereda IRT.- 5. kurzus 1 Informatikai Rendszerek Tervezése 4. Előadás: Genetikus algoritmusok Illyés László 1 Tartalom Bevezető A kanonikus genetikus
Sapientia - Erdélyi Magyar TudományEgyetem (EMTE) Csíkszereda IRT.- 5. kurzus 1 Informatikai Rendszerek Tervezése 4. Előadás: Genetikus algoritmusok Illyés László 1 Tartalom Bevezető A kanonikus genetikus
A PKU azért nem hal ki, mert gyógyítják, és ezzel növelik a mutáns allél gyakoriságát a Huntington kór pedig azért marad fenn, mert csak későn derül
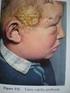 1 Múlt órán: Genetikai alapelvek, monogénes öröklődés Elgondolkodtató feladat Vajon miért nem halnak ki az olyan mendeli öröklődésű rendellenességek, mint a Phenylketonuria, vagy a Huntington kór? A PKU
1 Múlt órán: Genetikai alapelvek, monogénes öröklődés Elgondolkodtató feladat Vajon miért nem halnak ki az olyan mendeli öröklődésű rendellenességek, mint a Phenylketonuria, vagy a Huntington kór? A PKU
A genetikai sodródás
 A genetikai sodródás irányított, nem véletlenszerű Mindig a jobb nyer! természetes szelekció POPULÁCIÓ evolúció POPULÁCIÓ A kulcsszó: változékonyság a populáción belül POPULÁCIÓ nem irányított, véletlenszerű
A genetikai sodródás irányított, nem véletlenszerű Mindig a jobb nyer! természetes szelekció POPULÁCIÓ evolúció POPULÁCIÓ A kulcsszó: változékonyság a populáción belül POPULÁCIÓ nem irányított, véletlenszerű
Evolúciós algoritmusok
 Evolúciós algoritmusok Evolúció, mint kereső rendszer A problémára adható néhány lehetséges választ, azaz a problématér több egyedét tároljuk egyszerre. Ez a populáció. Kezdetben egy többnyire véletlen
Evolúciós algoritmusok Evolúció, mint kereső rendszer A problémára adható néhány lehetséges választ, azaz a problématér több egyedét tároljuk egyszerre. Ez a populáció. Kezdetben egy többnyire véletlen
BIOLÓGIA HÁZIVERSENY 1. FORDULÓ BIOKÉMIA, GENETIKA BIOKÉMIA, GENETIKA
 BIOKÉMIA, GENETIKA 1. Nukleinsavak keresztrejtvény (12+1 p) 1. 2. 3. 4. 5. 6. 7. 8. 9. 10. 11. 12. 1. A nukleinsavak a.-ok összekapcsolódásával kialakuló polimerek. 2. Purinvázas szerves bázis, amely az
BIOKÉMIA, GENETIKA 1. Nukleinsavak keresztrejtvény (12+1 p) 1. 2. 3. 4. 5. 6. 7. 8. 9. 10. 11. 12. 1. A nukleinsavak a.-ok összekapcsolódásával kialakuló polimerek. 2. Purinvázas szerves bázis, amely az
Példák a független öröklődésre
 GENETIKAI PROBLÉMÁK Példák a független öröklődésre Az amelogenesis imperfecta egy, a fogzománc gyengeségével és elszíneződésével járó öröklődő betegség, a 4-es kromoszómán lévő enam gén recesszív mutációja
GENETIKAI PROBLÉMÁK Példák a független öröklődésre Az amelogenesis imperfecta egy, a fogzománc gyengeségével és elszíneződésével járó öröklődő betegség, a 4-es kromoszómán lévő enam gén recesszív mutációja
Nagy Emese: Polimorfizmus és rokonsági körök vizsgálata kukoricában (Zea mays) Témavezetők: Cs. L. Marton G Gyulai
 Nagy Emese: Polimorfizmus és rokonsági körök vizsgálata kukoricában (Zea mays) Témavezetők: Cs. L. Marton G Gyulai BEVEZETÉS A molekuláris biológiai és genetikai módszerek gyors fejlődése egyre inkább
Nagy Emese: Polimorfizmus és rokonsági körök vizsgálata kukoricában (Zea mays) Témavezetők: Cs. L. Marton G Gyulai BEVEZETÉS A molekuláris biológiai és genetikai módszerek gyors fejlődése egyre inkább
A genomikai oktatás helyzete a Debreceni Egyetemen
 A genomikai oktatás helyzete a Debreceni Egyetemen Bálint Bálint L. GNTP Oktatás és Tudásmenedzsment Munkabizottság, 2009. június 10. Tények Debreceni Egyetemről 21000 nappali és 33000 összes hallgató
A genomikai oktatás helyzete a Debreceni Egyetemen Bálint Bálint L. GNTP Oktatás és Tudásmenedzsment Munkabizottság, 2009. június 10. Tények Debreceni Egyetemről 21000 nappali és 33000 összes hallgató
NÖVÉNYGENETIKA. Az Agrármérnöki MSc szak tananyagfejlesztése TÁMOP /1/A
 NÖVÉNYGENETIKA Az Agrármérnöki MSc szak tananyagfejlesztése TÁMOP-4.1.2-08/1/A-2009-0010 A citológia és a genetika társtudománya Citogenetika A kromoszómák eredetét, szerkezetét, genetikai funkcióját,
NÖVÉNYGENETIKA Az Agrármérnöki MSc szak tananyagfejlesztése TÁMOP-4.1.2-08/1/A-2009-0010 A citológia és a genetika társtudománya Citogenetika A kromoszómák eredetét, szerkezetét, genetikai funkcióját,
A humán mitokondriális genom: Evolúció, mutációk, polimorfizmusok, populációs vonatkozások. Egyed Balázs ELTE Genetikai Tanszék
 A humán mitokondriális genom: Evolúció, mutációk, polimorfizmusok, populációs vonatkozások Egyed Balázs ELTE Genetikai Tanszék Endoszimbiotikus gén-transzfer (Timmis et al., 2004, Nat Rev Gen) Endoszimbiotikus
A humán mitokondriális genom: Evolúció, mutációk, polimorfizmusok, populációs vonatkozások Egyed Balázs ELTE Genetikai Tanszék Endoszimbiotikus gén-transzfer (Timmis et al., 2004, Nat Rev Gen) Endoszimbiotikus
Bioinformatika - egészséges környezet, egészséges élelmiszer
 CESCI - III. SZENTGOTTHÁRDI SZLOVÉN MAGYAR FÓRUM Bioinformatika - egészséges környezet, egészséges élelmiszer Pannon Bio-Innováció Kft Taller János, PhD ügyvezető Szentgotthárd, 2017. május 23. 1 Pannon
CESCI - III. SZENTGOTTHÁRDI SZLOVÉN MAGYAR FÓRUM Bioinformatika - egészséges környezet, egészséges élelmiszer Pannon Bio-Innováció Kft Taller János, PhD ügyvezető Szentgotthárd, 2017. május 23. 1 Pannon
SZAKMAI ZÁRÓJELENTÉS
 SZAKMAI ZÁRÓJELENTÉS a Solanum stoloniferum alapú PVY immunitás gén (Rysto) molekuláris genetikai vizsgálata című tematikus OTKA pályázatról Nyilvántartási szám: TO37827 Keszthely 2002-2005. Zárójelentés
SZAKMAI ZÁRÓJELENTÉS a Solanum stoloniferum alapú PVY immunitás gén (Rysto) molekuláris genetikai vizsgálata című tematikus OTKA pályázatról Nyilvántartási szám: TO37827 Keszthely 2002-2005. Zárójelentés
A SZIE, MKK GENETIKA ÉS BIOTECHNOLÓGIAI INTÉZETÉNEK EREDMÉNYEI A NÖVÉNYNEMESÍTÉS TUDOMÁNYOKBAN ÉS A NEMESÍTÉS UTÁNPÓTLÁS NEVELÉSBEN
 A SZIE, MKK GENETIKA ÉS BIOTECHNOLÓGIAI INTÉZETÉNEK EREDMÉNYEI A NÖVÉNYNEMESÍTÉS TUDOMÁNYOKBAN ÉS A NEMESÍTÉS UTÁNPÓTLÁS NEVELÉSBEN Heszky László és Kiss Erzsébet Keszthely 2013 PARADIGMAVÁLTÁS AZ ÉLETTUDOMÁNYOKBAN
A SZIE, MKK GENETIKA ÉS BIOTECHNOLÓGIAI INTÉZETÉNEK EREDMÉNYEI A NÖVÉNYNEMESÍTÉS TUDOMÁNYOKBAN ÉS A NEMESÍTÉS UTÁNPÓTLÁS NEVELÉSBEN Heszky László és Kiss Erzsébet Keszthely 2013 PARADIGMAVÁLTÁS AZ ÉLETTUDOMÁNYOKBAN
A Hardy Weinberg-modell gyakorlati alkalmazása
 1 of 6 5/16/2009 2:59 PM A Hardy Weinberg-modell gyakorlati alkalmazása A genotípus-gyakoriság megoszlásának vizsgálata 1. ábra. A Hardy Weinberg-egyensúlyi genotípus-gyakoriságok az allélgyakoriság Számos
1 of 6 5/16/2009 2:59 PM A Hardy Weinberg-modell gyakorlati alkalmazása A genotípus-gyakoriság megoszlásának vizsgálata 1. ábra. A Hardy Weinberg-egyensúlyi genotípus-gyakoriságok az allélgyakoriság Számos
Nincs öntermékenyítés, de a véges méret miatt a párosodó egyedek bizonyos valószínűséggel rokonok, ezért kerül egy
 Véges populációméret okozta beltenyésztettség incs öntermékenyítés, de a véges méret miatt a párosodó egyedek bizonyos valószínűséggel rokonok, ezért kerül egy utódba 2 IBD allél Előadásról: -F t (-/2)
Véges populációméret okozta beltenyésztettség incs öntermékenyítés, de a véges méret miatt a párosodó egyedek bizonyos valószínűséggel rokonok, ezért kerül egy utódba 2 IBD allél Előadásról: -F t (-/2)
Sodródás Evolúció neutrális elmélete
 Sodródás Evolúció neutrális elmélete Egy kísérlet Drosophila Drosophila pseudoobscura 8 hím + 8 nőstény/tenyészet 107 darab tenyészet Minden tenyészet csak heterozigóta egyedekkel indul a neutrális szemszín
Sodródás Evolúció neutrális elmélete Egy kísérlet Drosophila Drosophila pseudoobscura 8 hím + 8 nőstény/tenyészet 107 darab tenyészet Minden tenyészet csak heterozigóta egyedekkel indul a neutrális szemszín
10. Genomika 2. Microarrayek és típusaik
 10. Genomika 2. 1. Microarray technikák és bioinformatikai vonatkozásaik Microarrayek és típusaik Korrelált génexpresszió mint a funkcionális genomika eszköze 2. Kombinált megközelítés a funkcionális genomikában
10. Genomika 2. 1. Microarray technikák és bioinformatikai vonatkozásaik Microarrayek és típusaik Korrelált génexpresszió mint a funkcionális genomika eszköze 2. Kombinált megközelítés a funkcionális genomikában
Kromoszómák, Gének centromer
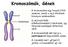 Kromoszómák, Gének A kromoszóma egy hosszú DNS szakasz, amely a sejt életének bizonyos szakaszában (a sejtosztódás előkészítéseként) tömörödik, így fénymikroszkóppal láthatóvá válik. A kromoszómák két
Kromoszómák, Gének A kromoszóma egy hosszú DNS szakasz, amely a sejt életének bizonyos szakaszában (a sejtosztódás előkészítéseként) tömörödik, így fénymikroszkóppal láthatóvá válik. A kromoszómák két
BEVEZETÉS CÉLKITŰZÉS
 BEVEZETÉS A molekuláris biológiai és genetikai módszerek gyors fejlődése egyre inkább tért hódít a növénynemesítés különböző területein, így a kukoricanemesítésben is. A növényi fenotípusos jellemzők és
BEVEZETÉS A molekuláris biológiai és genetikai módszerek gyors fejlődése egyre inkább tért hódít a növénynemesítés különböző területein, így a kukoricanemesítésben is. A növényi fenotípusos jellemzők és
ÁRPA KROMOSZÓMÁK AZONOSÍTÁSA ÚJ BÚZA X ÁRPA HIBRIDEK UTÓDVONALAIBAN IN SITU HIBRIDIZÁCIÓ ÉS SSR- MARKEREK SEGÍTSÉGÉVEL
 Hagyomány és haladás a növénynemesítésben ÁRPA KROMOSZÓMÁK AZONOSÍTÁSA ÚJ BÚZA X ÁRPA HIBRIDEK UTÓDVONALAIBAN IN SITU HIBRIDIZÁCIÓ ÉS SSR- MARKEREK SEGÍTSÉGÉVEL KRUPPA KLAUDIA, CSEH ANDRÁS ÉS LÁNGNÉ MOLNÁR
Hagyomány és haladás a növénynemesítésben ÁRPA KROMOSZÓMÁK AZONOSÍTÁSA ÚJ BÚZA X ÁRPA HIBRIDEK UTÓDVONALAIBAN IN SITU HIBRIDIZÁCIÓ ÉS SSR- MARKEREK SEGÍTSÉGÉVEL KRUPPA KLAUDIA, CSEH ANDRÁS ÉS LÁNGNÉ MOLNÁR
AZ ÁRPA SZÁRAZSÁGTŰRÉSÉNEK VIZSGÁLATA: QTL- ÉS ASSZOCIÁCIÓS ANALÍZIS, MARKER ALAPÚ SZELEKCIÓ, TILLING
 Hagyomány és haladás a növénynemesítésben AZ ÁRPA SZÁRAZSÁGTŰRÉSÉNEK VIZSGÁLATA: QTL- ÉS ASSZOCIÁCIÓS ANALÍZIS, MARKER ALAPÚ SZELEKCIÓ, BÁLINT ANDRÁS FERENC 1, SZIRA FRUZSINA 1, ANDREAS BÖRNER 2, KERSTIN
Hagyomány és haladás a növénynemesítésben AZ ÁRPA SZÁRAZSÁGTŰRÉSÉNEK VIZSGÁLATA: QTL- ÉS ASSZOCIÁCIÓS ANALÍZIS, MARKER ALAPÚ SZELEKCIÓ, BÁLINT ANDRÁS FERENC 1, SZIRA FRUZSINA 1, ANDREAS BÖRNER 2, KERSTIN
DNS-szekvencia meghatározás
 DNS-szekvencia meghatározás Gilbert 1980 (1958) Sanger 3-1 A DNS-polimerázok jellemzői 5'-3' polimeráz aktivitás 5'-3' exonukleáz 3'-5' exonukleáz aktivitás Az új szál szintéziséhez kell: templát DNS primer
DNS-szekvencia meghatározás Gilbert 1980 (1958) Sanger 3-1 A DNS-polimerázok jellemzői 5'-3' polimeráz aktivitás 5'-3' exonukleáz 3'-5' exonukleáz aktivitás Az új szál szintéziséhez kell: templát DNS primer
Diagnosztikai célú molekuláris biológiai vizsgálatok
 Diagnosztikai célú molekuláris biológiai vizsgálatok Dr. Patócs Attila, PhD MTA-SE Molekuláris Medicina Kutatócsoport, Semmelweis Egyetem II. sz. Belgyógyászati Klinika Laboratóriumi Medicina Intézet Genetikai
Diagnosztikai célú molekuláris biológiai vizsgálatok Dr. Patócs Attila, PhD MTA-SE Molekuláris Medicina Kutatócsoport, Semmelweis Egyetem II. sz. Belgyógyászati Klinika Laboratóriumi Medicina Intézet Genetikai
Szakmai zárójelentés
 Szakmai zárójelentés Kései szárazságtűrésben szerepet játszó génjelöltek asszociációs vizsgálata árpában EcoTILLING módszerrel című OTKA pályázat (OTKA azonosító: 72366) Kutatásaink elsődleges célja a
Szakmai zárójelentés Kései szárazságtűrésben szerepet játszó génjelöltek asszociációs vizsgálata árpában EcoTILLING módszerrel című OTKA pályázat (OTKA azonosító: 72366) Kutatásaink elsődleges célja a
A Leuce szekcióba tartozó hazai nyárak dunántúli természetes eredetű állományainak populációgenetikai vizsgálata
 Nyugat-magyarországi Egyetem Erdőmérnöki Kar Doktori (PhD) értekezés tézisei A Leuce szekcióba tartozó hazai nyárak dunántúli természetes eredetű állományainak populációgenetikai vizsgálata Benke Attila
Nyugat-magyarországi Egyetem Erdőmérnöki Kar Doktori (PhD) értekezés tézisei A Leuce szekcióba tartozó hazai nyárak dunántúli természetes eredetű állományainak populációgenetikai vizsgálata Benke Attila
Szent István Egyetem. A kenyérbúza (Triticum aestivum L.) környezeti adaptációját meghatározó genetikai komponensek vizsgálata
 Szent István Egyetem A kenyérbúza (Triticum aestivum L.) környezeti adaptációját meghatározó genetikai komponensek vizsgálata Doktori (PhD) értekezés tézisei Kiss Tibor Martonvásár 2016 A doktori iskola
Szent István Egyetem A kenyérbúza (Triticum aestivum L.) környezeti adaptációját meghatározó genetikai komponensek vizsgálata Doktori (PhD) értekezés tézisei Kiss Tibor Martonvásár 2016 A doktori iskola
Az orvosi biotechnológiai mesterképzés megfeleltetése az Európai Unió új társadalmi kihívásainak a Pécsi Tudományegyetemen és a Debreceni Egyetemen
 Az orvosi biotechnológiai mesterképzés megfeleltetése az Európai Unió új társadalmi kihívásainak a Pécsi Tudományegyetemen és a Debreceni Egyetemen Azonosító szám: TÁMOP-4.1.2-08/1/A-2009-0011 Az orvosi
Az orvosi biotechnológiai mesterképzés megfeleltetése az Európai Unió új társadalmi kihívásainak a Pécsi Tudományegyetemen és a Debreceni Egyetemen Azonosító szám: TÁMOP-4.1.2-08/1/A-2009-0011 Az orvosi
Bakteriális identifikáció 16S rrns gén szekvencia alapján
 Bakteriális identifikáció 16S rrns gén szekvencia alapján MOHR ANITA SIPOS RITA, SZÁNTÓ-EGÉSZ RÉKA, MICSINAI ADRIENN 2100 Gödöllő, Szent-Györgyi Albert út 4. info@biomi.hu, www.biomi.hu TÖRZS AZONOSÍTÁS
Bakteriális identifikáció 16S rrns gén szekvencia alapján MOHR ANITA SIPOS RITA, SZÁNTÓ-EGÉSZ RÉKA, MICSINAI ADRIENN 2100 Gödöllő, Szent-Györgyi Albert út 4. info@biomi.hu, www.biomi.hu TÖRZS AZONOSÍTÁS
Humán genom variációk single nucleotide polymorphism (SNP)
 Humán genom variációk single nucleotide polymorphism (SNP) A genom ~ 97 %-a két különböző egyedben teljesen azonos ~ 1% különbség: SNP miatt ~2% különbség: kópiaszámbeli eltérés, deléciók miatt 11-12 millió
Humán genom variációk single nucleotide polymorphism (SNP) A genom ~ 97 %-a két különböző egyedben teljesen azonos ~ 1% különbség: SNP miatt ~2% különbség: kópiaszámbeli eltérés, deléciók miatt 11-12 millió
Őszi búzafajták magas hozamának megőrzése környezeti stressz hatása alatt
 Őszi búzafajták magas hozamának megőrzése környezeti stressz hatása alatt WHEATSTR OM-00887/2009 Záró Jelentés: Időszak: 2009. október 1. - 2011. szeptember 30. Támogatott szervezet: Vetőmag Szövetség
Őszi búzafajták magas hozamának megőrzése környezeti stressz hatása alatt WHEATSTR OM-00887/2009 Záró Jelentés: Időszak: 2009. október 1. - 2011. szeptember 30. Támogatott szervezet: Vetőmag Szövetség
y ij = µ + α i + e ij
 Elmélet STATISZTIKA 3. Előadás Variancia-analízis Lineáris modellek A magyarázat a függő változó teljes heterogenitásának két részre bontását jelenti. A teljes heterogenitás egyik része az, amelynek okai
Elmélet STATISZTIKA 3. Előadás Variancia-analízis Lineáris modellek A magyarázat a függő változó teljes heterogenitásának két részre bontását jelenti. A teljes heterogenitás egyik része az, amelynek okai
Növényvédelmi Tudományos Napok 2014
 Növényvédelmi Tudományos Napok 2014 Budapest 60. NÖVÉNYVÉDELMI TUDOMÁNYOS NAPOK Szerkesztők HORVÁTH JÓZSEF HALTRICH ATTILA MOLNÁR JÁNOS Budapest 2014. február 18-19. ii Szerkesztőbizottság Tóth Miklós
Növényvédelmi Tudományos Napok 2014 Budapest 60. NÖVÉNYVÉDELMI TUDOMÁNYOS NAPOK Szerkesztők HORVÁTH JÓZSEF HALTRICH ATTILA MOLNÁR JÁNOS Budapest 2014. február 18-19. ii Szerkesztőbizottság Tóth Miklós
A Multi Locus Sequence Typing (MLST) alkalmazhatósága az élelmiszermikrobiológiában
 A Multi Locus Sequence Typing (MLST) alkalmazhatósága az élelmiszermikrobiológiában Sipos Rita, Lukács Alena, Simon Janka, Szántó-Egész Réka, Micsinai Adrienn 2100 Gödöllő, Szent-Györgyi Albert út 4. info@biomi.hu,
A Multi Locus Sequence Typing (MLST) alkalmazhatósága az élelmiszermikrobiológiában Sipos Rita, Lukács Alena, Simon Janka, Szántó-Egész Réka, Micsinai Adrienn 2100 Gödöllő, Szent-Györgyi Albert út 4. info@biomi.hu,
Természetvédelmi biológia
 Természetvédelmi biológia 1. A természetvédelmi biológia meghatározása, a biológiai sokféleség értelmezése A természetvédelmi biológia (konzervációbiológia) fı céljai 1. Az emberi tevékenység fajok populációra,
Természetvédelmi biológia 1. A természetvédelmi biológia meghatározása, a biológiai sokféleség értelmezése A természetvédelmi biológia (konzervációbiológia) fı céljai 1. Az emberi tevékenység fajok populációra,
Kvantitatív genetikai alapok április
 Kvantitatív genetikai alapok 2018. április A vizsgálható tulajdonságok köre: egyed - szám Egyedek morfológiai tulajdonságai: testméretek, arányok, testtömeg Egyedek fiziológiai tulajdonságai: vérnyomás,
Kvantitatív genetikai alapok 2018. április A vizsgálható tulajdonságok köre: egyed - szám Egyedek morfológiai tulajdonságai: testméretek, arányok, testtömeg Egyedek fiziológiai tulajdonságai: vérnyomás,
Biomolekuláris nanotechnológia. Vonderviszt Ferenc PE MÜKKI Bio-Nanorendszerek Laboratórium
 Biomolekuláris nanotechnológia Vonderviszt Ferenc PE MÜKKI Bio-Nanorendszerek Laboratórium Az élő szervezetek példája azt mutatja, hogy a fehérjék és nukleinsavak kiválóan alkalmasak önszerveződő molekuláris
Biomolekuláris nanotechnológia Vonderviszt Ferenc PE MÜKKI Bio-Nanorendszerek Laboratórium Az élő szervezetek példája azt mutatja, hogy a fehérjék és nukleinsavak kiválóan alkalmasak önszerveződő molekuláris
Genetikus algoritmusok
 Genetikus algoritmusok Zsolnai Károly - BME CS zsolnai@cs.bme.hu Keresőalgoritmusok osztályai Véletlent használó algoritmusok Keresőalgoritmusok Kimerítő algoritmusok Dinamikus programozás BFS DFS Tabu
Genetikus algoritmusok Zsolnai Károly - BME CS zsolnai@cs.bme.hu Keresőalgoritmusok osztályai Véletlent használó algoritmusok Keresőalgoritmusok Kimerítő algoritmusok Dinamikus programozás BFS DFS Tabu
Dr. Máthéné Dr. Szigeti Zsuzsanna és munkatársai
 Kar: TTK Tantárgy: CITOGENETIKA Kód: AOMBCGE3 ECTS Kredit: 3 A tantárgyat oktató intézet: TTK Mikrobiális Biotechnológiai és Sejtbiológiai Tanszék A tantárgy felvételére ajánlott félév: 3. Melyik félévben
Kar: TTK Tantárgy: CITOGENETIKA Kód: AOMBCGE3 ECTS Kredit: 3 A tantárgyat oktató intézet: TTK Mikrobiális Biotechnológiai és Sejtbiológiai Tanszék A tantárgy felvételére ajánlott félév: 3. Melyik félévben
NÖVÉNYNEMESÍTÉS. Az Agrármérnöki MSc szak tananyagfejlesztése TÁMOP /1/A
 NÖVÉNYNEMESÍTÉS Az Agrármérnöki MSc szak tananyagfejlesztése TÁMOP-4.1.2-08/1/A-2009-0010 Előadás áttekintése Növénynemesítés fogalma és célja Növénynemesítés feladatai Növénynemesítés társtudományai A
NÖVÉNYNEMESÍTÉS Az Agrármérnöki MSc szak tananyagfejlesztése TÁMOP-4.1.2-08/1/A-2009-0010 Előadás áttekintése Növénynemesítés fogalma és célja Növénynemesítés feladatai Növénynemesítés társtudományai A
4. EREDMÉNYEK EREDMÉNYEK 46
 EREDMÉNYEK 46 4. EREDMÉNYEK 4.1. Gabona genotípusok réztoleranciájának vizsgálata A Martonvásári Gabona Génbankban tárolt búza és búzarokonfajok réztoleranciájának tesztelésével célunk Cu-toleráns és Cu-érzékeny
EREDMÉNYEK 46 4. EREDMÉNYEK 4.1. Gabona genotípusok réztoleranciájának vizsgálata A Martonvásári Gabona Génbankban tárolt búza és búzarokonfajok réztoleranciájának tesztelésével célunk Cu-toleráns és Cu-érzékeny
Diverzitás és stabilitás. Mi a biodiverzitás?
 Diverzitás és stabilitás Szemethy László, Phd egyetemi docens SZIE VMI Szemethy.Laszlo@MKK.SZIE.hu Mi a biodiverzitás? a sokféleség sokfélesége (JNP) tudományos paradigma (tudományterület is) a diverzitás
Diverzitás és stabilitás Szemethy László, Phd egyetemi docens SZIE VMI Szemethy.Laszlo@MKK.SZIE.hu Mi a biodiverzitás? a sokféleség sokfélesége (JNP) tudományos paradigma (tudományterület is) a diverzitás
Biológiai feladatbank 12. évfolyam
 Biológiai feladatbank 12. évfolyam A pedagógus neve: A pedagógus szakja: Az iskola neve: Műveltségi terület: Tantárgy: A tantárgy cél és feladatrendszere: Tantárgyi kapcsolatok: Osztály: 12. Felhasznált
Biológiai feladatbank 12. évfolyam A pedagógus neve: A pedagógus szakja: Az iskola neve: Műveltségi terület: Tantárgy: A tantárgy cél és feladatrendszere: Tantárgyi kapcsolatok: Osztály: 12. Felhasznált
DNS viszgálatok, számítási módszerek
 DNS viszgálatok, számítási módszerek Apasági vizsgálatok Kizárás: -a gyereknél az apától örökölt allél nem egyezik a feltételezett apáéval - 3 kizárás esetén az apaság kizárható -100% Anya: 12-13, kk.
DNS viszgálatok, számítási módszerek Apasági vizsgálatok Kizárás: -a gyereknél az apától örökölt allél nem egyezik a feltételezett apáéval - 3 kizárás esetén az apaság kizárható -100% Anya: 12-13, kk.
KÖZPONTI STATISZTIKAI HIVATAL. Szóbeli vizsgatevékenység
 KÖZPONTI STATISZTIKAI HIVATAL A vizsgarészhez rendelt követelménymodul azonosító száma, megnevezése: 2144-06 Statisztikai szervezői és elemzési feladatok A vizsgarészhez rendelt vizsgafeladat megnevezése:
KÖZPONTI STATISZTIKAI HIVATAL A vizsgarészhez rendelt követelménymodul azonosító száma, megnevezése: 2144-06 Statisztikai szervezői és elemzési feladatok A vizsgarészhez rendelt vizsgafeladat megnevezése:
2. SZ. SZAKMAI ÖSSZEFOGLALÓ PIR 2
 Az Országos Onkológiai Intézet Norvég Finanszírozási mechanizmus keretében elnyert Kutatás Fejlesztési Pályázata Közös stratégia kifejlesztése molekuláris módszerek alkalmazásával a rák kezelésére Magyarországon
Az Országos Onkológiai Intézet Norvég Finanszírozási mechanizmus keretében elnyert Kutatás Fejlesztési Pályázata Közös stratégia kifejlesztése molekuláris módszerek alkalmazásával a rák kezelésére Magyarországon
Altruizmus. Altruizmus: a viselkedés az adott egyed fitneszét csökkenti, de másik egyed(ek)ét növeli. Lehet-e önző egyedek között?
 Altruizmus Altruizmus: a viselkedés az adott egyed fitneszét csökkenti, de másik egyed(ek)ét növeli. Lehet-e önző egyedek között? Altruizmus rokonok között A legtöbb másolat az adott génről vagy az egyed
Altruizmus Altruizmus: a viselkedés az adott egyed fitneszét csökkenti, de másik egyed(ek)ét növeli. Lehet-e önző egyedek között? Altruizmus rokonok között A legtöbb másolat az adott génről vagy az egyed
Prenatalis diagnosztika lehetőségei mikor, hogyan, miért? Dr. Almássy Zsuzsanna Heim Pál Kórház, Budapest Toxikológia és Anyagcsere Osztály
 Prenatalis diagnosztika lehetőségei mikor, hogyan, miért? Dr. Almássy Zsuzsanna Heim Pál Kórház, Budapest Toxikológia és Anyagcsere Osztály Definíció A prenatális diagnosztika a klinikai genetika azon
Prenatalis diagnosztika lehetőségei mikor, hogyan, miért? Dr. Almássy Zsuzsanna Heim Pál Kórház, Budapest Toxikológia és Anyagcsere Osztály Definíció A prenatális diagnosztika a klinikai genetika azon
molekuláris (RAPD, SSR, AFLP és SCAR) jellemzése.
 1 Különböző nyár, búza és kender genotípusok molekuláris (RAPD, SSR, AFLP és SCAR) jellemzése. Doktori értekezés Törjék Ottó Gödöllő 2001. 2 Doktori iskola: Vezetője: Tudományága: Program: Programvezető:
1 Különböző nyár, búza és kender genotípusok molekuláris (RAPD, SSR, AFLP és SCAR) jellemzése. Doktori értekezés Törjék Ottó Gödöllő 2001. 2 Doktori iskola: Vezetője: Tudományága: Program: Programvezető:
Altruizmus. Altruizmus: a viselkedés az adott egyed fitneszét csökkenti, de másik egyed(ek)ét növeli. Lehet-e önző egyedek között?
 Altruizmus Altruizmus: a viselkedés az adott egyed fitneszét csökkenti, de másik egyed(ek)ét növeli. Lehet-e önző egyedek között? Altruizmus rokonok között A legtöbb másolat az adott génről vagy az egyed
Altruizmus Altruizmus: a viselkedés az adott egyed fitneszét csökkenti, de másik egyed(ek)ét növeli. Lehet-e önző egyedek között? Altruizmus rokonok között A legtöbb másolat az adott génről vagy az egyed
10. CSI. A molekuláris biológiai technikák alkalmazásai
 10. CSI. A molekuláris biológiai technikák alkalmazásai A DNS mint azonosító 3 milliárd bázispár az emberi DNS-ben (99.9%-ban azonos) 0.1%-nyi különbség elegendő az egyedek megkülönböztetéséhez Genetikai
10. CSI. A molekuláris biológiai technikák alkalmazásai A DNS mint azonosító 3 milliárd bázispár az emberi DNS-ben (99.9%-ban azonos) 0.1%-nyi különbség elegendő az egyedek megkülönböztetéséhez Genetikai
Prof. Dr. Szabad János Tantárgyfelelős beosztása
 Tantárgy neve Genetika Tantárgy kódja BIB 1506 Meghírdetés féléve 5 Kreditpont 4 Összóraszám (elmélet + gyakorlat) 3+0 Számonkérés módja Kollokvium Előfeltétel (tantárgyi kód) BIB 1411 Tantárgyfelelős
Tantárgy neve Genetika Tantárgy kódja BIB 1506 Meghírdetés féléve 5 Kreditpont 4 Összóraszám (elmélet + gyakorlat) 3+0 Számonkérés módja Kollokvium Előfeltétel (tantárgyi kód) BIB 1411 Tantárgyfelelős
Norvég Finanszírozási Mechanizmus által támogatott projekt HU-0115/NA/2008-3/ÖP-9 ÚJ TERÁPIÁS CÉLPONTOK AZONOSÍTÁSA GENOMIKAI MÓDSZEREKKEL
 Norvég Finanszírozási Mechanizmus által támogatott projekt HU-0115/NA/2008-3/ÖP-9 ÚJ TERÁPIÁS CÉLPONTOK AZONOSÍTÁSA GENOMIKAI MÓDSZEREKKEL KÖZÖS STRATÉGIA KIFEJLESZTÉSE MOLEKULÁRIS MÓDSZEREK ALKALMAZÁSÁVAL
Norvég Finanszírozási Mechanizmus által támogatott projekt HU-0115/NA/2008-3/ÖP-9 ÚJ TERÁPIÁS CÉLPONTOK AZONOSÍTÁSA GENOMIKAI MÓDSZEREKKEL KÖZÖS STRATÉGIA KIFEJLESZTÉSE MOLEKULÁRIS MÓDSZEREK ALKALMAZÁSÁVAL
Genetikai kölcsönhatások rendszerbiológiája
 Genetikai kölcsönhatások rendszerbiológiája Papp Balázs www.brc.hu/sysbiol MTA Szegedi Biológiai Kutatóközpont Biokémiai Intézet Szintetikus és Rendszerbiológiai Egység Mikrobiális rendszerbiológia főbb
Genetikai kölcsönhatások rendszerbiológiája Papp Balázs www.brc.hu/sysbiol MTA Szegedi Biológiai Kutatóközpont Biokémiai Intézet Szintetikus és Rendszerbiológiai Egység Mikrobiális rendszerbiológia főbb
Szakmai zárójelentés. 1. Strukturális genomika Az RPS13 gén szekvenciájához homológ régió deléciója M. sativa-ban
 Szakmai zárójelentés 1. Strukturális genomika 1. 1. Az RPS13 gén szekvenciájához homológ régió deléciója M. sativa-ban A diploid lucerna (Medicago sativa) genetikai térképének megszerkesztése során csoportunk
Szakmai zárójelentés 1. Strukturális genomika 1. 1. Az RPS13 gén szekvenciájához homológ régió deléciója M. sativa-ban A diploid lucerna (Medicago sativa) genetikai térképének megszerkesztése során csoportunk
A kromoszómák kialakulása előtt a DNS állomány megkettőződik. A két azonos információ tartalmú DNS egymás mellé rendeződik és egy kromoszómát alkot.
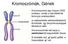 Kromoszómák, Gének A kromoszóma egy hosszú DNS szakasz, amely a sejt életének bizonyos szakaszában (a sejtosztódás előkészítéseként) tömörödik, így fénymikroszkóppal láthatóvá válik. A kromoszómák két
Kromoszómák, Gének A kromoszóma egy hosszú DNS szakasz, amely a sejt életének bizonyos szakaszában (a sejtosztódás előkészítéseként) tömörödik, így fénymikroszkóppal láthatóvá válik. A kromoszómák két
Etológia. Irányzatok a biológiában. Pongrácz Péter, PhD Etológia Tanszék
 Etológia Irányzatok a biológiában Pongrácz Péter, PhD Etológia Tanszék Etológia helye a biológia vizsgálódás szintjei szerint Szerveződési szintek Populációk Egyedek Neurális szabályozás Sejtek DNS Tudományág
Etológia Irányzatok a biológiában Pongrácz Péter, PhD Etológia Tanszék Etológia helye a biológia vizsgálódás szintjei szerint Szerveződési szintek Populációk Egyedek Neurális szabályozás Sejtek DNS Tudományág
Statisztika I. 11. előadás. Előadó: Dr. Ertsey Imre
 Statisztika I. 11. előadás Előadó: Dr. Ertsey Imre Összefüggés vizsgálatok A társadalmi gazdasági élet jelenségei kölcsönhatásban állnak, összefüggnek egymással. Statisztika alapvető feladata: - tényszerűségek
Statisztika I. 11. előadás Előadó: Dr. Ertsey Imre Összefüggés vizsgálatok A társadalmi gazdasági élet jelenségei kölcsönhatásban állnak, összefüggnek egymással. Statisztika alapvető feladata: - tényszerűségek
2006/1. XVIII. évfolyam 1. szám AZ MTA MEZÕGAZDASÁGI KUTATÓINTÉZETÉNEK KÖZLEMÉNYEI
 2006/1 XVIII. évfolyam 1. szám AZ MTA MEZÕGAZDASÁGI KUTATÓINTÉZETÉNEK KÖZLEMÉNYEI 2 2006/1 Eseménynaptár Bemutatók Július 14 15-én nagy érdeklõdés kísérte az Országos Kalászos Gabona Bemutatót. A közel
2006/1 XVIII. évfolyam 1. szám AZ MTA MEZÕGAZDASÁGI KUTATÓINTÉZETÉNEK KÖZLEMÉNYEI 2 2006/1 Eseménynaptár Bemutatók Július 14 15-én nagy érdeklõdés kísérte az Országos Kalászos Gabona Bemutatót. A közel
Adatelemzési eljárások az idegrendszer kutatásban Somogyvári Zoltán
 Adatelemzési eljárások az idegrendszer kutatásban Somogyvári Zoltán MTA KFKI Részecske és Magfizikai Intézet, Biofizikai osztály Az egy adatsorra (idősorra) is alkalmazható módszerek Példa: Az epileptikus
Adatelemzési eljárások az idegrendszer kutatásban Somogyvári Zoltán MTA KFKI Részecske és Magfizikai Intézet, Biofizikai osztály Az egy adatsorra (idősorra) is alkalmazható módszerek Példa: Az epileptikus
[Biomatematika 2] Orvosi biometria
![[Biomatematika 2] Orvosi biometria [Biomatematika 2] Orvosi biometria](/thumbs/54/33134304.jpg) [Biomatematika 2] Orvosi biometria 2016.02.29. A statisztika típusai Leíró jellegű statisztika: összegzi egy adathalmaz jellemzőit. A középértéket jelemzi (medián, módus, átlag) Az adatok változékonyságát
[Biomatematika 2] Orvosi biometria 2016.02.29. A statisztika típusai Leíró jellegű statisztika: összegzi egy adathalmaz jellemzőit. A középértéket jelemzi (medián, módus, átlag) Az adatok változékonyságát
PÉCSI TUDOMÁNYEGYETEM. Agrobacterium rezisztencia térképezése szőlőben. Kuczmog Anett
 PÉCSI TUDOMÁNYEGYETEM Biológia Doktori Iskola Genetika program Agrobacterium rezisztencia térképezése szőlőben PhD értekezés Kuczmog Anett Témavezető: Dr. Putnoky Péter egyetemi tanár PÉCS, 2012. 1. BEVEZETÉS
PÉCSI TUDOMÁNYEGYETEM Biológia Doktori Iskola Genetika program Agrobacterium rezisztencia térképezése szőlőben PhD értekezés Kuczmog Anett Témavezető: Dr. Putnoky Péter egyetemi tanár PÉCS, 2012. 1. BEVEZETÉS
NÖVÉNYNEMESÍTÉS. Az Agrármérnöki MSc szak tananyagfejlesztése TÁMOP /1/A
 NÖVÉNYNEMESÍTÉS Az Agrármérnöki MSc szak tananyagfejlesztése TÁMOP-4.1.2-08/1/A-2009-0010 Előadás áttekintése A növénynemesítés és molekuláris biológia története A biotechnológia jelentősége a növénynemesítésben
NÖVÉNYNEMESÍTÉS Az Agrármérnöki MSc szak tananyagfejlesztése TÁMOP-4.1.2-08/1/A-2009-0010 Előadás áttekintése A növénynemesítés és molekuláris biológia története A biotechnológia jelentősége a növénynemesítésben
A magyar populáció genetikai elemzése nemi kromoszómális markerek alapján
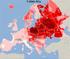 A magyar populáció genetikai elemzése nemi kromoszómális markerek alapján DOKTORI ÉRTEKEZÉS TÉZISEI Készítette: Vágó-Zalán Andrea ELTE TTK Biológia Doktori Iskola Iskolavezető: Prof. Dr. Erdei Anna Klasszikus
A magyar populáció genetikai elemzése nemi kromoszómális markerek alapján DOKTORI ÉRTEKEZÉS TÉZISEI Készítette: Vágó-Zalán Andrea ELTE TTK Biológia Doktori Iskola Iskolavezető: Prof. Dr. Erdei Anna Klasszikus
A hazai dohánytermesztés biológiai alapjai
 A hazai dohánytermesztés biológiai alapjai Dr. Varga Lajos Agroport-D Kft. A hazai dohánytermesztés biológiai alapjai Fajtanemesítés Fajtaelismerés Hazai nemesítésű fajták, fajtahasználat Vetőmagtermesztés
A hazai dohánytermesztés biológiai alapjai Dr. Varga Lajos Agroport-D Kft. A hazai dohánytermesztés biológiai alapjai Fajtanemesítés Fajtaelismerés Hazai nemesítésű fajták, fajtahasználat Vetőmagtermesztés
Egy mezofil lomberdei faj, a szártalan kankalin (Primula vulgaris Huds.) európai léptékű filogeográfiája, különös tekintettel a Kárpát-medencére
 Egy mezofil lomberdei faj, a szártalan kankalin (Primula vulgaris Huds.) európai léptékű filogeográfiája, különös tekintettel a Kárpát-medencére Laczkó Levente Témavezető: Sramkó Gábor posztdoktori kutató
Egy mezofil lomberdei faj, a szártalan kankalin (Primula vulgaris Huds.) európai léptékű filogeográfiája, különös tekintettel a Kárpát-medencére Laczkó Levente Témavezető: Sramkó Gábor posztdoktori kutató
Domináns-recesszív öröklődésmenet
 Domináns-recesszív öröklődésmenet Domináns recesszív öröklődés esetén tehát a homozigóta domináns és a heterozigóta egyedek fenotípusa megegyezik, így a három lehetséges genotípushoz (példánkban AA, Aa,
Domináns-recesszív öröklődésmenet Domináns recesszív öröklődés esetén tehát a homozigóta domináns és a heterozigóta egyedek fenotípusa megegyezik, így a három lehetséges genotípushoz (példánkban AA, Aa,
Mintavétel fogalmai STATISZTIKA, BIOMETRIA. Mintavételi hiba. Statisztikai adatgyűjtés. Nem véletlenen alapuló kiválasztás
 STATISZTIKA, BIOMETRIA. Előadás Mintavétel, mintavételi technikák, adatbázis Mintavétel fogalmai A mintavételt meg kell tervezni A sokaság elemei: X, X X N, lehet véges és végtelen Mintaelemek: x, x x
STATISZTIKA, BIOMETRIA. Előadás Mintavétel, mintavételi technikák, adatbázis Mintavétel fogalmai A mintavételt meg kell tervezni A sokaság elemei: X, X X N, lehet véges és végtelen Mintaelemek: x, x x
Az evolúció folyamatos változások olyan sorozata, melynek során bizonyos populációk öröklődő jellegei nemzedékről nemzedékre változnak.
 Evolúció Az evolúció folyamatos változások olyan sorozata, melynek során bizonyos populációk öröklődő jellegei nemzedékről nemzedékre változnak. Latin eredetű szó, jelentése: kibontakozás Időben egymást
Evolúció Az evolúció folyamatos változások olyan sorozata, melynek során bizonyos populációk öröklődő jellegei nemzedékről nemzedékre változnak. Latin eredetű szó, jelentése: kibontakozás Időben egymást
