Új picornavírusok azonosítása és meghatározása molekuláris biológiai módszerekkel
|
|
|
- Márton Rácz
- 7 évvel ezelőtt
- Látták:
Átírás
1 Új picornavírusok azonosítása és meghatározása molekuláris biológiai módszerekkel Doktori (Ph.D.) értekezés Pankovics Péter Témavezető: 1 Dr. Reuter Gábor Ph.D., Med. Habil. Programvezető: 2 Prof. Dr. Emődy Levente 1 Baranya Megyei Kormányhivatal, Népegészségügyi Szakigazgatási Szerve, Regionális Virológiai Laboratórium, Gastroenterális Vírusok Nemzeti Referencia Laboratóriuma 2 Pécsi Tudományegyetem, Általános Orvostudományi Kar, Orvosi Mikrobiológiai és Immunitástani Intézet Pécs, 2013
2 TARTALOMJEGYZÉK RÖVIDÍTÉSEK JEGYZÉKE... 5 BEVEZETÉS... 6 ÁLTALÁNOS BEVEZETÉS... 6 IRODALMI ÁTTEKINTÉS... 8 PICORNAVIRIDAE CSALÁD NEMZETSÉGEI... 8 Morfológia és genomszerkezet... 9 A virális nukleinsav mérete, felépítése... 9 A virális fehérjék és funkcióik...11 Fizikai-kémiai tulajdonságok...13 Biológiai tulajdonságok...13 A Picornaviridae családon belüli új nemzetség és vírusfaj meghatározásának feltételei...14 KOBUVÍRUS NEMZETSÉG...14 A prototípus kobuvírus, az Aichi vírus és a szarvasmarha kobuvírus felfedezése...14 A kobuvírusok jellegzetességei...15 A kobuvírusok genetikai sokszínűsége...17 Az Aichi vírus átvitele, szeroepidemiológiája...17 Az Aichi vírusfertőzés klinikai jellegzetességei...18 Aichi vírusok emberekben...18 A szarvasmarha kobuvírus jelentősége...19 A kobuvírus laboratóriumi diagnosztikája...19 A kobuvírusok gazdafajai...20 ENTEROVÍRUS NEMZETSÉG...20 SAPELOVÍRUS NEMZETSÉG...21 CÉLKITŰZÉSEK...22 ANYAGOK ÉS MÓDSZEREK...23 MINTÁK GYŰJTÉSE ÉS SZÁRMAZÁSI HELYE...23 Házi sertés (Sus scrofa domestica) minták...23 Szarvasmarha (Bos taurus) minták...24 Bárány (Ovis aries) minták...25 Humán minták...25 Háziasított fürj (Coturnix coturnix) minták...26 RNS EXTRAKCIÓ...27 REVERZ TRANSZKRIPCIÓ-POLIMERÁZ LÁNCREAKCIÓ (RT-PCR)...28 FELHASZNÁLT PRIMEREK LISTÁJA...28 GÉLELEKTROFORÉZIS...32 RT-PCR PRIMER-WALKING TECHNIKA /3 RACE MÓDSZER...32 DIREKT SZEKVENCIA MEGHATÁROZÁS...33 FILOGENETIKAI ANALÍZIS
3 EREDMÉNYEK...37 A SERTÉS KOBUVÍRUS FELFEDEZÉSE...37 A SERTÉS KOBUVÍRUS (SWINE/S-1-HUN/2007) TELJES GENOMJÁNAK MEGHATÁROZÁSA...39 Az S-1-HUN vírus nem kódoló régióinak (UTR) elemzése...41 Az S-1-HUN vírus kódoló régiójának elemzése...42 A sertés kobuvírus kimutatása új, általános kobuvírus szűrőprimer segítségével...46 A SERTÉS KOBUVÍRUS FERTŐZÉS NYOMON KÖVETÉSE ÉS VÍRUS EVOLÚCIÓJA...46 A SZARVASMARHA KOBUVÍRUS ELSŐ EURÓPAI KIMUTATÁSA...48 KOBUVÍRUS LEÍRÁSA ÉS TELJES GENOMJÁNAK MEGHATÁROZÁSA JUHOKBÓL...49 AICHI VÍRUS (HUMÁN KOBUVÍRUS) KIMUTATÁSA MAGYARORSZÁGON...52 ÚJ PICORNAVÍRUS AZONOSÍTÁSA HÁZIASÍTOTT FÜRJ (COTURNIX COTURNIX) FÉCESZMINTÁJÁBÓL AZ UNIV- KOBU SZŰRŐ -PRIMEREKKEL...54 A fürj picornavírus nem kódoló régióinak elemzése...56 A fürj picornavírus kódoló régiójának elemzése...58 A fürj picornavírus további keresése...61 ENTEROVÍRUS C109 (EV-C109) ELSŐ EURÓPAI KIMUTATÁSA...61 Az L87/HUN/2007 rekombinációs vizsgálata...64 Az enterovírus C109 fertőzés klinikai esetismertetése...64 MEGBESZÉLÉS...66 A SERTÉS KOBUVÍRUS FELFEDEZÉSE ÉS LEÍRÁSA...67 A SERTÉS KOBUVÍRUS FERTŐZÉS MOLEKULÁRIS EPIDEMIOLÓGIÁJA ÉS IN VIVO EVOLÚCIÓS VIZSGÁLATA...69 A SZARVASMARHA KOBUVÍRUS ELSŐ EURÓPAI KIMUTATÁSA...71 KOBUVÍRUS KIMUTATÁSA ÉS TELJES GENOMJÁNAK MEGHATÁROZÁSA BÁRÁNYBÓL...71 AZ AICHI VÍRUS (HUMÁN KOBUVÍRUS) KIMUTATÁSA MAGYARORSZÁGON...72 ÚJ PICORNAVÍRUS AZONOSÍTÁSA HÁZIASÍTOTT FÜRJ FÉCESZ MINTÁKBÓL...74 ÚJ ENTEROVÍRUS C109 (EV-C109) ELSŐ EURÓPAI KIMUTATÁSA, TELJES GENOMJÁNAK MEGHATÁROZÁSA, GYERMEKKORI LÉGÚTI FERTŐZÉSBŐL...75 ÚJ EREDMÉNYEK ÖSSZEFOGLALÁSA...77 KÖSZÖNETNYILVÁNÍTÁS...79 MELLÉKLETEK FELHASZNÁLT VEGYSZEREK...80 IRODALOMJEGYZÉK...81 PUBLIKÁCIÓS JEGYZÉK...94 FÜGGELÉK
4 Felfedezni valamit, annyit tesz mint: Látni, amit mindenki lát, és gondolni, amit még senki nem gondolt / Szent-Györgyi Albert / 4
5 RÖVIDÍTÉSEK JEGYZÉKE BAMKH NSzSz aa bp cdns DNS dntp ELISA EIA EMCV EV FMDV g ICTV IGR IRES kda L M MgCl 2 min ml mm M-MLV-RT mrna mtsai. NaCl NaAc NH 4 Ac nm nt ORF PBS PCR PEV PTE ÁOK QPV RACE RNS RT RT-PCR sec ssrns SV TBE UTR UV VP VPg μg μl μm Baranya Megyei Kormányhivatal, Népegészségügyi Szakigazgatási Szerve aminosav (amino acid) bázispár (base pair) komplementer dezoxiribonukleinsav (complement dezoxynucleic acid) dezoxiribonukleinsav (dezoxynucleic acid) dezoxiribonukleotid trifoszfát (dezoxynucleotid triphosphate) enzimhez kapcsolt immunesszé (enzyme-linked immunosorbent assay) enzim immunesszé (enzyme immunoassay) encephalomyocarditis vírus (encephalomyocarditis virus) enterovírus (enterovirus) száj és körömfájás vírus (foot-and-mouth disease virus) gramm (gram) Nemzetközi Vírustaxonómiai Bizottság (International Committee on Taxonomy of Viruses) intergenikus régió (intergenic region) belső riboszóma kötő hely (internal ribosomal entry site) kilodalton (kilodalton) "leader" fehérje (leader protein) mól (mol) magnézium-klorid (magnesium chloride) perc (minutum) milliliter (millilitre) millimól (millimol) egér leukémia vírus reverz transzkriptáz (moloney murine leukemia virus reverse transcriptase) hírvivő ribonukleinsav (messenger ribonucleic acid) munkatársai nátrium-klorid (sodium chloride) nátrium-acetát (sodium acetate) ammónium-acetát (ammonium acetate) nanométer (nanometre) nukleotid (nucleotide) nyílt leolvasási keret (open reading frame) foszfát pufferrel kiegészített fiziológiás sóoldat (phosphate buffered saline) polimeráz láncreakció (polymerase chain reaction) serttés enterovírus (porcine enterovírus) Pécsi Tudományegyetem, Általános Orvostudományi Kar fürj picornavírus (Quail picornavirus) cdns végek felsokszorozása (Rapid amplification of cdna ends) ribonukleinsav (ribonucleic acid) reverz transzkripció (reverse transcription) reverz transzkripció polimeráz láncreakció (reverse transcription polymerase chain reaction) másodperc (secundum) egyszálú ribonukleinsav (single-stranded ribonucleic acid) majom picornavírus (simian picornavirus) Tris-borát-EDTA puffer (tris-borate-edta buffer) nem kódoló szakasz (untranslated region) ultraviola sugárzás (ultraviolet light) virális fehérje (viral protein) genomhoz kötött virális fehérje (virion protein genome linked) mikrogramm (microgram) mikroliter (microliter) mikromól (micromol) 5
6 BEVEZETÉS Általános bevezetés A picornavírusok számos emberi és állati megbetegedést okozhatnak, melyek morbiditása és mortalitása rendkívül változatos. A legkorábbi virális eredetűnek tartott emberi betegségről szóló feljegyzés is a picornavírusokhoz köthető. Az i.e közé tehető egyiptomi [68] temetkezési domborművön látható pap jobb lábfejének petyhüdt bénulása a poliomyelitis következménye lehetett, amit az Enterovírus nemzetségbe tartozó picornavírus, a poliovírus okozhatott (1. ábra). 1. ábra: i.e (i.e. 14. század) körüli temetkezési fedőkő. A domborművön a poliomyelitis legkorábbi ismert ábrázolása látható. Ancient Life: An Ancient Scourge 2005; A poliomyelitis fertőzés első klinikai jellemzése von Heine (1840) és Medin nevéhez fűződik, de a vírust 1909-ben Landsteiner és Popper írta le [68; 78]. Majd 1898-ban Loeffler és Frosch felfedezte a száj- és körömfájás betegség (foot-and-mouth disease virus, FMDV) kórokozóját, amely az első megismert állati picornavírus volt. A picornavírusok jelentős szerepet töltöttek be a modern virológia fejlődésében is ban John Franklin Endersnek, Thomas Huckle Wellernek és Frederick Chapman Robbinsnak sikerült a poliovírust nem neurális eredetű sejtkultúrán tenyésztenie, amiért hat évvel később a Nobel díjat is megkapták [68]. A fejlődő szövettenyésztési, állatkísérleti eljárásokkal és emberi önkéntesek bevonásával 6
7 további picornavírusok felfedezése következett. Az amerikai egyesült államokbeli New York államban található Coxsackie városban Gilbert Dalldorf és Grace Sickles 1948-ban paralítikus tüneteket mutató gyermekek székletmintáiból izolálták először a coxsackie vírusokat. Ismételten Frederick Robbisnak és munkatársainak sikerült 1951-ben egy teljesen új picornavírust, az echovírust (enteric cytopathic human orphan) izolálniuk egészséges gyermekek mintáiból [68; 78]. Ugyancsak a picornavírusok közé tartoznak a közönséges nátha kórokozói, a rhinovírusok [68], melyből jelenleg több mint 130 szerotípust ismerünk [68]. Ma a poliovírusok, a coxsackievírusok, az echovírusok és a rhinovírusok a picornavírusok Enterovírus nemzetségébe tartoznak. E vírusokon kívül számtalan új picornavírust ismerünk, melyek száma az elmúlt 10 évben különösen megszaporodott. 7
8 IRODALMI ÁTTEKINTÉS Picornaviridae család nemzetségei A Nemzetközi Vírustaxonómiai Bizottság (ICTV, International Committee on Taxonomy of Viruses) hivatalosan tizenkét nemzetséget különített el 2010-ben a Picornaviridae családban: Aphthovírus, Avihepatovírus, Cardiovírus, Enterovírus, Erbovírus, Hepatovírus, Kobuvírus, Parechovírus, Sapelovírus, Senecavírus, Teschovírus és Tremovírus (2. ábra) [31; 32; 33; 53; 54] ban további öt nemzetséggel bővült a család: Aquamavírus, Cosavírus, Dicipivírus, Megrivírus és Salivírus [34; 35; 55; 56]. Minden egyes nemzetség számos (jelenleg összesen 37) vírusfajt foglal magában [35]. 2. ábra: A Picornaviridae család hivatalosan elismert nemzetségei 2010-tól (nemzetségnév döntve) és 2013-tól (nemzetségnév macskakörmök között). A gyökér nélküli filogenetikai fa a Picornaviridae családba tartozó vírusok P1 kapszid régiója alapján, Neighbor-Joining módszerrel készült [31; 32; 33; 53]. 8
9 Morfológia és genomszerkezet A Picornaviridae családba tartozó vírusok lipid burokkal nem rendelkező, elektronmikroszkópos képen ikozaéderes szimmetriát mutató vírusok. A víruspartikulák nm átmérőjűek. E kis méretről kapta a víruscsalád a pico (latin) = kicsi nevet. A család elnevezésének második fele a virális genom típusából, az RNS (ribonukleinsav) angol rövidítéséből RNA ered ( pico + rna = picorna ) [48; 56; 87]. Elektronmikroszkópos képen a picornavírusok többsége jellegtelen, felszínük kevéssé strukturált néhány kivételtől eltekintve, mint például a kobuvírusok (3. ábra) [132; 138] és a parechovírusok, ezért elkülönítésük ezzel a módszerrel meglehetősen nehéz. 3. ábra: A poliovírus 1 (PV-1, J02281) és az Aichi vírus (A846/88, AB010145) elektronmikroszkóppal készített felvétele [138]. A virális nukleinsav mérete, felépítése A Picornaviridae családba pozitív polaritású, egyszálú RNS genomú vírusok (+ssrns) tartoznak. A genom nukleotid (nt) hosszúságú, egy nyílt leolvasási kerettel rendelkezik, amiről egyetlen polyprotein íródik át. A picornavírus genom G+C (guanin+citozin) tartalma 35%-tól egészen 60%-ig változik. A picornavírus RNS genom 5 9
10 végéhez kovalensen kötődik egy a vírus által kódolt (3B régió) kisméretű fehérje, a VPg (Virion Protein genome linked, genomhoz kötött virális fehérje) (4. ábra) [48; 56; 87; 115]. 4. ábra: A picornavírus genom általános felépítése. Felül az RNS genom sematikus képe látható a genom 5 végéhez kovalensen kapcsolódó VPg fehérjével, az 5 nem kódoló résszel (5 UTR), a fehérje kódoló szakasszal (polyprotein), a 3 nem kódoló régióval (3 UTR), valamint a 3 végen található poly(a) farokkal. Az L (leader) fehérjét nem minden picornavírus kódolja. Az ábra alsó részén a picornavírus polyprotein hasítási helyek láthatók a hasítási köztes és végső termékeinek megadásával [87]. A VP0 nem minden picornavírus esetén hasítódik VP4 és VP2 fehérjékre. A picornavírus genom kódoló és a genom két végén elhelyezkedő nem kódoló régiókra (UTR=UnTranslated Regions) osztható (4. ábra). A picornavírusok 5 UTR-ja változatos (~ nt) hosszúságú és bonyolult másodlagos RNS szerkezettel jellemezhető. Ezen belül is mind strukturálisan, mind a funkció szempontjából kiemelendő a belső riboszóma kötő hely (IRES=Internal Ribosomal Entry Site). E genomszakasznak az RNS transzkripcióban és transzlációban is fontos szerepe van. Az IRES másodlagos szerkezete, a kezdő kodon helyzete és a különböző sejttípusokban megfigyelt aktivitás alapján jelenleg 5 különböző IRES típust ismerünk a picornavírusoknál. Az I-es típusú IRES az enterovírusoknál és rhinovírusoknál [143], a II-es típusú IRES a cardiovírusoknál és aphthovírusoknál [143], a III-as IRES a hepatitis A vírusnál [6; 7], IV-es típusú IRES a sertés teschovírusnál [41] és újabban az V-ös típusú IRES pedig egyes kobuvírusok genomjában találhatjuk meg [117; 143]. Érdemes megjegyezni, hogy a IV-es típusú belső riboszómakötő hely (IRES IV) prototípusát Hellen és de Breyne a Flaviviridae családba tartozó Pestivirus és Hepacivirus (hepatitis C vírus) nemzetségekbe sorolható vírusoknál írta le; innen a teljes 10
11 elnevezés: hepacivirus/pestivirus-szerű (HP) IV-es típusú IRES [28]. Ez az IRES típus figyelhető meg például a porcine (sertés) teschovírusnál (Teschovírus), az avian (madár) encephalomyelitis vírusnál (Tremovírus), a duck (kacsa) hepatitis vírus 1-nél (Avihepatovírus), az avian (madár), porcine (sertés), simian (majom) sapelovírusnál (Sapelovírus), és a Seneca Valley vírusnál (Senecavírus). Az egyes IRES típusokon belül a nukleotid szekvenciák azonossága alacsony (< 40%), de az egyes riboszóma kötődésben szerepet játszó konzervatív motívumok jelenléte mellett a másodlagos szerkezet formai hasonlóságot is mutat. A picornavírus genom 3 végén szintén egy eltérő hosszúságú (~ nt), másodlagos struktúrával jellemezhető, nem kódoló régió (UTR) található, melynek végénél egy változó hosszúságú polyadenin (poly(a)) farok található (4. ábra). A virális fehérjék és funkcióik A picornavírusok kódoló régiója három állandó részre osztható: P1, P2 és P3. Egyes picornavírusok (Aphthovírus, Cardiovírus, Erbovírus, Kobuvírus, Sapelovírus, Senecavírus és Teschovírus) a P1 régió előtt 5 irányban még egy Leader proteinnek (L-protein) elnevezett fehérjét is kódolnak [27; 31; 108; 144]. Az L-fehérje funkciója nem minden vírus esetében ismert. Az aphthovírusoknál és az erbovírusoknál [27; 108; 144] az L-fehérje proteáz aktivitással rendelkezik, papain-szerű cisztein fehérje hasító enzimek, amelyek képesek leállítani a gazdasejtek fehérje szintetizáló folyamatait [27; 144]. A cardiovírusok L-fehérjéje cinkhez kötődik és foszforilálódik a fertőzés folyamán, valamint részt vesz a virális genom átírásának a szabályozásában [27]. A kobuvírusok L-fehérjéjéről egyelőre csak feltételezik, hogy szerepe lehet az RNS replikációban és enkapszidációban [108; 144]. A senecavírus L- fehérje funkciója jelenleg nem ismert, de az biztosnak látszik, hogy hiányoznak belőle a proteolítikus aktivitáshoz szükséges katalitikus részek és nem tartalmaz cinkkötő motívumot [C-X-H-X(6)-C-X(2)-C] és tirozin foszforilációs motívumot [K-X(2)-E-X(2)-Y] sem. [27]. A sapelo- és teschovírusok L-fehérjéjének funkciója jelenleg egyáltalán nem ismertek [27; 144]. A P1 régió kódolja a kapszid fehérjéket, melyet a 3C proteáz enzim VP0 (VP4, VP2), VP3 és VP1 régiókra hasít [27]. A VP0 régió - a parechovírusok, kobuvírusok, a kacsa hepatitis 1 vírus és a seal (fóka) picornavírus kivételével - VP4 és VP2 fehérjékre darabolódik fel [27; 62]. A kapszid fehérjék közül a VP1 felelős leginkább az antigenitásért, ezért általában a genom legváltozékonyabb, illetve legváltozatosabb része. Ez a fehérje képezi az alapját a picornavírusok osztályozásának is. A kapszidfehérjéknek ezen kívül számos funkciója van: (1) védi a virális RNS-t a környezeti RNáz enzimek bontó hatásaival szemben, 11
12 (2) meghatározza a szövet és gazdasejt tropizmusát, hogy felismerjék a számukra megfelelő sejtfelszíni receptorokat, (3) áthatol a célsejt membránján és a virális RNS molekulát a sejt citoplazmájába juttatja, (4) kiválogatja és becsomagolja a sejtben elkészült RNS molekulákat [144]. A P2 és P3 régiók fehérje termékei a fehérjék hasításában (2A pro, 3C pro, 3CD pro ) és a virális genom replikációjában (2B, 2C, 3AB, 3B VPg, 3CD pro, 3D pol ) játszanak nélkülözhetetlen szerepet. A 2A génrégió a picornavírusok által kódolt fehérjék között nagymértékben eltérő mérettel és (sokszor ismeretlen) funkcióval is rendelkezhetnek. A 2A pro enzimnek, amely egy tripszin-szerű cisztein proteáz, az enterovírusoknál, a cardiovírusoknál és az aphthovírusoknál elsődlegesen a P1 és P2 régió határon történő (ön)hasítás a feladata [30]. A hepatovírusoknál, parechovírusoknál és az Aichi vírusnál a 2A fehérje nem rendelkezik ismert proteolitikus aktivitással [110]. A kobuvírusoknál, parechovírusoknál és a madár encephalomyelitis vírusnál a 2A a H-box/NC (H-rev107) sejtfehérje családdal van kapcsolatban [30; 110; 135] Az utóbbi vírusoknál az elsődleges hasítási funkciót a P1 és a P2 régió között a 3C pro enzim látja el. Minden picornavírus genom kódol egy kimotripszin-szerű cisztein proteáz 3C pro enzimet, amely elsődlegesen a 2C és 3A fehérjerégiók határán hasítja el a prekurzor molekulát, de a hepatovírusoknál és a parechovírusoknál a 2A és 2B régiók között is képes hasítani. A legfontosabb tulajdonsága, hogy - ellentétben a többi picornavírus proteinázzal - a 3C pro képes a P1 és P2 prekurzor molekuláit is hasítani. Ide tartozik a 3CD pro enzim is, ahol az autokatalitikus darabolódás elmarad. A 3C pro és a 3CD pro enzimek mindegyike hasonló hatékonysággal képes a P2 és P3 prekurzor régiók feldolgozására [87]. A 2B fehérjének a picornavírusoknál szerepe van a virális RNS replikáció helyét biztosító, intracelluláris membránnal körülhatárolt vezikulumok létrehozásában, [15], valamint a virális RNS intracelluláris membránokhoz (endoplazmatikus retikulum és Golgi apparátus) való kötődésében [17]; innen ered a fehérje másik neve: viroporin [58]. A 2C fehérje az RNS szintézishez szükséges replikációs komplexet membránhoz kapcsolja [15], és NTPase és RNS-kötő aktivitást is mutat [69; 103]. Azonban a 2C fehérjének a tényleges biológiai funkciói és virális replikációban betöltött szerepe még nem tisztázott [121]. A 3A fehérjének a vírusreplikációban számos alapvető funkciója lehet. Az RNS szintézis elindításánál a membránokhoz kötődve replikációs komplexeket alakít ki [124]. Feltételezhetően a 3AB prekurzor fehérjekomplex kofaktor szerepet tölthet be más fontos szintetikus folyamatban, így a 3D pol polimeráz és a 3CD fehérjék működését fokozhatja [59; 12
13 70]. Enterovírusoknál a 3A fehérje gátolja a fehérje transzportot az endoplazmatikus retikulum és a Golgi apparátus között. A 3B régióról fordítódó VPg fehérje a picornavírusok 5 végéhez kovalensen kötődik. A VPg jelenléte vagy hiánya nem befolyásolja a virális RNS fertőzőképességét, azonban azt feltételezik, hogy fontos szerepe van a transzkripcióban [87]. A 3D pol fehérje funkcióját tekintve egy RNS-függő RNS polimeráz. [87]. A nem szerkezeti fehérjék közül a 2C, a 3C proteázok, valamint a 3D polimeráz minden picornavírusnál tartalmaz esszenciális konzervatív aminosav motívumokat [144]. Fizikai-kémiai tulajdonságok A picornavírusok környezeti hatásokkal szemben igen ellenállóak. Nem érzékenyek éterre, chloroformra, etanolra (96%) vagy nem ionos detergensekre. A hőérzékenységük változatos, 4ºC-on hetekig, -20ºC alatt évekig is életképesek maradnak, de a hőmérséklet és a behatási idő emelésével csökken a fertőzőképességük. A picornavírusok azonban inaktiválhatóak ionizáló sugárzással, UV-fénnyel vagy photodinamikus festékek jelenlétével, mint például természetes vörös vagy a proflavin, valamint erős kémiai vegyszerekkel, mint például a fenol, a formaldehid (0,3%), 0,1 M HCl, vagy a klór tartalmú fertőtlenítőszerek [68; 87]. Biológiai tulajdonságok A picornavírusok rendkívül elterjedtek az élővilágban. A jelenlegi ismereteink szerint a legnépesebb víruscsaládnak tekinthetők, jelen vannak a halaktól a főemlősökig. A vírusfajok átvitele egyik gazdaszervezetről a másikra horizontálisan történhet, ahol kulcsfontosságú a fekális-orális és a légúti átvitel. Ízeltlábú vektorszervezetekkel történő átvitelre is van példa: az EMCV vírusát először szúnyogokban és egyes vérszívó ízeltlábúakban (atka, kullancs) találták meg, valamint a poliovírust legyekből is kimutatták. A vírusfertőzés következményeként jellemző a gazdaszervezetben előidézett citolízis, de találunk irodalmi példákat néhány vírusfaj esetén a perzisztens fertőzésre is [56; 68; 78]. Az ismert emberi megbetegedést okozó picornavírusok (Enterovírus, Hepatovírus, Kobuvírus és Parechovírus) változatos kórképek kialakításában játszhatnak szerepet. Az enterovírusok a légzőszervrendszert fertőzve alsó- és felső légúti betegségeket (pl.: közönséges megfázás) okozhatnak [78; 140]. Az idegrendszert fertőzve poliomyelitist, meningitist, meningoenchephalitist és akut flaccid paralízist, az emésztőszervrendszert fertőzve pedig 13
14 gastroenteritist is előidézhetnek [78]. Az enterovírusok okozhatnak myocarditist, pericarditist, herpanginát, exanthemával járó megbetegedéseket, valamint akut haemorrhagiás conjunctivitist is [78]. A parechovírusok és a kobuvírusok hasmenéses megbetegedésekben játszhatnak szerepet, de a parechovírusok újszülöttkori lázas fertőzéseket, alsó- és felső légúti, valamint központi idegrendszeri (aseptikus meningitis, enchephalitis, encephalomyelitis) kórképeket is előidézhetnek [8; 78; 100; 132; 133; 134; 135; 136; 137]. A hepatovírusok pedig emberekben hepatitist okoznak [89; 90]. A Picornaviridae családon belüli új nemzetség és vírusfaj meghatározásának feltételei A vírusfajok filogenetikailag rokon vírusok, melyek (1) (szűk) gazdaszervezethez és a sejt receptorokhoz való kötődés tekintetében, (2) a replikáció, az enkapszidáció és a genetikai rekombináció folyamataiban és (3) a genomok szerveződésében azonosak. A különböző nemzetségekbe tartozó vírusfajok aminosav szekvenciái között legalább 58%-os különbség található [48; 56]. Az ICTV Picornaviridae Study Group taxonómiai osztályozása szerint egy adott picornavírus nemzetségről beszélünk, ha (1) az L, a 2A, 2B és 3A vírus fehérjék a nemzetség tagjai között homológok, (2) a nemzetség tagjai alapvetően szerkezetileg homológ IRES-sel (azonos IRES típus) rendelkeznek. (Amennyiben az első szabály igaz, akkor 2. szabályt nem kell figyelembe venni. Érdemes megjegyezni, hogy a különböző nemzetségek tagjainál megfigyelhetünk hasonló típusú IRES szerkezeteket.) (3) Egy nemzetségbe tartozó vírusfajok genomjainak P1, P2 és P3 régiói filogenetikailag kapcsolatban kell lenniük és P1 > 40%, P2 > 40% és P3 > 50% aminosav szintű hasonlóságot kell mutatniuk [48; 56]. Kobuvírus nemzetség A prototípus kobuvírus, az Aichi vírus és a szarvasmarha kobuvírus felfedezése Yamashita és munkacsoportja 1991-ben egy új virális kórokozót azonosított kagylófogyasztáshoz köthető gastroenteritis járványból a japán Aichi prefektúrában [48; 132]. A heveny hasmenéses járvány székletmintáiból elektronmikroszkópos vizsgálattal kb. 30 nm nagyságú vírusszerű partikulákat lehetett látni, melyek BS-C-1 sejtvonalon citopátiás elváltozást is okoztak [135]. A feltételezett kórokozó nem hasonlított sem az enterovírusokhoz, sem pedig egyik ismert gastroenteritist okozó vírushoz (rotavírus, adenovírus, calicivírus, astrovírus) sem [133; 134]. A felfedezést követő nyolc évvel később 14
15 sikerült csak az új vírusfaj (mely időközben az Aichi vírus nevet kapta; márciustól Aichivírus A) teljes genetikai állományát meghatározni, mely egy új picornavírus nemzetség ma már tudjuk első, prototípus tagját képviseli [107; 135] ban ugyancsak Yamashita és munkatársai az Aichi vírus rokonát szarvasmarháktól származó mintákból mutatták ki [138]. A laboratóriumi szövettenyésztési munka során először egy citopátiás hatást okozó ágenst fedeztek fel, miután a Vero sejtekhez a HeLa sejttenyésztéshez 30 éve használt tápfolyadékot hozzáadták. A citopátiás hatást okozó ágenst az Aichi vírus antiszérum nem neutralizálta, de tulajdonságaiban hasonló volt az Aichi vírushoz. A vírus feltételezésük szerint a szarvasmarhák szérumából készített tápfolyadékból került bele, mely szarvasmarha fécesszel kontaminálódott. A vírust szarvasmarhák féceszmintáiból is kimutatták [138]. Az Aichi vírus (Aichi vírus A846/88; AB040749) és a bovine (szarvasmarha) kobuvírus (U-1; AB084788; márciustól Aichivírus B) 2005-től, mint két vírusfaj egy új picornavírus nemzetségbe, a Kobuvírus nemzetségbe tartoznak. A kobuvírus név a japán kobu szóból ered, jelentése dudor, csomó, mely a vírus elektronmikroszkópos képén látható dudorra vagy csomóra emlékeztető felszíni struktúráira utal (3. ábra). A kobuvírusok jellegzetességei A kobuvírusok, hasonlóan a Picornaviridae család többi nemzetségéhez kisméretűek (28-30 nm átmérő) (3. ábra). [132; 138]. Az Aichi vírus 8280 nt, a szarvasmarha kobuvírus (U-1) 8374 nt hosszúságú a poly(a) farok nélkül. A kobuvírus polyprotein az Aichi vírusnál 2432 aminosav, az U-1 kobuvírusnál 2463 aminosav hosszúságú [135; 138]. A kobuvírusok rendelkeznek L-fehérjével - melynek funkciója ismeretlen és a VP0 nem hasítódik. (4. ábra). A kobuvírusok genomjának G+C aránya más picornavírusokhoz képest magas. A kobuvírusok 5 UTR nukleotid hossza az Aichi vírus esetén 576 nt, a szarvasmarha kobuvírus esetén 808 nt. Az 5 UTR másodlagos szerkezete és az IRES típusa e két kobuvírus esetén 2012-ig nem volt ismert. Érdekes, hogy a két vírusgenom 5 UTR-jének első 108 nukleotidja nagy nuklotid és másodlagos szerkezeti hasonlóságot mutat. E genomrész három hajtűkanyarral jellemezhető (stem-loop: SL-A, SL-B és SL-C), mely talán az egész nemzetség jellegzetes bélyege. Feltehetően az RNS replikációban a régiónak nélkülözhetetlen szerepe lehet [108; 109]. Az 5 UTR többi része a két kobuvírus genomban nagymértékben különbözik. Az 5 UTR-hez hasonlóan a 3 UTR genetikai hasonlósága is nagyon alacsony a két kobuvírus között. 15
16 Az Aichi vírus L régiója (170 aa) 47% nukleotid azonosságot mutat az U-1 (187 aa) kobuvíruséhoz. A P1 polyprotein az Aichi vírusnál 846 aminosav, a szarvasmarha kobuvírusnál (U-1) 857 aminosav. Az Aichi vírus P1 régióját összehasonlítva az U-1 kobuvírus P1 régiójával a VP0/VP3/VP1 régiókban 60/63/48% nukleotid azonosságokat lehet megfigyelni. A P1 szerkezeti régión belül a VP1 az Aichi vírusnál 267 aminosav, míg a szarvasmarha kobuvírusnál 253 aminosav hosszúságú. A VP1 a kobuvírusok esetében is a legfőképp kitett és immunológiailag domináns fehérje, ami a víruson belül a legváltozatosabb képet mutatja. A P2 és P3 régiók méretei is különböznek a két vírusnál. Az Aichi vírus 2A fehérjének nukleotid szekvencia azonossága alacsony, 48% az U-1 vírushoz képest. A kobuvírusok 2B régiójának a funkciója jelenleg nem ismert. A nukleotid szintű azonosság ebben a régióban a két kobuvírus között 57%. A kobuvírusok 2C régiójának nukleotid azonossága 66%. A 2C fehérjében található a feltételezett picornavírus helikáz erősen konzervatív nukleotid kötő doménje, a GXXGXGKT motívum. A kobuvírusoknak egy aminosav hosszúságú 3A fehérjéje és egy aminosav hosszúságú 3B (VPg: az 5 UTR 5 végéhez kovalensen kapcsolódó polipeptid) fehérjéje van. A két kobuvírus között a 3A régióban 54% azonosság, míg a 3B régióban 60% nukleotid azonosság van. A 3C pro fehérje - mely a polyprotein feldarabolásában játszik szerepet - egy cisztein proteáz, melynek katalitikus triádja a konzervatív hisztidin, aszparaginsav és cisztein. Végül a 3D régió kódolja az RNS-függő RNS polymerázt, mely számos konzervatív aminosav motívumot (KDELR, YGDD és FLKR) tartalmaz. A két kobuvírus között ebben a régióban van a legmagasabb (69%) nukleotid szintű azonosság. Összességében a két legmagasabb szintű nukleotid azonosságot (66% és 69%) a 2C és a 3D régiókban figyelhetjük meg. Az Aichi vírusnál az L és a 2A fehérjék szerepe ismeretlen. Az L fehérjének nincsen autokatalitikus hatása és nem játszik szerepet a polyprotein feldarabolódásában, viszont az RNS replikációban és a nukleinsav kapszidból történő kiszabadulásában közreműködhet. A kobuvírus 2A fehérje konzervatív motívuma a H-box/NC és transzmembrán domén, mely a H-rev107 sejtfehérjék családjának a jellemzőit mutatja, ezért a sejtosztódás szabályozásban és a virális RNS replikációban játszhat szerepet. A kobuvírusoknál a nyílt leolvasási keret után egy viszonylag hosszú nukleotid hosszúságú 3 UTR régió és egy poly(a) farok kapcsolódik. A 3 UTR másodlagos szerkezete jelenleg még nem ismert. 16
17 A kobuvírusok genetikai sokszínűsége A GenBank adatbázisában, 2008-ban 4 Aichi és 1 szarvasmarha kobuvírus teljes genom és néhány részleges, elsősorban a 3C/3D kapocsrégiót érintő szekvencia volt elérhető. Az Aichi vírus VP1 szekvenciák (~30db) közötti nukleotid (> 87%) és aminosav (> 90%) szintű azonosságok alapján ezeket a kobuvírusokat A, B és C genotípusokba sorolhatjuk [2; 48; 56; 136]. Az Aichi vírusnak 1 szerotípusa ismert, és hasonlóképpen a szarvasmarha kobuvírushoz, itt is magas nukleotid (> 86%) és aminosav (> 96%) azonosságok figyelhetők meg. Az A genotípusú Aichi vírus gyakori Európában és Ázsiában, míg a B genotípusát kimutatták már az amerikai, az ázsiai és az európai kontinenseken is. Az Aichi vírus C genotípust pedig az afrikai kontinensen azonosították. Kobuvírusok közötti rekombinációról nem tudunk. Aichi vírus elleni antitestek nem mutathatóak ki a szarvasmarha szérumból, míg a szarvasmarha U-1 vírus elleni antitestek sem mutathatóak ki a humán szérumból [135; 138]. Ennek alapján a két kobuvírus faj nemcsak különböző genotípusnak tekinthető, hanem különböző antigén típust (szerotípus) is képvisel. Az Aichi vírus átvitele, szeroepidemiológiája Az Aichi vírus első szeroprevalencia vizsgálatát Japánban végezték [133], ahol a szeroprevalencia a 19. életkorban 50% a 39. életkorban 83%-os. Németországban a szeroprevalencia hasonlóan magas, de már sokkal fiatalabb életkorban (19 év) elérte a 83%-os értéket [77]. Franciaországban a 972 főtől vett szérumminta vizsgálata során a 7 hónap és 9 év közötti korcsoportban a 25%-os, a éves vagy magasabb életkorú embereknél 85%-os szeroprevalencia értékeket találtak [26]. Spanyolországban az Aichi vírus szeroprevalenciája a 2-4 éves korcsoportban 41%, 30 éves életkorban 95% [102]. Összegezve elmondható, hogy a populáció 80-90%-a rendelkezik Aichi vírus elleni antitestekkel éves korra, ami általános átfertőzöttséget jelent a különböző emberi közösségekben. Az Aichi vírusfertőzés valószínűleg hosszú távú védettséget nem ad, az újrafertőződések gyakoriak lehetnek. A kobuvírusok elsődlegesen fekális-orális úton terjedhetnek. Előfordulhat közvetlen kontaktus során emberről-emberre (piszkos kéz), szennyezett élelmiszerrel (tenger gyümölcsei) [2; 61; 84; 132] vagy vízzel (szennyvízzel) [1] történő átvitel is, más picornavírusokhoz hasonlóan [86; 111]. A vírus képes akár járványos formában is megjelenni. 17
18 Az Aichi vírusfertőzés klinikai jellegzetességei A tünetmentes fertőzések sokkal gyakoribbak lehetnek, mint a klinikai tüneteket indukáló megbetegedések. Az Aichi vírus jelenlegi tudásunk szerint hasmenéses megbetegedést okoz emberekben. Az Aichi vírus fő klinikai tünetei a gyomor és bélrendszeri zavarok, hasmenés, de előfordulhat még hasi fájdalom, hányinger, hányás és láz is [132; 137]. A Japánban folytatott három éves vizsgálat során 397 gyermeket vizsgáltak különböző tünetekkel, mint például gastroenteritis, légzőszervi megbetegedés, exanthema, meningitis, enchephalitis, lázas megbetegedés és Kawasaki betegség [133]. Közülük csak egy gyermek mintájában mutatták ki a vírust, akinek alsó légúti tünetei voltak. Aichi vírusok emberekben Néhány tanulmány foglalkozott csak az Aichi vírus kimutatásával és prevalenciájával. A témában eddig megjelent közleményeket a 1. táblázatban foglaltuk össze. 1. táblázat: A kobuvírusok kimutatásának irodalmi áttekintése különböző gazdaszervezetekben Gazdaszervezet Kobuvírus faj Prototípus vírus Ország Referencia Humán Aichi vírus A846/88 (AB040749) Japán [132; 133; 134] Németország Brazília [77] Banglades Thaiföld [84] Vietnám Franciaország [2] Tunézia [112] Szarvasmarha Szarvasmarha kobuvírus U-1 (AB084788) Japán [138] Thaiföld [44] Az Aichi vírus jelenlétét már minden kontinensen kimutatták. A legtöbb vizsgálat és kimutatás Ázsiában volt. Az Aichi vírus alacsony előfordulási gyakorisággal van jelen a sporadikus gastroenterális megbetegedésekben. Pakisztánban [134] és Franciaországban [2] végzett vizsgálatok szerint az Aichi vírust összesen 5 (2,2%) / 222, illetve 4 (0,9%) / 457 hasmenéses tünettel rendelkező gyermek székletmintájából tudták csak kimutatni. A Japánt, Bangladest, Thaiföldet és Vietnámot felölelő tanulmányban [84] összesen 28 (3%) esetben igazolták a vírus jelenlétét 912 gyermektől és felnőttől származó hasmenéses székletmintából. Franciaországban a 4 Aichi fertőzés közül 2 esetben más enterális kórokozót is ki lehetett 18
19 társfertőzésként mutatni [2]. Hasonló eredménye volt a tunéziai tanulmánynak is, ahol a 22 (3,5%) Aichi fertőzöttből 16 esetben volt csak egyedüli kórokozó az Aichi vírus [112]. Elsőként Japánban vizsgáltak Aichi vírus járványokat 1987 és 1998 között; a 37 gastroenterális járványból 12 (32%) esetben az Aichi vírus volt a kórokozó [136]. A 12 járványból származó 268 széklet mintából 54 (20,1%) esetben igazolták a virális RNS jelenlétét és majdnem mindnél osztriga volt a fertőzés forrása. Franciaországban összesen 110 gastroenterális járványból 6 (5,4%) járványnál igazolták RT-PCR módszerrel az Aichi vírus jelenlétét [2]. Érdekességképpen megemlíthető, hogy a hat járványban gyűjtött 566 székletminta közül mindössze 9 (1,6%) esetben mutatták ki a virális RNS-t. Azonban ezt a 6 enterális járványt is osztrigával, tengeri gyümölcsök fogyasztásával vagy más fertőző forrással hozták kapcsolatba. Hollandiában viszont az előzőleg norovírus negatív, 188 gastroenterális járványból egyszer sem tudták az Aichi vírust kimutatni [116]. A szarvasmarha kobuvírus jelentősége Japánban folytatott szeroepidemiológiai vizsgálatok kimutatták, hogy 72 szarvasmarha szérum közül 43 (59,7%) szérum minta mutatott pozitivitást az U-1 vírus elleni neutralizációs kísérletekben [138]. Az U-1 elleni antitesteket más fajokban, emberben, majomban, sertésben, lóban, kutyában és macskában nem mutattak ki. A vizsgálataink kezdetéig összesen két ázsiai országban foglalkoztak a szarvasmarha kobuvírusok kimutatásával (1. táblázat). A szarvasmarha kobuvírus RNS-t szarvasmarha bélsár mintákból tudták kimutatni Japánban 12 (16,7%) / 72 széklet mintából [138] és Thaiföldön 6 (8,3%) / 72 széklet mintából [44]. Az első esetben egészséges, míg a második esetben hasmenéses szarvasmarháktól származtak a minták. Valószínűsíthető, hogy a szarvasmarha kobuvírus általános fertőzést okoz és endemikusan van jelen a szarvasmarha állományokban. A szarvasmarha kobuvírusnak a gastroenterális vagy egyéb tünetekkel fennálló kapcsolata ezidáig nem bizonyított. A kobuvírus laboratóriumi diagnosztikája A prototípus Aichi vírus citopátiás hatást mutat BS-C-1 (Afrikai zöldmajom vesesejt) és Vero (Afrikai zöldmajom vesehámsejt) sejtvonalakon, de sem HeLa, HEL vagy RD humán sejtvonalakon, sem pedig újszülött egerekben nem mutat sejtkárosító hatást [132]. A szarvasmarha kobuvírus tenyésztése azonban nem járt sikerrel [138]. A vírustenyésztés 19
20 időigényes és érzékenysége vitatott [133]. Elektronmikroszkópos technikával a kobuvírust nehéz elkülöníteni más kerek struktúrát mutató vírusoktól. Japánban a tenyésztéses módszernél sokkal érzékenyebb EIA módszert fejlesztettek ki. A módszerben Aichi vírus elleni monoklonális antitestesteket használtak a vírus antigének székletmintákból történő kimutatására. A virális antigén kimutatásán alapuló EIA módszernél a szerológiai vizsgálatok még érzékenyebbek az Aichi vírus kimutatására [133], de kereskedelmi forgalomban ilyen tesztek még nem elérhetők. Az igazi áttörést a diagnosztikában az RT-PCR módszer jelenti. Az első szűrőprimer párt a kobuvírusok (Aichi és szarvasmarha kobuvírus) 3C/3D kapocsrégiójára tervezték [136]. A kobuvírusok gazdafajai A vizsgálataink kezdetéig az ember és a szarvasmarha volt ismert, mint a kobuvírus gazdaszervezete [132; 138]. Enterovírus nemzetség Az Enterovírus (EV) nemzetség szintén a Picornaviridae víruscsalád tagja. A nemzetség tagjai sokszínű képet mutatnak mind az ide tartozó vírusok (náthavírusok, humán és állati enterovírusok, echovírusok, coxsackievírusok és poliovírusok), mind az általuk okozott megbetegedések tekintetében; kezdve a tünetmentes vagy enyhe felső-légúti (közönséges megfázás) és gastroenterális fertőzésektől egészen a néha súlyos megbetegedésekig, mint például az agyvelőgyulladás, agyhártyagyulladás vagy a heveny petyhüdt bénulás [36; 78]. Az enterovírusok kis, kerek 30 nm átmérőjű vírusok. A vírusgenom átlagosan nukleotid hosszúságú. Egy nyílt leolvasási keretet (ORF) tartalmaz, amelyről egy ~2200 aminosav hosszúságú polyprotein fordítódik át [78]. A polyprotein további régiókra P1, P2 és P3 osztható. A P1 régió tartalmazza a virális kapszid szerkezeti fehérjéit (VP4, VP2, VP3 és a VP1) [78]. A VP1 alapján a humán enterovírusok (EV) márciustól - négy fajba sorolhatóak: EV-A, EV-B, EV-C (ide tartoznak a poliovírusok) és az EV-D [73; 78; 104; 120]. Az Enterovírus nemzetségen belüli, újonnan felfedezett emberi megbetegedést okozó enterovírusok száma rohamosan növekszik, ezt bizonyítja az EV-C csoportba tartozó, 2008 februárjában felfedezett enterovírus, az EV-C109 (GQ865517), melyet gyerekek légúti fertőzéseiből mutattak ki Nicaraguában [140]. Az EV-C109 érdekessége, hogy ez egy 20
21 rekombináns vírus: a virális genom 5 végén található nem kódoló régió (UTR) úgy tűnik az EV-A és az EV-C között létrejött rekombináció eredménye [106; 140]. A vírus kóroki szerepe és globális elterjedtsége egyelőre ismeretlen, mivel Nicaraguán kívül ezt a vírust még nem mutatták ki máshol. Sapelovírus nemzetség A Picornaviridae családban a Sapelovírus nemzetség 2006 óta hivatalosan elfogadott új nemzetség [52]. Az evolúciós összehasonlító elemzések alapján jelenleg három faj tartozik a nemzetségbe, melyekről a nemzetség a nevét kapta (SAPELO = S: Simian sapelovirus; A: Avian sapelovirus; P: Porcine sapelovirus; EL: enteric-like [125]. Eredetileg a majom picornavírusokat (SV) először az es években számos emlős szövetből azonosították és jelenleg ~13-14 majom picornavírus szerotípus ismert. A majom picornavírusokat legelőször az enterovírusokhoz sorolták, majd a VP1 filogenetikai összehasonlító elemzése alapján három szerotípus (SSV1-3) az enterovírusoktól a sapelovírusokhoz került [74; 75; 125]. Az madár sapelovírust (régebbi nevén kacsa picornavírus (DPV), TW90A) először hepatitises tüneteket mutató kacsák belső szerveiből azonosították [125]. Végül egy korábban a sertés enterovírusokhoz tartozó vírus, a sertés enterovírus 8 (PEV-8) is átkerült az enterovírusoktól a sapelovírusokhoz [51; 57; 125]. Jelenleg tehát 3 vírusfaj, a majom, a sertés és a madár sapelovírus tartozik a Sapelovírus nemzetségbe. 21
22 CÉLKITŰZÉSEK Célunk volt a calicivírusok (norovírus, sapovírus) molekuláris epidemiológiai vizsgálata során házi sertések féceszmintáiból véletlenül felfedezett (sertés) kobuvírus genetikai módszerekkel történő vizsgálata és meghatározása. Célunk volt a kobuvírus nemzetségbe tartozó porcine (sertés) kobuvírus teljes nukleotid sorrendjének meghatározása, jellemzése és filogenetikai összehasonlítása az ismert GenBank adatbázisban szereplő referencia picornavírus szekvenciákkal. Célunk volt az 5 UTR és IRES másodlagos szerkezetének meghatározása. Célunk volt a sertés kobuvírus molekuláris epidemiológiai vizsgálata és nyomon követése sertés fécesz/szérum mintapárokból és a sertés kobuvírus virémia lehetőségének keresése. Célunk volt a sertés kobuvírus evolúciós vizsgálata, a teljes genom alapján. Célunk volt a kobuvírus kimutatása más fajba tartozó haszonállatok (pl.: szarvasmarha, juh) féceszmintáiból. Célunk volt az Aichi vírus kimutatása gyermekektől származó széklet- és légúti mintákból. Célunk volt a kobuvírusok konzervatív génrégiójára újonnan tervezett UNIV-kobu- R/UNIV-kobu-F szűrőprimerpár alkalmazásával a kobuvírusok keresése emberi széklet és fürj bélsár mintákból. Célunk volt egyes molekuláris biológiai/genetikai módszerek (RT-PCR, Long-Range RT- PCR, primer walking PCR, 5 /3 RACE RT-PCR) laboratóriumi beállítása és fejlesztése a picornavírusok teljes genomjának meghatározása érdekében. 22
23 ANYAGOK ÉS MÓDSZEREK Minták gyűjtése és származási helye A vizsgálati mintákat 2000 és 2011 között gyűjtöttük. A vizsgált állatoktól fécesz, szérum és tojás felszínéről vett mintákat használtunk. Humán minták esetén székletet és nasopharyngeális váladékot vizsgáltunk. A széklet és bélsármintákat hasmenéses betegektől, illetve tünetmentes és tünetekkel rendelkező állatoktól gyűjtöttük. Az állati székletminták házi sertés (Sus scrofa domestica), szarvasmarha (Bos taurus), juh (Ovis aries) és házi fürjtől (Coturnix coturnix) származtak. Részletesen, a humán és állati minták típusát, a minták származási helyét, a mintagyűjtés idejét, a mintaszámot és az életkorokat alább, a táblázatokban foglaltuk össze. Házi sertés (Sus scrofa domestica) minták A házi sertésektől (Sus scrofa domestica) származó székletmintákat egy keletmagyarországi (Ebes) sertéstelepről gyűjtöttük. Első mintagyűjtésünk februárjában történt. Összesen féceszmintát gyűjtöttünk 10 napnál fiatalabb, 3 hetes, 3 hónapos és 6 hónapos egészséges malacoktól. A következő mintavételezés 21 hónappal később történt ugyanerről a farmról, novemberben. Összesen fécesz és szérumminta párt gyűjtöttünk az állatoktól. A vérmintákat a nyaki vénából, zárt vérvételi vákuumcsővel vettük. Hasonlóan az előző mintavételezéshez itt is négy különböző, az előző felosztással megegyező korcsoportokból származtak a minták (2. táblázat). Az állatok klinikai tünetet nem mutattak. 23
24 2. táblázat: Házi sertésektől (Sus scrofa domestica) származó minták Mintagyűjtés ideje Mintagyűjtés helye Minta típusa Mintaszám (db) db Mintaszám Életkor Tünetek február 60 Fécesz Ebes telep 60 (Kelet-Magyarország) november Szérum <10 nap 15 3 hetes 15 3 hónapos 15 6 hónapos 15 <10 nap 15 3 hetes 15 3 hónapos 15 6 hónapos 15 <10 nap 15 3 hetes 15 3 hónapos 15 6 hónapos klinikailag tünetmentes állatok Szarvasmarha (Bos taurus) minták A szarvasmarháktól (Bos taurus) származó székletmintákat egy közép-magyarországi (Aba), 870 állatot számláló szarvasmarha telepről gyűjtöttük februárban. Négy eltérő korcsoportból, összesen 32 bélsármintát vizsgáltunk. Hat szarvasmarha az 1-9 napos, négy a napos, öt a 6-7 hónapos és 17 az 1-7,6 éves korcsoportba tartozott (3. táblázat) februárban a mintavételt megismételtük a telepen. Ekkor összesen 26 szarvasmarha féceszmintát gyűjtöttünk 20 napnál fiatalabb állatoktól. A mintavételezés időpontjaiban (2002. és 2008.) az állatoknak hasmenéses megbetegedése nem volt. 3. táblázat: Szarvasmarháktól (Bos taurus) származó minták Mintagyűjtés ideje Mintagyűjtés helye Minta típusa Mintaszám (db) db Mintaszám Életkor Tünetek nap február Aba telep nap Fécesz hónapos (Közép Magyarország) ,6 éves február <20 nap Klinikailag tünetmentes állatok 24
25 Bárány (Ovis aries) minták Juh (Ovis aries) bélsármintákat gyűjtöttünk márciusban, közel 400 állatot számláló közép-magyarországi (Tárnok) telepről. A telepen magyar merinó típusú nőstény juhokat pároztatnak német feketefejű kosokkal. Összesen 8 állattól vettünk féceszmintát (4. táblázat). Az állatok 3 hétnél fiatalabbak voltak és a mintavételezés időpontjában klinikai tüneteket, hasmenést nem mutattak. 4. táblázat: Bárányoktól (Ovis aries) származó minták Mintagyűjtés ideje Mintagyűjtés helye Minta típusa Mintaszám (db) Mintaszám db Életkor Tünetek március Tárnok telep (Közép Magyarország) Fécesz 8 8 <3 hetes Klinikailag tünetmentes állatok Humán minták október és december között 65 székletmintát vizsgáltunk gastroenterális tüneteket (hasmenés) mutató baranya megyei gyermekektől, (5. táblázat) melyeket háziorvosok, kórházi járó- és fekvőbeteg gyermekosztályok orvosai küldték laboratóriumunkba rutin rotavírus vizsgálatra. A székletminták negatívak voltak a rutin tenyésztéses bakteriológiai vizsgálatokkal (Salmonella spp., Shigella spp., Campylobacter spp., E. coli, Y. enterocolitica, S. aureus és C. perfingens), valamint rotavírusra (Rotavirus- Adenovirus, CerTest-Biotec, Spanyolország), adenovírusra (Rotavirus-Adenovirus, CerTest- Biotec, Spanyolország) és calicivírusra (norovírus és sapovírus RT-PCR vizsgálat). Összesen 92 légúti (nasopharyngeális aspirátum) mintát gyűjtöttünk a Kaposi Mór Oktató Kórház, Tüdőgyógyászati Osztály kezelése alatt álló gyermekektől (5. táblázat). A minták két légúti szezonból (2005. október május 15.; illetve október május 15.) származtak. Az első légúti szezonban 43, a második légúti szezonban pedig 49 mintát gyűjtöttünk. A betegek kiválasztásának két szempontja a 10 év alatti életkor és a klinikailag alsó-felső légúti fertőzéses eredetűnek tekinthető tünetek, illetve kórképek pharyngitis, tracheitis, bronchitis, brochiolitis, croup és pneumonia voltak. A gyermekek között fekvő és járó betegek, egészséges és alapbetegséggel küzdők egyaránt voltak. A 25
26 vizsgálatokhoz a fertőzés heveny fázisában gyűjtött nasopharyngeális aspirátumot használtuk. A klinikai és epidemiológiai adatokat prospektív módon, formanyomtatványokon gyűjtöttük. 5. táblázat: Humán betegektől származó minták Mintagyűjtés ideje Mintagyűjtés helye Minta típusa Mintaszám (db) Mintaszám db Életkor Tünetek októbertől decemberig Baranya megye Széklet hetestől 12 évesig Hasmenéses tünetek októbertől májusig októbertől Kaposi Mór Oktató Kórház Somogy megye Nasopharyngeális aspirátum évestől 10 évesig Felső és alsólégúti tünetek és alapbetegségek májusig Háziasított fürj (Coturnix coturnix) minták Háziasított fürjtől (Coturnix coturnix) júliusban egy féceszmintát gyűjtöttünk egy dél-kelet magyarországi családi baromfitenyészetből (6. táblázat). A baromfiudvarban kb. 20 állatot tartottak egy 2x3 méteres fedett ketrecben. Az állatok két évesnél fiatalabbak voltak. Az állatok között elhullást tapasztalt a gazda bármiféle klinikai tünet megjelenése nélkül. A mintavételezést megismételtük a farmon áprilisban és összesen 10 fürjtől vettünk féceszmintát áprilisban kereskedelmi forgalomban kapható, étkezési felhasználásra árusított fürjtojásokat (n = 12 db) is vásároltunk. A tojások felszínéről fiziológiás sóoldattal átitatott tupferrell mintát vettünk a vizsgálatainkhoz. 26
27 6. táblázat: Háziasított fürjtől (Coturnix coturnix) származó minták Mintagyűjtés ideje Mintagyűjtés helye Minta típusa Mintaszám (db) db Mintaszám Életkor Tünetek július 1 1 Fécesz Dél-Kelet április Magyarország Tojás <2 év Elhullás, klinikai tünetek nélkül RNS extrakció A székletmintákat PBS-sel 10-50%-ra hígítottuk, majd μl mennyiségű székletszuszpenziót 1,5 ml-es Eppendorf csőbe (Eppendorf AG, Németország) mértük. A bemért székletszuszpenzióval egyenlő mennyiségű Genetronnal (1,1,2-trikloro-trifluoretán, Sigma-Aldrich, Amerikai Egyesült Államok) tisztítottuk és szobahőmérsékleten centrifugáltuk rpm-en 5 percig (Jouan CR3i, Franciaország). A részlegesen tisztított felülúszóból (színtelen vizes fázis) 150 μl térfogatot 500 μl TRIzol Reagenssel (Life Technologies, Amerikai Egyesült Államok) kevertük el. A szérum és légúti garatmosó mintákatból 150 μl térfogatot használtunk fel és 500 μl TRIzol LS Reagenssel (Life Technologies, Amerikai Egyesült Államok) kevertük el. Ezt követően a mintákat 5 percig szobahőmérsékleten inkubáltuk, majd 100 μl kloroformot (Reanal, Magyarország) adtunk hozzá. Keverővel 15 másodperces keverés után szobahőmérsékleten 5 percig inkubáltuk a csöveket, majd rpm-en 15 percig centrifugáltuk 4 C-on. Centrifugálás után kapott három fázis közül a felső vizes fázissal dolgoztunk tovább. A felülúszóból minimum 2x150 μl mennyiséget mértünk át steril 1,5 ml-es Eppendorf csövekbe. Az RNS kicsapását a vizes fázisból az előzőleg átvitt térfogattal egyenlő mennyiségű izopropanollal (Acidum-2 Kft, Magyarország) végeztük. Enyhe keverést követően az oldatot szobahőmérsékleten 10 percig inkubáltuk, majd rpm-en 10 percig 4 C-on centrifugáltuk. A kicsapott RNS fölül a vizes-izopropanolos oldatot pipettával eltávolítottuk. A csapadékot egyszer 75%-os etanollal mostuk, majd rpm-en 2 percig 4 C-on centrifugáltuk. A kinyert RNS-t szobahőmérsékleten történő 5-10 perces szárítás után 20 μl nukleáz-mentes vízben (Nuclease Free Water, Promega, Amerikai Egyesült Államok) oldottuk fel. A mintákat a további felhasználásig -80ºC-on tároltuk minimalizálva a többszöri ismételt felolvasztás és lefagyasztásokat. 27
28 Reverz transzkripció-polimeráz láncreakció (RT-PCR) A virális RNS felsokszorozását két lépésben valósítottuk meg. Először a reverz transzkripció (RT) során a virális RNS-t komplementer DNS-sé írtuk át. Az RT elegy 50 μl-es végtérfogatban a következő alkotórészeket tartalmazta: 10 X PCR puffer (100 mm Tris-HCl (ph: 8,3); 15 mm MgCl 2, 500 mm KCl, 0,01% gelatin; Sigma-Aldrich, Amerikai Egyesült Államok), 25 mm MgCl 2 (Zenon Biotechnológia Kft., Magyarország), 10 mm dntp (Promega, Amerikai Egyesült Államok), RNasin (40 U/μl, Promega, Amerikai Egyesült Államok), 10 U/μl M-MLV-RT (200 U/μl, Promega, Amerikai Egyesült Államok), reverz primer (0,1 μg/μl) és 3-5 μl RNS. A reverz transzkripció 42 C-on 60 percig zajlott (PTC- 100TM, MJ Research, Inc., Amerikai Egyesült Államok). Negatív kontrollként 3-5 μl nukleáz mentes vizet használtunk. Második lépésben a polimeráz láncreakció (PCR) történt meg. A PCR elegyet az RT elegy térfogatával megegyező mennyiségben (50 μl) készítettük el (teljes térfogat: 100 μl). A PCR reakcióelegy sorrendben a következő anyagokat tartalmazta: 10 X PCR puffer, 25 mm MgCl 2, Dupl-A-Taq TM DNS Polimeráz (5 U/μl, Zenon Biotechnológia Kft., Magyarország) és forward primer (0,1 μg/μl). A PCR készülékben a denaturáció 94 C-on 3 percig zajlott, majd 40 ciklusból álló láncreakciót végeztünk 95 C-on 1 perces, 42 C-on 1 perces, és 72 C-on 1 perces lépésekkel. A végső extenzió 72 C-on 10 percig tartott. A primer olvadási hőmérsékleteket (mealting temperature, T m ) az IDT (Integrated DNA Technologies, Belgium) honlapján ( található kalkulátor segítségével határoztuk meg. A PCR reakcióban az annealing hőmérsékleteket (T a ) a T a = T m - 5ºC képlet szerint állítottuk be. Az elongáció idejét a PCR termékek számított hosszának figyelembe vételével módosítottuk. Felhasznált primerek listája A vizsgálatok kiindulópontjaként a szűrővizsgálatokra használt p289/p290 oligonukleotid párokat Jiang és mtsai tervezték 1999-ben a norovírusok és a sapovírusok (calicivírusok) RNS-függő RNS polimerázt kódoló gén konzervatív szakaszára [37]. A primerek pozíciója a prototípus Norwalk vírus genomban: p289: nukleotid; p290: nukleotid pozícióknál vannak. A termék a norovírusok esetén 319 bázispár, a sapovírusok esetén pedig 331 bázispár nagyságú, mely magába foglalja a calicivírusok polimeráz régiójára jellemző konzervatív GLPSG aminosav motívumot [10]. A kobuvírusokra az UNIV-Kobu-F és UNIV-Kobu-R szűrő primerpárt a humán (Aichi), a szarvasmarha (U-1) 28
29 és sertés (S1-HUN) kobuvírusok RNS-függő RNS polimerázt kódoló gén konzervatív szakaszára terveztük (5. ábra). A várható PCR termék 216 bázispár hosszúságú. A fent említett oligonukleotid szűrőprimerek és a dolgozatban bemutatásra kerülő teljes picornavírus genom meghatározásához használt további primerek nevét, szekvenciáját és a meghatározott vírus nevét a 7. táblázatban foglaltuk össze. A primereket az IDT (Integrated DNA Technologies, Belgium) szintetizálta. 5. ábra: Az UNIV-Kobu-R és UNIV-Kobu-F primerek szekveciái és kötődési helyük. A felső ábrán az UNIV-Kobu-F szekvenciája és a szekvenciában használt bázis analóg (Y: C és T kékkel jelölve), míg az alsó ábrán az UNIV-Kobu-R szekvenciája és a szekvenciában használt bázis analóg (R: A és G pirossal jelölve) látható. Az UNIV primerek kötődési pozícióinak kezdetét és végét mind a három prototípus kobuvírusnál megadtuk. A szekvenciák végén található 5 és 3 jelölések a szekvenciák irányát jelentik. 29
30 7. táblázat: A dolgozatban felhasznált oligonukleotidok listája Oligonukleotid neve Szekvencia Vírus p TGA CAA TGT AAT CAT CAC CAT-3 p GAT TAC TCC AAG TGG GAC TCC AC-3 Norovírus/Sapovírus UNIV-Kobu-F 5 -TGG AYT ACA AG(/R) TGT TTT GAT GC-3 UNIV-Kobu-R 5 -ATG TTG TTR ATG ATG GTG TTG A-3 Kobuvírus Z20-F GCC AACTCTTCC TYA AAC AC-3 Szarvasmarha kobuvírus Z20-F GGR TAC CCC TGG AAC ACC-3 (Aba-Z20/2002/HUN, Z20-F TGC CTG ATG GTT TCG ACG TA-3 FJ225406) Z20-F CCA ACA TCC TGA CTT CTC TCC T-3 S1-F-0 5'-TTT GAA AAG GGG GTG GGG G-3' S-1-F30 excly 5'-CCT CAC CCT CTT TTC CGG-3' TB3-sheepkobu-160-F 5'-GTA CTT CTT CGC CTG GGT-3' TB3-sheepkobu-730-F 5'-GGC TAA TTG TCT ACG GTG-3' TB3-sheepkobu-1140-F 5'-CCT CCT CTC CCT TGA CTG TGA-3' TB3-sheepkobu-1920-F 5'-CCA CCC AGC ATG GTA ACG-3' Juh kobuvírus TB3-sheepkobu-2472-F 5'-CCT TGG AGG TTC CAT AC-3' (TB3-HUN/2009, TB3-sheepkobu-3000-F 5'-CAT CTG CTA CTC ACC ACC-3' GU245693) TB3-sheepkobu-4425-R 5'-GTG CAG TTG TGG GAA GC-3' TB3-sheepkobu-4920-R 5'-TCA GGA AGG CAG AGA TCC-3' TB3-sheepkobu-4930-R 5'-AGC AAG CCA GGT CAG GAA-3' TB3-sheepkobu-5360-F 5'-CAT CCA AGC GCT AAG CTC-3' TB3-sheepkobu-6120-F 5'-CCA ACT CGT TGA TGC CAT-3' TB3-sheepkobu-7500-R 5'-CAT CGT GTT CCA AGG ATA C-3' F2010-UTR-277-R 5'-CCC AAA CGT ACA GTT TGC GGA G-3' Fürj picornavírus F2010-UTR-379-R 5'-CAC ATT GTG GTC GAA TCT CAT GCC-3' (QPV1/HUN/2010, F R 5'-TCA CTC GCA ACA TGC CGT-3' JN674502) F2010-5UTR-10F 5'-CCT CAC CAT ACC TTT TTC-3' F R 5'-AAC AGC AAT GCG TCG TAC-3' F R 5'-ATT GTT CAG CAG ATG GGC-3' F R 5'-TCC TAA ACA ACC CTG GTG-3' F R 5'-CGA AAC GTC CCG GAT TCT CGA-3' F R 5'-GTA GGC CAA TGT CCC AGA-3' F F 5'-ACA TTT CAG GTG CCA GGT-3' F R 5'-CCA TAT GTG TTC CCA GCA TC-3' F R 5'-CAC TGC CGT AGC AGG TCC-3' F R 5'-GAT GGT TCA GCA GTA GTA ACA-3' F R 5'-GGA TTC GGA CAC TAG ACC-3' F R 5'-ACT AAC TTT GTG ACC CAC TC-3' 30
31 F R F R F R F R F R F R F F F R F F F R F R F F EV109 VP1 123F EV109 VP1 363R L R L R L R L F L R L F L F L Flong L F L87-3D-F L F L F L R L R L87-45-F L87-4-F L F L R L R L Rlong L R L R 5'-TGT CTC TAT CCT GAC CAA-3' 5'-CCT TTA TAA CCA TCA AAA TAC-3' 5'-GTG TCA ATG AAA AAC CTC CG-3' 5'-CGA GGC ACC TAT CTT TCA CAG-3' 5'-GCC ACT CTT TGT GGG GTA-3' 5'-GTT ATT CCC ATG GTG TTG TA-3' 5'-GAA TTC ATC ATT GAC CAC ATG TG-3' 5'-GGA GCA ACC TGA TGG CAT TC-3' 5'-TAC TTG AAG GAT GAA TTG-3' 5'-GTT TTC AAA ATG ATA TTG TTG-3' 5'-GTG GTC AAT GAT GAA TTC GAC ATC-3' 5'-CGG ACA AAG GGG AAA TCT TTA C-3' 5'-GGA GAC TGG AGC AAC TAG TAA AG-3' 5'-GGT GAA CAT TTC CAA TTT CCT ACG-3' 5'-CAC GCT CTC CAA CGT GTA-3' 5'-TTA TGG GGA TTG CTG TGG-3' 5'-GAT CTT GCC AGT GGC CAT-3' 5'-AAG GTC AGA GTG CAC GGT-3' 5'-AAG AGA TCC TTG GCG TTG-3' 5'-TGC AGC TAG TGG CGA CGG-3' 5'-GCA AAG GTT AGA GTG TAC-3' 5'-ACC CAA GCA CAT AAG GGT GTG GTG-3' 5'-TTG CAT CCA TAC GAA CCT ACG-3' 5'-CTC ACC ATC TGT ACA AGA AC-3' 5'-TTC AAG ACC AAA CAC CGC ATT GA-3' 5'-TCC TCC GGC CCC TGA ATG-3' 5'-CGC TAT AGA CTT GCC AGT ACC-3' 5'-GCT TGG CAA TTG CTC TAC C-3' 5'-GCG TTA GTA CTC TGG TAT-3' 5'-TAA AAC AGC CTG GGG GT-3' 5'-TGG AAG CTT TGT TTC AAG G-3' 5'-TAC ACA ACA ATT GTC ACC-3' 5'-GTC AAC CTC AAG TCT AGG ATC-3' 5'-GTC CAG TGA CAT ZAG TTG CCC AGC-3' 5'-GCA TCC CCC CTT TGA CAC AG-3' 5'-AAT GGA TGT GCC TGA GCA TCC-3' Enterovírus C109 (L87/HUN/2007, JN900470) 31
32 Gélelektroforézis A termékeket 1,5%-os agaróz gélben (NuSieveR 3:1 Agarose, FMCR BioProducts, Amerikai Egyesült Államok), perc hosszan V állandó feszültséggel, Minicell EC370M (Electroforetic Gel System, E-C Apparatus Co., Amerikai Egyesült Államok) típusú futtatókádban választottuk el. A futtatáshoz 0,5 X Tris-Borát-EDTA enyhén lúgos (ph: 8,0) puffert használtunk [105]. Az agaróz gélt etidium-bromiddal (Sigma-Aldrich, Amerikai Egyesült Államok) festettük és 320 nm hullámhosszúságú ultraviola fénnyel világítottuk át, majd Canon típusú fényképezőgéppel dokumentáltuk. A minták gélbe méréséhez Gel Loading Solution-t (Sigma-Aldrich, Amerikai Egyesült Államok) használtunk és minden gélfuttatás során pozitív kontrollt és 100 bp DNS markert (Promega, Amerikai Egyesült Államok) használtunk. RT-PCR primer-walking technika A primer walking technika során a már ismert szekvencia végétől számított 100 bázis távolságra tervezett szekvencia specifikus oligonukleotidokkal és az ismert szakasztól a legközelebbi konzervatív nukleotid (vagy aminosav) régióra tervezett degenerált oligonukleotidokkal történt a genom amplifikációja. A módszer segítségével bázis hosszúságú darabokat határoztunk meg. Másik lehetőségként a meglévő, de nem folytonos virális genom szakaszait kötöttük össze specifikus primerpárok segítségével. 5 /3 RACE módszer A virális genom 5 és 3 régióinak meghatározása a rapid amplification of cdna ends (5 /3 RACE) módszerrel történt (5 /3 RACE Kit, 2nd generation, Roche Diagnostic GmbH, Németország) a gyártó utasításai szerint. Röviden összefoglalva a virális nukleinsav 3 végének amplifikációja során 20 µl végtérfogatban állítottuk össze az RT reakció elegyet: 4 µl cdns szintézis puffer (VIAL 1), 2 µl dntp (VIAL 3), 1 µl oligodt-anchor primer (VIAL 8), 5 µl virális RNS, 1 µl reverz transzkriptáz enzim (VIAL 2) és 7 µl nukleáz mentes víz. Az elegyet óvatosan kevertük, majd centrifugáltuk és inkubáltuk 55 C-on 60 percig. A reverz transzkriptáz enzimet 85 C-on 5 perc hosszan inaktiváltuk. A PCR mixet 50 µl végtérfogatban mértük össze: 1 µl cdns-t, 1 µl PCR anchor primert (VIAL 9), 1 µl specifikus primert (0,1 µg/µl), 1 µl dntp (VIAL 3), 5 µl 32
33 10 X reakció puffert, 0,5 µl Taq DNS polimerázt (5 U/µl), majd 40,5 µl nukleáz mentes vízzel egészítettük ki. A virális RNS 5 végének a felsokszorozására génspecifikus oligonukleotidokat használtunk a cdns szintézishez az RT reakció paraméterei szerint. A cdns 3 végéhez poly(a) csoportot szintetizáltunk az alábbiak szerint: 19 µl cdns mintát, 2,5 µl 10 X reakció puffert (VIAL 5) és 2,5 µl datp (2 mm, VIAL 4) használtunk. A reakcióelegy összekeverése és centrifugálása után az elegyet inkubáltuk 94 C-on 3 percig, majd rögtön jégbe tettük. Hozzáadtunk 1 µl terminális Transzferáz enzimet (80 U/µl, VIAL 6) és összekeverés után 37 C-on 20 percig inkubáltuk. Végül a terminális transzferáz enzimet inaktiváltuk 70 C-on 10 percig történő melegítéssel és felhasználásig jégben tároltuk. Végül az 5 RACE PCR reakciót 50 µl végtérfogatban állítottuk össze: 5 µl da jelölt cdns, 1 µl oligo dt-anchor primer (VIAL 8), 1 µl génspecifikus primer (0,1 µg/µl), 1 µl dntp (VIAL 3), 5 µl 10 X reakció puffer, 0,5 µl Taq DNS polimeráz (5 U/µl) és 36,5 µl nukleáz mentes víz. Direkt szekvencia meghatározás A kapott PCR-termékekből 20 µl mennyiséget 20 µl NH 4 Ac-al (4 M) kevertünk el steril 0,5 ml-es PCR (Eppendorf AG, Németország) csövekbe, majd 120 µl 4ºC-os abszolút (96%) etanolt (Merck KGaA, Németország) adtunk hozzá. Vortexeltük a mintákat, majd rpm-en 15 percig 4 C-on centrifugáltuk. Következő lépésként a PCR terméket mostuk 200 µl 70%-os 4ºC-os etanollal és rpm-en centrifugáltuk 10 percig 4 C-on. Végül az etanolt eltávolítottuk és a PCR csöveket szobahőmérsékleten szárítottuk. A PCR termék erősségétől függően µl nukleáz mentes vízben oldottuk vissza. A szekvenálási reakcióhoz 10 µl végtérfogatban 2,3 µl nukleáz mentes vízhez, 0,7 µl reverz/forward RT-PCR-nél használt primerhez (0,1 µg/µl), 2 µl BigDye Terminator mixhez (v. 1.1., PE Applied Biosystems, Egyesült Királyság) 5 µl tisztított PCR terméket adtunk hozzá. A reakció körülményei 96 C-on 1 perces denaturáció után 25 cikluson keresztül 96 C-on 15 másodperc, 50 C-on 5 másodperc és 60 C-on 4 perc voltak. A szekvenálási reakció végén a reakcióelegyet 4 C on tartottuk. A kapott termékeket ismét tisztítottuk az alábbiak szerint: 5 µl NaAc-ot (3 M) és 100 µl 4ºC-os abszolút etanolt (96%) mértünk össze, majd a 20 µl szekvenálási reakciót 40 µl nukleáz mentes vízzel hígítva hozzámértük. A kapott 165 µl össztérfogatú elegyet rpm-en 30 percig 4 C-on centrifugáltuk. A felülúszó dekantálása után 300 µl 70%-os etanollal mostuk és rpm-en 10 percig 15 C-on centrifugáltuk. A felülúszó eltávolítása után a pelletet 50 C-on 3 percig hőblokkban 33
34 szárítottuk, majd 20 µl Hi-Di-ben (Hi-Di TM Formamide, Applied Biosystems, Amerikai Egyesült Államok) oldottuk vissza, majd 95 C-on 2 percig denaturáltuk és rögtön jégbe tettük 15 percre. A mintákat kapilláris automata szekvenátoron (ABI PRISM 310 Genetic Analyzer, Applied Biosystems, Amerikai Egyesült Államok) futtattuk. A molekuláris módszerek megkövetelik a ribonukleázmentes eszközök, vegyszerek és környezet használatát. A nukleinsav kivonási eljárás során különös gondot fordítottunk az eltérő minták feldolgozása során a keresztszennyeződések megakadályozására. A molekuláris genetikai vizsgálatok során a különböző munkafolyamatokat (RNS kivonás, molekuláris reagensek összemérése, molekuláris reakciók kivitelezése, az amplifikált nukleinsav láthatóvá tétele és tisztítása, szekvencia meghatározási lépések) térben elkülönítve és egyirányba végeztük. A munka során egyszer használatos, autoklávozott eszközöket, védőkesztyűt és védőruházatot használtunk. Filogenetikai analízis A szekvencia elemzéshez használt referencia szekvenciákat a GenBank (NCBI, National Center for Biotechnology Information, adatbázisából, illetve a picornavírus honlapról ( gyűjtöttük ki. A felhasznált referencia és saját szekvenciák neveit és azonosító számait a 8. táblázatban foglaltuk össze. A letöltött és a szekvenálás során kapott szekvenciákat a ClustalX (ver. 2.1) ingyenesen elérhető szoftverrel illesztettük [123]. A többszörös illesztés módszerét alkalmazva első lépésként a programban meghatározott algoritmus minden szekvenciát összehasonlít egymással (pairwise alignment). Második lépésként a program a szekvenciákat hasonlóságuk alapján megközelítőleg csoportokba rendezi és egy filogenetikai fához hasonló dendrogram fájlban tárolja. A végső illesztést a 2. lépésnél készített dendrogram alapján véglegesíti. Az illesztett szekvenciákat a GeneDoc ( Multiple Sequence Alignment Editor and Shading Utility v , 2006) szintén ingyenesen elérhető programmal elemeztük [72]. A filogenetikai elemzéshez a MEGA4 és 5 (Molecular Evolutionary Genetics Analysis, v. 4.0/v. 5.0) programot használtunk [118; 119]. A statisztikai módszer beállításainál az UPGMA, a neighbor-joining és Maximum-Likelihood módszereket használtunk. Az alkalmazott módszert és annak pontos beállításait minden filogenetikai fánál feltüntettük. 34
35 8. táblázat: A dolgozatban felhasznált szekvenciák és azonosító számuk Szekvencia neve Azonosító száma Szekvencia neve Azonosító száma Aba-Z20/2002/Hungary FJ "bat kobu-like virus" HM AEV AJ Avian sapelo virus NC_ Aichi AB BAY-1-03-DEU AY Aichi A846/88 AB CMB02 EF BAY/1/03/DEU AY CMB03 EF BEV-1 D00214 CMB06 EF CMB07 EF CMB07 EF CV-A16 U05876 CMB10 EF CV-A21 D00538 CMB11 EF CV-B3 M16572 Duck hepatitis A virus-1 DQ EMCV M81861 Duck1 EU ERAV L43052 Klassevirus GQ ERBV X96871 N-2 AB EV-70 D00820 N-5 AB FMDV-O X00871 PEV-8 NC_ Goiania/GO/03/01/Brasil DQ PEV-9 Y14459 HAV M14707 Pigeon picornavirus A FR HPeV-1 L02971 Pigeon picornavirus B FR HRV-14 K02121 PTV AJ HRV-2 X02316 PV-1 J02281 Human rhinovirus 2 X02316 QPV1/HUN/2010 JN K-3 AB S-1-HUN/2007/Hungary EU K-35 AB Seal picornavirus EU K-36 AB Simian sapelo vírus AY K-38 AB SPV1 AY K-4 AB SVV DQ K-44 AB HUN298/2000/Hungary FJ K-49 AB TB3-HUN/2009/Hungary GU K-55 AB TMEV M20301 K-6 AB Turdivirus 1 GU K-60 AB Turdivirus 2 GU LV AF Turkey hepatitis virus HM K-30-HUN/2008/Hungary GQ U-1 AB L87/HUN/2007 JN BAN DQ G13 (South Africa/50) AF G12 (South Africa/51) AF Flores (Mexico/52) AF BAN EF IH35 (New York/55) AF BAN EF G9 (South Africa/50) AF Belgium-1 (Belgium/51) AF Mahoney (Ohio/41) V01149 Sabin2 (P712, Ch, 2ab) X00595 P3/Leon 12 a1b X00925 Fin05-2 FJ Fin04-7 FJ BAN EF USA-GA EF EH24-70 (Singapore/70) D90457 Coe (California/54) D00538 BAN EF Kuykendall-A (California/52) AF USA DQ Chulman (New York/55) AF NIH-8663 (Japan/52) AF CL EU Tompkins (NY/47) AF
36 NICA GQ BAN AY CIV AY Del Carmen (Philippines/53) AY Toluca-1 (Mexico/59) AY Pinf7-54A DQ BAN AY /04 (DRC) EF Fermon (California/62) AY Duck picornavirus (TW90A) AY
37 EREDMÉNYEK A sertés kobuvírus felfedezése A vizsgálataink kezdeti célja a sertés calicivírusok (norovírusok és sapovírusok) kimutatása és molekuláris epidemiológiai elemzése volt házi sertések féceszmintáiból RT- PCR módszerrel az Európai Unió által támogatott EVENT pályázat (Enteric Virus Emergence, New Tools, SP22-CT , keretében [91, 98] februárban összesen 60 egészséges sertéstől (Sus scrofa domestica) gyűjtöttünk székletmintát Kelet-Magyarországról (Ebes). A calicivírusok molekuláris biológiai kimutatásához a p289/p290 oligonukleotid párt használtuk, melyet a calicivírusok (noro- és sapovírusok) RNS-függő RNS-polimeráz génrégiójára Jiang és mtsai terveztek [37]. A PCR-termék norovírusok esetén 319 nt, míg sapovírusok esetén 331 nt hosszúságú. A 60 mintából összesen 2 (3,3%) mintából sikerült a sapovírusra specifikus méretű (331 nt) terméket kapni (6. ábra). Azonban az agaróz gélképen a 60 mintából 21 (35%) mintában következetesen egy calicivírusokra nem specifikus méretű kb nt erős PCRterméket is láttunk (6. ábra). Direkt szekvencia elemzéssel meghatároztuk az aspecifikus méretű PCR-termék (1065 nt) bázissorrendjét. A szekvencia (swine/s-1-hun/2007, EU787450, NC_011829) a GenBank adatbázisával összehasonlítva, a kobuvírus nemzetségbe tartozó szarvasmarha (U-1 vírus) és humán (Aichi vírus) kobuvírus 3C pro (87 nt), illetve 3D pol (978 nt) kódoló génrégióival mutattak azonosságot: a nukleotid és az aminosav azonosság 73 és 79% volt az U-1, 69 és 70% volt az Aichi kobuvírusokhoz. 37
38 6. ábra: PCR-termékek 1,5%-os agaróz gélben futtatott gélelektroforetikus képe. Az RT-PCR reakciók az általános calicivírus primerekkel (p289/p290) történtek. A képen sertés sapovírusra (5. és 6. zseb) specifikus méretű (S: 331 nt) és kb nt hosszúságú aspecifikus PCR-termékek (1, 3, 4, és 5. zseb) láthatók. Az aspecifikus PCR-termékek (K: ~1100 nt) szekvenciái a kobuvírusokhoz mutattak hasonlóságot a GenBankban. Az 5. zsebben kettős (sertés sapovírus és kobuvírus) fertőzés látható. C- a negatív kontrollt, míg a C+ a pozitív kontroll (sertés sapovírus) jelöli. Az M a 100 bázispáros molekuláris markert jelenti. 38
39 A sertés kobuvírus (swine/s-1-hun/2007) teljes genomjának meghatározása Az egyik 10 napnál fiatalabb sertés féceszmintából meghatároztuk a kobuvírus teljes genomjának nukleotid sorrendjét (7. ábra). Az S-1-HUN vírusgenom teljes hossza 8210 nukleotid. Az S-1-HUN kódoló régiója 7467 nukleotid hosszúságú. Erről íródik át egy 2488 aminosav hosszúságú polyprotein. A virális genom 5 végén 576 nukleotid hosszúságú, míg a 3 végen pedig egy 167 nukleotid hosszúságú nem kódoló régió található (7-8. ábra). A genom 5 végéhez egy VPg kapcsolódik, a 3 végén pedig egy poly(a) farok található. 7. ábra: A sertés kobuvírus teljes genomjának meghatározása primer walking módszerrel. Fent, a sertés kobuvírus génrégiói és azok nukleotid hosszúságai láthatók, megadva az egyes génrégiók kezdő nukleotidjának pozícióját a genomban. A fekete téglalapok az amplifikált PCR-termékek elhelyezkedését ábrázolják, ahol a végeken lévő szám a listában szereplő primerek sorszámát jelentik. A oligonukleotidok nevében levő R: reverz, F: forward, a primer irányát, míg a primer nevében található X/X szám a sertés kobuvírus (S-1-HUN) genomban levő nukleotid helyzetét jelöli. A primerek az Aichi (AB040749), a szarvasmarha U-1 (AB084788) és a sertés S-1-HUN (EU787450) kobuvírusok aktuálisan elérhető, és illesztett konzervatív szekvenciáira lettek tervezve. 39
40 A genom nukleotid bázisainak aránya; A (adenin): 20,5%, G (guanin): 20,9%, C (citozin): 31,5%, míg az U (uracil): 27,1%. A kódoló régióról fordítódó polyprotein lehetséges hasítási helyei a 3A/3B és 3B/3C pro régiókban rendre Q/G (az U-1 vírusnál Q/A) és Q/S (az U-1 vírusnál Q/A) voltak. Az S-1-HUN további aminosav hasítási helyei az U-1 vírussal megegyeznek (8. ábra). 8. ábra: Az S-1-HUN teljes genetikai felépítése és szerkezeti összehasonlítása a humán (Aichi) és szarvasmarha (U-1) kobuvírusokkal összehasonlítva. A P1 régió a virális szerkezeti régiókat, míg a P2 és P3 a nem szerkezeti fehérjéket kódolja. Minden egyes génrégió nukleotid (felső szám) és aminosav (alsó szám) hosszúságát feltüntettük. Az S-1-HUN és a két ismert kobuvírus faj génrégiói között a százalékos nukleotid és aminosav (nukleotid/aminosav %) hasonlóságot jelöltük. A potenciális N-terminális aminosav hasítási helyeket, az egyes génrégiókat elválasztó határvonal felett tüntettük fel. 40
41 Az S-1-HUN vírus nem kódoló régióinak (UTR) elemzése Az S-1-HUN vírus 5 UTR régiója 576 nukleotid hosszúságú, ami 168 és 232 nukleotiddal rövidebb, mint az Aichi vírus (744 nt) és az U-1 vírus (808 nt) esetén. Az S-1- HUN 5 UTR régiójának lehetséges másodlagos szerkezete a 9. ábrán látható. Az első 108 nukleotid sorrendje és szerkezete nagyon hasonló a szarvasmarha (U-1) és humán (Aichi) kobuvírusokéhoz, az Aichi vírushoz való azonossága 86%. Szerkezetileg az S-1-HUN-nál is három stem-loop (SL) hurok (SL-A, SL-B és SL-C) rajzolható fel (9. ábra). Az 5 UTR régióban összesen kilenc AUG motívum azonosítható, ezek a 215., 343., 355., 376., 387., 401., 515., 550. és az 577. nukleotidoknál találhatóak. A kódoló régióval azonos leolvasási keretbe eső, első AUG motívum ( nt) tekinthető a start kodonnak. Ezt az AUG motívumot azonban nem előzi meg polypirimidin szakasz. Egy hosszabb, pirimidinben gazdag régió (UUUCUCU) az RNS molekulán a 459 és 465 nt pozíciói között található. Az S-1-HUN 5 UTR régiójának , valamint a nukleotidok közötti részén a duck hepatitis vírus 1 (EU395440) és a sertés teschovírus (AY392537) belső riboszóma kötő helyeivel kaptunk 79% és 74%-os nt hasonlóságot a GenBank adatbázisában. E hasonlósági adatok és az S-1-HUN IRES elemeinek potenciális másodlagos RNS szerkezete alapján (9. ábra) az S-1-HUN vírusnak hepacivirus/pestivirus-szerű (HP), a IV-es B-típushoz hasonló belső riboszómakötő helye (IRES IVB) lehet. Az S-1-HUN IRES-ében megtalálhatók a IV-es típusú IRES-re jellemző konzervatív szerkezeti elemek, így a II-es domén és a III-as domén (és ennek részei: III 1, III 2, III 3, III 4, IIIa, IIIb, IIIc, IIId, IIIe, IIIf) (9. ábra). A másodlagos szerkezeti modell szerint a pseudoknot stem1, stem2, linker és spacer régiók 9, 8, 5 és 9 nukleotid hosszúságúak. Megtalálhatók a konzervatív nt motívumok a IIId doménben (GCGUGGCGGGAAUCCACGC ), a IIIe doménben (UACUGCUUGAUAGAGUC ), és jelen van az A-A nem párosodó nukleotid (mispair) a spirális III-as domén, III 1 régiójában (5 AGAG/5 UACU). Az S-1-HUN vírus 3 UTR régiója 167 nukleotid hosszúságú, ami 70 és 7 nukleotiddal rövidebb, mint az Aichi vírus és az U-1 vírus esetén. Annak ellenére sem kaptunk szekvencia hasonlóságot a GenBank adatbázisában, hogy az S-1-HUN 3 UTR régiójának valószínűsített másodlagos szerkezetében, az U-1 víruséhoz hasonlóan három hajtűszerű stem struktúra található (ábra nélkül). 41
42 9. ábra: A sertés kobuvírus (S-1-HUN) 5 UTR és IRES régiójának potenciális másodlagos RNS szerkezete. A szerkezet megrajzolásához az Mfold programot használtunk [146]. A genom 5 végén található a három stem-loop szerkezeti elem (SL-A, SL-B, SL-C) [107], mely minden eddig ismert kobuvírusnál jelen van. Az 5 UTR 3 végén pedig a IV-típusú IRES (Internal Ribosomal Entry Site: belső riboszóma kötő hely) szerkezete látható [28]. A konzervatív doméneket római II és III-al jelöltük. A domének alegységeit (II 1, II 2, III 1, III 2, stb. és III a, III b stb.) az irodalomban szereplő nevezéktan szerint neveztük el. A konzervált nukleotid motívumokat és a polyproteint kezdő AUG start kodont szürke alapszínnel emeltük ki. Az S-1-HUN vírus kódoló régiójának elemzése Az S-1-HUN kódoló régiójának felépítése tipikus picornavírus szerkezetet mutat. A genom kódoló részének elején egy 585/(195aa) nukleotid hosszúságú L-fehérjét kódoló gén található (8. ábra). Az L-fehérje 8 és 25 aminosavval hosszabb az U-1 és az Aichi vírusok L- fehérjéhez képest. Az S-1-HUN L-fehérje 37%-os azonosságot mutat az U-1 L-proteinhez (8. ábra). A három kobuvírus ismeretében, az L-proteinekben két konzervatív aminosav motívum (PEDXLXDS és LPG) azonosítható. A kobuvírus L-proteinben a picornavírusoknál eddig ismert fehérje funkciók nem ismerhetők fel. 42
43 10. ábra: Az Aichi vírus, a szarvasmarha kobuvírus (U-1) és a sertés kobuvírus (S-1-HUN) génrégióinak (L; szerkezeti régiók; nem szerkezeti régiók) százalékos nukleotid és aminosav különbségének összehasonlítása. Az S-1-HUN P1 régiója 2529/(843 aa) nukleotid hosszúságú, ami 42 és 9 nukleotiddal rövidebb az U-1 és Aichi vírusok P1 régióinál. A VP0 és a VP1 régiók méretei változóak 366/367/370 aminosav és 254/267/253 aminosav az S-1-HUN, az U-1 és az Aichi vírusoknál. Az S-1-HUN és az U-1 vírusok közötti aminosav azonosság a VP0, VP3 és VP1 régiókban rendre 70%, 61% és 48%, míg az S-1-HUN és az Aichi vírusok közötti hasonlóság ennél alacsonyabb (8. és 10. ábra). A legnagyobb különbségek minden esetben a VP1 régióban figyelhetők meg. A sertés kobuvírus P2 régiója 1998/(666 aa) nukleotid, míg a P3 régiója 2355/(784 aa) nukleotid hosszúságú. A P2 fehérjék (2A-2C; nem szerkezeti fehérjék) 68%-ban az U-1 vírushoz és 59%-ban az Aichi vírushoz hasonlítanak (10. ábra). A 2A régió 408/(136 aa) nukleotid hosszú. Részletezve, az S-1-HUN 2A aminosav szekvenciája 69% és 53% azonosságot mutat az U-1 és Aichi vírusok 2A fehérjéihez. A 2A-ban találhatóak a HWAL és 43
44 NCXHFV konzervatív aminosav motívumok, melyek a H-box/NC fehérjecsaládhoz hasonlóak. A 2B 585/(195 aa) nukleotid hosszú. Egy 90 (30 aa) nukleotid hosszúságú egymás után ismétlődő aminosav motívum AANRVAESIETTAS(/T)V(/A)VREADLARSTLNISM ismerhető fel benne. A tandem motívum következtében növekszik a 2B régió hossza 585 nukleotidra az Aichi- és U-1 vírusok 495 nukleotid hosszúságú 2B régiójához képest (8. ábra). A 2C régióban 68-77% között változik az azonossági arány a három kobuvírus között. Az S-1-HUN kobuvírus 2C régiójában szintén megtalálható a GXXGXGKT/S (GPPGTGKS) konzervatív aminosav motívum (picornavírus helicase). A P3 régióban (3A-3D) átlagosan 71% aminosav azonosság számolható az U-1 vírussal és 63% az Aichi vírussal összehasonlítva. A P3 régión belül azonban jelentős eltérések tapasztalhatók: a 3A fehérje 26%-os, míg a 3D fehérje 81%-os azonosságot mutat az U-1 kobuvírushoz (10. ábra). Az S-1-HUN 3A régiója 270/(90 aa) nukleotid hosszúságú, ami 4, illetve 5 aminosavval rövidebb a két prototípus vírusfaj 3A régiójának aminosav hosszúságánál. A sertés kobuvírus 3B régió (VPg fehérjét kódoló rész) 102/(34 aa) nukleotid hosszúságú, ami viszont hosszabb 4, illetve 7 aminosavval hosszabb, mint az U-1 vírus (30 aa) és Aichi vírus (27 aa) azonos részei. Az S-1-HUN 3B (VPg) régiójának 3. aminosav pozíciójánál egy konzervatív picornavírus tirozin aminosav figyelhető meg. Az 576/(192 aa) nukleotid hosszúságú 3C pro fehérje (szerin típusú proteáz) a 43., 86. és 145. aminosavjainál hisztidin, aszparaginsav és cisztein aminosavak találhatóak, melyek a konzervatív katalitikus triádot alkotják. A 3C pro fehérje C-teminális részénél megtalálható konzervatív aminosav motívum a GXCGG, melynek módosult változata a sertés kobuvírusban is jelen van GLCGA (U-1 vírus: GMCGA; Aichi vírus: GLCGS) funkcióját tekintve a proteáz enzim aktív helyének része lehet. A tripszinszerű proteáz szubsztrát kötő helyének kialakításában vehet részt az a hisztidin maradék, ami az S-1-HUN 3C pro fehérje 163. aminosav pozíciójában található. A sertés kobuvírus 1407/(468 aa) nukleotid hosszú 3D régiója pedig tartalmazza a picornavírusokra jellemző, konzervatív aminosav motívumokat (KDELR, YGDD, FLKR). A szintén konzervatív GLPSG aminosav motívum helyett az S-1-HUN GNPSG motívumot tartalmaz. A kobuvírus kódoló régiójában nem találtunk genotípusok közötti rekombinációra utaló jelet a Simplot szoftverrel. A legmagasabb szekvencia azonosság is csak 74% volt a 2C és 3D régiókban az S-1-HUN, az U-1 és az Aichi kobuvírusok között. Az S-1-HUN kobuvírus és a reprezentatív picornavírusok teljes polyproteinjeinek [(L), P1, P2 és P3)] alapján készített filogenetikai elemzés a 11. ábrán látható. A filogenetikai fa megerősíti, hogy a sertés kobuvírus a szarvasmarha kobuvírustól és az Aichi vírustól különbözik, új vírusfajt alkothat. 44
45 11. ábra: A sertés kobuvírus (S-1-HUN, EU787450) filogenetikai elemzése a kobuvírus nemzetség és a reprezentatív picornavírusok teljes polyproteinjeinek ( L, P1, P2 és P3) alapján. A filogenetikai fa ClustalX (ver. 2.1) szoftverrel illesztett, aminosav szekvenciák alapján készült. Az ágrajz Neighbour-Joining statisztikai módszerrel készült (MEGA ver. 4.1). A bootstrap értékeket 1000 ismétlésre állítva. A referencia szekvenciák GenBank azonosítói: CV-B3: coxsackievírus B3; EV-70: enterovírus 70; CV-A21: coxsackievírus A21; PV-1: poliovírus 1; CV-A16: coxsackievírus A16; BEV-1: bovine enterovírus 1; PEV-9: porcine enterovírus 9; HRV-14: humán rhinovírus 14; HRV-2: humán rhinovírus 2; PTV: porcine teschovírus; ERBV: equine rhinitis B vírus; FMDV-0: foot-and-mouth disease vírus O; ERAV: equine rhinitis A vírus; EMCV: encephalomyocarditis vírus; TMEV: Theiler s murine encephalomyelitis vírus; Aichi: humán kobuvírus; U-1: bovine kobuvírus; HPeV-1: humán parechovírus 1; LV: Ljungan vírus; HAV: hepatitis A vírus; AEV: avian encephalomyelitis vírus. 45
46 A sertés kobuvírus kimutatása új, általános kobuvírus szűrőprimer segítségével Az Aichi, U-1 és S-1-HUN kobuvírusok legkonzervatívabb 3D régiójára egy általános kobuvírus szűrő -primerpárt (UNIV-kobu-F/UNIV-kobu-R) terveztünk (5. ábra és 7. táblázat). A februárban a kelet-magyarországi Ebes sertéstelepről gyűjtött 60 féceszmintát ezzel a primerpárral ismételten megvizsgáltuk. Összesen 39 (65%) esetben sikerült a sertés kobuvírust kimutatni az egészséges állatok féceszmintáiból. A tíz napos állatok közül 15/15 (100%) esetben, a 3 hetes állatok közül 14/15 (93,3%) esetben, a 3 hónapos állatok közül 3/15 (20%) esetben végül a 6 hónapos állatok közül 7/15 (46,7%) esetben tudtuk a sertés kobuvírus 3D régiójának 216 nukleotid hosszúságú szakaszát RT-PCR módszerrel felamplifikálni. A sertés kobuvírus fertőzés nyomon követése és vírus evolúciója A sertés kobuvírus endemikus elterjedtségét és in vivo evolúcióját vizsgáltuk az ebesi sertéstelepen, ahol a prototípus S-1-HUN vírust eredetileg kimutattuk novemberében 21 hónappal az első mintavételezés után ismételten 60 fécesz és 60 szérummintát vizsgáltunk (a mintapárok azonos állattól származtak) RT-PCR módszerrel. A sertés kobuvírus RNS-t összesen 32 (53,3%) esetben azonosítottuk a bélsármintákban, míg a vírus 16 (26,6%) esetben a szérumból is ki lehetett mutatni (9. táblázat). 9. táblázat: A sertés kobuvírus RNS incidenciája, házi sertésektől gyűjtött fécesz és szérumminta párokban. Az állatok ugyanarról a magyarországi farmról származnak. A korcsoportonként gyűjtött mintákat (15 minta/korcsoport) RT-PCR módszerrel vizsgáltuk ban a sertésektől széklet-szérum párokat gyűjtöttünk. A minták típusa és a mintavételek ideje február november Korcsoport Fécesz Fécesz Szérum <10 nap 15/15 (100%) 8/15 (53,3%) 1/15 (6,7%) <3-4 hét 14/15 (93,3%) 13/15 (86,7%) 7/15 (46,7%) <3 hónap 3/15 (20%) 5/15 (33,3%) 0/15 (0%) <6 hónap 7/15 (46,7%) 6/15 (40%) 8/15 (53,3%) 39/60 (65%) 32/60 (53,3%) 16/60 (26,6%) 46
47 A négy életkori korcsoportból gyűjtött féceszmintáknál minden korcsoportból legalább egy esetben azonosítottuk a vírust, a szérumminták esetén kizárólag a 3 hónapos korcsoportból nem sikerült a vírust kimutatni. A 10 napnál fiatalabb állatok szérum mintáiban 1 esetben, a 3-4 hetes állatok szérum mintáiban 7 esetben és a 6 hónapos állatok szérummintáiban pedig 8 esetben kaptunk specifikus méretű PCR-terméket. A sertés kobuvírust 7 esetben csak a szérumból és 23 esetben pedig csak a bélsárból lehetett azonosítani. Kilenc olyan mintapár volt (szérum-fécesz), ahol mind a féceszben és a szérumban is megtaláltuk a sertés kobuvírus RNS-t. A kobuvírus 3D régiójának 173 bp hosszúságú szakasza azonosnak bizonyult mind a 2007-ben és 2008-ban gyűjtött széklet és szérumminták esetén is. A 2008-ban a 3-4 hetes korcsoportból gyűjtött, kobuvírust tartalmazó székletminták közül az egyiknek meghatároztuk a teljes genomszekvenciáját (kobuvirus/swine/k-30-hun/2008/hungary; GQ249161) és összehasonlítottuk a prototípus kobuvirus/swine/s-1-hun/2007/hungary vírus szekvenciájával (10. táblázat). A nem szerkezeti régióban (2A-3D) 74 (1,7%; 1,14%-2,43% a 3C és 3D régiók között) nukleotid cserét tudtunk meghatározni a két kobuvírus összehasonlítása után. A nukleotid mutációk aminosav változást is eredményeztek 13 (0,9%) esetben (2, 2, 5, 2 és 2 aa változás a 2A, 2B, 2C, 3C és 3D régiókban). A szerkezeti régiókban ezzel szemben 36 (1,42%) nukleotid cserét találtunk, ami összesen 6 (0,71%) aminosav változást eredményezett a VP0 (3 aa csere) és a VP3 (3 aa csere) régiókban. A legmagasabb és a legalacsonyabb nukleotid szintű mutációs változást az L és a 3D régiókban találtuk (10. táblázat). A vizsgált 21 hónapos időintervallum alatt a legmagasabb szintű aminosav változást a 2C (1,49%), a 2A (1,47%), és a VP3 (1,34%) kódoló régiókban figyeltük meg (10. táblázat). Aminosav változást a VP1, 3A és 3B régiókban nem tapasztaltunk. 47
48 10. táblázat: A sertés kobuvírus kobuvirus/swine/k-30-hun/2008/hungary (GQ249161) nukleotid és aminosav összehasonlító elemzése a prototípus sertés kobuvírussal kobuvirus/swine/s-1-hun/2007/hungary (EU787450). A februárban és novemberben gyűjtött székletminták ugyanazon a sertéstelepen nevelt állatoktól származnak. UTR: nem kódoló régió; L: leader régió; : 2A-3D; : VP0-VP1 Természetes, in vivo mutációs változások azonosítása két sertés kobuvírus szekvencia alapján Nukleotid Aminosav Génrégió Hossz Különbség Különbség (%) Hossz Különbség Kölünbség (%) 5 UTR ,17 L , ,51 VP , ,82 VP , ,34 VP , A , ,47 2B , ,02 2C , ,49 3A , B , C , ,04 3D , ,42 3 UTR ,6 Nem szerkezeti régió , ,89 Szerkezeti régió , ,83 Teljes genom , ,86 A szarvasmarha kobuvírus első európai kimutatása A februárban gyűjtött 32 szarvasmarha székletminta közül összesen 2 (6,25%), az 1-7,6 éves korcsoportba tartozó állat bélsármintájában mutattunk ki szarvasmarha kobuvírust. A második mintavételezés (2008) során gyűjtött mintákból kobuvírust nem volt kimutatható. A két kobuvírus 3D régióinak részleges szekvenciái azonosak voltak. A szarvasmarha kobuvírus (kobuvirus/bovine/aba-z20/2002/hungary; FJ225406) 89%-94% nukleotid és 96%-100% aminosav azonosságokat mutatott a GenBank adatbázisban található 19 ismert ázsiai szarvasmarha kobuvírus szekvenciával. Az Aba-Z20 vírus (FJ225406) 93% nukleotid szintű azonosságot mutatott a prototípus U-1 vírus 3D/3 UTR (862 nt) szakaszon, míg kizárólag a 3 UTR (174 nt) rész hasonlósága 95% volt. A GenBank adatbázisban elérhető 48
49 szarvasmarha kobuvírus szekvenciák és a magyarországi Aba-Z20 kobuvírus 3D régióinak nukleotid szintű filogenetikai összehasonlítását a 12. ábra szemlélteti. 12. ábra: A szarvasmarha kobuvírus (kobuvirus/bovine/aba-z20/2002/hungary; FJ225406) filogenetikai összehasonlítása az ismert kobuvírusokkal. Az Aichi kobuvírust és az S-1-HUN sertés kobuvírust külső csoportként használtuk. A filogenetikai fa a kobuvírusok 3D régióinak 455 nukleotid hosszúságú nukleotid szekvenciái alapján készült. A saját szekvenciát vastagon szedve emeltük ki. A filogenetikai fa megrajzolásához Neighbor-Joining módszert használtunk (MEGA 4.1; A bootstrap értékeket 1000 ismétlésre állítottuk és az 50%-nál nagyobb értékeket az elágazási csomópontoknál tüntettük fel. A referencia szekvenciákat a GenBank adatbázisából gyűjtöttük. Kobuvírus leírása és teljes genomjának meghatározása juhokból A juhoktól (Ovis aries) márciusban vett 8 féceszminta közül 5 (62,5%) esetben tudtunk kobuvírust kimutatni RT-PCR módszerrel az UNIV-kobu primerekkel. A 216 nukleotid hosszúságú részleges 3D régiót elemezve mind az 5 juh kobuvírus szekvenciája azonos volt. A kimutatott juh kobuvírus (kobuvirus/sheep/tb3-hun/2009/hungary; 49
50 GU245693) 3D (688 nt) és 3 UTR (174 nt) régióit összehasonlítva az Aichi kobuvírussal 59% nukleotid és 77% aminosav, míg a sertés kobuvírussal (S-1-HUN) összehasonlítva 66% nukleotid és 84% aminosav azonosságokat találtunk. Viszont a szarvasmarha kobuvírusokhoz (U-1) nagyfokú (89% nukleotid és 97% aminosav) azonosságot kaptunk a 862 nukleotid hosszú 3D/3 UTR régió elemzése során Az adatbázisban elérhető szarvasmarha, sertés és humán kobuvírusok, valamint a TB3-HUN 3D/3 UTR 862 nukleotid hosszúságú átfedő szekvenciáinak filogenetikai elemzése a 13. ábrán látható. A TB3-HUN a szarvasmarha kobuvírusokhoz mutat legközelebbi rokonságot. 13. ábra: A juh kobuvírus (kobuvirus/sheep/tb3-hun/2009/hungary, GU245693) filogenetikai összehasonlítása a humán, szarvasmarha és sertés kobuvírusokkal a 3D/3 UTR régió alapján. A filogenetikai fa megrajzolásához Neighbor-Joining módszert használtunk, (MEGA 4.1; A bootstrap értékeket 1000 ismétlésre állítottuk és az 50%-nál nagyobb értékeket az elágazási csomópontoknál tüntettük fel. A referencia szekvenciákat a GenBank adatbázisából származnak. A juhból kimutatott kobuvírus szekvenciát vastagon szedve emeltük ki. 50
51 A TB3-HUN kobuvírus teljes genomja 8378 nt hosszúságú. Az 5 UTR 797 nt hosszúságú és a másodlagos szerkezete a szarvasmarha (U-1) kobuvírus 5 UTR-hez hasonlít. A TB3-HUN kódoló régiója 7407/(2469 aa) nukleotid hosszúságú. Az L-fehérjéje 564/(188 aa) nukleotid hosszúságú, ami 1 aminosavval hosszabb az U-1 kobuvírusnál. A P1, P2 és P3 régiói 2571/(857 aa), 1902/(634 aa) és 5013/(1671 aa) nukleotid hosszúságúak. A 3A régió 12/(4 aa) nukleotiddal hosszabb, míg a 3D régió 3/(1 aa) nukleotiddal rövidebb, mint az U-1. Az egyes régiók közötti hasítási helyek megegyeznek az U-1 vírussal kivéve a 3B/3C határán találtunk (Q/A Q/S) változást. A TB3-HUN és az U-1 kobuvírus kódoló génrégióiban megfigyelhető nukleotid/aminosav hosszúságbeli különbségek az 14. ábrán láthatóak. 14. ábra: A TB3-HUN juh kobuvírus genom kódoló régióinak összehasonlítása az U- 1 szarvasmarha kobuvírussal. Felül a téglalapok (fekete, zöld és piros) a P1, P2 és P3 régiókat jelölik. Minden egyes génrégió nukleotid (felső szám) és aminosav (alsó szám) hosszúságát feltüntettük. A TB3-HUN adott génrégión belüli eltérését az U-1 genomtól vastagon narancssárgával szedve jelöltük. 51
52 A humán, a szarvasmarha, a sertés és a juh kobuvírus L, P1, P2 és P3 kódoló régióinak egymáshoz való hasonlóságát az 11. táblázat foglalja össze. A TB3-HUN (juh) kobuvírus a kobuvírus nemzetségbe tartozik és az U-1 (szarvasmarha) kobuvírushoz mutat közeli rokonságot. 11. táblázat: A prototípus kobuvírusok kódoló régióinak (L, P1, P2, P3) összehasonlítása a százalékos aminosav azonosság alapján. HUMÁN AICHI (AB040749) SZARVASMARHA U-1 (AB084788) SERTÉS S-1-HUN (EU787450) JUH TB3-HUN (GU245693) HUMÁN AICHI (AB040749) SZARVASMARHA U-1 (AB084788) SERTÉS S-1-HUN (EU787450) L P1 P2 P3 L P1 P2 P3 L P1 P2 P3 L P1 P2 P3 L 100% 26% 23% 28% P1 100% 48% 53% 48% P2 100% 61% 59% 61% P3 100% 62% 63% 62% L 100% 34% 54% P1 100% 61% 81% P2 100% 68% 88% P3 100% 71% 92% L 100% 36% P1 100% 60% P2 100% 67% P3 100% 71% Aichi vírus (humán kobuvírus) kimutatása Magyarországon Összesen 65 (35 lány és 30 fiú) gyermek székletmintáját gyűjtöttük össze 2 hetes és 12 éves életkor között 2000-ben. A székletmintákból bakteriális kórokozó, illetve rota-, adeno-, calicivírus (norovírus és sapovírus) nem volt kimutatható. A 65-ből 1 (1,5%) székletmintából lehetett Aichi vírust (HUN298/2000/HUN; FJ225407) az UNIV-kobu primerekkel RT-PCR módszerrel kimutatni. A székletminta egy járóbeteg szakellátáson október 23-án megjelent 3 éves kislánytól származott. Az anamnézis alapján a gyermeknek október 2-án torokgyulladása volt, ami október 20-ára obstruktív bronchitisszé súlyosbodott, enyhe lázzal és száraz köhögéssel társulva. A mintavételezés napján október 23-án orrfolyás, gennyes kötőhártya-gyulladása és lázmentes hasmenése is volt. Október 25-én ismét láz (39,2ºC) és mellkasröntgennel alátámasztott gócos-tüdőgyulladást jelentkezett, emelkedett vörösvérsejt süllyedéssel (46 mm/h). A hasmenése három napig tartott. Légúti bakteriális kórokozó kimutatása irányában nem történtek vizsgálatok. A november 3-án készült ellenőrző mellkasröntgen már a normális tüdőképet mutatta. Az anémia és az emelkedett vörösvérsejt 52
53 süllyedési érték egészen december 24-ig fennálltak. A gyermek külföldön nem járt, tengeri eredetű élelmiszert nem fogyasztott. A részleges 3D régió alapján a HUN298/2000/HUN 96% nukleotid és 98% aminosav azonosságot mutatott a prototípus Japán A846/88 Aichi kobuvírushoz és 97% nukleotid, valamint 100% aminosav azonosságot mutatott a Németországban talált BAY-1-03-DEU Aichi vírushoz. A szekvencia és filogenetikai elemzés alapján a HUN298/2000/HUN az A genotípusú Aichi vírusok csoportjába tartozhat (15. ábra). 15. ábra: Filogenetikai fa az első hazánkban kimutatott Aichi vírus kobuvirus/human/szigetvar-hun298/2000/hungary, FJ és a kobuvírus nemzetség reprezentatív tagjainak felhasználásával. A hazai Aichi vírus szekvencia vastagon szedve látható. A törzsfán az A és B genotípusú Aichi vírus szekvenciák is szerepelnek. A filogenetikai fa az RNS-függő RNS polimeráz gén 173 nukleotid hosszú szakasza alapján készült Neighbor-Joining módszerrel (MEGA ver. 4.1). A bootstrap értékeket 1000 ismétlésre állítottuk és az 50%-nál nagyobb értékeket az elágazási csomópontoknál tüntettük fel. A referencia szekvenciákat a GenBank adatbázisból töltöttük le. 53
54 Új picornavírus azonosítása háziasított fürj (Coturnix coturnix) féceszmintájából az UNIV-KOBU szűrő -primerekkel Háziasított fürjtől (Coturnix coturnix) júliusában vett féceszmintából az UNIVKobu-F/UNIV-Kobu-R szűrő - primerpárral egy erős, specifikus méretű (216 bp) PCR terméket kaptunk (16. ábra). 16. ábra: Háziasított fürj féceszmintájának RT-PCR vizsgálata UNIV-Kobu-F/UNIV-Kobu-R szűrő-primerekkel és a PCR-termék agaróz gélelektroforetikus képe. A zsebek számozása balról jobbra haladva: 1. zseb: 216 bp hosszú, specifikus méretű PCR-termék háziasított fürj féceszmintájából (nyíl), 2. zseb: 100 bp molekuláris marker (Promega, Amerikai Egyesült Államok). A PCR termékeket 1,5%-os agaróz gélben futtattuk. A szekvencia meghatározást követően a GenBank adatbázisában 81%-os nukleotid azonosságot találtunk a humán enterovírus 99 (USA-Ok ) 3D régiójának részleges (74 nukleotid hosszú) szakaszával. Aminosav szinten a legközelebbi hasonlóságot (58%) a majom picornavírus 17 (YP_ ) 3D régiójának aminosav szekvenciájával kaptunk. A picornavírus (QPV1-HUN/2010, JN674502) teljes genomját meghatároztuk. A teljes RNS szekvencia 8159 nukleotid hosszúságú, melynek az 5 végen egy VPg, a 3 végen pedig egy poly(a)n farok van (17. ábra). A quail (fürj) picornavírus genomja a picornavírusokhoz 54
55 hasonló genetikai felépítéssel és jellemzőkkel rendelkezik: 5 UTR-L-P1[VP ]-P2[2A- B-C]-P3[3A-B-C-D]-3 UTR. 17. ábra: A háziasított fürj féceszmintájában azonosított picornavírus QPV1-HUN/2010, JN genomjának szerkezete. A P1 régió a strukturális a P2 és P3 régiók pedig a nemszerkezeti fehérjéket kódoló régiókat tartalmazzák. Minden egyes génrégió négyzetében feltüntettük a nukleotid (felső szám) és az aminosav (alsó szám) hosszúságokat, valamint a valószínűsített N-terminális hasítási helyeket (felül a génrégiók határain). A konzervatív picornavírus aminosav motívumokat és genombeli helyeiket a motívum első aminosavjának a pozíciójával adtuk meg és nyíllal jelöltük. Az L fehérjében megfigyelt 34 aminosav hosszúságú ciszteinben gazdag ismétlődő génszakaszt egymással illesztve kiemeltük. 55
56 A fürj picornavírus nem kódoló régióinak elemzése A fürj picornavírus 5 nem kódoló régiója 494 nukleotid hosszúságú. A GenBank nukleotid BLAST programot használva részleges szekvencia hasonlóságot találtunk 6 további picornavírus 5 UTR szekvenciájával (18. ábra). A fürj picornavírus 86% nukleotid azonosságot mutatott a denevér picornavírus 1-gyel (NC16A, HQ595340) a 11. és a 46. nukleotidok közötti szakaszon (18. ábra). Ezen kívül, egy 20 nukleotid hosszúságú konzervatív szakaszon, a nukleotidok között, teljes azonosságot találtunk a fóka picornavírussal (EU142040), a pigeon (galamb) picornavírus B-vel (FR727144), a kacsa hepatitis vírus 1-gyel (DQ249299) és a turkey (pulyka) hepatitis vírussal (HQ189775). (18. ábra). A kacsa hepatitis vírus 1, a fóka picornavírus és a bat (denevér) picornavírus 1 IRES szerkezete ismert, azonban mindegyik IVB típusú IRES-sel jellemezhető. Ennek alapján valószínűsítettük, hogy a fürj picornavírus IRES-e is IV-es típusú lehet. Az Mfold modellezés alátámasztotta a feltételezésünk, de fontos szerkezeti különbségek figyelhetők meg a valószínűsített modellben [146]. A IV-es típusú IRES szerkezet tartalmazza a II-es és III-as doméneket (III 1, III 2, III 3, III 4, IIIa, IIId, IIIe, és IIIf) (18. ábra). A pseudoknot stem 1 és 2, a linker és a spacer szerkezeti régiók hosszúsága 11, 8, 3 és 8 nukleotid hosszúak a modellben. A IIId és a IIIe hajtűszerű szerkezetek konzervatív motívumai a GAGGGAAUGGGAAACCCUC és a GGGUGACAGCCU megfigyelhetőek (18. ábra). A III 1 helikális doménben A-G mispair (UGA 210 G/UG 438 CA) található. A IV-es típusú IRES-től eltérő a III-as domén csúcsi része. A szerkezet nem egy IIIb hurokkal végződik, hanem helyette egy hosszú másodlagos szerkezet található, a csúcsi részén egy 8 -as szerű másodlagos szerkezettel, ahol a fent említett konzervatív, 20 nukleotid hosszúságú nukleotid motívum található (18. ábra). Az eddig ismert IRES szerkezetek között ez a forma egyedi. Érdekes, hogy a kacsa hepatitis vírus 1 és a fóka picornavírus esetén a csúcsi 8 -as szerkezet a 20 nukleotid hosszú konzervatív motívummal (melynek funkciója nem ismert) a II-es domén előtt található az 5 UTR-ben. 56
57 18. ábra: A quail (fürj) picornavírus (JN674502) 494 nukleotid hosszúságú 5 nem kódoló régiójának (UTR) valószínűsített másodlagos RNS szerkezete az Mfold program alapján [146]. (A): A IV-es típusú IRES III. doménjének helikális részeit (III 1, III 2 stb) és a hajtűszerű szerkezeti részeit (IIIa, IIIb, stb.), a korábbi szabályoknak megfelelően [28] jelöltük. A konzervatív nukleotid motívumokat, a polyproteint indító start kodont és a 20 konzervatív nukleotidot tartalmazó, 8 -as szerű csúcsi, másodlagos struktúrát szürke alappal ábrázoltuk. (B): A 20 nukleotid hosszú, 8 -as szerű csúcsi struktúra nukleotid illesztése más madár és a fóka picornavírus szekvenciákkal. A fürj picornavírus III-as domén csúcsi részén a 301. nukleotidtól a 328. nukleotidig tartó 20 nukleotid hosszúságú motívum azonos a seal (fóka) picornavírus (EU142040), a pigeon (galamb) picornavirus B (FR727144), a duck (kacsa) hepatitis 1 vírus (DQ249299), és a turkey (pulyka) hepatitis vírus (HQ189775) megadott szakaszaival. A fürj picornavírus 5 UTR hasonlóságát (a 11. és a 46. nukleotidok között) a bat (denevér) picornavírus 1-el (NC16A, HQ595340) vastag fekete vonallal emeltük ki. 57
58 A 3 UTR 216 nukleotid hosszúságú. Részleges szekvencia azonosságot (76%) találtunk a fürj picornavírus nukleotidjai közötti szakaszon a kacsa sapelovírus 3 UTR szekvenciájával a GenBank/BLAST keresést használva (19. ábra). A fürj picornavírus 3 UTR régiójának lehetséges másodlagos RNS szerkezete a 21. ábrán látható. 19. ábra: A fürj picornavírus 216 nukleotid hosszú 3 nem kódoló régiójának (UTR) a valószínűsített másodlagos RNS szerkezete az Mfold program alapján [146]. A fürj picornavírus 3 UTR régióban található nukleotid azonosságot az avian (madár) picornavírushoz vastag fekete vonallal jelöltük, feltüntetve a hasonlóság százalékos mértékét. A fürj picornavírus kódoló régiójának elemzése A 7449 nukleotid hosszúságú ORF egy 2482 aminosav hosszúságú polyproteint kódol. A kódoló régió a 495. nukleotid pozícióban, az AUG start kodonnál kezdődik. A genom elején az 5 UTR (1-494 nt), a végén pedig a 3 UTR (216 nt) található. Az L fehérje 1170 nt/(390 aa) hosszúságú. A teljes P1 (2571 nt/(857 aa)), P2 (1374 nt/(458 aa)) és a P3 (2334 nt/(777 aa)) hosszúságú régiók 43%, 39% és 47% aminosav hasonlóságot mutatnak a GenBank adatbázisában szereplő legközelebbi rokon szekvenciával a madár sapelovírussal (korábbi nevén duck picornavírus, TW90A, DPV: AY563023). A VP1 régió 295 aminosav hosszúságú és 35%-os hasonlóságot mutat a madár sapelovírushoz. A QPV1-HUN genom elemzése során összesen nyolc konzervatív picornavírusokra jellemző aminosav motívumot találtunk (18. ábra). (I.) A VP4 régió aminosav pozíciók közötti GQLSST peptid motívum, melynek az N-terminális vége (G 391 ) a picornavírusoknál mirisztilált. (II.) A 2C régió NTP kötő helyén az aminosav pozíciók közötti GXXGXGKS/T (GSPGQGKS) aminosav motívum. (III.) A szintén 2C régióban az aminosav pozíciók közötti DDLXQ (DDLGQ) motívum. (IV.) A 3C pro régióban a cisztein aktív helyének motívuma GXCG (GWCG) az aminosavnál fedezhető fel. (V.) A 3D pol aktív részének YGDD motívuma a aminosav pozíciók között valamint (VI.) a nagyon 58
59 konzervatív aminosav motívumok a KDELR a , (VII.) a PSG a és (VIII.) az FLKR a aminosav pozíciók között találhatók (17. ábra). Az L fehérje ciszteinben (C) gazdag (9,5%) és 5 olyan szakaszt tartalmaz, ahol 3 vagy 4 cisztein található egymás mellett. Továbbá az L fehérje egy 34 aminosav (102 nukleotid) hosszúságú ismétlődő motívumot is tartalmaz, az első motívum a aminosavak között, míg a második ismétlődés a aminosavak között van (17. ábra). Az ismétlődések között 54% nukleotid és 50% aminosav azonosságokat találtunk. Az ismétlődő szakaszban ismétlődésenként 6-7 ciszteinben (C) gazdag rész és számos hisztidin (H) aminosav található. Érdekes módon az első ismétlődés egy 3D-régióra jellemző aminosav motívumot (YGDD) kódol a aminosavak pozíciók között (17. ábra). A 2A régió rendkívül rövid, összesen 11 aminosavból áll. A NetPicoRNA programmal megvizsgáltuk a polyprotein lehetséges aminosav hasítási helyeit [5]. Az aminosav hasítási pontok a Q/G (Glutamin/Glicin) határoknál voltak minden esetben, kivéve a VP4/VP2 határon (Q 454 /S) és a VP2/VP3 határon (E 713 /G) (17. ábra). A QPV1-HUN/2010 vírus kódoló régiójának bázis összetétele 28,08% A, 24,86% G, 20,78% C és 26,27% U volt. A fürj picornavírus filogenetikai elemzése a reprezentatív picornavírusokkal az 20. ábrán látható. A fürj picornavírus a galamb picornavírus A és B vírusokkal (FR727145, FR727144) (önálló nemzetségbe nem besorolt új picornavírusok) a sapelovírusokkal (Sapelovírus nemzetség) mutat rokonságot. 59
60 20. ábra: Gyökér nélküli filogenetikai fa a quail (fürj) picornavírus QPV1-HUN/2010, JN674502; fekete nyíllal kiemelve és a 2010-ben elfogadott 12 picornavírus nemzetség reprezentatív vírusfajainak felhasználásával. A filogenetikai fa a vírusok teljes kódoló régiójának ( L, P1, P2 és P3) aminosav szekvenciái alapján készült. A pigeon (galamb) picornavirus A csak részleges (P3) szekvenciája érhető el a GenBankban. A referencia szekvenciákat a GenBank adatbázisából gyűjtöttük. A madarakban eddig talált picornavírusokat fekete körrel jelöltük. A filogenetikai fa a MEGA ver. 5 szoftverrel készült a Neighbor-Joining statisztikai módszer és a JTT helyettesítési modell felhasználásával. A bootstrap értékeket 1000 ismétlésre állítottuk és a valószínűségi értékeket a csomópontoknál százalékban adtuk meg. 60
61 A fürj picornavírus további keresése Az első mintavételezés után 9 hónappal, ugyanarról a fürjtelepről gyűjtött 10 féceszmintából 3-ban sikerült a fürj picornavírust kimutatni. A részleges 3D szekvenciák 99%-os nukleotid azonosságot mutattak a QPV1-HUN/2010 vírushoz képest. A szintén ben, kereskedelmi forgalomban kapható fürj tojások felszínéről a fürj picornavírus RNS-t nem tudtuk RT-PCR módszerrel kimutatni. Enterovírus C109 (EV-C109) első európai kimutatása Összesen 92 garatmosó folyadékot vizsgáltuk RT-PCR módszerrel két légúti szezon ( és ) alatt. A gyermekek átlagéletkora 3,7 év volt és 60%-uk 2 évesnél fiatalabb volt. A 92 mintából egy esetben (1,1%) kaptunk specifikus méretű PCR-terméket az UNIV-kobu-R/UNIV-kobu-F kobuvírus primerekkel (21. ábra). A szekvencia meghatározást követően a GenBank adatbázisában 84%-os nukleotid azonosságot találtunk a coxsackievírus A17 (G12, AF499639) 3D régiójának részleges (216 nukleotid hosszú) szakaszával. Aminosav szinten a legközelebbi azonosságot (94%) az enterovírus C104 (AGA18499) 3D régiójának aminosav szekvenciájával kaptunk. Az adatbázisban 2010-ben elérhető prototípus enterovírus C109 (NICA , GQ965517) ismeretében a VP1 meghatározásához készített specifikus primerekkel [140] a 92 mintát ismételten vizsgálva szintén egy esetben (1,1%) kaptunk specifikus méretű (240 nukleotid hosszú) PCR-terméket (22. ábra). A termék 94/96% nukleotid és aminosav azonosságot mutatott a prototípus enterovírus C109 (NICA ) vírushoz. 61
62 21. ábra: Az enterovírus C109 (L87/HUN/2007, JN900470) első hazai kimutatása gyermekkori légúti fertőzésből. Az ábrán (felül) az EV109 teljes kódoló régiója (P1, P2 és P3) valamint az 5 UTR és a 3 UTR elhelyezkedése látható. [A] Az UNIV-kobu-F/UNIV-kobu-R primerekkel felsokszorozott PCR-termékek agaróz gélelektroforetikus képe. M: 100 bp DNS marker (Promega, Amerikai Egyesült Államok), 1-10 zseb: gyermekkori légúti betegminták. A 8. zsebben az L87/HUN/2007 vírus specifikus méretű PCR-termékét fekete nyíllal jelöltük és helyzetét a genomban jelöltük. A fekete téglalap az UNIV-kobu-F/UNIV-kobu-R primerek által felsokszorozott terméket jelöli a genomban (nem méretarányos). [B]: A 123F/363R primerekkel felsokszorozott PCR-termékek agaróz gélelektroforikus képe. Az első zsebben a 100 bp DNS marker a 2. zsebben az L87/HUN/2007 vírus PCR-terméke látható a gyermekkori légúti mintából. A szürke téglalap a 123F/363R primerek által felsokszorozott terméket jelöli a genomban (nem méretarányos). A futtatáshoz 1,5%-os agaróz gélt használtunk. Az enterovírus nemzetség osztályozásához használt VP1 kapszid fehérjét szaggatott vonallal jelöltük. Az L87/HUN/2007 (JN900470) enterovírus teljes genomját meghatároztuk. A teljes RNS szekvencia 7354 nukleotid hosszúságú, melynek az 5 végéhez egy VPg, a 3 végéhez pedig egy poly(a) farok csatlakozik (21. ábra). A enterovírus genomja a picornavírusokhoz hasonló genetikai felépítéssel és jellemzőkkel rendelkezik: 5 UTR-P1[VP ]-P2[2A-BC]-P3[3A-B-C-D]-3 UTR (21. ábra). Az L87/HUN/2007 nem rendelkezik L fehérjével a VP4 régió előtt. A teljes VP1 génrégió (891/(297 aa) nukleotid) 92% nukleotid és 96% aminosav azonosságot mutat a prototípus enterovírus C109 (NICA ) vírushoz (22. ábra). Az L87/HUN/2007 és a NICA vírusok P1, P2 és P3 régióinak összehasonlítása során 92/98%, 93/98% és 93/97%-os azonosságokat kaptunk nukleotid és aminosav szinten. 62
63 Az L87/HUN/2007 5UTR régiója 663 nukleotid hosszúságú, aminek a azonossága 96%-os a NICA vírus 5 UTR régiójához. 22. ábra: A reprezentatív humán enterovírusok teljes VP1 kapszid régióinak aminosav szintű filogenetikai elemzése. A filogenetikai fa elkészítéséhez a Picornaviridae honlapon ( található enterovírus szekvenciákat használtuk fel. Az enterovírus C csoportba tartozó L87/HUN/2007 (JN900470) vírust halványszürke téglalappal jelöltük. A szekvenciák neveit és GenBank referencia számait a filogenetikai fán tüntettük fel. A filogenetikai fát a MEGA5 szoftvercsomagban található UPGMA statisztikai módszerrel, az evolúciós távolságokat a Jones-Taylor-Thornton (JTT) mátrix alapú modellel számoltuk ki. Az 50%-nál alacsonyabb bootstrap értékeket a fán nem tüntettük fel. 63
64 Az L87/HUN/2007 rekombinációs vizsgálata Az L87/HUN/2007 vírus ugyanolyan rekombinációs mintázatot mutat, mint a prototípus: nem-ev-c-szerű nukleotid szekvencia az 5 UTR részen és EV-C-szerű nukleotid szekvencia a kódoló régióban és a 3 UTR régióban (23. ábra). 23. ábra: Az L87/HUN/2007 enterovírus (EV-C109) bootscan elemzése. Az elemzéshez a Neighbor-Joining/Kimura 2 statisztikai módszert alkalmaztunk a SimPlot program segítségével. Az elemzés további beállításai: window: 200, Step: 20 bp, GapStrip: On, Reps: 1000 voltak. Az ábrán középen az enterovírusok részleges sematikus genom felépítése látható. Az 5 UTR és a VP4 (kódoló régió) határán az EV-C és EV-A csoportú enterovírusok közötti rekombináció látható. Az enterovírus C109 fertőzés klinikai esetismertetése A 2,5 éves fiúgyermeket január 23-án vették fel a Kaposi Mór Oktató Kórház Pulmonológiai Osztályára. A gyermek erősen köhögött, ziháltan vette a levegőt és folyt az orra. A testhőmérséklete 38,1 C volt. Az édesanya elmondása alapján a tünetei 4 nappal a kórházi felvétel előtt kezdődtek (12. táblázat). A fizikai vizsgálat során a tüdők felett zörej hallatszódott. Melkasröntgen-felvételen kiterjedt kétoldali peribroncho-vascularis infiltráció és néhány foltos árnyék látszódott a jobb oldali tüdőfélben. Az emelkedett (23 mm/h) vörösvértest süllyedés mellett más laboratóriumi paraméterek (pl., fehérvérsejtszám, C-reaktív fehérje (CRP), stb.) a normál értéktartományon belül voltak. A klinikai diagnózis 64
65 orrnyálkahártya gyulladás, bronchitis és pneumónia voltak. Alapbetegségként meg kell említeni a krónikus obstruktív légcsőhurutot. A kórházi felvételt követően a betegsége 4. napján, január 23-án történt a garatmosó folyadék vétele. Nozokómiális bakteriális felülfertőződést valószínűsítettek, azonban a rutin laboratóriumi torokleoltás bakteriális kórokozót nem mutatott ki. A felvételt követő 13. napon a beteget jó egészségi állapotban hazaengedték. A légúti mintából a EV-C109-en kívül további virális kórokozót (légúti óriássejtes vírus (RSV), influenza, humán metapneumovírus (hmpv) és anellovírust) nem tudtunk azonosítani. 12. táblázat: A hazai enterovírus C109 (EV-C109) fertőzés jellemző tüneteinek összehasonlítása a nicaraguai gyermekek (n = 5) klinikai jellegzetességeivel. Esetünkben a gyermeknél ziháló légzés is megfigyelhető volt. Nicaragua január - április M agyarország január Láz Nátha Köhögé s Torokfájás Hányás/Hasi fájdalom N yirokcsomó duzzanat Fejfájás Ziháló légzés 38,1 C Beteg 1 Beteg 2 Beteg 3 Beteg 4 Beteg 5 Beteg 1 (3 éves) (3 éves) (3 éves) (11 éves) (11 éves) (2,5 é ve s) 65
66 MEGBESZÉLÉS A Picornaviridae család a jelenleg ismert legnépesebb víruscsalád. Csak az elmúlt 4 évben 8-ról 17-re emelkedett a picornavírus nemzetségek száma. A Picornaviridae családba 2010-ben 12 nemzetség és 29 vírusfaj tartozott [32; 33], mely februárban további 5 nemzetséggel (Aquamavírus, Cosavírus, Dicipivírus, Megrivírus és Salivírus) és 8 vírusfajjal bővült [31; 35]. Az újonnan felfedezett picornavírusok száma rohamosan növekszik [50], jelenleg is számos új picornavírus vár rendszertani besorolásra, több közülük potenciálisan új nemzetség első tagja. E mellett a változások érintik a picornavírusok alapdefinícióját is. Az eredeti meghatározás szerint, minden picornavírus genom egy leolvasási kerettel (ORFs) rendelkezik ben azonban kutyák féceszmintájából olyan új dicisztronos genomú picornavírust (Cadicivírus A, Dicipivírus nemzetség) fedeztek fel, melynek genomja a P1 és a P2/P3 régiók között egy IRES-sel rendelkező intergenikus régiót (IGR) is tartalmaz [131] A doktori értekezésben a Kobuvírus, az Enterovírus és egy jelenleg még be nem sorolt picornavírus nemzetségbe tartozó picornavírusokról, köztük új picornavírus fajokról számoltunk be emberi és állatoktól származó mintákból. A pozitív, egyszálú RNS genomú picornavírusok sikeres azonosításának kezdetben véletlen, módszertani okai voltak. A molekuláris módszer (RT-PCR) során alkalmazott primerek aspecificitása vezetett, új, eddig nem ismert vírusok azonosításához. A kezdeti lépésben a p289/p290 jelű általános calicivírus primerpárral a sertés kobuvírust, majd az UNIV-kobu-R/UNIV-kobu-F primerpárral a kobuvírusok, Aichi, szarvasmarha, juh és sertés kobuvírus mellett a fürj picornavírus és az EV-C109 azonosítását tette lehetővé. Olyan a calicivírusok és a kobuvírusok genomrészeire specifikusnak hitt primerekkel (p289/p290, UNIV-kobu-R/UNIV-kobu-F) dolgoztunk, melyekről a használat során derült ki, hogy addig ismeretlen vírusok kimutatására is alkalmasak. E szerencsés események időrendi sorrendjét érdemes külön ábrán szemléltetni (24. ábra). 66
67 24. ábra: Az értekezésben bemutatott picornavírusok azonosításának időrendi sorrendje (a megjelent közlemények címeivel) [81; 82; 92; 93; 94; 95; 96; 97; 99] az alkalmazott p289/p290 (szaggatott vonal), majd az UNIV-Kobu-R/UNIV-Kobu-F (szaggatott vonal) primerek segítségével. A sertés kobuvírus felfedezése és leírása Vizsgálataink kezdetén két hivatalosan elfogadott vírusfaj az Aichi és a szarvasmarha kobuvírus, (2013. februártól: Aichivírus A és Aichivírus B néven) tartozott a Kobuvírus nemzetségbe, melyet emberi [132] és szarvasmarha [138] széklet mintákból mutattak ki. A házi sertésekben a kobuvírus jelenléte nem volt ismert. A sertésekből kimutatott picornavírus (S-1-HUN) a teljes nukleotid és a kódoló régió aminosav (L, P1, P2 és P3) szekvenciáinak elemzése alapján a kobuvírusok közé tartozik. A filogenetikai elemzés során az S-1-HUN a Kobuvírus nemzetségen belül, de az Aichi (Aichivírus A) és szarvasmarha (Aichivírus B) kobuvírusoktól eltérő filogenetikai vonalon helyezkedik el. A sertésekből kimutatott kobuvírus, sertés kobuvírus, a kobuvírus nemzetségen belül egy új vírusfajt képvisel, melyet az ICTV márciusban Aichivírus C néven elfogadott [35; 55; 56]. Az Aichi vírus, a szarvasmarha és sertés kobuvírusok genomjai közötti hasonlóság mellett azonban ki kell emelni a sertés kobvírus nem kódoló 5 UTR részének különbözőségét. Bár az S-1-HUN RNS 5 UTR első 108 nukleotidja és a stem-hurok domének másodlagos szerkezete (három stem-hurok SL-A, SL-B és SL-C) nagyon hasonlóak az ismert kobuvírusokéhoz, az 5 UTR 3 vége benne az IRES pedig különbözik. Míg az Aichi vírus és a szarvasmarha kobuvírus V-ös típusú IRES-sel rendelkezik melyet 2012-ben határoztak 67
68 meg [117] addig a sertés kobuvírusnak IV-es típusú IRES-e van. A IV-es típusú IRES-t [hepacivirus/pestivirus-szerű (HP)] először a Flaviviridae családba tartozó Pestivírus és Hepacivírus (hepatitis C vírus) nemzetségekbe tartozó vírusoknál írták le [28]. A IV-es típusú IRES-t a picornavírusok körében is Hellen és de Breyne modellezte először 2007-ben [28]. Ezt a szerkezetet lehet megtalálni például a sertés teschovírusnál, a madár encephalomyelitis vírusnál, a kacsa hepatitis vírus 1-nél, a kacsa picornavírusnál, a sertés sapelovírusnál, a Seneca Valley vírusnál és a majom picornavírusnál is [28; 41]. Bár bizonyíték még nincs rá, de feltételezhető, hogy a HP-szerű, IV-es típusú IRES rekombináció mechanizmusával cserélődhettek ki az akár egymással rokonságban nem álló víruscsaládok között. Ezt a feltevést erősíti, hogy az esetleges rekombináció pontja az RNS kódoló és nem kódoló régiói között tapasztalható. Ez azt is jelentheti, hogy az egyes genomrégiók modulárisan cserélődhetnek, így egymástól független evolúciós órával is jellemezhetők a picornavírusoknál. Hogy a sertés kobuvírus miért rendelkezik a többi kobuvírustól eltérő IRES-sel jelenleg még nem tudni. Mindenesetre ez az észlelet valószínűleg jelentősebb, mint azt jelenleg gondoljuk, hiszen ez az első eset, hogy egy adott picornavírus nemzetségen belül 2 különböző IRES-sel jellemezhető vírusfajjal találkozunk, mely talán a jövőre vonatkoztatva nem kivétel, hanem általános tulajdonság is lehet [79; 117]. További, kiemelésre érdemes hasonlóságok és különbségek találhatók a három kobuvírus faj között. Ezek az L-fehérjében, a kapszid, a 2A és a 3B fehérjékben vannak [114]. Az aminosav hasonlóság az L-fehérjében a legalacsonyabb az S-1-HUN, az Aichi és az U-1 vírusok között. Összesen két konzervatívnak tekinthető aminosav motívumot (PEDXLXDS és a LPG) lehet megfigyelni a három virális L-fehérjében, melyek funkciója nem ismert [135]. Az S-1-HUN kobuvírusban nem található meg sem a száj és körömfájás vírusának L- fehérjéjében leírt konzervatív papain-szerű thiol proteáz katalitikus diádja (ciszterin és hisztidin) [25], sem pedig az encephalomyocarditis vírusnál leírt cink-kötő motívum (C-H-C- C) [14]. A kapszid fehérjék közül a VP1 fehérjében a legalacsonyabb a százalékos hasonlóság az S-1-HUN, az Aichi vagy U-1 vírusok között. Az eddigi egyetlen közleményben, ahol vizsgálták az Aichi vírus elleni antitest jelenlétét más állatfajokban, Aichi vírus elleni antitestet nem mutattak ki sertésekben és szarvasmarhákban, illetve fordítva, szarvasmarha U- 1 vírus elleni antitestet sem mutattak ki humán és sertés szérum mintákban [138]. Ez az eredmény közvetett módon erősíti azt, hogy a sertés kobuvírus nemcsak különálló genetikai, hanem különálló antigén típust (szerotípus) is alkothat. 68
69 Az S-1-HUN, U-1 és Aichi vírusoknál is megtalálhatóak a H-box/NC motívumok a 2A fehérjékben [30]. A nem szerkezeti 2B fehérjében a 195 aminosav hosszúságú szakaszon belül egy 30 aminosav (90 nukleotid) hosszúságú egymást követő ismétlődő (tandem) szakasz azonosítható az S-1-HUN-ban. Ilyen sem az Aichi vírusban, sem az U-1 vírusban nem található. A 2B az enterovírusoknál egy viroporin [19; 60; 65; 129], mely a sejten belül, a membrántranszport folyamatok elősegítésében vesz részt. Nem kizárt, hogy ez a fehérje a kobuvírusoknál is hasonló funkciót lát el. Talán az S-1-HUN vírusnál a pórusképző domén megkettőződését látjuk a 2B-ben, mely fehérjeszerkezeti modellvizsgálatokkal lehetne bizonyítani. A 2B-ben található tandem ismétlődés egyébként a májban azonosított IQmotívumot tartalmazó GTP-ase aktiváló 2 fehérje C-terminális részéhez (IQGAP2) 23%-ban hasonlít [9]. Hogy a 2B fehérje és az IQGAP2 fehérjék közötti hasonlóság szignifikáns-e az jelenleg nem ismert. A picornavírusok körében a kódolt fehérje motívumok egymást követő ismétlődése egyelőre ritkán felismert jelenség [42], a fürj picornavírus L-proteinjében ismertünk fel hasonlót (lásd később). Olyanra van példa (aphthovírusoknál: foot-and-mouthdisease vírus: FMDV), hogy az adott génrégió (3B) háromszor (3B 1, 3B 2, 3B 3 ) ismétlődik a genomban egymás után [23], de ennek aminosav összetétele egymástól eltérő. Mind a három kobuvírus fajra jellemző, hogy a genomjukban más picornavírusokkal összehasonlítva magas pirimidin bázisok aránya [93; 138]. E jellegzetesség talán a kobuvírusokra jellemző bélyeg lehet [93; 138]. A sertés kobuvírus fertőzés molekuláris epidemiológiája és in vivo evolúciós vizsgálata A vizsgált sertés farmon a látszólag egészséges sertésekben a sertés kobuvírus igen magas (átlagosan 65%) százalékban volt jelen, melyet a székletükkel ürítettek. A kimutatási arány a 10 napnál fiatalabb állatoknál 100% volt. A 21 hónappal később ugyanazon a telepen megismételt mintavétel hasonló eredményre vezetett. A fécesz minták több mint fele sertés kobuvírus pozitív volt a különböző korcsoportokban. Ez azt jelenti, hogy a sertés kobuvírus endémiásan jelen van a vizsgált sertéstelepen. A második mintavétel során 27%-ban a szérum mintákból is kimutatható volt a virális RNS (vagy fertőző kobuvírus partikulák?). Ez pedig felveti annak a lehetőségét, hogy a kobuvírusok kiléphetnek az emésztőszervrendszerből a keringésbe és virémiát okozhatnak immunkompetens vírusfertőzött gazdaszervezetekben. További vizsgálatok elvégzése szükséges annak meghatározására, hogy ez a virémia aktív vagy passzív-e, illetve aktív virémia esetén melyek lehetnek a további célszervek. A virémia bizonyítása azért is fontos, mert ez az első eset, amikor a kobuvírus a véráramból kimutatásra 69
70 került. Ez az észlelet más megvilágításba helyezi a szarvasmarha kobuvírus felfedezését is, hisz leírásakor Yamashita és mtsai a sejttenyészet szarvasmarha fécesszel való kontaminálódását adták meg a szarvasmarha kobuvírus lehetséges forrásának [32; 138]. A virémia ismeretének birtokában valószínűbb, hogy a sejttenyésztéshez használt bovin szérum albumin (virémia!) tartalmazhatta a szarvasmarha kobuvírust. Így nem zárható ki annak a lehetősége sem, hogy a kobuvírusok általában is okozhatnak virémiát. A sertés kobuvírus kóroki szerepe jelenleg nem ismert. A sertés kobuvírus virémia azonban más megvilágításba helyezi nemcsak a szarvasmarha kobuvírus fertőzéseket, hanem az Aichi vírus pathogenezist is emberben. A fentiek alapján a valószínű domináns fekális-orális terjedés mellett elméletileg a kobuvírus terjedhet emlőből szoptatás során vagy vér útján is. Az átviteli utak bizonyításai további vizsgálatokat igényelnek. A nyomonkövetéses vizsgálattal első ízben történt meg egy kobuvírus in vivo gazdaszervezetek közötti evolúciójának elemzése. A sertéstelepen az egymáshoz közel élő, fertőzésre fogékony fiatal gazdaszervezetek generációi folyamatosan fertőződnek. Ennek következtében a vírusreplikáció és a vírus gazdaszervezeten kívül (nem replikatív virális alak) eltöltött ideje között eltelt időintervallumnak rövidnek kell lennie. A vizsgált két virális genom közötti nukleotid változás aránya megegyezett más picornavírusoknál leírt mutációs értékekkel [16; 20; 39], sugallva ezzel a vírus és a gazdaszervezet jó adaptációs kapcsolatát. Az alkalmazkodást a különböző génrégiókban található nukleotid változások is alátámasztják. Több nukleotid mutációt számoltunk össze a nem szerkezeti, mint a szerkezeti régiókban, ami azonban közel azonos gyakorisággal okozott nem szinonim aminosav változást a két régióban. A sertés kobuvírus felfedezése óta már számos közlemény számolt be a sertés kobuvírusok kimutatásáról [3; 4; 13; 21; 24; 45; 46; 64; 76; 83; 101; 113; 126; 127; 128; 141; 142; 145] megerősítve ezzel eredményeinket. Valószínűsíthető, hogy a sertés kobuvírus fertőzés nem köthető konkrét földrajzi régióhoz, hanem széles körben elterjedt a sertések körében világszerte. A sertés kobuvírus kimutatásáról szóló közlemények jelentek meg ben Kínában [141] és Thaiföldön [45], majd 2010-ben Japánban [46] és Dél-Koreában [83], 2011-ben Brazíliában és Hollandiában [4], végül 2013-ban a Cseh Köztársaságban [21] és az Amerikai Egyesült Államokban [126]. A klinikai tüneteket nem mutató sertések fécesz mintáinak 30%, 99%, 45% és 52%-ában volt jelen a sertés kobuvírus Kínában, Thaiföldön, Japánban és Koreában. Úgy néz ki, hogy a sertés kobuvírus, az eddig ismert vírusok közül, a leggyakrabban jelen lévő vírus a sertések feceszében. A sertés kobuvírus jelenléte a székletmintákban az életkor előrehaladtával csökkent minden fenti vizsgálatban. A sertés 70
71 kobuvírus jelenlétét 2013-ban az Egyesült Államokban is igazolták molekuláris módszerrel [126]. A 114 hasmenéses és 46 látszólag egészséges sertés fécesz mintáját vizsgálva azt tapasztalták, hogy a hasmenéses tüneteket mutató sertések 21,9%-ka, míg a látszólag egészséges állatok 21,7%-ka volt fertőzött sertés kobuvírussal. Úgy tűnik az első vizsgálatok alapján, hogy a sertés kobuvírus fertőzés talán nem jár a sertéseknél hasmenéssel. De egy friss, in vitro fertőzéses kísérlet azt mutatja, hogy a sertés kobuvírus előfordulási gyakorisága a hasmenéses tüneteket mutató sertéseknél szignifikánsan (P < 0,001) nagyobb volt [145], de a hasmenéses sertéseknél társfertőzésként a porcine epidemic diarrhea vírus (PEDV) is jelen volt hasonló gyakorisággal [145]. Összefoglalva, a sertés kobuvírus világszerte, a seréstelepek endemikus vírusa, elsősorban a fiatal állatok körében. A szarvasmarha kobuvírus első európai kimutatása A szarvasmarha kobuvírust (Aichivírus B) először 2003-ban Japánban, klinikailag egészséges állatok fécesz és szérum mintáiból [138], majd 2008-ban diarrhea-ban szenvedő szarvasmarhákból azonosították Koreában [44]. A vizsgálatainkkal először sikerült a szarvasmarha kobuvírust Európában kimutatni. Időközben Belgiumból is beszámoltak a szarvasmarha kobuvírus kimutatásáról [67]. Feltételezhető, hogy a szarvasmarha kobuvírus is világszerte elterjedt, de kóroki szerepe még nem tisztázott [44; 138]. A vizsgált farmon a klinikailag egészséges szarvasmarhák székletmintáiból a szarvasmarha kobuvíruson kívül további két fekális-orális úton terjedő RNS vírust, a szarvasmarha norovírust (GIII/1 és GIII/2 genotípusok) [91] és a szarvasmarha rotavírust is ki tudtuk párhuzamosan mutatni. A szarvasmarha kobuvírus jelentőségének, gyakoriságának és változatosságának a feltérképezéséhez további epidemiológiai és molekuláris biológiai vizsgálatok szükségesek a szarvasmarha állományok körében. Érdekességképpen megemlíthető, hogy a szarvasmarha kobuvírus szekvenciát (3D régió) sertések féceszmintáiból is azonosítottak Japánban [46]. Kobuvírus kimutatása és teljes genomjának meghatározása bárányból Kobuvírust juhból korábban még nem mutattak ki. A hazai juhok fécesz mintáiból azonosított kobuvírus a szarvasmarha kobuvírus (Aichivírus B) szekvenciákhoz áll a legközelebb. A filogenetikai elemzés alapján is a szarvasmarha kobuvírus fajba sorolható. Az 71
72 eredmény fontos kérdéseket vet fel: (a) jelen lehet-e és (b) kórokozó-e ez a nagyon hasonló kobuvírus a két különböző állatfajban (szarvasmarha és juh), (c) vagy az eredmény egy természetes kontamináció következménye? Azt, hogy a juh a szarvasmarha kobuvírus gazdaszervezete lehet alátámasztja, hogy: i) a fiatal egészséges bárányok székletében magas arányban jelen volt a kobuvírus, ii) a legkonzervatívabb genetikai régió (3D) szerint készült filogenetikai fán a kobuvírus egy szarvasmarha kobuvírus alcsoportban helyezkedik el, iii) valamint a genetikai rokonság a két potenciális kérődző gazdaszervezet (szarvasmarha és juh) között. Az egy kórokozó jelenléte 2 különböző gazdafajban jelen esetben szarvasmarhában és a juhban - jól ismert jelenség: példa lehet a bluetongue ( kéknyelv ) vírus, az adenovírusok, a juh herpeszvírus 2-es típusa és a száj és körömfájás vírus (picornavírus), melyek mindkét állatfajban kórokozók [122]. Ennek ellenére a természetes keresztkontamináció lehetőségét sem zárhatjuk ki. Elképzelhetőnek tartjuk a vírus passzív átviteli módját is a juhokba, mivel egy szarvasmarha telep helyezkedik el a vizsgált juh telep mellett. Ez lehetővé teszi a kobuvírusok fekál-orál úton történő átvitelét a két telep állatai között. Ezért egyelőre egyik lehetőséget (gazda és passzív vírushordozás) sem tudjuk elvetni, kizárni. A kobuvírusok jelentőségének, gyakoriságának és változatosságának felmérésére további molekuláris biológiai és epidemiológiai vizsgálatok lennének szükségesek juhokban. Az Aichi vírus (humán kobuvírus) kimutatása Magyarországon Az Aichi vírus (Aichivírus A) 1989-es (Japán) felfedezését [132; 135] követően csak 2006-ban kezdték el az Aichi vírus keresését Ázsián kívüli más földrajzi régiókban, így Németországban és Brazíliában [77], Franciaországban [2] és Tunéziában [112]. Magyarországon először sikerült Aichi vírust kimutatnunk egy enterális részben ezzel egyidőben légúti tüneteket mutató gyermek székletmintájából. [94]. Bár a kimutatási arány (1,5%) alacsony ez nem tér el az eddig közölt tanulmányok alacsony kimutatási gyakoriságaitól a szórványos gastroenteritisekben. Pakisztánban és Franciaországban 222 esetből 5 (2,2%), illetve 457-ből 4 (0,9%) esetben tudták igazolni az Aichi vírus jelenlétét hasmenéses tüneteket mutató gyermekek székletmintáiból [2; 134]. A közelmúltban megjelent tanulmányokban Japánból, Bangladesből, Thaiföldről és Vietnámból származó hasmenéses tüneteket mutató felnőttektől és gyerekektől összesen 912 székletmintát vizsgáltak, amiből 28 (3%) esetben tudták az Aichi vírus jelenlétét igazolni [84]. Magyarországon a között, a 75 ismeretlen eredetű (nem bakteriális és nem rota-/noro-/sapovírus járvány) gastroenterális járványból összesen 233 székletmintából egy esetben sem tudtunk Aichi 72
73 vírust kimutatni. Egy Sanghajban végzett tanulmány is megerősíti az eredményeket miszerint 445 akut hasmenéses 6 év alatti gyermek székletmintáiból csak 8 (1,8%) esetben tudták az Aichi vírus RNS-t kimutatni [139]. Egy korábbi 2005-ben, végzett finnországi felmérés szerint viszont Aichi vírust nem találtak a vizsgált 68 hasmenéses székletmintában [38]. Ugyanakkor szintén Finnországban 2010-ben nagyobb mintaszám (N=1063) mellett 5 (0,5%) gyermek székletéből tudták igazolni az Aichi vírust molekuláris módszerrel, ami szintén a 3%-os előfordulási érték alatt van [40]. Ki kell térni az Aichi vírus kórokozó-képességére. Franciaországban a 4 molekuláris módszerrel igazolt Aichi vírus fertőzöttnél 2 esetben társfertőzést is észleltek [2]. Egy tunéziai tanulmányban pedig a 22 (3,5%) RT-PCR módszerrel igazolt Aichi vírus fertőzött beteg közül 16 esetben találták csak az Aichi vírust egyedüli fertőző ágensnek, míg 6 esetben más kórokozó jelenléte is beigazolódott [112]. A dolgozatban leírt 3 éves gyereknél az Aichi vírus vírusürítésének szakaszában az enterális tünetek mellett jelen voltak légúti tünetek és gennyes kötőhártya-gyulladás is. Más ismert enterális kórokozót a székletből nem lehetett kimutatni, de molekuláris laboratóriumi vizsgálatot nem végeztünk a légúti mintából. Ennek alapján nem zárhatjuk ki a véletlen virális/bakteriális társ- vagy felülfertőződés jelenlétét az enterális tünetek mellett a légúti tünetek hátterében. Japán kutatók 397 hasmenéses, aszeptikus meningitis és felső légúti megbetegedésben szenvedő gyermek közül 1 gyermek esetben számoltak be arról, hogy Aichi vírus antigént találtak a székletmintában, akinek alsó légúti tünetei voltak, de hasmenése nem volt [133]. Az alacsony víruskimutatással ellentétben azonban már éves életkorra magas (80-95%) a szeroprevalencia Aichi vírus esetén, világszerte [2; 26; 77; 133], jelezve az általános kitettséget, cirkulációt és fertőződést a különböző emberi közösségekben. Ez talán azt jelentheti, hogy az Aichi vírusfertőzés csak ritkán jár súlyos klinikai tünetekkel. A kobuvírusok emberben, szarvasmarhában és sertésben immár igazoltan jelen vannak a különböző földrajzi régiókban. Az utóbbi egy évben pedig egerekből [22; 71; 85], kutyákból [12; 18; 43; 63] és macskákból [12] is sikerült kobuvírusokat kimutatni. Az Egyesült Királyságban végzett friss kísérlet során kutya és macska szérum mintákban Aichi vírus elleni antitesteket találtak [12]. A kutya kobuvírus és az egér kobuvírus pedig az Aichi vírustól szinte megkülönböztethetetlen (mindegyik az Aichi vírus vírusfajba tartozik). A nagyfokú egyezés miatt nem elképzelhetetlen, hogy az Aichi vírus emberben tulajdonképpen egy zoonózis! 73
74 Új picornavírus azonosítása háziasított fürj fécesz mintákból Bár az ismert madárfajok száma, több mint , de csak 8 picornavírust írtak le madarakból 2012-ig, legtöbbet kacsákból. Ezek a madár encephalomyelitis vírus (Tremovírus nemzetség) [66], a madár sapelovírus (Sapelovírus nemzetség, régebbi nevén kacsa picornavírus) [125] és a kacsa hepatitis vírus (Avihepatovírus nemzetség) [47], valamint rigókból (Turdidae madár család) a turdivírus 1 és 2 [130], pulykákból a turkey (pulyka) hepatitis vírus [29] és 2010-ben, a galamb picornavírus A és B [49]. A turdivírusok és a galamb picornavírusok rendszertani helyzete még nem tisztázott. A dolgozatban egy háziasított fürj (Coturnix coturnix) széklet mintájából egy új picornavírus kimutatását és genetikai elemzését mutattuk be. A fürj picornavírust (QPV-1) az általános kobuvírus primerpárral véletlenül azonosítottuk. A legfontosabb különbségek a picornavírusok között és így a fürj picornavírus esetén is a nem kódoló régiókban (5 UTR és 3 UTR) és az L, illetve a 2A régiókban vannak [87]. A fürj picornavírus 5 UTR régiójában található IRES szerkezet felépítése legjobban a IV-es típusú IRES-re hasonlít, bár jelentős szerkezeti különbségek figyelhetőek meg. Ezek: a III-as domén rendkívül hosszú másodlagos szerkezete és a csúcsi részen található jelenleg ismeretlen funkcióval rendelkező 20 nukleotid hosszúságú, konzervatív nukleotid motívum 8 -as szerű szerkezeti struktúrában. Érdekes, hogy ez a szerkezeti elem négy különböző madárfajból (kacsa, pulyka, galamb és a fürj) leírt picornavírusban és a fókában azonosított fóka picornavírusban is megtaláltuk. E madár eredetű picornavírusok három különböző picornavírus nemzetséget képviselnek. Ez az eredmény felveti annak a lehetőségét, hogy ez a 20 nukleotid hosszúságú 5 UTR/IRES motívum fontos szereppel rendelkezhet a madár eredetű picornavírusok replikációjában. A fürj picornavírus L-protein szintén egyedi. Az L-fehérje ciszteinben gazdag és egy ismétlődő aminosav motívum is megtalálható benne. A kódolt aminosav motívum ismétlődésére a sertés kobuvírus esetén már láttunk példát. Az ismétlődő motívum tartalmaz egy a 3D régióra jellemző konzervatív motívumot (YGDD), valamint megfigyelhető egy potenciális katalitikus elem (cisztein és hisztidin aminosavakkal), melyet a száj- és körömfájás betegség vírusának (FMDV) L-fehérjéjében írtak le először [25]. Az L-proteinen megtalálható még egy feltételezhetően cink-kötő domén (C-H-C-C) is, amit pedig az encephalomyocarditis vírus L-proteinjében azonosítottak [14]. Ez az összetett szerkezeti/funkcióbeli összefüggés az L-proteinben az egyik lehetséges magyarázata annak, hogy miért olyan rövid a fürj picornavírus 2A fehérjéje, amelynek általában szintén szerepe van a proteolitikus hasításban és vagy az L-protein nélküli picornavírusok RNSreplikációjában. A picornavírusoknál az egyelőre ismeretlen funkcióval rendelkező 2A fehérje 74
75 mérete változatos lehet, az eddig megismert vírusokban 9 és 305 aminosav hosszúságok között változik. A fürj picornavírusban a 2A fehérje mindössze 11 aminosavból áll, funkciója ismeretlen. A jelenlegi rendszertani szabályok szerint ( definitions/genus_definition.htm) egy picornavírus akkor alkothat új nemzetséget, ha a P1, P2 és P3 régiókban az aminosav azonosság az ismert picornavírusok megfelelő régióival összehasonlítva kevesebb, mint 40%, 40% és 50%. A szabályok értelmében a fürj picornavírus egy új picornavírus faj és valószínűleg egy új picornavírus nemzetség határán helyezkedik el. A 2010-ben leírt két új picornavírus (galamb picornavírus A és B) filogenetikailag rokon vírusok a fürj picornavírussal, de a P1, P2 és P3 régiókat összehasonlítva itt is csak nagyon alacsony 34%, 35% és 43% aminosav azonosságokat kapunk. Nagyon valószínű, hogy számos, egyelőre ismeretlen picornavírus található még madarakban. Továbbra is nyitott kérdés, hogy ezek az új picornavírusok egy madár eredetű picornavírus csoportot alkotnak-e közeli filogenetikai rokonságban a Sapelovírus és Enterovírus nemzetségek tagjaival, valamint hogy ezek a vírusok rendelkeznek e zoonótikus potenciállal. További kísérletek szükségesek ahhoz, hogy jellemezni tudjuk a fürj picornavírus biológiáját, ökológiáját, földrajzi elterjedtségét, patogenezisét és klinikai jelentőségét. Ez azért is fontos, mert a háziasított fürj húsa és tojása egyaránt kereskedelmi forgalomba kerül, mint alapanyag, és mint emberi élelmiszer. Általában is fontos kérdésként merül fel az utóbbi időben a madarak jelentősége a fertőző betegségek terjesztésében. A madarak evolúciós eredete, fajgazdagsága és széles vándorlási szokásaik, valamint az, hogy bizonyos madárfajok közeli szomszédságban élnek az emberrel és más állatokkal, a madarak potenciálisan fontosabb szerepét vetítik elénk a fertőző betegségek terjesztésében, mint azt előzőleg gondoltuk. Ennek tudatában a madarak hordozói és forrásai lehetnek ismert vagy ismeretlen virális kórokozóknak, melyek felderítése további vizsgálatokat igényelnek. Új enterovírus C109 (EV-C109) első európai kimutatása, teljes genomjának meghatározása, gyermekkori légúti fertőzésből A fertőző légúti megbetegedések továbbra is a vezető morbiditási és mortalitási okok között szerepelnek a világon [36; 78]. Az egyre érzékenyebb mikrobiológiai diagnosztikus módszerek alkalmazásával ebben a csoportban is növekszik az ismertté váló kórokozók száma [120]. A két légúti szezonban gyűjtött légúti mintákból többek között sikerült az első 75
76 humán metapneumovírust és a légúti óriássejtes vírus (RSV) két típusát is korábban hazánkban molekuláris módszerekkel azonosítani (13. táblázat). 13. táblázat: Két légúti szezonban, légúti fertőzésben szenvedő gyermekek (n = 92), légúti mintáiból kimutatott vírusok összefoglalása. Gyakoriság Minta jele Vírus Referencia (%) (GenBank azonosító száma) Influenza A vírus 2,2% - [80] Humán metapneumovírus HUN-L23 1,1% [88] (hmpv) (DQ518967) Légúti óriássejtes vírus (RSV) 25% (EU EU849704) [80] Anellovírus 57,6% (HQ HQ677765) [11] Enterovírus C109 1,1% L87/HUN/2007 (JN900470) [82] A mintákat az univerzális kobuvírus primerrel megvizsgálva kobuvírust nem, de egy akkor még ismeretlen humán picornavírust sikerült azonosítani. Időközben, Yozwiak és munkatársai nicaraguai gyermekek heveny légúti fertőzéseiből egy új, rekombináns C-típusú humán enterovírus (EV-C109) felfedezéséről számoltak be 2010-ben [140]. Tudomásunk szerint, az EV-C109-et eddig más földrajzi régióból még nem mutatták ki, így a hazánkban azonosított EV-C109 egyben az első európai kimutatásnak is tekinthető. A hazai EV-C109-es vírus egy évvel korábban gyűjtött és archivált garatmosó folyadékból származik, mint a nicaraguai EV-C109 vírus. Az EV-C109 vírust Nicaraguában 310 gyermek légúti mintájából 5-ben (1,6%) tudták kimutatni molekuláris módszerrel. A fertőzött gyermekek megbetegedéseinek vezető tünetei a köhögés, torokfájás, a láz és a nátha voltak, de a fejfájás, nyirokcsomó duzzanat mellett bélrendszeri panaszokat hányás, hasi fájdalom is megfigyeltek. A magyarországi kimutatási arány (1,1%) összhangban van a nicaraguai minták pozitivitási arányával. Ez azt jelenti, hogy kb. minden 100. orvosi ellátást igénylő gyermekkori légúti fertőzésben az EV-C109-nek szerepe lehet az egyes légúti szezonokban. Az EV-C109-et légúti kórokozónak tarthatjuk egyes influenza-szerű tünetekkel járó, heveny légúti fertőzésekben, továbbá szerepe lehet a kórházi kezelést igénylő, gyermekkori súlyos alsó légúti betegségekben is. Az eredmény felveti az EV-C109 molekuláris diagnosztikájának és molekuláris epidemiológiai nyomon követésének szükségességét. 76
77 ÚJ EREDMÉNYEK ÖSSZEFOGLALÁSA Az általános calicivírus primerekkel (p289/p290) véletlenül azonosítottunk egy új picornavírus fajt sertések fécesz mintáiból. A sertés kobuvírus a Kobuvírus nemzetségbe tartozik, melyet az ICTV márciusában Aichivírus C néven fogadott el új vírusfajként. Meghatároztuk a sertés kobuvírus genomjának teljes nukleotid sorrendjét. Elemeztük az S-1-HUN nem kódoló régióit (UTRs) és meghatároztuk az S-1-HUN belső riboszómakötő helyének (IRES) másodlagos RNS szerkezetét. Elemeztük az S-1-HUN kódoló régióit és összehasonlítottuk az Aichi vírus és az U-1 prototípus kobuvírus szekvenciákkal. A 2B genomrégióban egy ismétlődő szekvenciát (tandem repeat) azonosítottunk. Molekuláris epidemiológiai módszerekkel vizsgáltuk a sertés kobuvírus jelenlétét különböző korcsoportú sertések fécesz mintáiban. Kimutattuk a sertés kobuvírus gyakori és endémiás jelenlétét a vizsgált sertésfarmon. Igazoltuk a sertés kobuvírus jelenlétét szérum mintákban, ezzel felvetettük a virémia lehetőségét a kobuvírus fertőzések során. Elvégeztük a sertés kobuvírus in vivo evolúciós vizsgálatát a 21 hónappal később gyűjtött fécesz mintából kimutatott vírus (K-30-HUN) és a prototípus (S-1-HUN) teljes genomjának összehasonlításával. Meghatároztuk az egyes régiókban történt nukleotid/aminosav változásokat és megadtuk azok gyakoriságát. Először mutattunk ki szarvasmarha kobuvírust (Aichivírus B) szarvasmarha fécesz mintákból Európában. Először mutattunk ki kobuvírust (Aichivírus B) juhok fécesz mintáiból. A vírus (TB3- HUN) teljes genomját meghatároztuk. Először mutattunk ki Aichi vírust (Aichivírus A) gyermekektől származó székletmintákból hazánkban. Azonosítottunk egy új picornavírust fürj picornavírus fürj fécesz mintákból, melynek teljes genomját meghatároztuk és elemeztük. 77
78 Először mutattuk ki egy új rekombináns enterovírust EV-C109 Európában 10 év alatti, légúti tünetekben szenvedő gyermektől származó légúti mintában. Sikeresen adaptáltunk és fejlesztettünk molekuláris biológiai/genetikai módszereket (RT- PCR, Long-Range RT-PCR, primer walking PCR, 5 /3 RACE RT-PCR) a picornavírusok kimutatása és teljes genomjának meghatározása érdekében. 78
79 KÖSZÖNETNYILVÁNÍTÁS Köszönetemet szeretném kifejezni témavezetőmnek, Dr. Reuter Gábornak, hogy kitartóan segítségemre volt a kutatómunka első lépésétől kezdve a módszerek elsajátításában, az eredmények értékelésében és a dolgozat elkészítésében. Köszönöm a segítségét Dr. Kiss István, Dr. Kecskeméti Sándor, Dr. Egyed László állatorvosoknak és Kocsis Jánosnak a sertés, szarvasmarha, juh és fürj vizsgálati minták összegyűjtésében és a laboratóriumba küldésében. Köszönöm Dr. Papp Gábornak a segítségét az Aichi vírust tartalmazó minta küldésében és a klinikai adatok összegyűjtésében. Köszönettel tartozom továbbá a Kaposi Mór Oktató Kórház orvosainak Dr. Szabó Hajnalkának, Dr. Székely Gyöngyinek és Dr. Gyurkovits Kálmánnak, hogy az epidemiológiai vizsgálatunkat támogatták és nélkülözhetetlen segítséget nyújtottak a humán légúti minták gyűjtésében, valamint az epidemiológiai és klinikai adatok rendelkezésünkre bocsátásában. Köszönet illeti Boldizsár Ákost és Dr. Boros Ákost a kísérletekben és a közlemények elkészítésében tett javaslataikért és észrevételeikért. Köszönettel tartozom a laboratórium valamennyi szakdolgozójának is. Köszönettel tartozom menyasszonyomnak Kocsis Zsuzsannának az angol nyelvű összefoglalók, közlemények nyelvtani javításában tett odaadó segítségéért. Köszönettel tartozom barátaimnak és családomnak, hogy támogattak, tartalék energiákkal láttak el. 79
80 MELLÉKLETEK FELHASZNÁLT VEGYSZEREK Foszfát pufferrel kiegészített fiziológiás sóoldat (PBS): 8,5 g NaCl, 0,315 g NaH 2 PO 4, 1,096 g Na 2 HPO 4 anyagokat fel kell oldani 1 liter ioncserélt desztillált vízben (ph: 7,4). 5 X Tris-Borate (TBE): 54 g Tris-Base, 27,5 g bórsav, 4,65 g Na 2 EDTA x 2H 2 O anyagokat fel kell oldani 1 liter ioncserélt desztillált vízben (ph: 7,4). 4 M NH 4 Ac (Ammónium-acetát, CH 3 COONH 4 ): M NH4Ac =77 g/mol, 3,0832 g NH 4 Acot fel kell oldani 10 ml ioncserélt desztillált vízben (ph: 7,4). 3 M NaAc (Nátrium-acetát, CH 3 COONa): M NaAc =136 g/mol, 4,08 g NaAc-ot fel kell oldani 10 ml ioncserélt desztillált vízben (ph: 7,4). 10 U/μl M-MLV-RT: Az M-MLV-RT enzim hígítását 200 U/μl törzsoldatból végezzük. 1 μl törzs enzimehez 19 μl nukleáz mentes vizet mérek és vortex-el alaposan összekeverem. 70%-os Etanol: A hígítást 96%-os abs. etanolból készítjük. 10 ml etanolhoz 3,71 ml nukleáz mentes vizet adunk majd alaposan összekeverjük. 75%-os Etanol: A hígítást 96%-os abs. etanolból készítjük. 10 ml etanolhoz 2,8 ml nukleáz mentes vizet adunk majd alaposan összekeverjük. 80
81 IRODALOMJEGYZÉK 1. Alcalá A, Vizzi E, Rodríguez-Díaz J, Zambrano JL, Betancourt W, Liprandi F Molecular detection and characterization of Aichi viruses in sewage-polluted waters of Venezuela. Appl Environ Microbiol. 76(12): Ambert-Balay K, Lorrot M, Bon F, Giraudon G, Kaplon J, Wolfer M, Lebon P, Gendrel D, Pothier P Prevalence and genetic diversity of Aichi virus strains in stool samples from community and hospitalized patients. J Clin Microbiol. 46(4): An DJ, Jeoung HY, Jeong W, Lee HS, Park JY, Kim B Porcine kobuvirus from pig stool in Korea. Virus Genes 42(2): Barry AF, Ribeiro J, Alfieri AF, van der Poel WH, Alfieri AA First detection of kobuvirus in farm animals in Brazil and the Netherlands. Infect Genet Evol. 11(7): Blom N, Hansen J, Blaas D, Brunak S Cleavage site analysis in picornaviral polyproteins: discovering cellular targets by neural networks. Protein Sci. 5(11): Brown EA, Zajac AJ, Lemon SM In vitro characterization o fan internal ribosomal entry site (IRES) present within the 5 nontranslated region of hepatitis A virus RNA: comparison with the IRES of encephalomyocarditis virus. J Virol. 68(2): Borman AM, Le Mercier P, Girard M, Kean KM Comparison of picornavirus IRES-driven internal initiation of translation in cultured cells of different origins. Nucleic Acids Res. 25(5): Boros A, Új M, Pankovics P, Reuter G Detection and characterization of human parechoviruses in archived cell cultures, in Hungary. J Clin Virol. 47(4): Brill S, Li S, Lyman CW, Church DM, Wasmuth JJ, Weissbach L, Bernards A, Snijders AJ The ras GTPase-activating protein related human protein IQGAP2 harbors a potential actin binding domain and interacts with calmodulin and Rho family GTPases. Mol Cell Biol. 16(9): Bruenn JA Relationships among the positive strand and double-strand RNA viruses as viewed through their RNA-dependent RNA polymerases. Nucleic Acids Res. 19(2):
82 11. Burián Z, Szabó H, Székely G, Gyurkovits K, Pankovics P, Farkas T, Reuter G Detection and follow-up of torque teno midi virus ("small anelloviruses") in nasopharyngeal aspirates and three other human body fluids in children. Arch Virol. 156(9): Carmona-Vicente N, Buesa J, Brown PA, Merga JY, Darby AC, Stavisky J, Sadler L, Gaskell RM, Dawson S, Radford AD Phylogeny and prevalence of kobuviruses in dogs and cats in the UK. Vet Microbiol. 164(3-4): Cao W, Zheng H, Zhang K, Jin Y, Lv L, Yang F, Liu X Complete genome sequence of the porcine kobuvirus variant CH/HNXX-4/2012. J Virol. 86(21): Chen HH, Kong WP, Roos RP The leader peptide of Theiler's murine encephalomyelitis virus is a zinc-binding protein. J Virol. 69(12): Cho MW, Teterina N, Egger D, Bienz K, Ehrenfeld E Membrane rearrangement and vesicle induction by recombinant poliovirus 2C and 2BC in human cells. Virology. 202(1): Cottam EM, Haydon DT, Paton DJ, Gloster J, Wilesmith JW, Ferris NP, Hutchings GH, King DP Molecular epidemiology of the foot-and-mouth disease virus outbreak in the United Kingdom in J Virol. 80(22): de Jong AS, de Mattia F, Van Dommelen MM, Lanke K, Melchers WJ, Willems PH, van Kuppeveld FJ Functional analysis of picornavirus 2B proteins: effects on calcium homeostasis and intracellular protein trafficking. J Virol. 82(7): Di Martino B, Di Felice E, Ceci C, Di Profio F, Marsilio F Canine kobuviruses in diarrhoeic dogs in Italy. Vet Microbiol. 166(1-2): Dodens JR, Kirkegaard K Inhibition of cellular protein secretion by poliovirus proteins 2B and 3A. EMBO J. 14(5): Domingo E, Holland JJ RNA virus mutations and fitness for survival. Annu Rev Microbiol 51: Dufkova L, Scigalkova I, Moutelikova R, Malenovska H. Prodelalova J Genetic diverity of porcine sapoviruses, kobuviruses, and astroviruses in asymptomatic pigs: an emerging new sapovirus GIII genotype. Arch Virol. 158(3): Farkas T, Fey B, Keller G, Martella V,. Egyed L Molecular detection of novel astrovirus in wild and laboratory mice. Virus Genes. 45(3): Fross S, Schaller H A tandem repeat gene in a picornavirus. Nucleic Acids Res. 10(20): Ge FF, Yang DQ, Ju HB, Wang J, Liu J, Liu PH, Zhou JP Epidemiological 82
83 survey of porcine epidemic diarrhea virus in swine farms in Sanghai, China. Arch Virol. 158(11): Gorbalenya AE, Koonin EV, Lai MM Putative papain-related thiol proteases of positive-strand RNA viruses. Identification of rubi- and aphthovirus proteases and delineation of a novel conserved domain associated with proteases of rubi-, alpha- and coronaviruses. FEBS Lett. 288(1-2): Goyer M, Aho LS, Bour JB, Ambert-Balay K, Pothier P Seroprevalence distribution of Aichi virus among a French population in Arch Virol. 153(6): Hales LM, Knowles NJ, Reddy PS, Xu L, Hay C, Hallenbeck PL Complete genome sequence analysis of Seneca Valley virus-001, a novel oncolytic picornavirus. J Gen Virol. 89(Pt 5): Hellen CU, de Breyne S A distinct group of hepacivirus/pestivirus-like internal ribosomal entry sites in members of diverse picornavirus genera: evidence for modular exchange of functional noncoding RNA elements by recombination. J Virol. 81(11): Honkavuori KS, Shivaprasad HL, Briese T, Street C, Hirschberg DL, Hutchison SK, Lipkin WI Novel picornavirus in Turkey poults with hepatitis, California, USA. Emerg Infect Dis. 17(3): Hughes PJ, Stanway G The 2A proteins of three diverse picornaviruses are related to each other and to the H-rev107 family of proteins involved in the control of cell proliferation. J Gen Virol. 81(Pt 1): ICTV Online. Official taxonomy: updates since the 8th report. default.aspx 32. ICTV Online ICTV Discussion forum of the International Committee on Taxonomy of Viruses ICTV Online ICTV Discussion forum of the International Committee on Taxonomy of Viruses ICTV Online ICTV Discussion forum of the International Committee on Taxonomy of Viruses ICTV Online ICTV Discussion forum of the International Committee on Taxonomy of Viruses Jacques J, Moret H, Minette D, Léveque N, Jovenin N, Deslée G, Lebargy F, Motte 83
84 J, Andréoletti L Epidemiological, molecular, and clinical features of enterovirus respiratory infections in French children between 1999 and J Clin Microbiol. 46(1): Jiang X, Huang PW, Zhong WM, Farkas T, Cubitt DW, Matson DO Design and evaluation of a primer pair that detects both Norwalk- and Sapporo-like caliciviruses by RT-PCR. J Virol Methods. 83(1-2): Jokela P, Joki-Korpela P, Maaronen M, Glumoff V, Hyypiä T Detection of human picornaviruses by multiplex reverse transcription-pcr and liquid hybridization. J Clin Microbiol. 43(3): Jorba J, Campagnoli R, De L, Kew O Calibration of multiple poliovirus molecular clocks covering an extended evolutionary range. J Virol. 82(9): Kaikkonen S, Räsänen S, Rämet M, Vesikari T Aichi virus infection in children with acute gastroenteritis in Finland. Epidemiol Infect. 138(8): Kaku Y, Chard LS, Inoue T, Belsham GJ Unique characteristics of a picornavirus internal ribosome entry site from the porcine teschovirus-1 talfan. J Virol. 76(22): Kapoor A, Victoria J, Simmonds P, Wang C, Shafer RW, Nims R, Nielsen O, Delwart E A highly divergent picornavirus in a marine mammal. J Virol. 82(1): Kapoor A, Simmonds P, Dubovi EJ, Qaisar N, Henriquez JA, Medina J, Shields S, Lipkin WI Characterization of a canine homolog of human Aichivirus. J Virol. 85(21): Khamrin P, Maneekarn N, Peerakome S, Okitsu S, Mizuguchi M, Ushijima H Bovine kobuviruses from cattle with diarrhea. Emerg Infect Dis. 14(6): Khamrin P, Maneekarn N, Kongkaew A, Kongkaew S, Okitsu S, Ushijima H Porcine kobuvirus in piglets, Thailand. Emerg Infect Dis. 15(12): Khamrin P, Maneekarn N, Hidaka S, Kishikawa S, Ushijima K, Okitsu S, Ushijima H Molecular detection of kobuvirus sequences in stool samples collected from healthy pigs in Japan. Infect Genet Evol. 10(7): Kim MC, Kwon YK, Joh SJ, Lindberg AM, Kwon JH, Kim JH, Kim SJ Molecular analysis of duck hepatitis virus type 1 reveals a novel lineage close to the genus Parechovirus in the family Picornaviridae. J Gen Virol. 87(Pt 11): King AMQ, Brown F, Christian P Picornaviridae. In: Virus taxonomy: seventh report of the International Committee on Taxonomy of Viruses, van Regenmortel M. H. 84
85 V., Faquet C. M., Bishop D. H. L., Carsten E. B., Estes M. K., Lemon S. M., Maniloff J., Mayo M. A., McGeoch D. J., Pringle C. R., Wickner R. B. (eds). Academic Press, Inc: New York, Kofstad T, Jonassen CM Screening of feral and wood pigeons for viruses harbouring a conserved mobile viral element: characterization of novel Astroviruses and Picornaviruses. PLoS One. 6(10):e Koonin EV, Wolf YI, Nagasaki K, Dolja VV The big bang of picorna-like virus evolution antedates the radiation of eukaryotic supergroups. Nat Rev Microbiol. 6(12): Knowles NJ, Buckley LS, Pereira HG Classification of porcine enteroviruses by antigenic analysis and cytopathic effects in tissue culture: description of three new serotypes. Arch Virol. 62(3): Knowles NJ, Hovi T, Hyypiä T, King AMQ, Lindberg AM, Minor PD, Pallansch MA, Palmenberg AC, Skern T, Stanway G, Yamashita T, Zell R Taxonomy of Picornaviridae: Current Situation and Future Proposals. EUROPIC Knowles NJ, Hovi T, Hyypiä T, King AMQ, Lindberg AM, Minor PD, Pallansch MA, Palmenberg AC, Skern T, Stanway G, Yamashita T, Zell R Taxonomy of Picornaviridae: Current situation and future proposals. EUROPIC Knowles NJ, Hovi T, Hyypiä T, King AMQ, Lindberg AM, Pallansch MA, Palmenberg AC, Skern T, Stanway G, Yamashita T, Zell R Taxonomy of the Picornaviridae: Expanding the family beyond 12 genera. EUROPIC Knowles NJ, Delwart E, Gorbalenya AE, Hovi T, Hyypiä T, King AMQ, Lindberg AM, Pallansch MA, Palmenberg AC, Reuter G, Simmonds P, Skern T, Stanway G, Yamashita T, Zell R Picornaviridae: the 9 th ICTV Report and Beyond. EUROPIC Knowles NJ, Hovi T, Hyypiä T, King AQM, Lindberg AM, Pallansch MA, Palmenberg AC, Simmonds P, Skern T, Stanway G, Yamashita T, Zell R Picornaviridae. In: Virus Taxonomy: Classification and Nomenclature of Viruses: Ninth Report of the International Committee on Taxonomy of Viruses. Ed: King AMQ, Adams MJ, Carstens EB, Lefkowitz EJ, San Diego: Elsevier, pp Krumbholz A, Dauber M, Henke A, Birch-Hirschfeld E, Knowles NJ, Stelzner A, 85
86 Zell R Sequencing of porcine enterovirus groups II and III reveals unique features of both virus groups. J Virol. 76(11): van Kuppeveld FJ, Hoenderop JG, Smeets RL, Willems PH, Dijkman HB, Galama JM, Melchers WJ Coxsackievirus protein 2B modifies endoplasmatic reticulum membrane and plasma membrane permeability and facilitates virus release. EMBO J. 16(12): Lama J, Paul AV, Harris KS, Wimmer E Properties of purified recombinant poliovirus protein 3AB as substrate for viral proteinases and as co-factor for RNA polymerase 3Dpol. J Biol Chem. 269(1): Lama J, Carrasco L Expression of poliovirus nonstructural protein sin Escherichia coli cells. Modification of membrane permeability induced by 2B and 3A. J Biol Chem. 267(22): Le Guyader FS, Le Saux JC, Ambert-Balay K, Krol J, Serais O, Parnaudeau S, Giraudon H, Delmas G, Pommepuy M, Pothier P, Atmar RL Aichi virus, norovirus, astrovirus, enterovirus, and rotavirus involved in clinical cases from a French oyster-related gastroenteritis outbreak. J Clin Microbiol. 46(12): Leong LEC, Cornell CT, Semler BL Processing determinants and functions of cleavage products of picornavirus polyproteins. p In: Semler BL, Wimmer E. (ed) Molecular biology of picornaviruses. ASM Press, Washington, D.C. 63. Li L, Pesavento PA, Shan T, Leutenegger CM, Wang C, Delwart E Viruses in diarrhoeic dogs include novel kobuviruses and sapoviruses. J Gen Virol. 92(Pt 11): Lin Y, Xiao S, Zeng Y, Song T, Zeng S, Chen H, Fang L Complete genome sequence of porcine kobuvirus strain WUH1. J Virol. 86(12): Madan V, Sánchez-Martínez S, Vedovato N, Rispoli G, Carrasco L, Nieva JL Plasma membrane-porating domain in poliovirus 2B protein. A short peptide mimics viroporin activity. J Mol Biol. 374(4): Marvil P, Knowles NJ, Mockett AP, Britton P, Brown TD, Cavanagh D Avian encephalomyelitis virus is a picornavirus and is most closely related to hepatitis A virus. J Gen Virol. 80 (Pt 3): Mauroy A, Scipioni A, Mathijs E, Thys C, Thiry E Molecular detection of kobuviruses and recombinant noroviruses in cattle in continental Europe. Arch Virol. 154(11): Minor P Picornaviruses. In: Topley&Wilson s Microbiology & Microbial 86
87 Infections, 10 th Edition, Virology vol.2, ed. Mahy, B. W. J., V. ter Meulen. ASM Press, Hodder Arnold 69. Mirzayan C, Wimmer E Biochemical studies on poliovirus polypeptide 2C: evidence for ATPase activity. Virology. 199(1): Molla A, Harris KS, Paul AV, Shin SH, Mugavero J, Wimmer E Stimulation of poliovirus proteinase 3Cpro-related proteolysis by the genome-linked protein VPg and its precursor 3AB. J Biol Chem. 269(43): Ng TF, Marine R, Wang C, Simmonds P, Kapusinszky B, Bodhidatta L, Oderinde BS, Wommack KE, Delwart E High variety of known and new RNA and DNA viruses of diverse origins in untreated sewage. J Virol. 86(22): Nicholas KB, Nicholas HBJ GeneDoc: a tool for editing and annotating multiple sequence alignment. Distributed by the author Oberste MS, Maher K, Kilpatrik DR, Pallansch MA Molecular evolution of the human enteroviruses: correlation of serotype with VP1 sequence and application to picornavirus classification. J Virol. 73(3): Oberste MS, Maher K, Pallansch MA Molecular phylogeny and proposed classification of the simian picornaviruses. J Virol. 76(3): Oberste MS, Maher K, Pallansch MA Genomic evidence that simian virus 2 and other six simian picornaviruses represent a new genus in Picornaviridae. Virology. 314(1): Okitsu S, Khamrin P, Thongprachum A, Hidaka S, Kongkaew S, Kongkaew A, Maneekarn N, Mizuguchi M, Hayakawa S, Ushijima H Sequence analysis of porcine kobuvirus VP1 region detected in pigs in Japan and Thailand. Virus Genes. 44(2): Oh DY, Silva PA, Hauroeder B, Diedrich S, Cardoso DD, Schreier E Molecular characterization of the first Aichi viruses isolated in Europe and in South America. Arch Virol. 151(6): Pallansch M, Roos R. Enteroviruses: polioviruses, coxsackieviruses, echoviruses, and newer enteroviruses. In Knipe DM, Howley PM, Griffin DE, Lamb RA, Martin MA, Roizman B, Straus SE (ed.), Fields Virology, 5th ed. Lippincott Williams & Wilkins, Philadelphia, PA Palmquist JM, Munir S, Taku A, Kapur A, Goyal SM Detection of porcine teschovirus and enterovirus type II by reverse transcription-polymerase chain reaction. J Vet Diagn Invest. 14(6):
88 80. Pankovics P, Szabó H, Székely G, Gyurkovits K, Reuter G [Detection and molecular epidemiology of respiratory syncytial virus type A and B strains in childhood respiratory infections in Hungary]. Orv Hetil. 150(3): Pankovics P, Boros A, and G. Reuter Novel picornavirus in domesticated common quail (Coturnix coturnix) in Hungary. Arch Virol. 157(2): Pankovics P, Boros A, Szabó H, Székely G, Gyurkovits K, Reuter G Human enterovirus 109 (EV109) in acute paediatric respiratory disease in Hungary. Acta Microbiol Immunol Hung. 59(2): Park SJ, Kim HK, Moon HJ, Song DS, Rho SM, Han JY, Nguyen VG, Park BK Molecular detection of porcine kobuviruses in pigs in Korea and their association with diarrhea. Arch Virol. 155(11): Pham NT, Khamrin P, Nguyen TA, Kanti DS, Phan TG, Okitsu S, Ushijima H Isolation and molecular characterization of Aichi viruses from fecal specimens collected in Japan, Bangladesh, Thailand, and Vietnam. J Clin Microbiol. 45(7): Phan TG, Kapusinszky B, Wang C, Rose RK, Lipton HL, Delwart E The fecal viral flora of wild rodents. PLoS Pathog. 7(9):e Pintó RM, Costafreda MI, Bosch A Risk assessment in shellfish-borne outbreaks of hepatitis A. Appl Environ Microbiol. 75(23): Racaniello VR Picornaviridae: the viruses and their replication. In: Knipe DM, Howley PM. (eds) Fields virology, 5th edn. Lippincott Williams & Wilkins, Philadelphia, pp Reuter G, Szabó H, Székely G, Gyurkovits K, Szücs G [First detection of human metapneumovirus in a child with respiratory syndrome in Hungary]. Orv Hetil. 147(48): Reuter G, Juhász A, Kosztolányi L, Lefler E, Fekete Z Co-circulation of genotype IA and new variant IB hepatitis A virus in outbreaks of acute hepatitis in Hungary 2003/2004. J Med Virol. 78(11): Reuter G, Pankovics P, Stefler D, Löveyné Dr M, Varga E, Kiss G, Szucs M, Fekete Z, Szucs G [Hepatitis A outbreak in Transdanubia (Hungary): molecular connections and epidemiological conclusions Orv Hetil. 148(22): Reuter G, Bíró H, Szűcs G Enteric caliciviruses in domestic pigs in Hungary. Arch Virol. 152(3): Reuter G, Boldizsár A, Kiss I, Pankovics P Candidate new species of 88
89 Kobuvirus in porcine hosts. Emerg Infect Dis. 14(12): Reuter G, Boldizsár A, Pankovics P Complete nucleotide and amino acid sequences and genetic organization of porcine kobuvirus, a member of a new species in the genus Kobuvirus, family Picornaviridae. Arch Virol. 154(1): Reuter G, Boldizsár A, Papp G, Pankovics P Detection of Aichi virus shedding in a child with enteric and extraintestinal symptoms in Hungary. Arch Virol. 154(9): Reuter G, Egyed L Bovine kobuvirus in Europe. Emerg Infect Dis. 15(5): Reuter G, Kecskémeti S, Pankovics P Evolution of porcine kobuvirus infection, Hungary. Emerg Infect Dis. 16(4): Reuter G, Boros A, Pankovics P, Egyed L Kobuvirus in domestic sheep, Hungary. Emerg Infect Dis. 16(5): Reuter G, Zimsek-Mijovski J, Poljsak-Prijatelj M, Di Bartolo I, Ruggeri FM, Kantala T, Maunula L, Kiss I, Kecskemeti S, Halaihel N, Buesa J, Johnsen C, Hjulsager CK, Larsen LE, Koopmans M, Böttiger B Incidence, diversity, and molecular epidemiology of sapoviruses in swine across Europe. J Clin Microbiol. 48(2): Reuter G, Boros A, Pankovics P Kobuviruses - a comprehensive review. Rev Med Virol. 21(1): Reuter G, Új M, Pankovics P, Kolozsi T, Mihály I, Liptai Z, Boros Á [Clinical significance and the first identification of human parechoviruses in Hungary]. Orv Hetil. 152(25): Reuter G, Nemes C, Boros A, Kapusinszky B, Delwart E, Pankovics P Porcine kobuvirus in wild boars (Sus scrofa). Arch Virol. 158(1): Ribes JM, Montava R, Téllez-Castillo CJ, Fernández-Jiménez M, Buesa J Seroprevalence of Aichi virus in a Spanish population from 2007 to Clin Vaccine Immunol. 17(4): Rodríguez PL, Carrasco L Poliovirus protein 2C contains two regions involved in RNA binding activity. J Biol Chem. 270(17): Romero JR. Enteroviruses. In Richman DD, Whitley RJ, Hayden FG. (ed.) Clinical Virology, 3rd ed. ASM Press, Washington, DC Sambrook J, Fritsch EF, Maniatis T Molecular clonong laboratory manual, Second edition. Cold Spring Harbor Laboratory Press. 89
90 106. Santti J, Hyypiä T, Kinnunen L, Salminen M Evidence of recombination among enteroviruses. J Virol. 73(10): Sasaki J, Kusuhara Y, Maeno Y, Kobayashi N, Yamashita T, Sakae K, Takeda N, Taniguchi K Construction of an infectious cdna clone of Aichi virus (a new member of the family Picornaviridae) and mutational analysis of a stem-loop structure at the 5' end of the genome. J Virol. 75(17): Sasaki J, Nagashima S, Taniguchi K Aichi virus leader protein is involved in viral RNA replication and encapsidation. J Virol. 77(20): Sasaki J, Taniguchi K The 5 -end sequence of the genome of Aichi virus, a picornavirus, contains an element critical for viral RNA encapsidation. J Virol. 77(6): Sasaki J, Taniguchi K Aichi virus 2A protein is involved in viral RNA replication. J Virol. 82(19): Sánchez G, Bosch A, Pintó RM Hepatitis A virus detection in food: current and future prospects. Lett Appl Microbiol. 45(1): Sdiri-Loulizi K, Gharbi-Khélifi H, de Rougemont A, Chouchane S, Sakly N, Ambert-Balay K, Hassine M, Guédiche MN, Aouni M, Pothier P Acute infantile gastroenteritis associated with human enteric viruses in Tunisia. J Clin Microbiol. 46(4): Sisay Z, Wang Q, Oka T, Saif L Prevalence and molecular characterization of porcine enteric caliciviruses and first detection of porcine kobuviruses in US swine. Arch Virol. 158(7): Stanway G, Hovi T, Knowles N, Hyypiä T Molecular and biological basis of picornavirus taxonomy. In: Semler BL, Wimmer E. (eds) Molecular biology of picornaviruses. ASM, Washington DC, pp Stanway G, Brown F, Christian P, Hovi T, Hyypiä T, King AMQ, Knowles N, Lemon SM, Minor PD, Pallansch MA, Palmenberg AC, Skern T Family Picornaviridae. In: Fauquet CM, Mayo MA, Maniloff J, Desselberger U, Ball LA. (eds) Virus taxonomy. Eight report of the International Committee on Taxonomy of Viruses. Elsevier. Academic Press. London, pp Svraka S, Duizer E, Vennema H, de Bruin E, van der Veer B, Dorresteijn B, Koopmans M Etiological role of viruses in outbreaks of acute gastroenteritis in The Netherlands from 1994 through J Clin Microbiol. 45(5): Sweeney TR, Dhote V, Yu Y, Hellen CU A distinct class of internal ribosomal 90
91 entry site in members of the Kobuvirus and proposed Salivirus and Paraturdivirus genera of the Picornaviridae. J Virol. 86(3): Tamura K, Dudley J, Nei M, Kumar S MEGA4: Molecular Evolutionary Genetics Analysis (MEGA) software version 4.0. Mol Biol Evol. 24(8): Tamura K, Peterson D, Peterson N, Stecher G, Nei M, Kumar S MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods. Mol Biol Evol. 28(10): Tapparel C, Junier T, Gerlach D, Van-Belle S, Turin L, Cordey S, Mühlemann K, Regamey N, Aubert JD, Soccal PM, Eigenmann P, Zdobnov E, Kaiser L New respiratory enterovirus and recombinant rhinoviruses among circulating picornaviruses. Emerg Infect Dis. 15(5): Teterina NL, Levenson E, Rinaudo MS, Egger D, Bienz K, Gorbalenya AE, Ehrenfeld E Evidence for fuctional protein interactions required for poliovirus RNA replication. J Virol. 80(11): Thiry E Clinical virology of ruminants. Paris: Wolters-Kluwer Thomson JD, Gibson TJ, Plewniak F, Jeanmougin F, Higgins DG The ClustalX windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools. Nucleic Acid Res. 25(24): Towner JS, Ho TV, Semler BL Determinants of membrane association for poliovirus protein 3AB. J Biol Chem. 271(43): Tseng CH, Tsai HJ Sequence analysis of a duck picornavirus isolate indicates that it together with porcine enterovirus type 8 and simian picornavirus type 2 should be assigned to a new picornavirus genus. Virus Res. 129(2): Verma H, Mor SK, Abdel-Glil MY, Goyal SM Identification and molecular characterization of porcine kobuvirus in U.S. swine. Virus Genes. 46(3): Wang C, Lan D, Hua X Porcine kobuvirus from pig stool specimens in Shanghai, China. Virus Genes. 43(3): Wang C, Lan D, Cui L, Yang Z, Yuan C, Hua X Molecular characterization of a porcine kobuvirus strain in China. Arch Virol. 157(3): Wang K, Xie S, Sun B Viral protein sas ion channels. Biochem Biophys Acta. 1808(2): Woo PC, Lau SK, Huang Y, Lam CS, Poon RW, Tsoi HW, Lee P, Tse H, Chan AS, Luk G, Chan KH, Yuen KY Comparative analysis of six genome sequences of three novel picornaviruses, turdiviruses 1, 2 and 3, in dead wild birds, and proposal of 91
92 two novel genera, Orthoturdivirus and Paraturdivirus, in the family Picornaviridae. J Gen Virol. 91(Pt 10): Woo PC, Lau SK, Choi GK, Huang Y, Teng JL, Tsoi HW, Tse H, Yeung ML, Chan KH, Jin DY, Yuen KY Natural occurence and characterization of two internal ribosome entry site elements in a novel virus, caninepicodicistrovirus, in the picornavirus-like superfamily. J Virol. 86(5): Yamashita T, Kobayashi S, Sakae K, Nakata S, Chiba S, Ishihara Y, Isomura S Isolation of cytopathic small round viruses with BS-C-1 cells from patients with gastroenteritis. J Infect Dis. 164(5): Yamashita T, Sakae K, Ishihara Y, Isomura S, Utagawa E Prevalence of newly isolated, cytopathic small round virus (Aichi strain) in Japan. J Clin Microbiol. 31(11): Yamashita T, Sakae K, Kobayashi S, Ishihara Y, Miyake T, Mubina A, Isomura S Isolation of cytopathic small round virus (Aichi virus) from Pakistani children and Japanese travelers from Southeast Asia. Microbiol Immunol. 39(6): Yamashita T, Sakae K, Tsuzuki H, Suzuki Y, Ishikawa N, Takeda N, Miyamura T, Yamazaki S Complete nucleotide sequence and genetic organization of Aichi virus, a distinct member of the Picornaviridae associated with acute gastroenteritis in humans. J Virol. 72(10): Yamashita T, Sugiyama M, Tsuzuki H, Sakae K, Suzuki Y, Miyazaki Y Application of a reverse transcription-pcr for identification and differentiation of Aichi virus, a new member of the Picornavirus family associated with gastroenteritis in humans. J Clin Microbiol. 38(8): Yamashita T, Ito M, Tsuzuki H, Sakae K Identification of Aichi virus infection by measurement of immunoglobulin responses in an enzyme-linked immunosorbent assay. J Clin Microbiol. 39(11): Yamashita T, Ito M, Kabashima Y, Tsuzuki H, Fujiura A, Sakae K Isolation and characterization of a new species of kobuvirus associated with cattle. J Gen Virol. 84(Pt 11): Yang S, Zhang W, Shen Q, Yang Z, ZhuJ, Cui L, Hua X Aichi virus strains in children with gastroenteritis, China. Emerg Infect Dis. 15(10): Yozwiak NL, Skewes-Cox P, Gordon A, Saborio S, Kuan G, Balmaseda A, Ganem D, Harris E, DeRisi JL Human enterovirus 109: a novel interspecies recombinant enterovirus isolated from a case of acute pediatric respiratory illness in 92
93 Nicaragua. J Virol. 84(18): Yu JM, Jin M, Zhang Q, Li HY, Li DD, Xu ZQ, Li JS, Cui SX, Yang SH, Liu N, Duan ZJ Candidate porcine Kobuvirus, China. Emerg Infect Dis. 15(5): Yu JM, Xu ZQ, Li BW, Zhang Q, Cui SX, Jin M, Duan ZJ Analysis and characterization of the complete genome of a member of a new species of kobuvirus associated with swine. Arch Virol. 156(5): Yu Y, Sweeney TR, Kafasla P, Jackson RJ, Pestova TV, Hellen CU The mechanism of translation initiation on Aichivirus RNA mediated by a novel type of picornavirus IRES. EMBO J. 30(21): Zell R, Seitz S, Henke A, Munder T, Wutzler P Linkage map of proteinprotein interactions of Porcine teschovirus. J Gen Virol. 86(Pt 10): Zhang Q, Hu R, Tang X, Wu C, He Q, Zhao Z, Chen H, Wu B Occurrence and investigation of enteric viral infections in pigs with diarrhea in China. Arch Virol. 158(8): Zuker M Mfold web server for nucleic acid folding and hybridization prediction. Nucleic Acids Res. 31(13):
94 PUBLIKÁCIÓS JEGYZÉK AZ ÉRTEKEZÉS ALAPJÁUL SZOLGÁLÓ KÖZLEMÉNYEK Reuter G., Boldizsár Á., Kiss I., Pankovics P.: Candidate new species of kobuvirus in porcine hosts. Emerg. Infect. Dis , (IF: 6,449) Reuter G., Boldizsár Á., Pankovics P.: Complete nucleotide and amino acid sequences and genetic organization of porcine kobuvirus, a member of a new species in the genus, Kobuvirus family Picornaviridae. Arch. Virol , (IF: 1,909) Reuter G., Boldizsár Á., Kiss I., Kecskeméti S., Pankovics P.: Sertés kobuvírus: egy új vírusfaj leírása a Picornaviridae család Kobuvírus nemzetségében. Magyar Állatorvosok Lapja, , (IF: 0,2) Reuter G., Boldizsár Á., Papp G., Pankovics P.: Detection of Aichi virus shedding in a child with enteric and extraintestinal symptoms in Hungary. Arch. Virol , (IF: 1,909) Reuter G., Kecskeméti S., Pankovics P.: Evolution of porcine kobuvirus infection, Hungary. Emerg. Infect. Dis , (IF: 6,859) Reuter G., Boros Á., Pankovics P., Egyed L.: Kobuvirus in domestic sheep, Hungary. Emerg. Infect. Dis , (IF: 6,859) Reuter G., Boros A., Pankovics P.: Kobuviruses a comprehensive review. Rev Med Virol , (IF: 7,2) Pankovics P., Boros A., Reuter G.: Novel picornavirus in domesticated common quail (Coturnix coturnix). Arch. Virol , (IF: 2,03) Pankovics P., Boros A., Szabó H., Székely G., Gyurkovits K., Reuter G.: Human enterovirus 109 (EV109) in acute paediatric respiratory disease in Hungary. Acta Microbiol. Immunol. Hung , (IF: 0,646) AZ ÉRTEKEZÉS ALAPJÁUL SZOLGÁLÓ ABSZTRAKTOK, ELŐADÁSOK ÉS POSZTEREK Reuter G., Boldizsár Á., Pankovics P.: Detection and characterization of kobuviruses (in family Picornaviridae) in new host species. Journal of Clinical Virology, 2009;46(Suppl. 1), S57. (PXII-14) (absztrakt) Pankovics P., Boros Á., Reuter G.: Novel picornavirus in domesticated common quail (Coturnix coturnix) in Hungary. Magyar Mikrobiológiai Társaság évi Nagygyűlése, október Keszthely, Magyarország (előadás) Reuter G., Boldizsár Á., Pankovics P.: Detection and characterization of kobuviruses (in family Picornaviridae) in new host species. European Society for Clinical Virology (ESCV), Istanbul, Törökország, szeptember (poszter) Reuter G., Boldizsár Á., Boros Á., Pankovics P.: Kobuviruses and a potential new picornavirus genus in different host species. 4 th European Congress of Virology, Como, Olaszország, április (poszter) 94
95 Reuter G., Boldizsár Á., Boros Á., Pankovics P.: Detection and characterization of kobuvirus (in familiy Picornaviridae) in human and in new host species in Hungary. 20 th European Congress of Clinical Microbiology, Bécs, Ausztria, április (poszter) EGYÉB KÖZLEMÉNYEK Reuter G., Pankovics P., Stefler D., Löveyné M.Á., Varga E., Kiss G., Szűcs M., Fekete Zs., Szűcs Gy. Hepatitis A járvány a Dél-Dunántúlon: molekuláris összefüggések, epidemiológiai következtetések [Hepatitis A outbreak in Transdanubia (Hungary): molecular connections and epidemiological conclusions 2006] Orv. Hetil. 2007, 148, 22: (IF: 0) Reuter G., Pankovics P., Szűcs Gy.: Genetic drifts of norovirus genotype GII-4 in 7 consecutive epidemic seasons in Hungary. J. Clinic. Virol , (IF: 3,32) Reuter G. Pankovics P., Szűcs Gy.: A GII4 genotípusú norovírus genetikai evolúciója (drift) és pandémiás potenciálja Hét hazai járványszezont felölelő tanulmány a calaicivírus járványok epidemiológiájának molekuláris szintű megértéséhez. Infektológia és Klinikai Mikrobiológia, , (IF: 0) Reuter G., Pankovics P., Egyed L.: Szarvasmarha-norovírusok (calicivírus) molekuláris kimutatása hazánkban. Magyar Állatorvosok Lapja, , (IF: 0,088) Pankovics P., Szabó H., Székely Gy., Gyurkovits K., Reuter G.: A légúti óriássejtes vírus A és B típusának molekuláris kimutatása és epidemiológiája gyermekkori légúti fertőzésekben. Orv. Hetil , (IF: 0) Pankovics P., Szabó H., Székely Gy., Gyurkovits K., Reuter G.: Detection and molecular epidemiology of respiratory syncytial virus type A and B strains in childhood respiratory infections in Hungary. Clinic. Exper. Medic. J , (IF: 0) Pankovics P., Kugler Z., Kátai A., Reuter G.: Sapovírus (GI2) okozta gastroenteritis járvány első hazai igazolása nemzetközi epidémia részjelensége? [First gastroenteritis outbreak caused by sapovirus (GI2) in Hungary part o fan international epidemic?] Orv. Hetil , (IF: 0) Reuter G., Pankovics P., Egyed L.: Detection of genotype 1 and 2 bovine noroviruses in Hungary. Vet. Rec , (IF: 1,504) Boros Á., Új M., Pankovics P., Reuter G.: Detection and characterization of human parechoviruses in archived cell cultures in Hungary. J. Clinic. Virol , (IF: 4,023) Reuter G., Pankovics P., Boros A.: Identification of a novel astrovirus in a domestic pig in Hungary. Arch Virol , (IF: 2,111) Pankovics P., Boros Á., Rovács M., Nagy E., Krisztián E., Vollain M., Reuter G.: Humán astrovirus okozta gastroenteritis járvány első hazai igazolása. [First detection of human astrovirus in gastroenteritis outbreak in Hungary]. Orv. Hetil , (IF: 0) 95
96 Boros Á., Pankovics P., Reuter G.: Characterization of a novel porcine enterovirus in domestic pig in Hungary. Infect Genet Evol , (IF: 3,128) Burian Z., Szabó H., Székely G., Gyurkovits K., Pankovics P., Farkas T., Reuter G.: Setection and follow-up of torque-teno midi virus ( small anelloviruses ) in nasopharyngeal aspirates and three other body fluid sin children. Arch Virol , (IF: 2,111) Reuter G., Új M., Pankovics P., Kolozsi T., Mihály I., Liptai Z., Boros Á.: [Clinical significance and the first identification of human parechoviruses in Hungary]. Orv. Hetil (IF: 0) Reuter G., Pankovics P., Delwart E., Boros A.: Identification of a novel astrovirus in domestic sheep in Hungary. Arch Virol , (IF: 2,03) Boros A., Pankovics P., Simmonds P., Reuter G.: Novel positive-sense, Singlestranded RNA (+ssrna) Virus with Di-cistronic Genome from Intestinal Content of Freshwater Carp (Cyprinus carpio). PLoS ONE ;6(12):e (IF: 4,092) Boros A., Nemes C., Pankovics P., Bíró H., Kapusinszky B., Delwart E., Reuter G.: Characterization of novel porcine enterovirus in wild boars in Hungary. Arch. Virol , (IF: 2,03) Reuter G., Nemes C., Boros A., Kapusinszky B., Delwart E., Pankovics P.: Astrovirus in wild boars (Sus scrofa) in Hungary. Arch. Virol , (IF: 2,03) Boros A., Nemes C., Pankovics P., Kapusinszky B., Delwart E., Reuter G.: Porcine teschovirus in wild boars in Hungary. Arch. Virol , (IF: 2,03) Boros A., Pankovics P., Knowles NJ., Reuter G.: Natural interspecies recombinant bovine/porcine enterovirus in sheep. J. Gen. Virol , (IF: 3,127) Boros A., Nemes C., Pankovics P., Kapusinszky B., Delwart E., Reuter G.: Identification and complete genome characterization of a novel picornavirus in turkey (Meleagris gallopavo). J. Gen. Virol , (IF: 3,127) Reuter G., Nemes C., Boros A., Kapusinszky B., Delwart E., Pankovics P.: Porcine kobuvirus in wild boars (Sus scrofa). Arch. Virol , (IF: 2,03) Reuter G., Pankovics P., Knowles NJ., Boros A.: Two closely related novel picornaviruses in cattle and sheep in Hungary from 2008 to 2009, proposed as members of a new genus in the family Picornaviridae. J. Virol , (IF: 5,076) Pankovics P., Boros Á., Nemes Cs., Delwart E., Reuter G.: Nebovírus (Caliciviridae) első hazai kimutatása hasmenéses borjú bélsármintájából. Magyar Állatorvosok Lapja , (IF: 0,146) Horváth K.B., Pankovics P., Battyáni Z., Kálmán E., Reuter G.: A probable etiological role of Merkel cell polyomavirus int he development of Merkel cell carcinoma. Orv. Hetil , (IF: 0) 96
97 EGYÉB ABSZTRAKTOK, ELŐADÁSOK ÉS POSZTEREK Reuter G., Krisztalovics K., Pankovics P., Szűcs Gy.: Ten-years Survelliance of Norovirus in Outbreaks of Gastroenteritis in Hungary: Molecular Epidemiology, Genetic Diversity and Evolution. 18 th European Congress of Clinical Microbiology and Infectious Diseases (ECCMID), 19-22/April/2008, Barcelona, Spain. (poszter) Pankovics P., Szabó H., Székely Gy., Gyurkovits K., Reuter G.: Molecular detection and epidemiology of type A and B respiratory syncytial viruses in childhood respiratory infections. Magyar Mikrobiológiai Társaság évi Nagygyűlése és a XI. Fermentációs Kollokvium, Absztraktfüzet 62pp. Hotel Helikon, Keszthely, , Magyarország (absztrakt) Pankovics P., Szabó H., Székely Gy., Gyurkovits K., Reuter G.: Molecular detection and epidemiology of type A and B respiratory syncytial viruses in childhood respiratory infections. Journal of Clinical Virology, 2009;46(Suppl. 1), S22. (PIV-17) (absztrakt) Pankovics P., Szabó H., Székely Gy., Gyurkovits K., Reuter G.: Molecular detection and epidemiology of type A and B respiratory syncytial viruses in childhood respiratory infections. Acta Microbiologica et Immunologica Hungarica, , (absztrakt) Pankovics P., Szabó H., Székely Gy., Gyurkovits K., Reuter G.: Molecular detection and epidemiology of type A and B respiratory syncytial viruses in childhood respiratory infections. European Society for Clinical Virology (ESCV), Isztanbul, Törökország, szeptember (poszter) Reuter G., Bíró H., Egyed L., Kiss L., Pankovics P., Szűcs Gy.: Genetic drift and pandemic potential of genotype GII4 norovirus strains in humans is seven consecutive epidemic seasons and the first detection of caliciviruses in animals (swine and cattle) in Hungary. Acta Microbiologica et Immunologica Hungarica, , (absztrakt) Pankovics P., Reuter G.: Hepatitis A járvány molekuláris biológiai nyomon követése és elemzése a Dél-Dunántúlon (2006). XXVIII. Országos Tudományos Diákköri Konferencia, Biológia szekció, Molekuláris Biológia 2. tagozat, április 6. Debrecen, Magyarország (előadás) Pankovics P., Reuter G., Stefler D., Löveyné-Móricz Á., Varga E., Kiss G., Szűcs M., Fekete Zs., Szűcs Gy.: Hepatitis A outbreak in South-Transdanubia: Characterization of endemic and imported HAV strains in Hungary. DIVINE- NET/EVENT (Enteric Virus Emergence, New Tools) Nemzetközi Konferencia, szeptember Pécs, Magyarország (előadás) Pankovics P., Szabó H., Székely Gy., Gyurkovits K., Reuter G.: Molecular detection and epidemiology of type A and B respiratory syncitial viruses in childhood respiratory infections. Magyar Mikrobiológiai Társaság évi Nagygyűlése és XI. Fermentációs Kollokvium, október Keszthely, Magyarország (előadás) Pankovics P., Szabó H., Székely Gy., Gyurkovits K., Reuter G.: Molecular detection and epidemiology of type A and B respiratory syncytial viruses in childhood 97
98 respiratory infections. Magyar Gyermekorvosok Társasága 53. Nagygyűlése június Eger, Magyarország (előadás) Pankovics P., Kugler Z., Kátai A., Reuter G.: First gastroenteritis outbreak caused by sapovirus in Hungary, part of an international epidemic? Magyar Mikrobiológiai Társaság évi Nagygyűlése, október Keszthely, Magyarország (előadás) Pankovics P., Boros Á., Szabó H., Székely Gy., Gyurkovits K., Reuter G.: Humán enterovírus 109 (EV109) első európai kimutatása gyermekkori akut légúti fertőzésből. Magyar Infektológiai és Klinikai Mikrobiológiai Társaság. Fővárosi Szent László Kórház, június 7. Budapest, Magyarország (előadás) Kassa Cs., Kolozsi T., Benyó G., Kállay K., Stréhn A., Sinkó J., Pankovics P., Mihály I., Reuter G., Kriván G.: Adenovírus infekciók csökkent immunitású gyermekekben diagnosztikus és terápiás lehetőségeink. Magyar Infektológiai és Klinikai Mikrobiológiai Társaság 40. Kongresszusa, Budapest, szeptember (előadás) Összesített impakt faktor: 88,1 Az értekezés alapjául szolgáló közlemények impakt faktora: 33,861 Összes független idézetek száma:
99 FÜGGELÉK 99
100 LETTERS which excluded false-positive results because of amplicon contamination. The second case was identified among the 86 sheep investigated. Placenta and fetal lung and liver exhibited necrotizing placentitis and vasculitis (Figure, panel A), interstitial pneumonia (Figure, panel B), and mixed cellular periportal hepatitis. Fetal liver was negative by parachlamydial 16S rrna real-time PCR and immunohistochemical analysis, but the fetal lung was positive by parachlamydial 16S rrna real-time PCR (Ct 40.7) and immunohistochemical tests (Figure, panel C), but negative with the tlc PCR. Fetal lung and liver were positive by realtime PCR for Chlamydiaceae (mean Ct for both organs 36.7), but negative by immunohistochemical tests. The placenta was positive for Chlamydiaceae by immunohistochemical tests and real-time PCR (mean Ct 23.3), and C. abortus was identified by ArrayTube Microarray. Brown (Chlamydiaceae) and red (Parachlamydia spp.) granular reaction was demonstrated within the necrotic lesions of the placenta by double immunohistochemical labeling (Figure, panel D). We report Parachlamydia infection in small ruminant abortion. C. abortus and Parachlamydia spp. were simultaneously present in an aborted sheep placenta. Parachlamydia spp. could be further detected in the lung of the aborted sheep fetus by realtime PCR and immunohistochemistry. Parachlamydia was also detected in a goat placenta. Thus, Parachlamydia spp. should be considered as a new abortigenic agent in sheep and goats. Persons in contact with small ruminants should be informed about the zoonotic potential of these abortigenic agents. Acknowledgments We are grateful to the laboratory staff (in particular, Carmen Kaiser) of the Institute of Veterinary Pathology, Vetsuisse Faculty, University of Zurich. This study was supported by the State Secretary for Education and Research, Bern, Switzerland (project number C ), as part of the COST Action 855 (Animal Chlamydiosis and Zoonotic Implications). G.G. is supported by the Leenards Foundation through a career award entitled Bourse Leenards pour la relève académique en médecine clinique à Lausanne. Silke Ruhl, Genevieve Goy, Nicola Casson, Rudolf Thoma, Andreas Pospischil, Gilbert Greub, and Nicole Borel Author affi liations: University of Zurich, Zurich, Switzerland (S. Ruhl, A. Pospischil, N. Borel); University of Lausanne, Lausanne, Switzerland (G. Goy, N. Casson, G. Greub); and Cantonal Laboratory of Veterinary Bacteriology, Chur, Switzerland (R. Thoma) DOI: /eid References 1. Longbottom D, Coulter LJ. Animal chlamydioses and zoonotic implications. J Comp Pathol. 2003;128: DOI: /jcpa Greub G, Raoult D. Parachlamydiaceae: potential emerging pathogens. Emerg Infect Dis. 2002;8: Borel N, Ruhl S, Casson N, Kaiser C, Pospischil A, Greub G. Parachlamydia spp. and related Chlamydia-like organisms and bovine abortion. Emerg Infect Dis. 2007;13: Chanton-Greutmann H, Thoma R, Corboz L, Borel N, Pospischil A. Abortion in small ruminants in Switzerland: investigations during two lambing seasons ( ) with special regard to chlamydial abortions [in German]. Schweiz Arch Tierheilkd. 2002;144: DOI: / Borel N, Thoma R, Spaeni P, Weilenmann R, Teankum K, Brugnera E, et al. Chlamydia-related abortions in cattle from Graubunden, Switzerland. Vet Pathol. 2006;43: DOI: /vp Everett KD, Bush RM, Andersen AA. Emended description of the order Chlamydiales, proposal of Parachlamydiaceae fam. nov. and Simkaniaceae fam. nov., each containing one monotypic genus, revised taxonomy of the family Chlamydiaceae, including a new genus and five new species, and standards for the identification of organisms. Int J Syst Bacteriol. 1999;49: Borel N, Kempf E, Hotzel H, Schubert E, Torgerson P, Slickers P, et al. Direct identification of chlamydiae from clinical samples using a DNA microarray a validation study. Mol Cell Probes. 2008;22: DOI: /j.mcp Casson N, Posfay-Barbe KM, Gervaix A, Greub G. New diagnostic real-time PCR for specific detection of Parachlamydia acanthamoebae DNA in clinical samples. J Clin Microbiol. 2008;46: DOI: /JCM Greub G, Berger P, Papazian L, Raoult D. Parachlamydiaceae as rare agents of pneumonia. Emerg Infect Dis. 2003;9: Casson N, Entenza JM, Greub G. Serological cross-reactivity between different Chlamydia-like organisms. J Clin Microbiol. 2007;45: DOI: / JCM Address for correspondence: Nicole Borel, Institute of Veterinary Pathology, Vetsuisse Faculty, University of Zurich, 8057 Zurich, Switzerland; n.borel@access.uzh.ch Candidate New Species of Kobuvirus in Porcine Hosts To the Editor: Picornaviruses (family Picornaviridae) are small, nonenveloped viruses with singlestranded, positive-sense genomic RNA, currently divided into 8 genera: Enterovirus, Aphthovirus, Cardiovirus, Hepatovirus, Parechovirus, Erbovirus, Teschovirus, and Kobuvirus (1). To date, the genus Kobuvirus consists of 2 species, Aichi virus and Bovine kobuvirus, each possessing 1 serotype. Aichi virus (strain A846/88) was first isolated from a stool sample obtained from a person with acute gastroenteritis in 1991 (2). Bovine kobuvirus (strain U-1) was detected in bovine 1968 Emerging Infectious Diseases Vol. 14, No. 12, December 2008
101 LETTERS sera and in feces samples from clinically healthy cattle in 2003 (3). Human and bovine kobuviruses were first isolated in Japan. Recently, kobuviruses have also been detected in humans in other countries in Asia (4), Europe (5,6), and South America (5) and in calves with diarrhea in Thailand (7). The Aichi virus and bovine kobuvirus genomes are approximately kb, respectively, and both have a typical picornavirus genome organization, including the L protein following structural (VP0, VP3, and VP1) and nonstructural (2A 2C and 3A 3D) regions (1,3). Genetic identity between Aichi and U-1 viruses ranges from 47.7% (3 untranslated region) through 70.8% (3D region) (3). In this study, we report a new candidate species of kobuvirus. Porcine kobuvirus was serendipitously detected in fecal samples from domestic pigs in Hungary. Fecal samples were collected in February 2007 from 15 healthy piglets (Sus scrofa domestica) <10 days of age from a farm in Ebes located in eastern Hungary. The aim of the study was to detect porcine calicivirus (norovirus and sapovirus) in domestic pigs by using reverse transcription PCR (RT- PCR), using the generic primer pairs p289/p290 designed for the calicivirus RNA-dependent RNA polymerase gene (319 nt for norovirus or 331 nt for sapovirus) (8). RNA isolation and RT-PCR were performed as described previously (9). PCR products were sequenced directly in both directions with the BigDye Terminator Cycle Sequencing Ready Reaction Kit (PE Applied Biosystems, Warrington, UK) by using the PCR primers and run on an automated sequencer (ABI PRISM 310 Genetic Analyzer; Applied Biosystems, Stafford, TX, USA). Phylogenetic analysis was conducted by using MEGA software version 4.0 (10). Complete nucleotide sequence of porcine kobuvirus (strain Kobuvirus/swine/S-1-HUN/2007/Hungary) was submitted to GenBank under accession no. EU Two (13.3%) of 15 samples were positive for porcine sapoviruses; however, a consequent nonspecific, 1,100-nt, strong, and single PCR product was found in all samples by agarose gel electrophoresis (data not shown). The nucleotide sequence of the 1,065-nt nonspecific PCR product was determined. Genetic and amino acid similarity was found to be bovine (U-1) and human Aichi kobuvirus 3C (87 nt) and 3D (978 nt) regions in GenBank database by using BLAST ( cgi). Nucleotide and amino acid identity of the 3C 3D regions were 73% 79% and 69% 70% to U-1 strain and Aichi virus, respectively. The phylogenetic tree confirmed that S-1-HUN belonged to kobuviruses and formed a distinct lineage (Figure). Cleavage sites for 3C and 3D was Q/S. Highly conserved amino acid motif KDELR in 3D (RNA-dependent RNA polymerase) region and high rate of cytidine (29%) and uracil (26%) nucleoside composition were seen in the 3C and 3D parts of the genome of strain S-1-HUN; both are suspected to be a typical skew of kobuviruses (3) K 3 (AB097152) K 6 (AB097154) K 4 (AB097153) N 5 (AB097163) K 49 (AB097159) K 35 (AB097155) K 36 (AB097156) U 1 (AB084788) Most picornavirus genera consist of >2 species (1). Our study reports detection of kobuvirus in domestic pigs. Serendipitously, the generic calicivirus primers p289 and p290, designed for a calicivirus RNAdependent RNA polymerase region, amplified a kobuvirus 3C/3D region when specimens were tested for porcine caliciviruses by RT-PCR. Comparison of the primers p289 and p290 and the S-1-HUN sequence showed that there are 12- and 11-bp homologous regions between the kobuvirus and the 3 end of the primer sequences, respectively. Reverse primer p289 designed for calicivirus (norovirus and sapovirus) conserved amino acid 3D motif YGDD, which is also present in kobuviruses. All apparently healthy animals <10 days of age carried the kobuvirus, which was excreted in the feces. These results indicate a general circulation and endemic infection of kobuvirus on the tested farm. In addition, because of its analogy to other picornaviruses and because bovine kobuvirus was first detected in culture medium that originated from K 38 (AB097157) K 60 (AB097161) CMB07 (EF659453) K 44 (AB097158) N 2 (AB097162) K 55 (AB097160) Kobuvirus/swine/S 1 HUN/2007/Hungary Bovine kobuvirus BAY/1/03/DEU (AY747174) AichiA846/88 (AB010145) Goiania/GO/03/01/Brazil (DQ028632) Human rhinovirus 2 Porcine kobuvirus Human kobuvirus Figure. Phylogenetic tree of porcine kobuvirus (Kobuvirus/swine/S-1-HUN/2007/Hungary, GenBank accession no. EU787450), based upon the 1,065-nt fragment of the kobuvirus 3C/3D regions. The phylogenetic tree was constructed by using the neighbor-joining clustering method; distance was calculated by using the maximum composite likelihood correction for evolutionary rate with help of the MEGA version 4.0 software (10). Bootstrap values (based on 1,000 replicates) for each node if >50% are given. Reference strains were obtained from GenBank. The human rhinovirus 2 strain (X02316) was included in the tree as an outgroup. Scale bar indicates nucleotide substitutions per site. Emerging Infectious Diseases Vol. 14, No. 12, December
102 LETTERS cattle sera (1,3), we cannot exclude the possibility that the S-1-HUN like kobuvirus can cause viremia (and generalized infection) in swine. S-1- HUN like virus may typically cause asymptomatic infections in swine. However, epidemiologic and molecular studies are required regarding the importance of this virus as a causative agent of some diseases of domestic pigs and related animals. Sequence analysis of the complete nucleotide and amino acid sequences of coding (L, P1, P2, and P3: 7,467 nt) and noncoding regions and the genetic organization strain indicate that S-1-HUN is a typical kobuvirus. Phylogenetic analysis shows that S-1 -HUN strain is genetically included in the genus Kobuvirus but is distinct from Aichi and bovine kobuviruses. Porcine kobuvirus strain S-1-HUN is a candidate for a new, third species of the genus Kobuvirus. This work was supported by grants from the Hungarian Scientific Research Fund (OTKA, F048433), and the Enteric Virus Emergence, New Tools (EVENT, SP22-CT ) funded by the European Union. Gábor Reuter, Ákos Boldizsár, István Kiss, and Péter Pankovics Author affi liations: ÁNTSZ Regional Institute of State Public Health Service, Pécs, Hungary (G. Reuter, Á. Boldizsár, P. Pankovics); and Veterinary Diagnostic Directorate, Debrecen, Hungary (I. Kiss) DOI: /eid References 1. Online ICTV. Discussion forum of the International Committee on Taxonomy of Viruses, ICTV 2008 Official Taxonomy [cited 2008 Oct 22]. Available from talk.ictvonline.org 2. Yamashita T, Kobayashi S, Sakae K, Nakata S, Chiba S, Ishihara Y, et al. Isolation of cytopathic small round viruses with BS-C-1 cells from patients with gastroenteritis. J Infect Dis. 1991;164: Yamashita T, Ito M, Kabashima Y, Tsuzuki H, Fujiura A, Sakae K. Isolation and characterization of a new species of kobuvirus associated with cattle. J Gen Virol. 2003;84: DOI: / vir Pham NT, Khamrin P, Nguyen TA, Kanti DS, Phan TG, Okitsu S, et al. Isolation and molecular characterization of Aichi viruses from fecal specimens collected in Japan, Bangladesh, Thailand, and Vietnam. J Clin Microbiol. 2007;45: DOI: /JCM Oh DY, Silva PA, Hauroeder B, Deidrich S, Cardoso DD, Schreier E. Molecular characterization of the first Aichi viruses isolated in Europe and in South America. Arch Virol. 2006;151: DOI: /s Ambert-Balay K, Lorrot M, Bon F, Giraudon H, Kaplon J, Wolfer M, et al. Prevalence and genetic diversity of Aichi virus strains in stool samples from community and hospitalized patients. J Clin Microbiol. 2008;46: DOI: / JCM Khamrin P, Maneekarn N, Peerakome S, Okitsu S, Mizuguchi M, Ushijama H. Bovine kobuviruses from cattle with diarrhea. Emerg Infect Dis. 2008;14: Jiang X, Huang PW, Zhong WM, Farkas T, Cubitt DW, Matson DO. Design and evaluation of a primer pair that detects both Norwalk- and Sapporo-like caliciviruses by RT-PCR. J Virol Methods. 1999;83: DOI: /S (99) Reuter G, Krisztalovics K, Vennema H, Koopmans M. Szűcs Gy. Evidence of the etiological predominance of norovirus in gastroenteritis outbreaks emerging new variant and recombinant noroviruses in Hungary. J Med Virol. 2005;76: DOI: /jmv Tamura K, Dudley J, Nei M, Kumar S. MEGA4: molecular evolutionary genetic analysis (MEGA) software version 4.0. Mol Biol Evol. 2007;24: DOI: /molbev/msm092 Address for correspondence: Gábor Reuter, Regional Laboratory of Virology, National Reference Laboratory of Gastroenteric Viruses, ÁNTSZ Regional Institute of State, Public Health Service, Szabadság út 7, H-7623 Pécs, Hungary; reuter.gabor@baranya.antsz.hu The opinions expressed by authors contributing to this journal do not necessarily reflect the opinions of the Centers for Disease Control and Prevention or the institutions with which the authors are affiliated. Human Case of Rickettsia felis Infection, Taiwan To the Editor: Rickettsia felis, the etiologic agent of flea-borne spotted fever, is carried by fleas worldwide (1). In the past decade, several human cases of R. felis infection have been reported (1 3). Clinical symptoms and biological data for R. felis infections are similar to those for murine typhus and other rickettsial diseases, which makes clinical diagnosis difficult (2). Patients with R. felis infections may have common clinical manifestations, such as fever, headache, myalgia, macular rash, and elevated levels of liver enzymes (4,5). Reportable rickettsioses in Taiwan include scrub typhus, epidemic typhus, and murine typhus. Although there are no known human cases of infections caused by spotted fever group (SFG) rickettsiae in Taiwan, novel strains of SFG rickettsiae have been isolated as recently described (6,7). In addition, evidence for R. felis infections in cat and cat flea populations has been identified by using immunofluorescence assay (IFA), PCR, and organism isolation (K.-H.Tsai et al., unpub. data). We report an indigenous human case of R. felis infection in Taiwan. In January 2005, a 27-year-old woman living in Fongshan City, Kaohsiung County, in southern Taiwan was admitted to Kaohsiung Medical University Hospital with a 4-day history of intermittent fever (37.8 C 38.0 C), chills, headache, and fatigue. Associated symptoms were frequent micturition and a burning sensation upon voiding. The patient was admitted with a possible urinary tract infection; urinalysis showed pyuria (leukocyte count 25 50/high-power field), compatible with the clinical diagnosis. During the 6-day hospital stay, the patient received daily intravenous firstgeneration cephalosporin (cefazolin); gentamicin was given only on the 1970 Emerging Infectious Diseases Vol. 14, No. 12, December 2008
103 Arch Virol (2009) 154: DOI /s ORIGINAL ARTICLE Complete nucleotide and amino acid sequences and genetic organization of porcine kobuvirus, a member of a new species in the genus Kobuvirus, family Picornaviridae Gábor Reuter Æ Ákos Boldizsár Æ Péter Pankovics Received: 22 October 2008 / Accepted: 26 November 2008 / Published online: 19 December 2008 Ó Springer-Verlag 2008 Abstract Kobuvirus is a new genus in the family Picornaviridae. Two species are currently known: Aichi virus (human kobuvirus) and Bovine kobuvirus (U-1). In this study, the complete nucleotide and amino acid sequences and genetic organization of porcine kobuvirus (Kobuvirus/ swine/s-1-hun/2007/hungary, EU787450) were determined. The structure of the S-1-HUN genome, VPg 5 0 UTR leader protein structural proteins (VP0, VP3, VP1) non-structural proteins (2A 2C, 3A 3D) 3 0 UTR poly(a) tail, was found to be typical of picornavirus. The 8210-nucleotide (nt)-long RNA genome contains a large open reading frame (7467 nt) encoding a potential polyprotein precursor of 2488 amino acids (aa) that has 57/56% and 63/64% nt/aa identity with Aichi virus and U-1, respectively. The 5 0 UTR contains a hepacivirus/pestiviruslike internal ribosomal entry site (IRES type IV group-blike) with conserved pseudoknot, II and IIIa f domains. A tandem repeat (a 30-amino-acid-long motif) was detected in 2B. Thirty-nine (65%) of the 60 fecal samples from pigs under the age of 6 months at the tested farm were positive (the incidence was 90% under the age of 3 weeks). Porcine kobuvirus belongs to a potential new species the third in the genus Kobuvirus. Nucleotide sequence data reported are available in the GenBank database under accession number EU G. Reuter (&) Á. Boldizsár P. Pankovics Regional Laboratory of Virology, ÁNTSZ Regional Institute of State Public Health Service, Szabadság út 7, 7623 Pécs, Hungary reuter.gabor@baranya.antsz.hu Introduction Picornaviruses (family Picornaviridae) are small, nonenveloped viruses with single-stranded, positive-sense genomic RNA and are divided into eight genera: Enterovirus, Aphthovirus, Cardiovirus, Hepatovirus, Parechovirus, Erbovirus, Teschovirus and Kobuvirus [8, 23]. Most picornavirus genera consist of two or more species [18]. To date, kobuviruses are comprised of two species: Aichi virus and Bovine kobuvirus, each possessing a single serotype. Aichi virus (strain A846/88) was isolated from a stool sample from a human with gastroenteritis in 1991 [25]. Bovine kobuvirus (strain U-1) was detected in bovine sera and also in feces samples from clinically healthy cattle in 2003 [28]. Both human and bovine kobuviruses were first isolated in Japan. Recently, kobuviruses have also been detected in humans in other countries in Asia [17], Europe [1, 15] and South America [15] and in calves with diarrhea in Southeast Asia [12]. The Aichi virus and the bovine kobuvirus genomes are 8280 and 8374 nucleotide (nt) long, respectively, and both encode a single polyprotein (2432 aa and 2463 aa). This polyprotein has a clear similarity with other picornaviruses. However, some picornaviruses have unique characteristics, such as the genome organization of kobuviruses. Kobuviruses have a leader (L) protein and only three structural proteins, VP0, VP3 and VP1 [26]. The kobuvirus 2A protein contains conserved H-box/NC motifs that are characteristic of a family of cellular proteins involved in the control of cell proliferation [7]. The genetic identity between Aichi and U- 1 viruses is between 47.7% (3 0 UTR) and 70.8% (3D region). In this study, a complete nucleotide sequence and a detailed genomic organization of a new kobuvirus (Kobuvirus/swine/S-1-HUN/2007/Hungary), which was serendipitously detected by RT-PCR in Hungary in fecal 123
104 102 G. Reuter et al. samples from healthy domestic pigs [20], are presented. Generic kobuvirus screening primers were also designed and used for an epidemiological study. Materials and methods Specimens Fecal samples were collected from 60 healthy piglets (Sus scrofa domestica) in four age groups (15 specimens per group under the age of 10 days, 3 weeks, 3 months and 6 months) from a farm located in eastern Hungary in February The aim of the study was to detect porcine calicivirus (norovirus and sapovirus) in domestic pigs by reverse transcription-polymerase chain reaction (RT-PCR) using the generic primer-pairs p289/p290 designed for the calicivirus RNA-dependent RNA polymerase gene (319 nt for norovirus or 331 nt for sapovirus) [9, 20]. RNA isolation and RT-PCR RNA isolation and RT-PCR were performed as described previously [19]. Briefly, nucleic acid was extracted from 300-ll stool suspensions using the TRIzol Reagent (Invitrogen). The RT reaction was performed in 50 ll of reaction mixture containing 109 PCR buffer (Sigma-Aldrich), dntp (10 mm), negative-sense primer, rrnasin (40 U/ll), M- MLV-RT (200 U/ll), nuclease-free water and 3 ll of the extracted RNA for 1 h at 42 C. For PCR, 50 ll of19 PCR buffer containing positive-sense primer and Taq polymerase (Dupl-A-Taq DNA Polymerase, ZenonBio, Szeged, Hungary) was added. The thermocycle program included 3 min at 94 C, 40 cycles for 30 s at 94 C, 90 s at 49 C (or 37 C) and 60 s at 72 C, and a final 10-min extension at 72 C, in a PTC-100 TM (MJ Research, Inc., Watertown, Massachusetts). For the determination of the complete nucleotide sequence of porcine kobuvirus strain S-1-HUN, sequencespecific oligonucleotides were designed on the basis of the conserved regions of Aichi (AB040749) and bovine U-1 (AB084788) kobuviruses and the actually available overlapping S-1-HUN nucleotide sequences (Fig. 1). The extreme 5 0 and 3 0 ends of the genome were determined using a5 0 /3 0 RACE kit (2nd generation, Roche) and oligo(dt) 15 (Promega), respectively (Fig. 1). Generic kobuvirus primers were selected based on the conserved sequences of the Aichi virus (human), U-1 (bovine) and S-1-HUN (swine): UNIVkobu-F (forward, 5 0 -TGGAYTACAAG(/R)TGTTTTGA 1 st nt position of the gene: Oligo d(t)-anchor primer (5 /3 RACE kit, Roche) 5 -GACCACGCGTATCGATGTCGACTTTTTTTTTTTTTTTTV-3 2 S-1-R-338/ CATCTCCTATCCCATTACTCA-3 3 S-1-F-1612/ CCCCTTCCCAACCCCGA-3 4 S-1-R-2655/ GTKCCGCGCATCGCTTCC-3 5 p290 [9] 5 -GATTACTCCAAGTGGGACTCCAC-3 6 p289 [9] 5 -TGACAATGTAATCATCACCATA-3 7 S-1-F-31/48 5 -CCTCACCCTCTTTTCCGG-3 8 S-1-F-1225/ GTTAACCGTAGGAGTCCAACC-3 9 S-1-F-2378/ AAGTTCACGATTGGCTTGAG-3 10 S-1-R-4009/ CGAGRGCMAGGGAGAAGCC-3 11 S-1-F-5184/ TGARTACTTCGATGGGTACAC-3 12 S-1-R-6629/ GGAATGATGATGGATTGTTGGT-3 13 S-1-F-7518/ CACTTCCATCATCAACACCATCA-3 14 oligo(dt) 15 (Promega) 5 -TTTTTTTTTTTTTTT-3 15 S-1-F-928/ TTGTCCGGCCATTCAGAGTC-3 16 S-1-F-1170/ CACACCAACATCTACGG-3 17 S-1-R-1735/ TCCAGAGCGGAATTTTGCAT-3 18 S-1-F-2965/ AACATTGAGTCTGGTTCTGCT-3 19 S-1-R-3757/ TGGGTGTACGTCATGCGCTG-3 20 S-1-F-4311/ CTCYCTTGCTGCTTCTGACAT-3 21 S-1-R-781/ GTTGACAGTAGAGACAGTTGTA-3 22 S-1-R-4472/ AGGACAAGAAGGTAACCAAAGA-3 23 S-1-F-4910/ CCATCACGCGCTGTCGCGA-3 24 S-1-R-6229/ GTCGCCCAGTCAAAGGTGTC-3 Fig. 1 List and positions of primers used in this study. The first and the last nucleotide positions (corresponding to the complete nucleotide sequence of porcine kobuvirus strain S-1-HUN) of the primers are in the primer names (X/X). Sequence-specific oligonucleotides were designed on the basis of the conserved regions of Aichi (AB040749) and bovine U-1 (AB084788) kobuviruses and the available overlapping S-1-HUN nucleotide sequences. R reverse and F forward in the primer names, nt nucleotide 123
105 Complete nucleotide and amino acid sequences and genetic organization of porcine kobuvirus 103 TGC, corresponding to nts of U-1 virus) and UNIV-kobu-R (reverse, 5 0 -ATGTTGTTRATGATGGT GTTGA, corresponding to nts of U-1 virus), amplifying a 216-nt region of the 3D protein. Amplicons were run in a 2% ethidium-bromide-stained agarose gel. Sequence and phylogenetic analysis PCR products were sequenced directly in both directions (at least twice) with BigDye Terminator Cycle Sequencing Ready Reaction Kit (PE Applied Biosystems, Warrington, UK) using the PCR primers and an automated sequencer (ABI PRISM 310 Genetic Analyzer, Applied Biosystems, Stafford, USA). Sequences were analysed using the ClustalX version 1.81 [24] and Genedoc version 2.6 programs [14]. A dendrogram was constructed using the neighborjoining clustering method in MEGA (version 3.1) [13]. Possible recombination events in the coding region were analysed using Simplot software (version 3.5.1, The secondary structures of the and 3 0 -terminal untranslated regions (UTR) were predicted using the Mfold program [29]. The complete sequence of porcine kobuvirus (strain Kobuvirus/swine/S-1-HUN/2007/Hungary) has been submitted to the GenBank/EMBL/DDBJ databases under accession number EU Results Fig. 2 Specific (S: 331 nt long) PCR product for porcine sapovirus (lane 5) and non-specific (K: *1100 nt long) PCR products for porcine kobuvirus (lanes 1, 3, 4, and 5) amplified by generic calicivirus primers p289/p290. Co-infection of porcine sapo- and kobuvirus is shown in lane 5. C- Negative control (porcine sapovirus), C? positive control (porcine sapovirus), M 100-bp molecular marker Two (3.3%) of 60 samples were positive using p289/p290 primers for porcine sapoviruses. However, a *1100-ntlong PCR product was consistently found in 21 (35%) specimens (Fig. 2). Nucleotide sequences of these nonspecific PCR-products had genetic similarity to bovine (strain U-1) and human (Aichi virus) kubovirus 3C pro /3D RNA-dependent RNA polymerase genes in GenBank [20]. The complete RNA genome of the S-1-HUN strain consists of 8210 nt, excluding the poly(a) tail. A large ORF of 7467 nt, which encodes a potential polyprotein precursor of 2488 aa, was found, preceded at the 5 0 end by 576 nt, and followed at the 3 0 end by 167 nt and a poly(a) tail (Fig. 3). The base composition of coding region was found to be 20.6% A, 20.6% G, 31.5% C and 27.3% U. Possible cleavage sites for 3A/3B and 3B/3C pro were Q/G (Q/A in strain U-1) and Q/S (Q/A in strain U-1), respectively. Other cleavage sites were found to be identical to those in bovine strain U-1 (Fig. 3). Analysis of S-1-HUN strain untranslated regions (UTRs) The 576-nt-long 5 0 UTR was 168 and 232 nt shorter than those of Aichi virus (744 nt) and U-1 (808 nt), respectively. The secondary structure of the extreme 5 0 UTR (the first 108 nt) is very similar to those of kobuviruses, particularly (86%) to human kobuvirus (Aichi virus). It forms stem-loop (SL) domains with three potential stem-loops: SL-A, SL-B and SL-C (Fig. 4). Nine AUG motifs were identified in the 5 0 UTR at nts 215, 343, 355, 376, 387, 401, 515, 550 and 577. The last in-frame AUG is the possible authentic initiation codon, at position This AUG codon is not preceded by a significant polypyrimidine tract. A longer pyrimidine tract (UUUCUCU) is located upstream in the RNA between nts 459 and 465, some 49 nts (spacer) upstream of the AUG motif at nt 515/516/517. The initiator AUG is located a further 59 nts downstream (at nt 577). This 59-nt region codes an AUG at position nt 550. In the GenBank database, sequence similarity (79% and 74%) was found between 3 0 the end of the S-1-HUN 5 0 UTR region (from nt 496 to nt 549 and from nt 496 to 568) and the internal ribosomal entry sites (IRESs) of duck hepatitis virus 1 (EU395440) and porcine teschovirus (AY392537), respectively. Based upon these data and the predicted secondary structure of the 5 0 UTR IRES element, S-1-HUN has a potential hepacivirus/pestivirus-like (HP) type IV group-b-like internal ribosomal entry site (IRES IVB-like) with pseudoknot and conserved elements in an upstream bent hairpin-like structure (87-nt-long domains II with variant motifs in loop E: CAA versus AGUA) and in domain III (III 1, III 2, III 3, III 4, IIIa, IIIb, IIIc, IIId, IIIe and IIIf) (Fig. 4). The predicted lengths of the pseudoknot stem 1, stem 2, linker and spacer are 9, 8, 5 and 9 nucleotides, respectively. The conserved nucleotide motifs of branching hairpins IIId and IIIe are: GCGUGGCGGGAAUCC ACGC and UACUGCUUGAUAGAGUC The mispairing in helical segment III 1 (5 0 AGAG/5 0 UACU) 123
106 104 G. Reuter et al. P1 P2 P3 5 -UTR L VP0 VP3 VP1 2A 2B 2C 3A 3B 3C 3D 3 -UTR Q/G Q/H Q/A Q/C Q/G Q/G Q/G Q/A Q/A Q/S U-1 (bovine) /81% 64/63% 40/41% 55/50% 70/77% 51/51% 65/69% 54/48% 62/61% 66/70% 49/37% S-1-HUN (porcine) Q/G Q/H Q/A Q/C Q/G Q/G Q/G Q/G Q/S Q/S * /73% 56/53% 34/26% 53/43% 64/68% 49/48% 53/53% 48/39% 60/57% 60/62% 35/24% Q/G Q/H Q/T Q/G Q/G Q/G Q/G Q/A Q/G Q/S Aichi (human) * two copies of a 30-amino-acid (90 nucleotides) long tandem repeat in 2B region Fig. 3 Genome organization of porcine kobuvirus S-1-HUN and comparison of the structure among human (Aichi) and bovine (U-1) kobuviruses. P1 represents viral structural proteins and P2 and P3 represent nonstructural proteins. Nucleotide (upper number) and amino acid (lower number) lengths are indicated in each gene box. G UC C UG AU GC CG II GU U CGC 3 UG AU GC CG III A A GU G UA 4 A GC Loop E CC A G 340 C A C A C C C AU G U A A U C U A AU CG III CG CG UA 3? A C G IIIa A A C CG C CG U U UU AU A U CA A II2 GCU CCCCUG U CGA GGGGAC UA A A UAC U C UC U AUA CG C C U CG CG CG C G 250 GUCAC C G C C C U C G G C SL-C C GC G C U U C A UC U C A U A A CG U A U CAGUGUG 170 U AU 400 IIIc CG GC C U A A UA CG C G G C C G CG CAUU G GC GC 300 U G C C G G U U A A U A UG C C III U 2 A GU U CG CG CG GU G C C C C C GC U A A U U C U U CG AU GC GC CG UG 497 U II G C CG U A G C G C CG C G C U U GC U U A CCUC G UA AU U A GCGUGGCU G 1 U G U G U C U G G C A U GC C C C C G G A GU UA C A U CG AU C G CU U UCA U A U CG CG C G C CG G U CC UA GC C AGUUCGCACCUA G G A GC U A 515 G UA AU III G CG 1 UG GU A U Stem1 U A AU U A A A 526 GC GC A U CG A 5 UUUGC UA A U U 376 GC U A UG A U AU G U G ACA UACCCUUACUCCACUCACCC UGCUACUUGACCUCGGAA GAAAAAGCACA CGCCCAUGAGU GCUU C G GGGUACUCA UGAG A U IIIe G C GC A GC UUAAA CG U A GC IIIf GC U G GC C G CG A U CG A G Stem2 C C C C C G C SL-A SL-B Nucleotide and amino acid identity between S-1-HUN and members of the two other species of kobuviruses are indicated in percentage (nucleotide/amino acid %) in each gene. Predicted N-terminal cleavage sites are indicated at the top of the border in each gene box II 459 UU A U GC IIIb GC UG UG GC A CAAGACACAUG IIId 577 Fig. 4 Predicted RNA secondary structure of the porcine kobuvirus strain S-1-HUN 5 0 UTR as determined using the Mfold program. The three extreme 5 0 terminal stem-loops (SL) are called SL-A, SL-B and SL-C [21], and the type-iv group-b-like IRES [6] has been annotated as previously proposed. Domains are labeled II and III; individual helical segments are labeled II 1,II 2, III 1, III 2, etc.; and individual hairpins are labeled IIIa and IIIb, etc. to maintain the continuity of the current nomenclature. The positions of conserved domains and the polyprotein AUG start codon are indicated by shaded boxes 123
107 Complete nucleotide and amino acid sequences and genetic organization of porcine kobuvirus 105 is A A in domain III (Fig. 4). The S-1-HUN strain 3 0 -UTR was 167 nt long and 70 nt and 7 nt bases shorter that of the Aichi virus and U-1 strain. No similar sequences were found in GenBank, but the predicted secondary structure of the 3 0 UTR of S-1-HUN consisted of three ds hairpin stems, analogous to U-1 strain (figure not shown). Analysis of S-1-HUN strain coding regions S-1-HUN has a leader (L) protein comprising 195 aa, which was 8 aa and 25 aa longer than those of the U-1 strain and Aichi virus, respectively (Fig. 3). The P1 polyprotein of strain S-1-HUN was 2529 nt long and 42 nt and 9 nt bases shorter that of U-1 strain and Aichi virus, respectively. VP0 and VP1 sequences varied in length: 366/367/370 aa for VP0 and 254/267/253 aa for VP1 in S- 1-HUN, U-1 and Aichi virus, respectively (Fig. 3). The amino acid identity of the VP0, VP3 and VP1 proteins was 70%, 61% and 48% between S-1-HUN and U-1, respectively, and lower between S-1-HUN and Aichi virus (Fig. 3). The P2 and P3 regions are 1998 nt/666 aa and 2355 nt/ 784 aa long, respectively. The amino acid identity of the P2 proteins (non-structural protein 2A 2C) is 68% and 59% to the U-1 strain and Aichi virus (Fig. 3). The amino acid sequence of the 2A region of the S-1-HUN strain had 69% and 53% identity with the U-1 strain and Aichi virus, and this conserved motif consisted of H-box/NC proteins (HWAL and NCXHFV) and a transmembrane domain. Within the 2B region, S-1-HUN encodes two copies of a 30-amino-acid (90-nucleotide)-long motif, AANRVAESI- ETTAS(/T)V(/A)VREADLARSTLNISM, one after the other (in tandem), which extends the 2B region (to 585 instead of 495 nucleotide in U1 or Aichi virus) (Fig. 3). The region 2C is conserved (68 77% aa identity) among the three kobuviruses. A highly conserved amino acid motif, GXXGXGKT (GPPGTGKS), in the nucleotidebinding domain of the putative picornavirus helicase was also found in the S-1-HUN 2C protein. The amino acid identity of the P3 proteins (non-structural proteins 3A 3D) is 71% and 63% to the U-1 strain and Aichi virus; however, the amino acid identity varied from 26% (Aichi virus 3A) to 81% (U-1 virus 3D) (Fig. 3). Protein 3A is smaller (90 aa versus 94 aa and 95 aa), and one copy of 3B (VPg) is longer in S-1-HUN than in U-1 and Aichi virus (34 aa versus 30 aa and 27 aa, respectively). A conserved picornavirus tyrosine residue was observed at the third aa position of S-1-HUN 3B (VPg). The 3C pro protein (serinetype protease) contains a conserved picornavirus catalytic triad formed by histidine, aspartic acid and cysteine, found in S-1-HUN at aa 43, aa 86 and aa 145, respectively. A motif, GXCGG, that is conserved at the C terminus of enterovirus and rhinovirus 3C pro was considered to form a CV-B3 (M16572) EV-70 (D00820) CV-A21 (D00538) PV-1 (J02281) CV-A16 (U05876) BEV-1 (D00214) PEV-9 (Y14459) HRV-14 (K02121) HRV-2 (X02316) PTV (AJ011380) ERBV (X96871) FMDV-O (X00871) ERAV (L43052) EMCV (M81861) TMEV (M20301) Aichi AB U-1 (AB084788) S-1-HUN (EU787450) HPeV-1 (L02971) LV (AF327920) HAV( M14707) AEV (AJ225173) Enterovirus Teschovirus Erbovirus Aphthovirus Cardiovirus Kobuvirus Parechovirus Hepatovirus Fig. 5 Relationship between kobuviruses including porcine kobuvirus (S-1-HUN, EU787450) in bold and other picornaviruses based on amino acid sequences of picornavirus complete coding regions ( L, P1, P2 and P3). The phylogenetic tree was constructed by using neighbor-joining clustering method in MEGA version 3.1. Bootstrap values (based on 1000 replicates) for each node are given if [50%. The scale bar indicates amino acid substitutions per site. Reference strains were obtained from GenBank. CV-B3: coxsackievirus B3, EV- 70: enterovirus type 70, CV-A21: coxsackievirus A21, PV-1 poliovirus type 1, CV-A16 coxsackievirus A16, BEV-1 bovine enterovirus type 1, PEV-9 porcine enterovirus type 9, HRV-14 human rhinovirus type 14, HRV-2 human rhinovirus type 2, PTV porcine teschovirus, ERBV equine rhinitis B virus, FMDV-O foot-and-mouth disease virus type O, ERAV equine rhinitis A virus, EMCV encephalomyocarditis virus, TMEV Theiler s murine encephalomyelitis virus, Aichi human Aichi virus, U-1: bovine kobuvirus, HPeV-1 human parechovirus type 1, LV Ljungan virus, HAV hepatitis A virus, AEV avian encephalomyelitis-like virus part of the active site, and this motif was present but altered to GMCGA in S-1-HUN (GLCGA in U-1 and GLCGS in Aichi virus). A histidine residue that probably participates in the substrate-binding pocket in the trypsine-like protease was found at aa 163 in S-1-HUN 3C pro. The highly conserved amino acid motifs KDELR, YGDD and FLKR were also seen in 3D region of porcine kobuvirus. No intergenotype recombination was observed in the kobuvirus coding regions. The highest sequence identity (up to 74%) was found in the 2C and 3D regions between S-1-HUN, U-1 and Aichi virus by Simplot. The phylogenetic relationships between the S-1-HUN and other representative picornavirus polyproteins (complete L, P1, P2 and P3 regions) are shown in Fig. 5. This confirmed that S-1-HUN is more closely related to kobuviruses, particularly to bovine kobuvirus, than to any other picornaviruses. Detection of kobuviruses by a new generic primer pair A 216-nt-long 3D region of Aichi virus (strain A846/88, kindly provided by T. Yamashita and strain Kobuvirus/ human/szigetvar-hun298/2000/hungary, FJ225407), 123
108 106 G. Reuter et al. porcine kobuvirus (strain Kobuvirus/swine/S-1-HUN/2007/ Hungary, EU787450) and bovine kobuvirus (strain Kobuvirus/bovine/Z20/2002/Hungary, FJ225406) was successfully amplified using kobuvirus primers UNIV-kobu-F/ UNIV-kobu-R (3 min at 94 C, 40 cycles for 30 s at 94 C, 90 s at 42 C and 60 s at 72 C, and a final 10-min extension at 72 C) (figure not shown). Using these primers for reamplification, 39 (65%) of the 60 fecal samples were positive for kobuvirus at the tested farm: 15 of 15 (100%), 14 of 15 (93.3%), 3 of 15 (20%) and 7 of 15 (46.7%) samples from swine under the age of 10 days, 3 weeks, 3 months and 6 months, respectively. Discussion Most picornavirus genera consist of two or more species [18]. This study reports on the genetic characterization and description of porcine kobuvirus, a new member of the genus Kobuvirus family Picornaviridae. S-1-HUN has a typical picornavirus genome organization (VPg-5 0 NTR-leader protein-structural proteinnonstructural protein-3 0 NTR-poly(A) tail). Interestingly, genetic ambivalence was found in the S-1-HUN 5 0 UTR sequence. The nucleotide sequence of the first 108 nt and the secondary structure of the stem-loop domains (three stem-loops: SL-A, SL-B and SL-C) are very similar, particularly between porcine and human kobuviruses, and probably distinctive to kobuviruses. These data support the essential role of this region in the formation of virus particles and in RNA replication [21]. On the other hand, the genetic identity of the 3 0 part of the 5 0 UTRs between strains S-1-HUN and U-1 and Aichi virus was very low. In addition, more genetic similarity and common motifs were found in the IRES element between porcine kobuvirus, porcine teschovirus [10] and duck hepatitis virus 1 than between porcine and human or bovine kobuvirus 5 0 UTRs. The secondary structures of the functionally important IRESs of human and bovine kobuviruses are not known [28]. By contrast, porcine kobuvirus S-1-HUN forms a potential hepacivirus/pestivirus-like (HP) type IV internal ribosomal entry site (IRES IV) described recently by Hellen and de Breyne as one of the new types of IRES among some picornaviruses [6]. Porcine kobuvirus S-1- HUN IRES is more closely related to type IV group-b-like IRESs; however, stem 1 is only 9 nt long (instead of 12 nt in a typical IVB-type IRES), which is characteristic for type-iv group-a IRESs [6]. Type-IV IRES elements described in members of the genera Hepacivirus (hepatitis C virus) and Pestivirus of the family Flaviviridae have also been identified in picornaviruses, including avian encephalomyelitits virus, duck hepatitis virus 1, duck picornavirus, porcine teschovirus, porcine entrovirus 8, Seneca Valley virus and simian picornavirus [6]. These authors concluded that HP-like IRESs have been exchanged between unrelated virus families by recombination, which supports the hypothesis that RNA viruses consist of modular coding and noncoding elements that can exchange and evolve independently. Also, this type of IRESs initiates translation independently of eukaryotic initiation factors (eifs) [6]. However, porcine kobuvirus S-1-HUN potentially belongs to the HP IRES group of viruses, which have conserved ribosome-binding regions in the pseudoknot and domains II and III (particularly hairpins IIId and IIIe). Further study is needed to provide biochemical evidence for this structure and the relationship between porcine kobuvirus and other kobuvirus IRESs. At this moment, it seems that members of the three kobuvirus species (human, bovine and porcine) contain genetically very different IRES sequences and probably form three different types of IRESs. Determination of whether the genetic similarity of porcine kobuvirus IRES can be attributed to recombination events and why this region is a potential promiscuous region (independent genetic entity) must await a detailed study including more kobuvirus field isolates and 5 0 UTR sequences. Co-infection was tested, and the sample used for this study was negative for porcine teschovirus by RT-PCR [16]. The major differences between kobuviruses and other picornaviruses are found in the coding region of the L protein, the absence of a VP0 cleavage site and a distinct form of the 2A protein [22]. The S-1-HUN strain probably also exhibited these features; however, PAGE analysis of purified capsid proteins including the possible intact VP0 was not done. Amino acid identities between S-1-HUN and U-1 and Aichi viruses are lower in the L protein than 3 in A and higher than in 3B. However, the porcine kobuvirus L protein is more closely related to bovine kobuvirus than to the human kobuvirus L protein; relatively low similarity has been found (with two potential conserved motifs: PEDxLxDS and LPG) between the three viral L proteins. The function of the kobuvirus L protein is not known. Neither the catalytic dyad (Cys and His), conserved in a papain-like thiol protease and found in foot-and-mouth disease virus L protein [5], nor a putative zinc-binding motif, Cys His Cys Cys, found in encephalomyocarditis virus [3], could be identified. The percentage identity of the VP1 protein between S-1- HUN and Aichi or U-1 strains was lower than that of the VP0 region. VP1 is the most exposed and immunodominant part of the picornavirus capsid proteins [28], and this protein is also the most variable structural protein among the three kobuviruses. In a previous study, antibody to Aichi virus was not detected in pigs and cattle, and conversely, antibody to bovine U-1 strain was not detected in human and pig sera [28]. This raises the possibility that 123
109 Complete nucleotide and amino acid sequences and genetic organization of porcine kobuvirus 107 porcine kobuvirus forms not only a distinct genetic but also a different antigenic type (serotype). Further study is necessary to determine the antigenic relationship between the three kobuviruses. A high degree of amino acid difference has been identified among 2A proteins of picornaviruses. Some picornaviruses (parechoviruses, avian encephalomyelitis virus) including kobuviruses contain conserved motifs (H-box/NC) that are characteristic of a family of cellular proteins (H-rev107) involved in control of cell proliferation [7]. Like U-1 and Aichi virus, S-1-HUN also encodes this H-box/NC motif in the 2A protein. A tandem repeat (two consecutive copies of a 30-aalong motif) was detected within the 195-aa-long nonstructural protein 2B (also called viroporin in enteroviruses). The nucleotide and amino acid similarities were 82% and 93%, respectively, between the two repeated sequences. Among picornaviruses, tandem repeat regions are known in aphthovirus (three copies of complete 3B); however, two unrelated 2As are found in seal picornavirus and duck hepatitis virus [11]. Interestingly, besides the genetic similarity of one copy of this 2B sequence motif of S-1-HUN to those of bovine (61%) and human (56%) kobuviruses, the repeated sequence has a genetic similarity (23%) to the C-terminus of the cellular IQ-motif-containing GTP-ase activating protein 2 (IQGAP2) detected only in liver [2], according to a BLAST search in GenBank. Homologues between human and picornavirus sequences are not unique. The entero/rhinovirus 3C pro protein (a cellular trypsin-like serine protease), the aphthovirus L protein (papain-like protease), and the parecho/kobuvirus 2A protein (H-box/NC, see above) have cellular homologues [7]. Further analysis is needed to find out whether this observation with kobuvirus 2B and IQGAP2 represents a significant similarity. The kobuvirus 2B function is not known; however, protein 2B has a different function in members of different picornavirus genera [4]. Low sequence similarity was detected among the three kobuviruses in 3 0 UTR region. In addition, no 3 0 UTR sequences similar to that of S-1-HUN were found in the GenBank database. Still, the predicted secondary structure of this relatively long non-translated region consisted of three ds hairpin stems with analogy to those in the U-1 strain. An atypical genome base composition has been identified in Aichi and U-1 viruses [28]. The pyrimidine content (31.5% C and 27.3% U) of S-1-HUN is similar to that of the U-1 strain and is also higher than that of other picornaviruses. The high C composition in the genome is suspected to be a typical trait of kobuviruses [28]. More than 60% of the apparently healthy pigs at the farm carried the virus. The incidence was 90% under the age of 3 weeks. These results indicate a general circulation and probably persistent infection of kobuvirus on the tested farm. On the other hand, based upon the analogy of the pathogenesis of many picornaviruses and the first detection of bovine kobuvirus in culture medium derived from cattle sera [28], we cannot exclude the possibility that kobuviruses in general, and S-1-HUN-like kobuvirus in swine, can cause viraemia (and generalized infection) and have target organ(s) inside the body. This hypothesis should be taken into consideration, too, in kobuvirus pathogenesis which of course needs confirmation since Yamashita and his coworkers suspect that culture medium supplemented with cattle sera had possibly been contaminated with cattle feces containing bovine kobuviruses [28]. S-1-HUN-like virus may typically be asymptomatic in swine; however, more epidemiological studies are required regarding the significance of this virus as a causative agent in any diseases in domestic pig and other animals. The screening primer pair (6261/6779) designed for human kobuvirus 3C/3D junctions [27] was negative for porcine kobuvirus. The kobuvirus primers (UNIV-kobu-F/ R) designed in this study gave PCR products for all three (human, bovine and porcine) known kobuviruses. The sensitive kobuvirus primers designed based upon the conserved 3D sequences of human, bovine and porcine kobuviruses give an opportunity for successful molecular epidemiological studies for the detection of kobuviruses in these species (in one test reaction) and probably in further animal species. Acknowledgments We thank Teruo Yamashita for kindly providing an Aichi virus prototype strain A846/88 in year We thank István Kiss and Sándor Kecskeméti for animal sampling. We thank Zsuzsanna Kocsis for English editing of the manuscript. This work was supported by grants from the Hungarian Scientific Research Fund (OTKA, F048433), and the project Enteric Virus Emergence, New Tools (EVENT, SP22-CT ) funded by the European Union. References 1. Ambert-Balay K, Lorrot M, Bon F, Giraudon H, Kaplon J, Wolfer M, Lebon P, Gendrel D, Pothier P (2008) Prevalence and genetic diversity of Aichi virus strains in stool samples from community and hospitalized patients. J Clin Microbiol 46: Brill S, Li S, Lyman CW, Church DM, Wasmuth JJ, Weissbach CW, Bernards A, Snijders AJ (1996) The Ras GTPase-activatingprotein-related human protein IQGAP2 harbors a potential actin binding domain and interacts with calmodulin and Rho family GTPases. Mol Cell Biol 16: Chen HH, Kong WP, Roos RP (1995) The leader peptide of Theiler s murine encephalomyelitis virus is a zinc-binding protein. J Virol 69: de Jong A, de Mattia F, van Dommelen M, Lanke K, Melchers WJG, Willems HGM, van Kuppeveld FJM (2008) Functional analysis of picornavirus 2B proteins: effects on calcium homeostasis and intracellular protein trafficking. J Virol 82: Gorbalenya AE, Koonin EV, Lai MM (1991) Putative papainrelated thiol proteases of positive-strand RNA viruses. 123
110 108 G. Reuter et al. Identification of rubi- and aphthovirus proteases and delineation of a novel conserved domain associated with proteases of rubi-, alpho- and coronaviruses. FEBS Lett 288: Hellen CUT, de Breyne S (2007) A distinct group of hepacivirus/ pestivirus-like internal ribosomal entry sites in members of diverse Picornavirus genera: evidence for modular exchange of functional noncoding RNA elements by recombination. J Virol 81: Hughes PJ, Stanway G (2000) The 2A proteins of three diverse picornaviruses are related to each other and to the H-rev107 family of proteins involved in the control of cell proliferation. J Gen Virol 81: ICTV Online, Discussion Forum of the International Committee on Taxonomy of Viruses, ICTV 2008 Official Taxonomy Jiang X, Huang PW, Zhong WM, Farkas T, Cubitt DW, Matson DO (1999) Design and evaluation of a primer pair that detects both Norwalk- and Sapporo-like caliciviruses by RT-PCR. J Virol Methods 83: Kaku Y, Chard LS, Inoue T, Belsham GJ (2002) Unique characteristics of a picornavirus internal ribosome entry site from the porcine teschovirus-1 Talfan. J Virol 76: Kapoor A, Victoria J, Simmonds P, Wang C, Shafer RW, Nims R, Nielsen O, Delwart E (2008) A highly divergent picornavirus in a marine mammal. J Virol 82: Khamrin P, Maneekarn N, Peerakome S, Okitsu S, Mizuguchi M, Ushijama H (2008) Bovine kobuviruses from cattle with diarrhea. Emerg Infect Dis 6: Kumar S, Tamura K, Jakobsen IB, Nei M (2001) MEGA2: molecular evolutionary genetics analysis software. Bioinformatics 17: Nicholas KB, Nicholas HB Jr (1997) GeneDoc: a tool for editing and annotating multiple sequence alignments 15. Oh DY, Silva PA, Hauroeder B, Deidrich S, Cardoso DD, Schreier E (2006) Molecular characterization of the first Aichi viruses isolated in Europe and in South America. Arch Virol 151: Palmquist JM, Munir S, Taku A, Kapu V, Goyal SM (2002) Detection of porcine teschovirus and enterovirus type II by reverse transcription-polymerase chain reaction. J Vet Diagn Invest 14: Pham NT, Khamrin P, Nguyen TA, Kanti DS, Phan TG, Okitsu S, Ushijama H (2007) Isolation and molecular characterization of Aichi viruses from fecal specimens collected in Japan, Bangladesh, Thailand, and Vietnam. J Clin Microbiol 45: Racaniello VR (2007) Picornaviridae: the viruses and their replication. In: Knipe DM, Howley PM (eds) Fields virology, 5th edn. Lippincott Williams & Wilkins, Philadelphia, pp Reuter G, Krisztalovics K, Vennema H, Koopmans M, Sz}ucs Gy (2005) Evidence of the etiological predominance of norovirus in gastroenteritis outbreaks emerging new variant and recombinant noroviruses in Hungary. J Med Virol 76: Reuter G, BoldizsárÁ, Kiss I, Pankovics P (2008) Candidate new species of Kobuvirus in porcine hosts. Emerg Infect Dis 12: Sasaki J, Kusuhara Y, Maeno Y, Kobayashi N, Yamashita T, Sakae K, Takeda N, Taniguchi K (2001) Construction of an infectious cdna clone of Aichi virus (a new member of the family Picornaviridae) and mutational analysis of a new stemloop structure at 5 0 end of the genome. J Virol 75: Stanway G, Hovi T, Knowles N, Hyypia T (2002) Molecular and biological basis of picornavirus taxonomy. In: Semler BL, Wimmer E (eds) Molecular biology of picornaviruses. ASM, Washington DC, pp Stanway G, Brown F, Christian P, Hovi T, Hyypiä T, King AMQ, Knowles NJ, Lemon SM, Minor PD, Pallansch MA, Palmenberg AC, Skern T (2005) Family Picornaviridae. In: Fauquet CM, Mayo MA, Maniloff J, Desselberger U, Ball LA (eds) Virus taxonomy. Eight Report of the International Committee on Taxonomy of Viruses. Elsevier/Academic Press, London, pp Thompson JD, Gibson TJ, Plewniak F, Jeanmougin F, Higgins DG (1997) The ClustalX windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools. Nucleic Acids Res 25: Yamashita T, Kobayashi S, Sakae K, Nakata S, Chiba S, Ishihara Y, Isomura S (1991) Isolation of cytopathic small round viruses with BS-C-1 cells from patients with gastroenteritis. J Infect Dis 164: Yamashita T, Sakae K, Tsuzuki H, Suzuki Y, Ishikawa N, Takeda N, Miyamura T, Yamazaki S (1998) Complete nucleotide sequence and genetic organization of Aichi virus, a distinct member of Picornaviridae associated with acute gastro-enteritis in humans. J Virol 72: Yamashita T, Sugiyama M, Tsuzuki H, Sakae K, Suzuki Y, Miyazaki Y (2000) Application of a reverse transcription-pcr for identification and differentiation of Aichi virus, a new member of the picornavirus family associated with gastroenteritis in humans. J Clin Microbiol 38: Yamashita T, Ito M, Kabashima Y, Tsuzuki H, Fujiura A, Sakae K (2003) Isolation and characterization of a new species of kobuvirus associated with cattle. J Gen Virol 84: Zucker M (2003) Mfold web server for nucleic acid folding and hybridization prediction. Nucleic Acids Res 31:
111 DISPATCHES Evolution of Porcine Kobuvirus Infection, Hungary Gábor Reuter, Sándor Kecskeméti, and Péter Pankovics Porcine kobuvirus was fi rst identifi ed in early 2007 in Hungary. Originally thought to be confi ned to the intestine, almost 2 years later the virus was found in the blood of clinically healthy pigs on the same farm. Porcine kobuvirus may be widely distributed on pig farms worldwide. Picornaviruses (family Picornaviridae) are small, nonenveloped viruses with single-stranded, positive-sense genomic RNA; they are divided into 12 genera: Aphthovirus, Avihepatovirus, Cardiovirus, Enterovirus, Erbovirus, Hepatovirus, Parechovirus, Sapelovirus, Senecavirus, Teschovirus, Tremovirus, and Kobuvirus (1). The genus Kobuvirus consists of 2 officially recognized species, Aichi virus and Bovine kobuvirus, and 1 candidate species, porcine kobuvirus (2 4). Aichi virus (strain A846/88) was first isolated in 1989 in Japan, from a fecal sample from a person with acute gastroenteritis (2). Aichi viruses have also been detected in human fecal samples in other countries in Asia (5); Europe (6,7), including Hungary; South America (6); and North Africa (8). Bovine kobuvirus (strain U-1) has been detected in culture medium derived from cattle serum suspected to be contaminated with cattle feces (3) and in fecal samples from cattle in 2003 in Japan (3) and in 2008 in Thailand (9) and Hungary (10). Porcine kobuvirus was first identified and the complete genome (S-1-HUN; EU787450) characterized from fecal samples from domestic pigs in Hungary (4,11). Porcine kobuvirus has also been recently (2009) reported from the People s Republic of China (12). Kobuvirus genomes are kb. Genome organization includes leader (L) protein following structural (VP0, VP3, and VP1) and nonstructural (2A-2C and 3A-3D) regions (1,3,4,11). Genetic identity on the coding region between Aichi, U-1, and S-1- HUN viruses varies from 35% (L protein) to 74% (3D region) (3,11). Kobuvirus infection has been thought to be confined to the intestine. To our knowledge, detection of kobuvirus in the infected host species serum (viremia) has not been reported. We report the endemic circulation and in vivo Author affi liations: ÁNTSZ Regional Institute of State Public Health Service, Pécs, Hungary (G. Reuter, P. Pankovics); and Central Agricultural Offi ce, Debrecen, Hungary (S. Kecskeméti) DOI: /eid evolution of porcine kobuvirus on a pig farm where the virus was originally discovered and virus escape from the gastrointestinal tract, resulting in viremia in domestic pigs. The Study In February 2007, a total of 39 (65%) of 60 fecal samples from clinically healthy domestic pigs on a farm in eastern Hungary were positive (by reverse transcription PCR) for porcine kobuvirus (11) (Table 1). In November 2008, 21 months later, we obtained 60 fecal and 60 serum sample pairs, again from apparently clinically healthy domestic pigs on the same farm. We divided these pigs, all <6 months of age with no diarrhea, into 4 age groups (Table 1). Blood samples were taken from the jugular vein by using closed vacuum sets. Fecal and serum samples were processed and tested separately for porcine kobuvirus by using the same methods (RNA isolation, reverse transcription PCR) and the same primers for screening (UNIV-kobu-F/UNIV-kobu-R for 3D) and for the complete genome analysis described previously (11). PCR products were sequenced directly in both directions with the BigDye Reaction Kit (Applied Biosystems, Warrington, UK) by using the PCR primers and an automated sequencer (ABI PRISM 310 Genetic Analyzer; Applied Biosystems, Stafford, TX, USA). The genome was assigned GenBank accession no. GQ Kobuvirus-positive fecal samples were inoculated into Vero cells. Porcine kobuvirus RNA was detected in 32 (53.3%) of the 60 fecal samples and 16 (26.6%) of the 60 serum samples collected in November 2008 (Table 1). Porcine kobuvirus was found in at least 1 fecal sample from each age group; from serum samples, viral RNA was detected in 1 sample from the 10-day group, 7 samples from the 3 4- week group, and 8 samples from the 6-month group. Porcine kobuvirus was identified in 9 sample pairs (feces and serum) collected at the same time. Only the serum or fecal samples were kobuvirus positive, for 7 and 23 animals, respectively. The 173-nt sequences of kobuvirus 3D region were genetically identical in all serum and fecal samples collected in 2007 and The complete genetic sequence of strain kobuvirus/ swine/k-30-hun/2008/hungary, detected in a fecal specimen from a 3 4-week-old pig in 2008, was determined and compared with the prototype strain kobuvirus/swine/s-1- HUN/2007/Hungary (Table 2). In the nonstructural 2A-3D regions, 74 (1.7%; 1.14% 2.43% between 3D and 3C) nucleotide substitutions could be detected between the prototype strains collected in 2007 (S-1-HUN) and 2008 (K-30- HUN), which led to 13 (0.9%) nonsynonymous amino acid changes (2, 2, 5, 2, and 2 aa in the 2A, 2B, 2C, 3C, and 3D regions). In structural regions, 36 (1.42%) nt substitutions were detected, leading to 6 (0.71%) nonsynonymous amino acid changes (3 3 aa in VP0 and VP3 regions). The highest 696 Emerging Infectious Diseases Vol. 16, No. 4, April 2010
112 Evolution of Porcine Kobuvirus Infection, Hungary Table 1. Incidence of porcine kobuvirus RNA in samples from domestic pigs, Hungary* Specimen, by date 2007 Feb 2008 Nov Pig age Feces Feces Serum <10 d 15 (100) 8 (53.3) 1 (6.7) <3 4 wk 14 (93.3) 13 (86.7) 7 (46.7) <3 mo 3 (20) 5 (33.3) 0 <6 mo 7 (46.7) 6 (40) 8 (53.3) Total 39/60 (65) 32/60 (53.3) 16/60 (26.6) *Values are no. (%) or no. positive/no. tested (%). All animals were on the same farm. For each type of sample and age group, 15 specimens were tested. In November 2008, specimens (feces and serum) were collected in pairs from the same animals. and the lowest rates of nucleotide mutations were seen in coding regions, L and 3D, respectively (Table 2). For amino acid levels, the highest changes in percentage within the 21-month period were found in 2C (1.49%), 2A (1.47%), and VP3 (1.34%) (Table 2). No amino acid changes were detected in regions VP1, 3A, and 3B. No cytopathic effect was seen in Vero cells after serial passages. Conclusions We report the endemic circulation and natural interhost evolution of porcine kobuvirus among domestic pigs on a pig farm within a 21-month period. This follow-up study indicates that domestic pigs are generally infected with porcine kobuvirus. It is likely that porcine kobuvirus is not restricted geographically and is widely distributed on pig farms worldwide. According to a veterinarian s clinical report, no clinical signs were associated with the endemic kobuvirus infection. In this closed-animal community, the susceptible young host generations were continuously infected; the length of the cycle interval when the virus is outside the host (nonreplicative viral form) is probably short. However, the number of nucleotide changes is consistent with rates observed for other picornaviruses (13 15), suggesting good adaptation of the virus host relationship. This adaptation is also supported by the level of the nucleotide changes in different gene regions. More nucleotide mutations were found in nonstructural than in structural regions, which leads to a nearly equal probability for nonsynonymous amino acid changes in these 2 regions. No available data confirm transmission mode, transmission efficiency, and pathogenesis of kobuviruses. Aichi virus has been reported to be shed in feces and maybe transmitted by food (especially seafood) in humans (2,5,7), suggesting fecal oral transmission. However, in the pigs, viral RNA or infectious kobuvirus particles were also present in serum of virus-infected pigs. This finding suggests that porcine kobuvirus (and possibly kobuviruses in general) escaped the gastrointestinal tract into the circulatory system in immunocompetent virus-infected hosts, resulting in viremia. Further study is needed to investigate whether the viremia was acquired passively or actively. In addition to direct fecal oral transmission, the possibility of kobuvirus transmission through breast-feeding (milk) and of bloodborne, foodborne, and zoonotic infections remains. Similar explanations for the pathogenesis of many picornaviruses exist. Escape from the gastrointestinal tract into the bloodstream was probably the situation for the bovine kobuvirus detected in culture medium supplemented with cattle serum (3), which was suspected of possibly being contaminated with feces containing bovine kobuviruses. Porcine kobuvirus viremia open these data to another Table 2. Natural interhost in vivo mutation changes in sequences of 2 porcine kobuvirus strains, Hungary* Nucleotides Amino acids Region Length Difference % Difference Length Difference % Difference 5 UTR L VP0 1, VP VP A B C 1, A B C D 1, UTR Nonstructural 4, , Structural 2, Complete genome 8, , *Strain length shown is for kobuvirus/swine/k-30-hun/2008/hungary (accession no. GQ249161) and is compared with prototype strain kobuvirus/swine/s-1-hun/2007/hungary (length not shown; accession no. EU787450), from samples collected from pigs on the same farm, November 2008 and February 2007 respectively. UTR, untranslated region; L, leader region. 2A-3D. VP0 VP1. Emerging Infectious Diseases Vol. 16, No. 4, April
113 DISPATCHES interpretation for bovine kobuvirus infections and for Aichi virus pathogenesis in humans. Aichi virus was identified in low incidence in fecal samples from humans with gastroenteritis, but seroprevalence was high (6,7). Knowledge about viremia and natural virus evolution is crucial for understanding the pathogenesis, transmission, immunology, and clinical manifestations of kobuvirus infection in general and especially in humans. This work was supported by grants from the Hungarian Scientific Research Fund (OTKA, F048433) and the project Enteric Virus Emergence, New Tools (EVENT, SP22-CT ) funded by the European Union. Dr Reuter is a medical virologist at the Regional Laboratory of Virology, ÁNTSZ Regional Institute of State Public Health Service, Pécs, Hungary. His research interests include molecular epidemiology of viral diseases, especially of viral gastroenteritis and enterically transmitted and food-borne viruses. References 1. International Committee on Taxonomy of Viruses. ICTV official taxonomy: updates since the 8th report [cited 2010 Feb 10]. talk.ictvonline.org/files/ictv_official_taxonomy_updates_since_ the_8th_report/m/default.aspx 2. Yamashita T, Kobayashi S, Sakae K, Nakata S, Chiba S, Ishihara Y, et al. Isolation of cytopathic small round viruses with BS-C-1 cells from patients with gastroenteritis. J Infect Dis. 1991;164: Yamashita T, Ito M, Kabashima Y, Tsuzuki H, Fujiura A, Sakae K. Isolation and characterization of a new species of kobuvirus associated with cattle. J Gen Virol. 2003;84: DOI: / vir Reuter G, Boldizsár Á, Kiss I, Pankovics P. Candidate new species of Kobuvirus in porcine hosts. Emerg Infect Dis. 2008;14: DOI: /eid Pham NT, Khamrin P, Nguyen TA, Kanti DS, Phan TG, Okitsu S, et al. Isolation and molecular characterization of Aichi viruses from fecal specimens collected in Japan, Bangladesh, Thailand, and Vietnam. J Clin Microbiol. 2007;45: DOI: /JCM Oh DY, Silva PA, Hauroeder B, Deidrich S, Cardoso DD, Schreier E. Molecular characterization of the first Aichi viruses isolated in Europe and in South America. Arch Virol. 2006;151: DOI: /s Ambert-Balay K, Lorrot M, Bon F, Giraudon H, Kaplon J, Wolfer M, et al. Prevalence and genetic diversity of Aichi virus strains in stool samples from community and hospitalized patients. J Clin Microbiol. 2008;46: DOI: /JCM Sdiri-Loulizi K, Gharbi-Khélifi H, de Rougemont A, Chouchane S, Sakly N, Ambert-Balay K, et al. Acute infantile gastroenteritis associated with human enteric viruses in Tunisia. J Clin Microbiol. 2008;46: DOI: /JCM Khamrin P, Maneekarn N, Peerakome S, Okitsu S, Mizuguchi M, Ushijama H. Bovine kobuviruses from cattle with diarrhea. Emerg Infect Dis. 2008;14: DOI: /eid Reuter G, Egyed L. Bovine kobuvirus in Europe. Emerg Infect Dis. 2009;15: DOI: /eid Reuter G, Boldizsár Á, Pankovics P. Complete nucleotide and amino acid sequences and genetic organization of porcine kobuvirus, a member of a new species in genus Kobuvirus, family Picornaviridae. Arch Virol. 2009;154: DOI: /s Yu J-M, Jin M, Zhang Q, Li H-Y, Li D-D, Xu Z-Q, et al. Candidate porcine Kobuvirus, China. Emerg Infect Dis. 2009;15: DOI: /eid Domingo E, Holland JJ. RNA virus mutations and fitness for survival. In: Ornston LN, Balows A, Greenberg EP, editors. Annual review of microbiology. Palo Alto (CA): Annual Reviews;1997;51: Cottam EM, Haydon DT, Paton DJ, Gloster J, Wilesmith JW, Ferris NP, et al. Molecular epidemiology of the foot-and-mouth disease virus outbreak in the United Kingdom in J Virol. 2006;80: DOI: /JVI Jorba J, Campagnoli R, De L, Kew O. Calibration of multiple poliovirus molecular clocks covering an extended evolutionary range. J Virol. 2008;82: DOI: /JVI Address for correspondence: Gábor Reuter, Regional Laboratory of Virology, National Reference Laboratory of Gastroenteric Viruses, ÁNTSZ Regional Institute of State Public Health Service, Szabadság út 7. H-7623 Pécs, Hungary; reuter.gabor@ddr.antsz.hu The opinions expressed by authors contributing to this journal do not necessarily reflect the opinions of the Centers for Disease Control and Prevention or the institutions with which the authors are affiliated. 698 Emerging Infectious Diseases Vol. 16, No. 4, April 2010
114 Reviews in Medical Virology R E V I E W Rev. Med. Virol. 2011; 21: Published online in Wiley Online Library (wileyonlinelibrary.com) DOI: /rmv.677 Kobuviruses a comprehensive review Gábor Reuter*, Ákos Boros and Péter Pankovics Regional Laboratory of Virology, National Reference Laboratory of Gastroenteric Viruses, ÁNTSZ Regional Institute of State Public Health Service, Pécs, Hungary SUMMARY Kobuviruses are members of the large and growing family Picornaviridae. Until now, two official, Aichi virus and Bovine kobuvirus, and one candidate kobuvirus species, porcine kobuvirus, have been identified in human, cattle and swine, respectively. In addition, kobu-like viruses were detected very recently in the bat. Aichi virus could be one of the causative agents of gastroenteritis in humans, and kobuviruses probably also cause diarrhoea in cattle and swine. Although Aichi virus has been detected relatively infrequently (0 3%) in human diarrhoea, high seroprevalence, up to 80 95% at the age of 30 40, was found indicating the general nature of infection in different human populations. In the previous years, much new information has accumulated relating to kobuviruses and their host species. This review summarises the current knowledge on kobuviruses including taxonomy, biology and viral characteristics, and covers all aspects of infection including epidemiology, clinical picture, host species diversity, laboratory diagnosis and it gives a summary about possible future perspectives. Copyright # 2011 John Wiley & Sons, Ltd. Received: 30 August 2010; Revised: 24 November 2010; Accepted: 10 December 2010 INTRODUCTION Acute gastroenteritis is a leading cause of morbidity worldwide and an important cause of death among children <5 years old especially in developing countries [1]. The majority of acute gastroenteritis is thought to be caused by viruses, among which rotaviruses, caliciviruses (norovirus and sapovirus), enteric adenoviruses and astroviruses figure prominently. On the other hand, there are many newly discovered enteric viruses, which are potentially also associated with acute gastroenteritis including kobuvirus. DISCOVERY OF THE PROTOTYPE KOBUVIRUS, AICHI VIRUS In Aichi Prefecture, Japan, in March 1989, stool specimens from patients suffering from gastroenteritis associated with consumption of raw oysters were found to contain a new cytopathic virus in BS-C-1 cells [2]. Eight years later, genetic Corresponding author: G. Reuter, Regional Laboratory of Virology, ÁNTSZ Regional Institute of State Public Health Service, H-7623 Szabadság út7. Pécs, Hungary. reuter.gabor@ddr.antsz.hu Abbreviations used: IRES, internal ribosomal entry site; UTR, untranslated region. analysis confirmed that this virus is a novel member of the family Picornaviridae [3]. The complete nucleotide sequence of this picornavirus (GenBank accession no AB040749) was determined by the discoverers, Drs T. Yamashita and N. Takeda, at the Aichi Prefectural Institute of Public Health, Aichi, Japan [3,4]. TAXONOMY OF KOBUVIRUSES The family Picornaviridae contains several important human and animal pathogens grouped into twelve genera: Aphthovirus, Avihepatovirus, Cardiovirus, Enterovirus, Erbovirus, Hepatovirus, Kobuvirus, Parechovirus, Sapelovirus, Senecavirus, Teschovirus and Tremovirus. Kobuviruses were classified into a new genus, Kobuvirus, in 1999 [5]. The name kobu is derived from the Japanese word for bump or knob, because the virion appears bumpy in the electron microscope [6]. Currently, two official (Aichi virus and Bovine kobuvirus) and one candidate kobuvirus species ( porcine kobuvirus ) are known. The prototype kobuvirus strains were detected in human (Aichi virus strain A846/88 AB040749), cattle (bovine kobuvirus strain U-1 AB084788) and swine (porcine kobuvirus strain S-1-HUN; EU787450). At present, Aichi virus is further divided into three genotypes, A, B and C [6,7]. Copyright # 2011 John Wiley & Sons, Ltd.
115 Kobuviruses 33 GENETIC STRUCTURE AND FUNCTION OF KOBUVIRUSES Kobuvirus is a small (30 nm in diameter), spherical, non-enveloped virus which has a single-stranded, positive sense RNA genome [2,8,9]. The prototype kobuviruses, Aichi virus, bovine kobuvirus and porcine kobuvirus have an 8280, 8374 and 8210 nucleotide long genome, respectively, excluding the poly(a) tail. The kobuviral genome encodes a single polyprotein (2432, 2463 and 2488 aa), which undergoes a cleavage cascade performed by virusencoded activities, to give the final virus proteins [3]. Predicted cleavage sites are similar to those in other picornaviruses, all being between a glutamine (Q) and another amino acid, frequently glycine (G), alanine (A) or serine (S) and less frequently histidine (H), cysteine (C) or threonine (T). All kobuviruses share essentially the same genome organisation. Nonstructural protein L (leader), is encoded at the N-terminus of the polyprotein followed by three structural capsid proteins VP0, VP3 and VP1 and seven nonstructural proteins 2A, 2B, 2C, 3A, 3B, 3C and 3D (Figure 1). The open reading frame encoding the kobuvirus polyprotein is preceded by a nucleotide 5 0 untranslated region (UTR). This is predicted to have a complex secondary/tertiary structure involved in genome replication. Interestingly, genetic ambivalence was found in the kobuviral 5 0 UTR sequences. The nucleotide sequence of the first 108 nucleotides and the secondary structure of the stem-loop domain (three stem-loops: SL-A, SL- B and SL-C) are very similar and probably distinctive to kobuviruses. These data support the essential role of this region in the production of infectious virus particles and in RNA replication [4]. On the other hand, the genetic identity of the 3 0 part of the 5 0 UTRs between Aichi virus, bovine and porcine kobuviruses is very low [9]. In addition, more genetic similarity and common motifs were found in the internal ribosomal entry site (IRES) element between porcine kobuvirus, porcine teschovirus and duck hepatitis virus 1 than between porcine and human or bovine kobuvirus 5 0 UTR. The secondary structures of the functionally important IRESs of human and bovine kobuviruses are not known [8]. In contrast, porcine kobuvirus S- 1-HUN forms a hepacivirus/pestivirus-like (HP) type IV IRES [9] described in members of the genera Hepacivirus (hepatitis C virus) and Pestivirus of the family Flaviviridae. Similar structures have also been identified in many picornaviruses, including avian encephalomyelitis virus, duck hepatitis virus 1, duck picornavirus, porcine teschovirus, porcine enterovirus 8, Seneca Valley virus and simian picornavirus [10]. These authors concluded that HP-like IRESs have been exchanged between unrelated virus families by recombination and support the hypothesis that RNA viruses consist of modular coding and non-coding elements that can exchange and evolve independently. At this moment it seems that the three known kobuvirus species (human, bovine and porcine) contain genetically highly different IRES sequences and form probably three different types of IRESs. The major differences between kobuviruses and other picornaviruses are found in the coding region of the L protein, the absence of a VP0 cleavage site and a distinct form of the 2A protein. The L protein comprises aa. The P1 polyprotein is 846, 857 and 843 aa long in Aichi virus, bovine kobuvirus strain U-1 and porcine kobuvirus S-1- HUN, respectively. VP1 sequences varied in length: 254/267/253 aa in S-1-HUN, U-1 and Aichi virus, respectively. VP1 is the most exposed and immunodominant part of the picornavirus capsid proteins [8] and this protein is the most variable structural protein among the three kobuviruses, too (Figure 2). The length of the P2 and P3 regions are also different in the three kobuvirus species. The amino acid identity of the S-1-HUN P2 proteins Figure 1. Genome organisation of kobuviruses: Aichi virus (A846/88), bovine kobuvirus (U-1) and porcine kobuvirus (S-1-HUN). P1 represents viral structure proteins (black boxes), P2 and P3 represents nonstructural proteins. Copyright # 2011 John Wiley & Sons, Ltd. Rev. Med. Virol. 2011; 21: DOI: /rmv
116 34 G. Reuter, A. Á. Boros and P. Pankovics Figure 2. Nucleotide and amino acid differences in percentage on different gene regions between kobuvirus species (black box: U-1 versus S-1-HUN; stripped box: Aichi virus versus S-1-HUN; dotted box: Aichi virus versus U-1). (non-structural protein 2A-2C) is 68% and 59% to U-1 strain and Aichi virus. The amino acid sequence of the 2A region of S-1-HUN strain had 69% and 53% identity with U-1 strain and Aichi viruses (Figure 2). Within the 2B region, S-1-HUN encodes a 90 nucleotide long tandem repeat [9]. Kobuvirus 2B function is not known, however, protein 2B has a different function in different picornavirus genera. The region 2C is conserved (68-77% aa identity) among the three kobuviruses. A highly conserved amino acid motif GXXGXGKT the nucleotide binding domain of the putative picornavirus helicase is present in 2C proteins. The amino acid identity of the S-1-HUN P3 protein (non-structural proteins 3A-3D) is 71% and 63% to U-1 strain and Aichi virus, however, the amino acid identity varied from 26% (Aichi virus 3B) to 81% (U-1 virus 3D). Protein 3A is aa long, and one copy of aa long 3B (VPg; a small polypeptide covalently attached to the 5 0 UTR) is present in kobuviruses (Figure 1). The 3C pro protein is a cysteine protease, which contains a conserved picornavirus catalytic triad formed by histidine, aspartic acid and cysteine. The consistent nature of the predicted cleavage sites suggested that all cleavages in the polyprotein are performed by this protease. Highly conserved amino acid motifs KDELR, YGDD and FLKR are present in kobuvirus 3D region, which is a viral RNA-dependent RNA polymerase. The highest nt sequence identity (up to 74%) was found at 2C and 3D regions between S- 1-HUN, U-1 and Aichi virus (Figure 2). Aichi virus L and 2A functions remain unknown [3]. L protein has no autocatalytic activity and is not involved in polyprotein cleavage, but is probably involved in both viral RNA replication and encapsidation [11]. It was also reported that the 2A proteins of kobuviruses, as well as human parechoviruses and avian encephalomyelitis virus, have conserved motifs (H-box/NC and transmembrane domain) that are characteristic of a family of cellular proteins (H-rev107) involved in the control of cell proliferation [12] and in viral RNA replication [13]. Kobuviruses have a long nt 3 0 UTR and a poly(a) tract following the open reading frame. The secondary structure of 3 0 UTR has not yet been determined. Interestingly, atypical genome base composition has been identified in Aichi, U-1 and S-1-HUN viruses [8,9]. The pyrimidine (C and U) content is higher in kobuviruses than in other picornaviruses. The high C composition in the genome is suspected to be a typical skew of kobuviruses. The viral particles are described to be relatively stable in vitro similar to other picornaviruses: stable in acidic conditions of ph 3.5, resistant to treatment with some chemicals including chloroform, ether and non-ionic detergents, however, the virus is thermolabile at 608C for 30 min [3]. GENETIC DIVERSITY AND RELATIONSHIPS AMONG KOBUVIRUSES To date, only 4, 1 and 3 complete Aichi, bovine and porcine kobuvirus genomes have been characterised, respectively (Figures 2 and 3). Most of the available partial sequences cover the 3C/3D junction since VP1 sequence comparison has been suggested as valuable in picornavirus classification. The nt/aa homology among the 30 available Aichi virus VP1 sequences representing genotype A, B and C and strains from all continents is more than 87/90%, respectively. By analogy with other picornaviruses, these levels of nt/aa Copyright # 2011 John Wiley & Sons, Ltd. Rev. Med. Virol. 2011; 21: DOI: /rmv
Új picornavírusok azonosítása és meghatározása molekuláris biológiai módszerekkel. Egyetemi doktori (Ph.D.) értekezés tézisei.
 Új picornavírusok azonosítása és meghatározása molekuláris biológiai módszerekkel Egyetemi doktori (Ph.D.) értekezés tézisei Pankovics Péter Témavezető: 1 Dr. Reuter Gábor Ph.D., Med. Habil. Programvezető:
Új picornavírusok azonosítása és meghatározása molekuláris biológiai módszerekkel Egyetemi doktori (Ph.D.) értekezés tézisei Pankovics Péter Témavezető: 1 Dr. Reuter Gábor Ph.D., Med. Habil. Programvezető:
Bírálat. Dr. Reuter Gábor Kamilló A burok nélküli, pozitív egyszálú RNS (+ssrns) genomú vírusok sokszínű világa című MTA doktori értekezéséhez
 SEM MEL WEI S EGY ETE M Általános Orvostudományi Kar Orvosi Mikrobiológiai Intézet Igazgató: Dr. Szabó Dóra egyetemi docens Bírálat Dr. Reuter Gábor Kamilló A burok nélküli, pozitív egyszálú RNS (+ssrns)
SEM MEL WEI S EGY ETE M Általános Orvostudományi Kar Orvosi Mikrobiológiai Intézet Igazgató: Dr. Szabó Dóra egyetemi docens Bírálat Dr. Reuter Gábor Kamilló A burok nélküli, pozitív egyszálú RNS (+ssrns)
ÉLELMISZERBIZTONSÁG 4.
 ÉLELMISZERBIZTONSÁG 4. Élelmiszerrel terjedő vírusok Összeállította: Dr. Simon László Nyíregyházi Főiskola Élelmiszerekkel terjedő vírusok A vírusok sejtparaziták, csak élő sejtekben képesek szaporodni.
ÉLELMISZERBIZTONSÁG 4. Élelmiszerrel terjedő vírusok Összeállította: Dr. Simon László Nyíregyházi Főiskola Élelmiszerekkel terjedő vírusok A vírusok sejtparaziták, csak élő sejtekben képesek szaporodni.
REGIONÁLIS VIROLÓGIAI LABORATÓRIUM GASTROENTERÁLIS VÍRUSOK NEMZETI REFERENCIA LABORATÓRIUMA
 REGIONÁLIS VIROLÓGIAI LABORATÓRIUM GASTROENTERÁLIS VÍRUSOK NEMZETI REFERENCIA LABORATÓRIUMA Nemzeti Akkreditáló Testület (NAT) által akkreditált vizsgálólaboratórium (NAT-1-1458/2006) Nemzeti Akkreditáló
REGIONÁLIS VIROLÓGIAI LABORATÓRIUM GASTROENTERÁLIS VÍRUSOK NEMZETI REFERENCIA LABORATÓRIUMA Nemzeti Akkreditáló Testület (NAT) által akkreditált vizsgálólaboratórium (NAT-1-1458/2006) Nemzeti Akkreditáló
Zárójelentés. Állati rotavírusok összehasonlító genomvizsgálata. c. OTKA kutatási programról. Bányai Krisztián (MTA ATK ÁOTI)
 Zárójelentés Állati rotavírusok összehasonlító genomvizsgálata c. OTKA kutatási programról Bányai Krisztián (MTA ATK ÁOTI) 2012 1 Az Állati rotavírusok összehasonlító genomvizsgálata c. programban azt
Zárójelentés Állati rotavírusok összehasonlító genomvizsgálata c. OTKA kutatási programról Bányai Krisztián (MTA ATK ÁOTI) 2012 1 Az Állati rotavírusok összehasonlító genomvizsgálata c. programban azt
A burok nélküli, pozitív, egyszálú RNS (+ssrns) genomú vírusok sokszínő világa
 MTA doktori értekezés tézisei A burok nélküli, pozitív, egyszálú RNS (+ssrns) genomú vírusok sokszínő világa Dr. Reuter Gábor Kamilló Pécs, 2013 TARTALOMJEGYZÉK BEVEZETÉS... 3 CÉLKITŐZÉSEK... 5 ANYAGOK
MTA doktori értekezés tézisei A burok nélküli, pozitív, egyszálú RNS (+ssrns) genomú vírusok sokszínő világa Dr. Reuter Gábor Kamilló Pécs, 2013 TARTALOMJEGYZÉK BEVEZETÉS... 3 CÉLKITŐZÉSEK... 5 ANYAGOK
Nemzeti Akkreditáló Testület. BŐVÍTETT RÉSZLETEZŐ OKIRAT (1) a NAT-1-1668/2012 nyilvántartási számú akkreditált státuszhoz
 Nemzeti Akkreditáló Testület BŐVÍTETT RÉSZLETEZŐ OKIRAT (1) a NAT-1-1668/2012 nyilvántartási számú akkreditált státuszhoz Az Országos Epidemiológiai Központ Virológiai főosztály (1097 Budapest, Albert
Nemzeti Akkreditáló Testület BŐVÍTETT RÉSZLETEZŐ OKIRAT (1) a NAT-1-1668/2012 nyilvántartási számú akkreditált státuszhoz Az Országos Epidemiológiai Központ Virológiai főosztály (1097 Budapest, Albert
Molekuláris biológiai eljárások alkalmazása a GMO analitikában és az élelmiszerbiztonság területén
 Molekuláris biológiai eljárások alkalmazása a GMO analitikában és az élelmiszerbiztonság területén Dr. Dallmann Klára A molekuláris biológia célja az élőlények és sejtek működésének molekuláris szintű
Molekuláris biológiai eljárások alkalmazása a GMO analitikában és az élelmiszerbiztonság területén Dr. Dallmann Klára A molekuláris biológia célja az élőlények és sejtek működésének molekuláris szintű
MTA DOKTORI ÉRTEKEZÉS A BUROK NÉLKÜLI, POZITÍV, EGYSZÁLÚ RNS
 MTA DOKTORI ÉRTEKEZÉS A BUROK NÉLKÜLI, POZITÍV, EGYSZÁLÚ RNS (+ssrns) GENOMÚ VÍRUSOK SOKSZÍNŐ VILÁGA DR. REUTER GÁBOR KAMILLÓ PÉCS, 2013 Látni, amit mindenki lát, és gondolni, amit még senki nem gondolt."
MTA DOKTORI ÉRTEKEZÉS A BUROK NÉLKÜLI, POZITÍV, EGYSZÁLÚ RNS (+ssrns) GENOMÚ VÍRUSOK SOKSZÍNŐ VILÁGA DR. REUTER GÁBOR KAMILLÓ PÉCS, 2013 Látni, amit mindenki lát, és gondolni, amit még senki nem gondolt."
A vírusok kutatásának gyakorlati és elméleti jelentősége
 Vírustan - virológia Jenner himlő elleni vakcina (1798) Pasteur veszettség elleni vakcina (1885) Ivanovszkij az első növénykórokozó vírus felfedezése (dohánymozaik vírus) (1892) Loeffler és Frosch száj-
Vírustan - virológia Jenner himlő elleni vakcina (1798) Pasteur veszettség elleni vakcina (1885) Ivanovszkij az első növénykórokozó vírus felfedezése (dohánymozaik vírus) (1892) Loeffler és Frosch száj-
A T sejt receptor (TCR) heterodimer
 Immunbiológia - II A T sejt receptor (TCR) heterodimer 1 kötőhely lánc lánc 14. kromoszóma 7. kromoszóma V V C C EXTRACELLULÁRIS TÉR SEJTMEMBRÁN CITOSZÓL lánc: VJ régió lánc: VDJ régió Nincs szomatikus
Immunbiológia - II A T sejt receptor (TCR) heterodimer 1 kötőhely lánc lánc 14. kromoszóma 7. kromoszóma V V C C EXTRACELLULÁRIS TÉR SEJTMEMBRÁN CITOSZÓL lánc: VJ régió lánc: VDJ régió Nincs szomatikus
(1) A T sejtek aktiválása (2) Az ön reaktív T sejtek toleranciája. α lánc. β lánc. V α. V β. C β. C α.
 Immunbiológia II A T sejt receptor () heterodimer α lánc kötőhely β lánc 14. kromoszóma 7. kromoszóma 1 V α V β C α C β EXTRACELLULÁRIS TÉR SEJTMEMBRÁN CITOSZÓL αlánc: VJ régió β lánc: VDJ régió Nincs
Immunbiológia II A T sejt receptor () heterodimer α lánc kötőhely β lánc 14. kromoszóma 7. kromoszóma 1 V α V β C α C β EXTRACELLULÁRIS TÉR SEJTMEMBRÁN CITOSZÓL αlánc: VJ régió β lánc: VDJ régió Nincs
Vírusok szerkezete, osztályozása, általános tulajdonságai és szaporodása
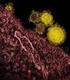 Vírusok szerkezete, osztályozása, általános tulajdonságai és szaporodása Történeti áttekintés Ramses kr.e. 1100 bőrlaesiok a múmián himlő Kanyaró és himlő pontos leírása: Rhazes kr.u. 900 Egyiptomi múmia
Vírusok szerkezete, osztályozása, általános tulajdonságai és szaporodása Történeti áttekintés Ramses kr.e. 1100 bőrlaesiok a múmián himlő Kanyaró és himlő pontos leírása: Rhazes kr.u. 900 Egyiptomi múmia
A mesterséges medencékhez kapcsolódó vízjárványok, USA
 A mesterséges medencékhez kapcsolódó vízjárványok, USA 1971-2002 Nemzetközi Fürdőhigiénés Konferencia Budapest 2005 március 10-11 Rebecca L. Calderon U.S. Environmental Protection Agency National Health
A mesterséges medencékhez kapcsolódó vízjárványok, USA 1971-2002 Nemzetközi Fürdőhigiénés Konferencia Budapest 2005 március 10-11 Rebecca L. Calderon U.S. Environmental Protection Agency National Health
2.6.16. VIZSGÁLATOK IDEGEN KÓROKOZÓKRA HUMÁN ÉLŐVÍRUS-VAKCINÁKBAN
 2.6.16. Vizsgálatok idegen kórokozókra Ph.Hg.VIII. - Ph.Eur.7.0 1 2.6.16. VIZSGÁLATOK IDEGEN KÓROKOZÓKRA HUMÁN ÉLŐVÍRUS-VAKCINÁKBAN 01/2011:20616 Azokhoz a vizsgálatokhoz, amelyekhez a vírust előzőleg
2.6.16. Vizsgálatok idegen kórokozókra Ph.Hg.VIII. - Ph.Eur.7.0 1 2.6.16. VIZSGÁLATOK IDEGEN KÓROKOZÓKRA HUMÁN ÉLŐVÍRUS-VAKCINÁKBAN 01/2011:20616 Azokhoz a vizsgálatokhoz, amelyekhez a vírust előzőleg
ADENOVÍRUSOK OKOZTA BETEGSÉGEK BAROMFIÁLLOMÁNYOKBAN
 ADENOVÍRUSOK OKOZTA BETEGSÉGEK BAROMFIÁLLOMÁNYOKBAN Benkő Mária 1, Ivanics Éva 2, Palya Vilmos 3, Nemes Csaba 4, Kecskeméti Sándor 5, Dán Ádám 2, Kaján Győző 1, Glávits Róbert 2 1 MTA Állatorvos-tudományi
ADENOVÍRUSOK OKOZTA BETEGSÉGEK BAROMFIÁLLOMÁNYOKBAN Benkő Mária 1, Ivanics Éva 2, Palya Vilmos 3, Nemes Csaba 4, Kecskeméti Sándor 5, Dán Ádám 2, Kaján Győző 1, Glávits Róbert 2 1 MTA Állatorvos-tudományi
Elérte hazánkat az influenzajárvány
 Az Országos Epidemiológiai Központ tájékoztatója az influenza figyelőszolgálat adatairól Magyarország 2015. 3. hét Elérte hazánkat az influenzajárvány A figyelőszolgálatban résztvevő orvosok jelentései
Az Országos Epidemiológiai Központ tájékoztatója az influenza figyelőszolgálat adatairól Magyarország 2015. 3. hét Elérte hazánkat az influenzajárvány A figyelőszolgálatban résztvevő orvosok jelentései
DNS-szekvencia meghatározás
 DNS-szekvencia meghatározás Gilbert 1980 (1958) Sanger 3-1 A DNS-polimerázok jellemzői 5'-3' polimeráz aktivitás 5'-3' exonukleáz 3'-5' exonukleáz aktivitás Az új szál szintéziséhez kell: templát DNS primer
DNS-szekvencia meghatározás Gilbert 1980 (1958) Sanger 3-1 A DNS-polimerázok jellemzői 5'-3' polimeráz aktivitás 5'-3' exonukleáz 3'-5' exonukleáz aktivitás Az új szál szintéziséhez kell: templát DNS primer
A Virológiai főosztály szolgáltatásainak jegyzéke
 Virológiai főosztály akkreditálási okiratának (NT-1-1473/2008) érvényessége: 2010. július 5. z újraakkreditálás NT-1-1668 eljárás nyilvántartási számon folyamatban van. Virológiai főosztály szolgáltatásainak
Virológiai főosztály akkreditálási okiratának (NT-1-1473/2008) érvényessége: 2010. július 5. z újraakkreditálás NT-1-1668 eljárás nyilvántartási számon folyamatban van. Virológiai főosztály szolgáltatásainak
Biológus MSc. Molekuláris biológiai alapismeretek
 Biológus MSc Molekuláris biológiai alapismeretek A nukleotidok építőkövei A nukleotidok szerkezete Nukleotid = N-tartalmú szerves bázis + pentóz + foszfát N-glikozidos kötés 5 1 4 2 3 (Foszfát)észter-kötés
Biológus MSc Molekuláris biológiai alapismeretek A nukleotidok építőkövei A nukleotidok szerkezete Nukleotid = N-tartalmú szerves bázis + pentóz + foszfát N-glikozidos kötés 5 1 4 2 3 (Foszfát)észter-kötés
Kapusinszky Beatrix. Elméleti és evolúcióbiológia Doktori Program Programvezető: Prof. Dr. Szathmáry Eörs, Ph.D., egyetemi tanár
 BÉNULÁST OKOZÓ POLIOVÍRUS 3 IZOLÁTUMOK MOLEKULÁRIS GENETIKAI VIZSGÁLATA, VALAMINT A HUMÁN NON-POLIO ENTEROVÍRUSOK MOLEKULÁRIS EPIDEMIOLÓGIÁJA MAGYARORSZÁGON Kapusinszky Beatrix Eötvös Loránd Tudományegyetem
BÉNULÁST OKOZÓ POLIOVÍRUS 3 IZOLÁTUMOK MOLEKULÁRIS GENETIKAI VIZSGÁLATA, VALAMINT A HUMÁN NON-POLIO ENTEROVÍRUSOK MOLEKULÁRIS EPIDEMIOLÓGIÁJA MAGYARORSZÁGON Kapusinszky Beatrix Eötvös Loránd Tudományegyetem
M. A. H. FOOD CONTROLL Kft. Mikrobiológiai vizsgáló Laboratóriuma Állategészségügyi Diagnosztikai Részleg BROILER PROGRAM
 BROILER PROGRAM Telepítés utáni mintavétel: élőállat-szállító autóról vett, lezárt csomagolású alompapír és 10 db útihulla (vagy 25 g meconium), mely a MSZ EN ISO 6579:2002/A1:2007 és a 180/2009 (XII:
BROILER PROGRAM Telepítés utáni mintavétel: élőállat-szállító autóról vett, lezárt csomagolású alompapír és 10 db útihulla (vagy 25 g meconium), mely a MSZ EN ISO 6579:2002/A1:2007 és a 180/2009 (XII:
In Situ Hibridizáció a pathologiai diagnosztikában és ami mögötte van.
 In Situ Hibridizáció a pathologiai diagnosztikában és ami mögötte van. Kneif Józsefné PTE KK Pathologiai Intézet Budapest 2017. 05. 26 Kromoszóma rendellenesség kimutatás PCR technika: izolált nukleinsavak
In Situ Hibridizáció a pathologiai diagnosztikában és ami mögötte van. Kneif Józsefné PTE KK Pathologiai Intézet Budapest 2017. 05. 26 Kromoszóma rendellenesség kimutatás PCR technika: izolált nukleinsavak
Az adenovírusok morfológiája I.
 Adenovírusok A vírusok Elnevezésük a latin virus szóból ered, amelynek jelentése méreg. A vírusok a legkisebb ismert entitások. Csak elektronmikroszkóppal tanulmányozhatóak, mert méretük 20-400 nanométerig
Adenovírusok A vírusok Elnevezésük a latin virus szóból ered, amelynek jelentése méreg. A vírusok a legkisebb ismert entitások. Csak elektronmikroszkóppal tanulmányozhatóak, mert méretük 20-400 nanométerig
Humán kórokozó vírusok előfordulása magyarországi felszíni- és fürdővizekben
 Eötvös Loránd Tudományegyetem Természettudományi Kar Környezettudományi Doktori Iskola Humán kórokozó vírusok előfordulása magyarországi felszíni- és fürdővizekben Kern nita Témavezető: Dr. Vargha Márta,
Eötvös Loránd Tudományegyetem Természettudományi Kar Környezettudományi Doktori Iskola Humán kórokozó vírusok előfordulása magyarországi felszíni- és fürdővizekben Kern nita Témavezető: Dr. Vargha Márta,
3. gyakorlat: nukleinsav-tisztítás, polimeráz láncreakció
 3. gyakorlat: nukleinsav-tisztítás, polimeráz láncreakció A vírus genetikai anyagának vizsgálata (direkt víruskimutatási módszer) biztosítja a legrészletesebb és legspecifikusabb információkat a kimutatott
3. gyakorlat: nukleinsav-tisztítás, polimeráz láncreakció A vírus genetikai anyagának vizsgálata (direkt víruskimutatási módszer) biztosítja a legrészletesebb és legspecifikusabb információkat a kimutatott
Változatlanul alacsony az influenza aktivitása
 Az Országos Epidemiológiai Központ Tájékoztatója az influenza surveillance adatairól Magyarország 2010. 5. hét Változatlanul alacsony az influenza aktivitása 2010. év 5. hetében hasonló volt az influenzaszerű
Az Országos Epidemiológiai Központ Tájékoztatója az influenza surveillance adatairól Magyarország 2010. 5. hét Változatlanul alacsony az influenza aktivitása 2010. év 5. hetében hasonló volt az influenzaszerű
A BVD és IBR mentesítés diagnosztikája : lehetőségek és buktatók. Pálfi Vilmos Budapest 2013. 02. 21.
 A BVD és IBR mentesítés diagnosztikája : lehetőségek és buktatók Pálfi Vilmos Budapest 2013. 02. 21. IBR és BVD mentesítés IBR mentesítés 1. ge negatív állomány kialakítása 2. IBR negatív állomány kialakítása
A BVD és IBR mentesítés diagnosztikája : lehetőségek és buktatók Pálfi Vilmos Budapest 2013. 02. 21. IBR és BVD mentesítés IBR mentesítés 1. ge negatív állomány kialakítása 2. IBR negatív állomány kialakítása
LABORATÓRIUMI ÁLLATOK OKOZTA ZOONOSISOK
 LABORATÓRIUMI ÁLLATOK OKOZTA ZOONOSISOK Prof. Dr. László Fodor Head of the department Dean A laboratóriumi állatok fertőző betegségei indukált betegségek fogékonyság spontán betegségek terjedés az állományon
LABORATÓRIUMI ÁLLATOK OKOZTA ZOONOSISOK Prof. Dr. László Fodor Head of the department Dean A laboratóriumi állatok fertőző betegségei indukált betegségek fogékonyság spontán betegségek terjedés az állományon
DNS munka a gyakorlatban. 2012.10.12. Természetvédelmi genetika
 DNS munka a gyakorlatban 2012.10.12. Természetvédelmi genetika Munka fázisok DNS kivonás elektroforézis (fakultatív lépés) PCR Elektroforézis Szekvenálás Szekvencia elemzés faji azonosítás; variabilitás,
DNS munka a gyakorlatban 2012.10.12. Természetvédelmi genetika Munka fázisok DNS kivonás elektroforézis (fakultatív lépés) PCR Elektroforézis Szekvenálás Szekvencia elemzés faji azonosítás; variabilitás,
Tovább nőtt az orvoshoz forduló betegek száma. Az influenza B vírus felelős a megbetegedések többségéért.
 Az Országos Epidemiológiai Központ tájékoztatója az influenza figyelőszolgálat adatairól Magyarország 2016. 6. hét Tovább nőtt az orvoshoz forduló betegek száma. Az influenza B vírus felelős a megbetegedések
Az Országos Epidemiológiai Központ tájékoztatója az influenza figyelőszolgálat adatairól Magyarország 2016. 6. hét Tovább nőtt az orvoshoz forduló betegek száma. Az influenza B vírus felelős a megbetegedések
A kvantitatív PCR alkalmazhatósága a fertőző bronchitis vakcinák hatékonysági vizsgálatában. Derzsy Napok, Sárvár, 2011 Június 2-3.
 A kvantitatív PCR alkalmazhatósága a fertőző bronchitis vakcinák hatékonysági vizsgálatában Pénzes Zoltán PhD, Soós Pál PhD, Nógrády Noémi PhD, Varga Mária, Jorge Chacón PhD, Zolnai Anna PhD, Nagy Zoltán
A kvantitatív PCR alkalmazhatósága a fertőző bronchitis vakcinák hatékonysági vizsgálatában Pénzes Zoltán PhD, Soós Pál PhD, Nógrády Noémi PhD, Varga Mária, Jorge Chacón PhD, Zolnai Anna PhD, Nagy Zoltán
BIOGÉN ELEMEK Azok a kémiai elemek, amelyek az élőlények számára létfontosságúak
 BIOGÉN ELEMEK Azok a kémiai elemek, amelyek az élőlények számára létfontosságúak A több mint száz ismert kémiai elem nagyobbik hányada megtalálható az élőlények testében is, de sokuknak nincsen kimutatható
BIOGÉN ELEMEK Azok a kémiai elemek, amelyek az élőlények számára létfontosságúak A több mint száz ismert kémiai elem nagyobbik hányada megtalálható az élőlények testében is, de sokuknak nincsen kimutatható
Fehérje expressziós rendszerek. Gyógyszerészi Biotechnológia
 Fehérje expressziós rendszerek Gyógyszerészi Biotechnológia Expressziós rendszerek Cél: rekombináns fehérjék előállítása nagy tisztaságban és nagy mennyiségben kísérleti ill. gyakorlati (therapia) felhasználásokra
Fehérje expressziós rendszerek Gyógyszerészi Biotechnológia Expressziós rendszerek Cél: rekombináns fehérjék előállítása nagy tisztaságban és nagy mennyiségben kísérleti ill. gyakorlati (therapia) felhasználásokra
Chlamydiaceae család 2014.12.02. Obligát intracelluláris baktérium. Replikációs ciklus: Antigenitás. Humán patogén chlamydiák
 Chlamydiaceae család Obligát intracelluláris baktérium Replikáció: élő szövetekben, élőlényekben Replikációs ciklus: Elemi test, reticularis test Antigenitás Csoportspecifikus LPS antigen Faj- és típusspecifikus
Chlamydiaceae család Obligát intracelluláris baktérium Replikáció: élő szövetekben, élőlényekben Replikációs ciklus: Elemi test, reticularis test Antigenitás Csoportspecifikus LPS antigen Faj- és típusspecifikus
Pulyka légzőszervi betegségek
 Pulyka A kórokozók Légzőszervi Gyakorlati helyzet Betegségek Alacsony patogenitású madárinfluenza XXIII. DERZSY NAPOK 2015. június 4-5. Zalakaros A jelenlegi megoldások Merial Avian Technical Services
Pulyka A kórokozók Légzőszervi Gyakorlati helyzet Betegségek Alacsony patogenitású madárinfluenza XXIII. DERZSY NAPOK 2015. június 4-5. Zalakaros A jelenlegi megoldások Merial Avian Technical Services
Megkezdődött hazánkban az influenzajárvány
 Az Országos Epidemiológiai Központ tájékoztatója az influenza figyelőszolgálat adatairól Magyarország 2017. 01. hét Megkezdődött hazánkban az influenzajárvány A figyelőszolgálatban résztvevő orvosok jelentései
Az Országos Epidemiológiai Központ tájékoztatója az influenza figyelőszolgálat adatairól Magyarország 2017. 01. hét Megkezdődött hazánkban az influenzajárvány A figyelőszolgálatban résztvevő orvosok jelentései
Posztvakcinációs rotavírus surveillance Magyarországon, 2007-2011
 Egyetemi doktori (Ph.D.) értekezés tézisei Posztvakcinációs rotavírus surveillance Magyarországon, 2007-2011 Antalné László Brigitta Témavezetők: Dr. Bányai Krisztián és Dr. Kónya József DEBRECENI EGYETEM
Egyetemi doktori (Ph.D.) értekezés tézisei Posztvakcinációs rotavírus surveillance Magyarországon, 2007-2011 Antalné László Brigitta Témavezetők: Dr. Bányai Krisztián és Dr. Kónya József DEBRECENI EGYETEM
Új temékek az UD-GenoMed Kft. kínálatában!
 Új temékek az UD-GenMed Kft. kínálatában! Műanyag termékek: SARSTEDT műanyag termékek teljes választéka Egyszer használats labratóriumi műanyag eszközök szövet és sejttenyésztéshez Vérvételi és diagnsztikai
Új temékek az UD-GenMed Kft. kínálatában! Műanyag termékek: SARSTEDT műanyag termékek teljes választéka Egyszer használats labratóriumi műanyag eszközök szövet és sejttenyésztéshez Vérvételi és diagnsztikai
Picornavírusok okozta betegségek
 Picornavírusok okozta betegségek Picornavirales rend Picornaviridae család 22-30 nm, köbös, burok nincs 4-5 struktúrprotein, VP1-5, 12 kapszomer ss+rns, lineáris, polya, 7-9 kb Csoport-, típus-specifikus
Picornavírusok okozta betegségek Picornavirales rend Picornaviridae család 22-30 nm, köbös, burok nincs 4-5 struktúrprotein, VP1-5, 12 kapszomer ss+rns, lineáris, polya, 7-9 kb Csoport-, típus-specifikus
Patogén mikroorganizmusok vizsgálata molekuláris biológiai módszerekkel
 Patogén mikroorganizmusok vizsgálata molekuláris biológiai módszerekkel Rohonczy Kata, Zoller Linda, Fodor Andrea, Tabajdiné, dr. Pintér Vera FoodMicro Kft. Célkitűzés Élelmiszerekben és takarmányokban
Patogén mikroorganizmusok vizsgálata molekuláris biológiai módszerekkel Rohonczy Kata, Zoller Linda, Fodor Andrea, Tabajdiné, dr. Pintér Vera FoodMicro Kft. Célkitűzés Élelmiszerekben és takarmányokban
TÉMAKÖRÖK. Ősi RNS világ BEVEZETÉS. RNS-ek tradicionális szerepben
 esirna mirtron BEVEZETÉS TÉMAKÖRÖK Ősi RNS világ RNS-ek tradicionális szerepben bevezetés BIOLÓGIAI MOLEKULÁK FEHÉRJÉK NUKLEINSAVAK DNS-ek RNS-ek BIOLÓGIAI MOLEKULÁK FEHÉRJÉK NUKLEINSAVAK DNS-ek RNS-ek
esirna mirtron BEVEZETÉS TÉMAKÖRÖK Ősi RNS világ RNS-ek tradicionális szerepben bevezetés BIOLÓGIAI MOLEKULÁK FEHÉRJÉK NUKLEINSAVAK DNS-ek RNS-ek BIOLÓGIAI MOLEKULÁK FEHÉRJÉK NUKLEINSAVAK DNS-ek RNS-ek
A rotavírus a gyomor és a belek fertőzését előidéző vírus, amely súlyos gyomor-bélhurutot okozhat.
 A rotavírus a gyomor és a belek fertőzését előidéző vírus, amely súlyos gyomor-bélhurutot okozhat. A rotavírus az egyik leggyakoribb okozója a súlyos hasmenésnek csecsemő és kisdedkorban. Évente világszerte
A rotavírus a gyomor és a belek fertőzését előidéző vírus, amely súlyos gyomor-bélhurutot okozhat. A rotavírus az egyik leggyakoribb okozója a súlyos hasmenésnek csecsemő és kisdedkorban. Évente világszerte
Virológia gyakorlatok
 Virológia gyakorlatok A virológia gyakorlatok célja, hogy a hallgatók betekintést nyerhessenek az állatorvosi virológiai diagnosztika különböző módszereibe. A hallgatók a gyakorlatokon esetfeldolgozás
Virológia gyakorlatok A virológia gyakorlatok célja, hogy a hallgatók betekintést nyerhessenek az állatorvosi virológiai diagnosztika különböző módszereibe. A hallgatók a gyakorlatokon esetfeldolgozás
Összefoglaló a 2018/2019. évi influenza szezon hetéről ( ) Baranya megye
 Összefoglaló a 218/219. évi influenza szezon 5-51. hetéről (218.12.1 218.12.23) Baranya megye Betegforgalmi adatok: A figyelőszolgálatban résztvevő orvosok jelentése alapján Baranya megyében 218. 5-51.
Összefoglaló a 218/219. évi influenza szezon 5-51. hetéről (218.12.1 218.12.23) Baranya megye Betegforgalmi adatok: A figyelőszolgálatban résztvevő orvosok jelentése alapján Baranya megyében 218. 5-51.
Élő metapneumovírus vakcina fejlesztése tojóállományok részére: ártalmatlansági és hatékonysági vizsgálatok. Hajdúszoboszló, június 2-3.
 Élő metapneumovírus vakcina fejlesztése tojóállományok részére: ártalmatlansági és hatékonysági vizsgálatok Dr. Kollár Anna, Dr. Soós Pál, Antalné Szalkai Teodóra, Dr. Tóth Ádám, Dr. Nagy Zoltán, Gubucz-Sombor
Élő metapneumovírus vakcina fejlesztése tojóállományok részére: ártalmatlansági és hatékonysági vizsgálatok Dr. Kollár Anna, Dr. Soós Pál, Antalné Szalkai Teodóra, Dr. Tóth Ádám, Dr. Nagy Zoltán, Gubucz-Sombor
Mutagenezis és s Karcinogenezis kutatócsoport. Haracska Lajos.
 Mutagenezis és s Karcinogenezis kutatócsoport SZBK Genetikai Intézete (429 dolgozó,, Tel: 62-599666) haracska@brc.hu Haracska Lajos www.brc.hu/lajoslab Evolúci ció és s karcinogenezis: közös k s gyökerek
Mutagenezis és s Karcinogenezis kutatócsoport SZBK Genetikai Intézete (429 dolgozó,, Tel: 62-599666) haracska@brc.hu Haracska Lajos www.brc.hu/lajoslab Evolúci ció és s karcinogenezis: közös k s gyökerek
Az Országos Epidemiológiai Központ tájékoztatója az influenza figyelőszolgálat adatairól Magyarország hét
 Az Országos Epidemiológiai Központ tájékoztatója az influenza figyelőszolgálat adatairól Magyarország 2015. 8. hét Az elmúlt héten tetőzött az influenzajárvány A figyelőszolgálatban résztvevő orvosok 2015.
Az Országos Epidemiológiai Központ tájékoztatója az influenza figyelőszolgálat adatairól Magyarország 2015. 8. hét Az elmúlt héten tetőzött az influenzajárvány A figyelőszolgálatban résztvevő orvosok 2015.
RNS-ek. 1. Az ősi RNS Világ: - az élet hajnalán. 2. Egy már ismert RNS Világ: - a fehérjeszintézis ben résztvevő RNS-ek
 RNS-ek RNS-ek 1. Az ősi RNS Világ: - az élet hajnalán 2. Egy már ismert RNS Világ: - a fehérjeszintézis ben résztvevő RNS-ek 3. Egy újonnan felfedezett RNS Világ: - szabályozó RNS-ek 4. Transzkripció Ősi
RNS-ek RNS-ek 1. Az ősi RNS Világ: - az élet hajnalán 2. Egy már ismert RNS Világ: - a fehérjeszintézis ben résztvevő RNS-ek 3. Egy újonnan felfedezett RNS Világ: - szabályozó RNS-ek 4. Transzkripció Ősi
norovírus járványok számának kiugrásában és az epidémiákban. Az egyes GII4 variánsok váltásai időben gyorsan és variáns szinten teljesen
 Az OTKA F048433 számú Magyarországi gastroenteritis járványokból kimutatott calicivírusok molekuláris szintű elemzése című pályázat összefoglalója (zárójelentés) A calicivírus vizsgálataink a 2005 évben,
Az OTKA F048433 számú Magyarországi gastroenteritis járványokból kimutatott calicivírusok molekuláris szintű elemzése című pályázat összefoglalója (zárójelentés) A calicivírus vizsgálataink a 2005 évben,
17.2. ábra Az immunválasz kialakulása és lezajlása patogén hatására
 11. 2016. nov 30. 17.2. ábra Az immunválasz kialakulása és lezajlása patogén hatására 17.3. ábra A sejtközötti térben és a sejten belül élő és szaporodó kórokozók ellen kialakuló védekezési mechanizmusok
11. 2016. nov 30. 17.2. ábra Az immunválasz kialakulása és lezajlása patogén hatására 17.3. ábra A sejtközötti térben és a sejten belül élő és szaporodó kórokozók ellen kialakuló védekezési mechanizmusok
Hamar Péter. RNS világ. Lánczos Kornél Gimnázium, Székesfehérvár, 2014. október 21. www.meetthescientist.hu 1 26
 Hamar Péter RNS világ Lánczos Kornél Gimnázium, Székesfehérvár, 2014. október 21. 1 26 Főszereplők: DNS -> RNS -> fehérje A kód lefordítása Dezoxy-ribo-Nuklein-Sav: DNS az élet kódja megkettőződés (replikáció)
Hamar Péter RNS világ Lánczos Kornél Gimnázium, Székesfehérvár, 2014. október 21. 1 26 Főszereplők: DNS -> RNS -> fehérje A kód lefordítása Dezoxy-ribo-Nuklein-Sav: DNS az élet kódja megkettőződés (replikáció)
NEMZETI NOSOCOMIALIS SURVEILLANCE RENDSZER NOSOCOMIALIS JÁRVÁNYOK, 2006 EPINFO 2007; 35:349-359.
 NEMZETI NOSOCOMIALIS SURVEILLANCE RENDSZER NOSOCOMIALIS JÁRVÁNYOK, 2006 EPINFO 2007; 35:349-359. Az ÁNTSZ Fővárosi és Megyei Intézetei az OEK Kórházi járványügyi osztályára a 2006. évben 143 nosocomialis
NEMZETI NOSOCOMIALIS SURVEILLANCE RENDSZER NOSOCOMIALIS JÁRVÁNYOK, 2006 EPINFO 2007; 35:349-359. Az ÁNTSZ Fővárosi és Megyei Intézetei az OEK Kórházi járványügyi osztályára a 2006. évben 143 nosocomialis
-pl. baktériumok és gombák toxinjai, mérgező növények, mérgező állati termékek, növényvédő szerek, különböző szennyező anyagok
 ÉLELEM ÚTJÁN TERJEDŐ MEGBETEGEDÉSEK = elfogyasztott ételben, italban levő mérgező hatású anyag (mikroorganizmus, mérgező növény, vegyi anyag) okoz Jellemzői: rövid lappangási idő heveny, robbanásszerű
ÉLELEM ÚTJÁN TERJEDŐ MEGBETEGEDÉSEK = elfogyasztott ételben, italban levő mérgező hatású anyag (mikroorganizmus, mérgező növény, vegyi anyag) okoz Jellemzői: rövid lappangási idő heveny, robbanásszerű
Kutatási terület: Haszonállatok egészségvédelme, állománydiagnosztika
 Dr. Abonyi Tamás Tanulmányok: Állatorvostudományi Egyetem, 1981. Kutatási terület: Haszonállatok egészségvédelme, állománydiagnosztika Korábbi munkahelyek: MTA Mezőgazdasági Kutató Intézet Kísérleti Gazdasága
Dr. Abonyi Tamás Tanulmányok: Állatorvostudományi Egyetem, 1981. Kutatási terület: Haszonállatok egészségvédelme, állománydiagnosztika Korábbi munkahelyek: MTA Mezőgazdasági Kutató Intézet Kísérleti Gazdasága
Levonulóban az influenzajárvány
 Az Országos Epidemiológiai Központ tájékoztatója az influenza figyelőszolgálat adatairól Magyarország 2016. 9. hét Levonulóban az influenzajárvány A figyelőszolgálatban résztvevő orvosok jelentéseinek
Az Országos Epidemiológiai Központ tájékoztatója az influenza figyelőszolgálat adatairól Magyarország 2016. 9. hét Levonulóban az influenzajárvány A figyelőszolgálatban résztvevő orvosok jelentéseinek
Antiszenz hatás és RNS interferencia (a génexpresszió befolyásolásának régi és legújabb lehetőségei)
 Antiszenz hatás és RNS interferencia (a génexpresszió befolyásolásának régi és legújabb lehetőségei) Az antiszenz elv története Reverz transzkripció replikáció transzkripció transzláció DNS DNS RNS Fehérje
Antiszenz hatás és RNS interferencia (a génexpresszió befolyásolásának régi és legújabb lehetőségei) Az antiszenz elv története Reverz transzkripció replikáció transzkripció transzláció DNS DNS RNS Fehérje
transzláció DNS RNS Fehérje A fehérjék jelenléte nélkülözhetetlen minden sejt számára: enzimek, szerkezeti fehérjék, transzportfehérjék
 Transzláció A molekuláris biológia centrális dogmája transzkripció transzláció DNS RNS Fehérje replikáció Reverz transzkriptáz A fehérjék jelenléte nélkülözhetetlen minden sejt számára: enzimek, szerkezeti
Transzláció A molekuláris biológia centrális dogmája transzkripció transzláció DNS RNS Fehérje replikáció Reverz transzkriptáz A fehérjék jelenléte nélkülözhetetlen minden sejt számára: enzimek, szerkezeti
DNS replikáció. DNS RNS Polipeptid Amino terminus. Karboxi terminus. Templát szál
 DNS replikáció DNS RNS Polipeptid Amino terminus Templát szál Karboxi terminus Szuper-csavarodott prokarióta cirkuláris DNS Hisztonok komplexe DNS hisztonokra történő felcsvarodása Hiszton-kötött negatív
DNS replikáció DNS RNS Polipeptid Amino terminus Templát szál Karboxi terminus Szuper-csavarodott prokarióta cirkuláris DNS Hisztonok komplexe DNS hisztonokra történő felcsvarodása Hiszton-kötött negatív
A LABORBAN ELÉRHETŐ GYORSTESZTEK ÉRTELMEZÉSE
 A LABORBAN ELÉRHETŐ GYORSTESZTEK ÉRTELMEZÉSE Dr. Kiss Virág 2016.11.20 A LABORBAN ELÉRHETŐ GYORSTESZTEK: 1. Clostridium difficile gyorsteszt 2. RSV gyorsteszt 3. Influenza gyorsteszt 4. H.pylori Ag gyorsteszt
A LABORBAN ELÉRHETŐ GYORSTESZTEK ÉRTELMEZÉSE Dr. Kiss Virág 2016.11.20 A LABORBAN ELÉRHETŐ GYORSTESZTEK: 1. Clostridium difficile gyorsteszt 2. RSV gyorsteszt 3. Influenza gyorsteszt 4. H.pylori Ag gyorsteszt
3. Kombinált, amelynek van helikális és kubikális szakasza, pl. a bakteriofágok és egyes rákkeltő RNS vírusok.
 Vírusok Szerkesztette: Vizkievicz András A XIX. sz. végén Dmitrij Ivanovszkij orosz biológus a dohány mozaikosodásának kórokozóját próbálta kimutatni. A mozaikosodás a levél foltokban jelentkező sárgulása.
Vírusok Szerkesztette: Vizkievicz András A XIX. sz. végén Dmitrij Ivanovszkij orosz biológus a dohány mozaikosodásának kórokozóját próbálta kimutatni. A mozaikosodás a levél foltokban jelentkező sárgulása.
Lymphoma sejtvonalak és gyerekkori leukémia (ALL) sejtek mikro RNS (mir) profiljának vizsgálata
 Lymphoma sejtvonalak és gyerekkori leukémia (ALL) sejtek mikro RNS (mir) profiljának vizsgálata Dr. Nemes Karolina, Márk Ágnes, Dr. Hajdu Melinda, Csorba Gézáné, Dr. Kopper László, Dr. Csóka Monika, Dr.
Lymphoma sejtvonalak és gyerekkori leukémia (ALL) sejtek mikro RNS (mir) profiljának vizsgálata Dr. Nemes Karolina, Márk Ágnes, Dr. Hajdu Melinda, Csorba Gézáné, Dr. Kopper László, Dr. Csóka Monika, Dr.
Dr. Nemes Nagy Zsuzsa Szakképzés Karl Landsteiner Karl Landsteiner:
 Az AB0 vércsoport rendszer Dr. Nemes Nagy Zsuzsa Szakképzés 2011 Az AB0 rendszer felfedezése 1901. Karl Landsteiner Landsteiner szabály 1901 Karl Landsteiner: Munkatársai vérmintáit vizsgálva fedezte fel
Az AB0 vércsoport rendszer Dr. Nemes Nagy Zsuzsa Szakképzés 2011 Az AB0 rendszer felfedezése 1901. Karl Landsteiner Landsteiner szabály 1901 Karl Landsteiner: Munkatársai vérmintáit vizsgálva fedezte fel
Fertőző betegségek járványtana. dr. Gyuranecz Miklós MTA ATK Állatorvos-tudományi Intézet
 Fertőző betegségek járványtana dr. Gyuranecz Miklós MTA ATK Állatorvos-tudományi Intézet Fogalmak Fertőző betegség: olyan betegség, melyet specifikus fertőző ágens vagy annak terméke hoz létre. Ezek közvetlenül
Fertőző betegségek járványtana dr. Gyuranecz Miklós MTA ATK Állatorvos-tudományi Intézet Fogalmak Fertőző betegség: olyan betegség, melyet specifikus fertőző ágens vagy annak terméke hoz létre. Ezek közvetlenül
Új temékek az UD- GenoMed Kft. kínálatában!
 Új temékek az UD- GenoMed Kft. kínálatában! Szolgáltatásaink: Medical Genomic Technologies Kft. Betegtoborzás és biobanking Bioinformatika o Adatelemzés/adatbányászás o Integrált adatbázis készítés Sejtvonal
Új temékek az UD- GenoMed Kft. kínálatában! Szolgáltatásaink: Medical Genomic Technologies Kft. Betegtoborzás és biobanking Bioinformatika o Adatelemzés/adatbányászás o Integrált adatbázis készítés Sejtvonal
A Legionella jelenlétének kimutatása
 A Legionella jelenlétének kimutatása Diagnosztikai lehetőségek Kari András Budapest 2016. 04.07. Legionella nemzetség: aerob coccoid-pálca Gram (gyengén festődik) kataláz +, oxidáz +, hippurátot hidrulizálja
A Legionella jelenlétének kimutatása Diagnosztikai lehetőségek Kari András Budapest 2016. 04.07. Legionella nemzetség: aerob coccoid-pálca Gram (gyengén festődik) kataláz +, oxidáz +, hippurátot hidrulizálja
Az Országos Epidemiológiai Központ tájékoztatója az influenza figyelőszolgálat adatairól Magyarország 2015. 7. hét
 Az Országos Epidemiológiai Központ tájékoztatója az influenza figyelőszolgálat adatairól Magyarország 2015. 7. hét Országosan nem változott az influenzaaktivitás A figyelőszolgálatban résztvevő orvosok
Az Országos Epidemiológiai Központ tájékoztatója az influenza figyelőszolgálat adatairól Magyarország 2015. 7. hét Országosan nem változott az influenzaaktivitás A figyelőszolgálatban résztvevő orvosok
Intenzíven terjed az influenza
 Az Országos Epidemiológiai Központ tájékoztatója az influenza figyelőszolgálat adatairól Magyarország 2015. 4. hét Intenzíven terjed az influenza A 4. naptári héten tovább nőtt az influenzás panaszok miatt
Az Országos Epidemiológiai Központ tájékoztatója az influenza figyelőszolgálat adatairól Magyarország 2015. 4. hét Intenzíven terjed az influenza A 4. naptári héten tovább nőtt az influenzás panaszok miatt
Influenzaszerű megbetegedések részaránya korcsoportonként, Baranya megye, 2018/2019.
 1. 3. 5. 7. 9. 11. 13. 15. 17. 19. részarány 52 2. 4. 6. 8. 1. 12. 14. 16. 18. 2. betegforgalom (fő) ISZM (fő) Összefoglaló a 218/219. évi influenza szezon 2. hetéről (219.1.7-219.1.13) Baranya megye Betegforgalmi
1. 3. 5. 7. 9. 11. 13. 15. 17. 19. részarány 52 2. 4. 6. 8. 1. 12. 14. 16. 18. 2. betegforgalom (fő) ISZM (fő) Összefoglaló a 218/219. évi influenza szezon 2. hetéről (219.1.7-219.1.13) Baranya megye Betegforgalmi
TRIPSZIN TISZTÍTÁSA AFFINITÁS KROMATOGRÁFIA SEGÍTSÉGÉVEL
 TRIPSZIN TISZTÍTÁSA AFFINITÁS KROMATOGRÁFIA SEGÍTSÉGÉVEL Az egyes biomolekulák izolálása kulcsfontosságú a biológiai szerepük tisztázásához. Az affinitás kromatográfia egyszerűsége, reprodukálhatósága
TRIPSZIN TISZTÍTÁSA AFFINITÁS KROMATOGRÁFIA SEGÍTSÉGÉVEL Az egyes biomolekulák izolálása kulcsfontosságú a biológiai szerepük tisztázásához. Az affinitás kromatográfia egyszerűsége, reprodukálhatósága
Intenzíven terjed az influenza
 Az Országos Epidemiológiai Központ tájékoztatója az influenza figyelőszolgálat adatairól Magyarország 2017. 3. hét Intenzíven terjed az influenza A figyelőszolgálatban résztvevő orvosok jelentései alapján
Az Országos Epidemiológiai Központ tájékoztatója az influenza figyelőszolgálat adatairól Magyarország 2017. 3. hét Intenzíven terjed az influenza A figyelőszolgálatban résztvevő orvosok jelentései alapján
13. RNS szintézis és splicing
 13. RNS szintézis és splicing 1 Visszatekintés: Az RNS típusai és szerkezete Hírvivő RNS = mrns (messenger RNA = mrna) : fehérjeszintézis pre-mrns érett mrns (intronok kivágódnak = splicing) Transzfer
13. RNS szintézis és splicing 1 Visszatekintés: Az RNS típusai és szerkezete Hírvivő RNS = mrns (messenger RNA = mrna) : fehérjeszintézis pre-mrns érett mrns (intronok kivágódnak = splicing) Transzfer
Tovább csökkent az influenzaszerű megbetegedések száma
 Az Országos Epidemiológiai Központ tájékoztatója az influenza figyelőszolgálat adatairól Magyarország 2017. 8. hét Tovább csökkent az influenzaszerű megbetegedések száma A figyelőszolgálatban résztvevő
Az Országos Epidemiológiai Központ tájékoztatója az influenza figyelőszolgálat adatairól Magyarország 2017. 8. hét Tovább csökkent az influenzaszerű megbetegedések száma A figyelőszolgálatban résztvevő
A SZARVASMARHA LÉGZŐSZERVI BETEGSÉG-KOMPLEXE
 VII. Praxismenedzsment Konferencia Budapest, 2013. november 23. A SZARVASMARHA LÉGZŐSZERVI BETEGSÉG-KOMPLEXE Fodor László Szent István Egyetem, Állatorvos-tudományi Kar, Járványtani és Mikrobiológiai Tanszék
VII. Praxismenedzsment Konferencia Budapest, 2013. november 23. A SZARVASMARHA LÉGZŐSZERVI BETEGSÉG-KOMPLEXE Fodor László Szent István Egyetem, Állatorvos-tudományi Kar, Járványtani és Mikrobiológiai Tanszék
BD Vacutainer Molekuláris Diagnosztikai termékei
 BD Vacutainer Molekuláris Diagnosztikai termékei Andrea Süle, PhD Termékspecialista BD Diagnostics, Preanalytical Systems MOLSZE XI. Nagygyőlés, Pécs, 2009 augusztus 27-29. BD A BD egy orvostechnológiai
BD Vacutainer Molekuláris Diagnosztikai termékei Andrea Süle, PhD Termékspecialista BD Diagnostics, Preanalytical Systems MOLSZE XI. Nagygyőlés, Pécs, 2009 augusztus 27-29. BD A BD egy orvostechnológiai
A 2013/1. SZÁM TARTALMA. Posta J., Rudiné Mezei A., Mihók S.: Díjugrató sportban a hasznos
 A 2013/1. SZÁM TARTALMA ÚJÉVI KÖSZÖNTŐ LÓ Posta J., Rudiné Mezei A., Mihók S.: Díjugrató sportban a hasznos élettartam értékelése különböző mérőszámokkal / 7 SZARVASMARHA Pankovics P., Boros Á., Nemes
A 2013/1. SZÁM TARTALMA ÚJÉVI KÖSZÖNTŐ LÓ Posta J., Rudiné Mezei A., Mihók S.: Díjugrató sportban a hasznos élettartam értékelése különböző mérőszámokkal / 7 SZARVASMARHA Pankovics P., Boros Á., Nemes
18/1998. (VI. 3.) NM rendelet a fertőző betegségek és a járványok megelőzése érdekében szükséges járványügyi intézkedésekről
 18/1998. (VI. 3.) NM rendelet a fertőző betegségek és a járványok megelőzése érdekében szükséges járványügyi intézkedésekről Dr. Bíró Zoltán állatorvos MH EK KJSZ ALAPFOGALMAK 1. egészségügyi ellátással
18/1998. (VI. 3.) NM rendelet a fertőző betegségek és a járványok megelőzése érdekében szükséges járványügyi intézkedésekről Dr. Bíró Zoltán állatorvos MH EK KJSZ ALAPFOGALMAK 1. egészségügyi ellátással
Az egész országot érinti az influenzajárvány Kiugróan magas volt az orvoshoz forduló betegek száma
 Az Országos Epidemiológiai Központ tájékoztatója az influenza figyelőszolgálat adatairól Magyarország 2015. 5. hét Az egész országot érinti az influenzajárvány Kiugróan magas volt az orvoshoz forduló betegek
Az Országos Epidemiológiai Központ tájékoztatója az influenza figyelőszolgálat adatairól Magyarország 2015. 5. hét Az egész országot érinti az influenzajárvány Kiugróan magas volt az orvoshoz forduló betegek
A malacok fontosabb felnevelési betegségei
 A malacok fontosabb felnevelési betegségei Varga János egyetemi tanár az MTA tagja Szent István Egyetem Állatorvos-tudományi Kar Mikrobiológia és Járványtan Tanszék 1581 Budapest, 146 Pf. 22. A szopós
A malacok fontosabb felnevelési betegségei Varga János egyetemi tanár az MTA tagja Szent István Egyetem Állatorvos-tudományi Kar Mikrobiológia és Járványtan Tanszék 1581 Budapest, 146 Pf. 22. A szopós
Marker koncepció (Bovilis BVD)
 Marker koncepció (Bovilis BVD) VI. MSDay Budapest 2013. február 21. Dr. Földi József Mi a marker vagy diva koncepció? Típusai BVDV NS3 (p80) marker vakcinák és tesztek A BVDV NS3 marker elv gyakorlati
Marker koncepció (Bovilis BVD) VI. MSDay Budapest 2013. február 21. Dr. Földi József Mi a marker vagy diva koncepció? Típusai BVDV NS3 (p80) marker vakcinák és tesztek A BVDV NS3 marker elv gyakorlati
Mangalica specifikus DNS alapú módszer kifejlesztés és validálása a MANGFOOD projekt keretében
 Mangalica specifikus DNS alapú módszer kifejlesztés és validálása a MANGFOOD projekt keretében Szántó-Egész Réka 1, Mohr Anita 1, Sipos Rita 1, Dallmann Klára 1, Ujhelyi Gabriella 2, Koppányné Szabó Erika
Mangalica specifikus DNS alapú módszer kifejlesztés és validálása a MANGFOOD projekt keretében Szántó-Egész Réka 1, Mohr Anita 1, Sipos Rita 1, Dallmann Klára 1, Ujhelyi Gabriella 2, Koppányné Szabó Erika
Tájékoztató az influenza figyelőszolgálat adatairól Magyarország hét. Járványosan terjed az influenza
 KÓRHÁZHIGIÉNÉS ÉS JÁRVÁNYÜGYI FELÜGYELETI FŐOSZTÁLY Tájékoztató az influenza figyelőszolgálat adatairól Magyarország 2018. 03. hét Járványosan terjed az influenza A figyelőszolgálatban résztvevő orvosok
KÓRHÁZHIGIÉNÉS ÉS JÁRVÁNYÜGYI FELÜGYELETI FŐOSZTÁLY Tájékoztató az influenza figyelőszolgálat adatairól Magyarország 2018. 03. hét Járványosan terjed az influenza A figyelőszolgálatban résztvevő orvosok
A BRDC KÓROKTANA ÉS TÜNETTANA AETIOLOGY AND CLINICAL SIGNS OF BRDC
 Nemzetközi Szarvasmarha Akadémia Szarvasmarha telepi BRDC menedzsment a gyakorlatban Budapest, 2012. november 30. A BRDC KÓROKTANA ÉS TÜNETTANA AETIOLOGY AND CLINICAL SIGNS OF BRDC Fodor László Szent István
Nemzetközi Szarvasmarha Akadémia Szarvasmarha telepi BRDC menedzsment a gyakorlatban Budapest, 2012. november 30. A BRDC KÓROKTANA ÉS TÜNETTANA AETIOLOGY AND CLINICAL SIGNS OF BRDC Fodor László Szent István
Az Országos Epidemiológiai Központ Tájékoztatója az influenza surveillance adatairól Magyarország hét
 Az Országos Epidemiológiai Központ Tájékoztatója az influenza surveillance adatairól Magyarország 2011. 15. A 15. naptári en 24%-kal csökkent az influenzaszerű megbetegedéssel orvoshoz fordulók száma az
Az Országos Epidemiológiai Központ Tájékoztatója az influenza surveillance adatairól Magyarország 2011. 15. A 15. naptári en 24%-kal csökkent az influenzaszerű megbetegedéssel orvoshoz fordulók száma az
Az Országos Epidemiológiai Központ Tájékoztatója az influenza surveillance adatairól Magyarország hét
 Az Országos Epidemiológiai Központ Tájékoztatója az influenza surveillance adatairól Magyarország 2011. 9. Csökken az influenzaszerű megbetegedések száma A 9. naptári en folytatódott az influenzaszerű
Az Országos Epidemiológiai Központ Tájékoztatója az influenza surveillance adatairól Magyarország 2011. 9. Csökken az influenzaszerű megbetegedések száma A 9. naptári en folytatódott az influenzaszerű
NUKLEINSAVAK. Nukleinsav: az élő szervezetek sejtmagvában és a citoplazmában található, az átöröklésben szerepet játszó, nagy molekulájú anyag
 NUKLEINSAVAK Nukleinsav: az élő szervezetek sejtmagvában és a citoplazmában található, az átöröklésben szerepet játszó, nagy molekulájú anyag RNS = Ribonukleinsav DNS = Dezoxi-ribonukleinsav A nukleinsavak
NUKLEINSAVAK Nukleinsav: az élő szervezetek sejtmagvában és a citoplazmában található, az átöröklésben szerepet játszó, nagy molekulájú anyag RNS = Ribonukleinsav DNS = Dezoxi-ribonukleinsav A nukleinsavak
J.1.sz.táblázat. A nem specifikus és specifikus járványokban megbetegedettek és meghaltak száma 2010-ben. véráramfertőzés
 száma % 1 Nosocomiális járványok 2010. Az ÁNTSZ regionális és kistérségi intézetei az OEK Kórházi Járványügyi Osztályára 2010. évben 185 nosocomiális járványt valamint 1 Salmonella typhi murium, 4 MRSA,
száma % 1 Nosocomiális járványok 2010. Az ÁNTSZ regionális és kistérségi intézetei az OEK Kórházi Járványügyi Osztályára 2010. évben 185 nosocomiális járványt valamint 1 Salmonella typhi murium, 4 MRSA,
Tovább csökkent az influenzaszerű megbetegedések száma
 Az Országos Epidemiológiai Központ tájékoztatója az influenza figyelőszolgálat adatairól Magyarország 2017. 6. hét Tovább csökkent az influenzaszerű megbetegedések száma 2017. február 612. között a figyelőszolgálatban
Az Országos Epidemiológiai Központ tájékoztatója az influenza figyelőszolgálat adatairól Magyarország 2017. 6. hét Tovább csökkent az influenzaszerű megbetegedések száma 2017. február 612. között a figyelőszolgálatban
TDK lehetőségek az MTA TTK Enzimológiai Intézetben
 TDK lehetőségek az MTA TTK Enzimológiai Intézetben Vértessy G. Beáta egyetemi tanár TDK mind 1-3 helyezettek OTDK Pro Scientia különdíj 1 második díj Diákjaink Eredményei Zsűri különdíj 2 első díj OTDK
TDK lehetőségek az MTA TTK Enzimológiai Intézetben Vértessy G. Beáta egyetemi tanár TDK mind 1-3 helyezettek OTDK Pro Scientia különdíj 1 második díj Diákjaink Eredményei Zsűri különdíj 2 első díj OTDK
DNS molekulák elválasztása agaróz gélelektroforézissel és kapilláris elektroforézissel
 DNS molekulák elválasztása agaróz gélelektroforézissel és kapilláris elektroforézissel Gyakorlat helye: BIOMI Kft. Gödöllő, Szent-Györgyi A. u. 4. (Nemzeti Agrárkutatási és Innovációs Központ épülete volt
DNS molekulák elválasztása agaróz gélelektroforézissel és kapilláris elektroforézissel Gyakorlat helye: BIOMI Kft. Gödöllő, Szent-Györgyi A. u. 4. (Nemzeti Agrárkutatási és Innovációs Központ épülete volt
Az egész országban terjed az influenza Kiugróan magas volt az orvoshoz forduló betegek száma
 Az Országos Epidemiológiai Központ tájékoztatója az influenza figyelőszolgálat adatairól Magyarország 2017. 4. hét Az egész országban terjed az influenza Kiugróan magas volt az orvoshoz forduló betegek
Az Országos Epidemiológiai Központ tájékoztatója az influenza figyelőszolgálat adatairól Magyarország 2017. 4. hét Az egész országban terjed az influenza Kiugróan magas volt az orvoshoz forduló betegek
TEMATIKA Biokémia és molekuláris biológia IB kurzus (bb5t1301)
 Biokémia és molekuláris biológia I. kurzus (bb5t1301) Tematika 1 TEMATIKA Biokémia és molekuláris biológia IB kurzus (bb5t1301) 0. Bevezető A (a biokémiáról) (~40 perc: 1. heti előadás) A BIOkémia tárgya
Biokémia és molekuláris biológia I. kurzus (bb5t1301) Tematika 1 TEMATIKA Biokémia és molekuláris biológia IB kurzus (bb5t1301) 0. Bevezető A (a biokémiáról) (~40 perc: 1. heti előadás) A BIOkémia tárgya
A preventív vakcináció lényege :
 Vakcináció Célja: antigénspecifkus immunválasz kiváltása a szervezetben A vakcina egy olyan készítmény, amely fokozza az immunitást egy adott betegséggel szemben (aktiválja az immunrendszert). A preventív
Vakcináció Célja: antigénspecifkus immunválasz kiváltása a szervezetben A vakcina egy olyan készítmény, amely fokozza az immunitást egy adott betegséggel szemben (aktiválja az immunrendszert). A preventív
KERINGŐ EXTRACELLULÁRIS VEZIKULÁK ÁLTAL INDUKÁLT GÉNEXPRESSZIÓS MINTÁZAT VIZSGÁLATA TROPHOBLAST SEJTVONALBAN
 2016. 10. 14. KERINGŐ EXTRACELLULÁRIS VEZIKULÁK ÁLTAL INDUKÁLT GÉNEXPRESSZIÓS MINTÁZAT VIZSGÁLATA TROPHOBLAST SEJTVONALBAN Kovács Árpád Ferenc 1, Pap Erna 1, Fekete Nóra 1, Rigó János 2, Buzás Edit 1,
2016. 10. 14. KERINGŐ EXTRACELLULÁRIS VEZIKULÁK ÁLTAL INDUKÁLT GÉNEXPRESSZIÓS MINTÁZAT VIZSGÁLATA TROPHOBLAST SEJTVONALBAN Kovács Árpád Ferenc 1, Pap Erna 1, Fekete Nóra 1, Rigó János 2, Buzás Edit 1,
Biológiai módszerek alkalmazása környezeti hatások okozta terhelések kimutatására
 Szalma Katalin Biológiai módszerek alkalmazása környezeti hatások okozta terhelések kimutatására Témavezető: Dr. Turai István, OSSKI Budapest, 2010. október 4. Az ionizáló sugárzás sejt kölcsönhatása Antone
Szalma Katalin Biológiai módszerek alkalmazása környezeti hatások okozta terhelések kimutatására Témavezető: Dr. Turai István, OSSKI Budapest, 2010. október 4. Az ionizáló sugárzás sejt kölcsönhatása Antone
A bioinformatika gyökerei
 A bioinformatika gyökerei 1944: Avery a transforming principle a DNS 1952: Hershey és Chase perdöntő bizonyíték: a bakteriofágok szaporodásakor csak a DNS jut be a sejtbe 1953: Watson és Crick a DNS szerkezete
A bioinformatika gyökerei 1944: Avery a transforming principle a DNS 1952: Hershey és Chase perdöntő bizonyíték: a bakteriofágok szaporodásakor csak a DNS jut be a sejtbe 1953: Watson és Crick a DNS szerkezete
Egy 10,3 kb méretű, lineáris, a mitokondriumban lokalizált DNS-plazmidot izoláltunk a
 Egy 10,3 kb méretű, lineáris, a mitokondriumban lokalizált DNS-plazmidot izoláltunk a Fusarium proliferatum (Gibberella intermedia) ITEM 2337-es törzséből, és a plazmidot pfp1- nek neveztük el. Proteináz
Egy 10,3 kb méretű, lineáris, a mitokondriumban lokalizált DNS-plazmidot izoláltunk a Fusarium proliferatum (Gibberella intermedia) ITEM 2337-es törzséből, és a plazmidot pfp1- nek neveztük el. Proteináz
KÓROKOZÓ VÍRUSOK ELŐFORDULÁSA MAGYARORSZÁGI FELSZÍNI- ÉS FÜRDŐVIZEKBEN. Doktori értekezés tézisei. Kern Anita
 KÓROKOZÓ VÍRUSOK ELŐFORDULÁSA MAGYARORSZÁGI FELSZÍNI- ÉS FÜRDŐVIZEKBEN Doktori értekezés tézisei Kern Anita Eötvös Loránd Tudományegyetem, Környezettudományi Doktori Iskola, Környezetbiológia Program,
KÓROKOZÓ VÍRUSOK ELŐFORDULÁSA MAGYARORSZÁGI FELSZÍNI- ÉS FÜRDŐVIZEKBEN Doktori értekezés tézisei Kern Anita Eötvös Loránd Tudományegyetem, Környezettudományi Doktori Iskola, Környezetbiológia Program,
Az ország valamennyi területét érintő influenza-járvány bontakozott ki
 Az Országos Epidemiológiai Központ Tájékoztatója az influenza surveillance adatairól Magyarország 2011. 4. Az ország valamennyi területét érintő influenza-járvány bontakozott ki A figyelőszolgálatra kijelölt
Az Országos Epidemiológiai Központ Tájékoztatója az influenza surveillance adatairól Magyarország 2011. 4. Az ország valamennyi területét érintő influenza-járvány bontakozott ki A figyelőszolgálatra kijelölt
Zoonózisok és élelmiszer eredetű események Európában az EFSA és az ECDC 2012 évi zoonózis jelentése alapján
 Zoonózisok és élelmiszer eredetű események Európában az EFSA és az ECDC 2012 évi zoonózis jelentése alapján Dr. Szeitzné Dr. Szabó Mária Kerekes Kata NÉBIH-ÉKI 2014. április 15. Miről lesz szó? Hogyan
Zoonózisok és élelmiszer eredetű események Európában az EFSA és az ECDC 2012 évi zoonózis jelentése alapján Dr. Szeitzné Dr. Szabó Mária Kerekes Kata NÉBIH-ÉKI 2014. április 15. Miről lesz szó? Hogyan
