SUPPLEMENTAL DATA. Structure and specificity of the Type VI secretion system ClpV-TssC interaction in enteroaggregative Escherichia coli.
|
|
|
- Róbert Fodor
- 4 évvel ezelőtt
- Látták:
Átírás
1 SUPPLEMENTAL DATA Structure and specificity of the Type VI secretion system ClpV-TssC interaction in enteroaggregative Escherichia coli. B. Douzi, Y.R. Brunet, S. Spinelli, V. Lensi, P. Legrand, S. Blangy, A. Kumar, L. Journet, E. Cascales & C. Cambillau
2 Supplemental Figure S1. Clustal W alignment of the EAEC and amino-acid sequences. The two full-length protein sequences were aligned using ClustalW on the Pôle Rhone-Alpes de BioInformatique (PRABI) server ( Identical residues are shown in red and indicated by a star. Similar residues are shown in blue or green. The red arrow indicates the position of the ClpV N-terminal domain C-terminus used in all the constructs VNNMENSAALLRRLNHYCARALEGAASLCQTRAHAEITPEHWLLKLLEQGEGDLTVLGRRYDWDMDAIWQSLLGWLDNQPRSVRSRPQLAQSLNALLKQA --MSIYLKPIINKLTPESRNTLDSAINYAISRSHHEVDCLHLLWKLLQEHKYIAEVLYEQSLFNPEWVLNAIESELIRINTVPQSSPVFSESMQTLLEKT.::.:*...:*:.*.. :*:* *: * * ***:: : **.: :: : : :::. *. :* * :::*:::**::: WMVASLQGE-EHIRSVHLLGALTENPHLVRCDGLWPLLTLSQSQLQRLSPLLDAQSDECPETLQDAEPVLPQGDSVTFIGRPVGADTAGIPSGDLPPVLQ WIHASTKWQIDHIDIPVFLSTMIN--FR---DSIFPLN VSDALCCDMDVAEELLISFSDEAEHSASH----RPT DRSS-----H *: ** : : :** :*.:: :. *.::** :* *.: *. * *.. :. * **... : GALDKFTRDITASAREGKIDPVSGRDTEIRQMVDILSRRRKNNPILVGDPGVGKTALVEGLALRIVEGNVPESLRPVTLRTLDLGLLQAGAGVKGEFEQR EYLSKYTENLSLRAETGKLDPVTGREKEVRQLIDILLRRRQNNPILTGEPGVGKSSIVEGLALQIASGRVPDVLKNVRIHALDMGALLAGASVRGEFENR *.*:*.::: *. **:***:**:.*:**::*** ***:*****.*:*****:::******:*..*.**: *: * :::**:* * ***.*:****:* LKNVIDAVQLSPAPVLLFIDEAHTLIGAGNQAGGADAANLLKPALARGELRTIAATTWSEYKQYLERDAALERRFQMVKVDEPDDETACLMLRSLKSRYA LKSLLTELNSLDGTAILFIDEAHSLIGAGGLPGQTDAANLLKPALARGELRIIAATTWGEYKKYFEKDGALARRFQIVKVAEPNQDVTAEMLRSLLPMME **.:: ::...:*******:*****..* :**************** ******.***:*:*:*.** ****:*** **:::.:. *****.
3 EHHNVHITDEAVRAAVTLSRRYLTERQLPDKAVDLLDTAAARVRMSLDTVPEQLTRIRSQLASLGMEKQALLEDIAVGHQNHGERLSAIEQEENVLMTAR KHHNVSIREEAITATVHLSDRYLHGRRQPDKSVSLLDTACSRVAVSQSTSPDAIQDVEASLVRYQGELALLTQ-----ERSDVLRQEMLANKIAQLEEEL :**** * :**: *:* ** *** *: ***:*.*****.:** :*.* *: : :.:.*. * * :.:.. *. : :: * DDLEQQYARECELTGELLESRSDISRQSETHHLQQALHDIQQNQPLLSVDVDVRTVAGVVADWTGVPLSSLMKDEQTELLHLEKDIGRRVVGQDVALESI EQLKSAWRHQSELVAKIQSS-DDIS--SKNMYRKELESAYKKDSPMVFECVDKNCVADVVSGWTGVPLGVCLDGEQQKASGLLRCLEQRVLGQRYAMSAI ::*:. : ::.**..::.*.*** *:. : :: :::.*:: **. **.**:.******. :..** : * : : :**:** *:.:* AQRLRAAKTGLTSGNGPQEVFLLVGPSGVGKTETALALADVMYGGEKSLITINLSEYQEPHTVSQLKGSPPGYVGYGQGGILTEAVRKRPYSVVLLDEVE ASQVLICRADLKDPVKPDGVFLLAGPSGTGKTETARALAEFVYGDENKLITINMTEFQEAHTVSTLKGAPPGYVGFGQGGTLTERVSHNPYSVILLDEIE *.::.::.*.. *: ****.****.****** ***:.:**.*:.*****::*:**.**** ***:******:**** *** * :.****:****:* KAHRDVLNLFYQVFDRGFMRDGEGREIDFRNTVILMTSNLGSDLLMQQLSEKPETTESELHELIRPLLRDHFQPALLARFQTVIYRPLTPSAMRTIVEMK KAHPDVLEFFFQIFDSGIIEDAEGKMVSFRDCLIIMTSNFASENITNIWNDG-ETNRDRIKEMLLPLFNEHFGAAFMGRTNLIPFTPLHSKTLRDIVLIK *** ***::*:*:** *::.*.**: :.**: :*:****:.*: : :.: **...::*:: **:.:**.*::.* : : : **..::* ** :*! LAQVCERLHCHYG--LSTSVDERVYDALTSACLLPDTGARNVESLLNQQLLPVLSRQLLSHMAAKQKPQALALAWSDEDGMVIELRQECAL- IDKICQRFEQASGQMYKIEYNDSLIDWITHHCQCDKSGARDIDSVLNSTVLPVLAR-YLTDSEDNRTPKKIRISVR-KNNITLRSSQFATRN : ::*:*:. *.. :: : * :* *.:***:::*:**. :****:* *:. ::.*: : :: ::.:.:. *.:
4 Supplemental Table S1. Strains, plasmids and oligonucleotides used in this study. Strains Strains Description and genotype Source E. coli K-12 DH5α F-, Δ(argF-lac)U169, phoa, supe44, Δ(lacZ)M15, rela, enda, thi, hsdr New England Biolabs W3110 F-, lambda- IN(rrnD-rrnE)1 rph-1 Laboratory collection BTH101 F-, cya-99, arad139, gale15, galk16, rpsl1 (Str R ), hsdr2, mcra1, mcrb1. Karimova et al., 2005 BL21(DE3) plys F-, minif lysy laci q (Cm R ) / fhua2 lacz::t7 gene1 [lon] ompt gal sula11 R(mcr-73::miniTn10--Tet S )2 [dcm] R(zgb-210::Tn10--Tet S ) enda1 Δ(mcrC-mrr) 114::IS10 New England Biolabs Enteroaggregative E. coli 17-2 WT enteroaggregative Escherichia coli Arlette Darfeuille-Michaud 17-2ΔclpV deleted of the clpv gene of the sci1 T6SS gene cluster (EC042_4530) Brunet et al., ΔclpV deleted of the clpv gene of the sci2 T6SS gene cluster (EC042_4577) M.S. Aschtgen 17-2ΔtssC deleted of the tssc gene of the sci1 T6SS gene cluster (EC042_4525) Brunet et al., 2015 Plasmids Vectors Description Source Expression vectors pua66-rrnb P rrnb ::gfpmut2 transcriptional fusion in pua66 Zaslaver et al., 2006 pask-iba37 cloning vector, Ptet, Amp R IBA Technology piba37- FLAG sci1 clpv, C-terminal FLAG tag cloned into pask-iba37 This study
5 piba37--nt FLAG sci1 clpv N-terminal fragment (residues 1-162), C-terminal FLAG tag cloned into pask-iba37(+) This study piba37--e24k FLAG clpv1 Glu24-to-Lys substitution into piba37- FLAG This study piba37--r87e FLAG clpv1 Arg87-to-Glu substitution into piba37- FLAG This study piba37--e24k-r87e FLAG clpv1 Glu24-to-Lys and Arg87-to-Glu substitutions into piba37- FLAG This study piba37- FLAG sci2 clpv, C-terminal FLAG tag cloned into pask-iba37 This study piba37--nt FLAG sci2 clpv N-terminal fragment (residues 1-147), C-terminal FLAG tag cloned into pask-iba37(+) This study pbad33 cloning vector, pacyc184 origin, Para, arac Cm R Guzman et al., 1995 pbad33-tssc1 VSV-G sci1 tssc, C-terminal VSV-G tag cloned into pbad33 This study pbad33-tssc1-e31k-k32e VSV-G tssc1 Glu31-to-Lys and Lys32-to-Glu substitutions into pbad33-tssc1 VSV-G This study pbad33-tssc1 h VSV-G Deletion of amino-acids of sci1 tssc into pbad33-tssc1 VSV-G This study pbad33-tssc2 VSV-G sci2 tssc, C-terminal VSV-G tag cloned into pbad33 This study pbad33-tssc2 h VSV-G Deletion of amino-acids 2-15 of sci2 tssc into pbad33-tssc2 VSV-G This study Bacterial Two-Hybrid vectors pt18-flag Bacterial Two Hybrid vector, ColE1 origin, Plac, T18 fragment of Bordetella pertussis CyaA, Amp R Battesti & Bouveret, 2008 pt18-pal Soluble region of E. coli K-12 Pal cloned downstream T18 in pt18-flag Battesti & Bouveret, 2008 p-t18 sci1 clpv cloned upstream T18 into pt18-flag This study pt18- sci1 clpv cloned downstream T18 into pt18-flag This study pt18--e24k clpv1 Glu24-to-Lys substitution into pt18- This study pt18--r87e clpv1 Arg87-to-Glu substitution into pt18- This study pt18--e24k-r87e clpv1 Glu24-to-Lys and Arg87-to-Glu substitutions into pt18- This study pt18- sci2 clpv cloned downstream T18 into pt18-flag This study pt25-flag Bacterial Two Hybrid vector, p15a origin, Plac, T25 fragment of Bordetella pertussis CyaA, Kan R Battesti & Bouveret, 2008 ptolb-t25 E. coli K-12 tolb cloned upstream T25 in pt25-flag Battesti & Bouveret, 2008 p-t25 sci1 clpv cloned upstream T25 into pt25-flag This study pt25- sci1 clpv cloned downstream T25 into pt25-flag This study ptssc1-t25 sci1 tssc cloned upstream T25 into pt25-flag Zoued et al., 2013 ptssc1-e31k-k32e-t25 tssc1 Glu31-to-Lys and Lys32-to-Glu substitutions into ptssc1-t25 This study ptssc2-t25 sci2 tssc (EC042_4564) cloned upstream T25 into pt25-flag This study All others BACTH constructs have been described in Zoued et al., 2013.
6 Oligonucleotides Name Destination Sequence (5' to 3 ) For plasmid construction a,b,c 5-pIBA37- FLAG piba- FLAG / piba--nt GACAAAAATCTAGAAATAATTTTGTTTAACTTTAAGAAGGAGATATACAAATGAATAACA TGGAAAATTCGGCAGCCCTGTTACG 3-pIBA37- FLAG piba- FLAG GATGGTGATGGTGATGCGATCCTCTGCTAGCTTATTTATCATCGTCGTCTTTATAATCTAACGC GCATTCCTGCCGCAGCTC 3- piba37--nter piba--nt GATGGTGATGGTGATGCGATCCTCTGCTAGCTTATTTATCATCGTCGTCTTTATAATCTAACGT CTCCGGACACTCATCAGACTG 5-pIBA37- FLAG piba- FLAG / piba--nt GACAAAAATCTAGAAATAATTTTGTTTAACTTTAAGAAGGAGATATACAAATGGTGAGTA TCTATCTGAAACCAATTATTAATAAATTAACTCCAGAAAG 3- piba37- FLAG piba- GATGGTGATGGTGATGCGATCCTCTGCTAGCTTATTTATCATCGTCGTCTTTATAATCATTACG AGTTGCAAATTGCGAACTACGTAATGTAATG 3-pIBA37--Nter piba--nt GATGGTGATGGTGATGCGATCCTCTGCTAGCTTATTTATCATCGTCGTCTTTATAATCAAGTA ACTCCTCAGCTACATCCATATCACAACAT 5-pBAD33-TssC1 VSV-G pbad33-tssc1 VSV-G CTCTCTACTGTTTCTCCATACCCGTTTTTTTGGGCTAGCAGGAGGTATTACACCATGCTGATG TCTGTACAGAAAGAAAAGAACGTTG 3-pBAD33-TssC1 VSV-G pbad33-tssc1 VSV-G GGTCGACTCTAGAGGATCCCCGGGTACCTTATTTTCCTAATCTATTCATTTCAATATCTGTATAC GCTTTTGCCTTCGGCATCTGCG 5-pBAD33-TssC2 VSV-G pbad33-tssc2 VSV-G CTCTCTACTGTTTCTCCATACCCGTTTTTTTGGGCTAGCAGGAGGTATTACACCATGACAGTT GCATCAACATTAGG 3-pBAD33-TssC2 VSV-G pbad33-tssc2 VSV-G GGTCGACTCTAGAGGATCCCCGGGTACCTTATTTTCCTAATCTATTCATTTCAATATCTGTATAA CTTACACTTGATGGTAAATCAGC T25T18C p-t18 / p-t25 CGGATAACAATTTCACACAGGAAACAGCTATGACCATGAATAACATGGAAAATTCGGCAG CCCT T18C p-t18 CCTCGCTGGCGGCTAAGCTTGGCGTAATTAACGCGCATTCCTGCCGCAG T25C p-t25 GTTTGCGTAACCAGCCTGATGCGATTGCTGTAACGCGCATTCCTGCCGCAG T25N pt25- GGCGGGCTGCAGATTATAAAGATGACGATGACAAGAATAACATGGAAAATTCGGCAGCCC T18N !!!! pt18- CGCCACTGCAGGGATTATAAAGATGACGATGACAAGAATAACATGGAAAATTCGGCAGCC CT T25T18N pt18- / pt25- CGAGGTCGACGGTATCGATAAGCTTGATATCGAATTCTAGTTATAACGCGCATTCCTGCCG CAG T25T18C-5-TssC2 ptssc2-t25 CGGATAACAATTTCACACAGGAAACAGCTATGACCATGACAGTTGCATCAACATTAGG
7 T25C-3-TssC2 pt25-tssc2 GTTTGCGTAACCAGCCTGATGCGATTGCTGACTTACACTTGATGGTAAATCAGC 5- petg--nter petg20a--nt GGGGACAAGTTTGTACAAAAAAGCAGGCTTAGAAAACCTGTACTTCCAGGGTGTGAATAA CATGGAAAATTC 3- petg--nter petg20a--nt GGACCACTTTGTACAAGAAAGCTGGGTCTTATTATAACGTCTCCGGACACTCA For site-directed mutagenesis d,e,f A-C1- h pbad33-tssc1 VSV-G GTGGTATCTGAAGCGCATGCCGGCAGTCCGGTATCTGCCCTGAGTGCAC A-C1- h pbad33-tssc1 VSV-G GTGCACTCAGGGCAGATACCGGACTGCCGGCATGCGCTTCAGATACCAC A-C2- h pbad33-tssc2 VSV-G AGCAGGAGGTATTACACCATGACAGATGATTGTCTTGAAGAG A-C2- h pbad33-tssc2 VSV-G CTCTTCAAGACAATCATCTGTCATGGTGTAATACCTCCTGCT A--E24K piba37- FLAG and pt18- CTGTGCCCGTGCACTGAAAGGCGCAGCCTCCCTTTG B--E24K piba37- FLAG and pt18- CAAAGGGAGGCTGCGCCTTTCAGTGCACGGGCACAG A--R87E piba37- FLAG and pt18- CCGTAGCGTACGCAGTGAACCGCAGCTTGCGCAGTC B--R87E piba37- FLAG and pt18- GACTGCGCAAGCTGCGGTTCACTGCGTACGCTACGG A-TssC1-E31K-K32E pbad33-tssc1 VSV-G and ptssc1-t25 GTATATGCTTCCCTGTTTAAAGAAATTAACCTGAGTCCGG B-TssC1-E31K-K32E pbad33-tssc1 VSV-G and ptssc1-t25 CCGGACTCAGGTTAATTTCTTTAAACAGGGAAGCATATAC a Sequence annealing on the target plasmid underlined. b FLAG or VSV-G epitope coding sequence italicized. c Additional Shine Dalgarno sequence italicized underlined. d Codons upstream and downstream the deletion italicized. e Nucleotide substitutions in bold. d Mutagenized codons underlined.
8 Supplemental Table S2. Data collection and refinement statistics DATA COLLECTION Soleil Proxima 1 Soleil Proxima 1 PDB 4HH5 4HH6 Proteins -Nt -Nt/TssC1-pept Space group, cell (Å, ) P , 40.9, 58.7, 65.6 P , 40.9, 46.85, 75.5 Resolution limits a (Å) ( ) ( ) Rmeas a (%) 5.2 (38) 8.6 (72.3) Nr. of observations a,b (9916) (1739) Nr. unique reflections a,b (1405 ) 5360 (388) Mean((I)/sd(I)) a 27.1 (5.5) 13.1 (2.2) Completeness a (%) 96.1 (92.1) 99.6 (98.5) Multiplicity a REFINEMENT Resolution a (Å) ( ) ( ) Nr of reflections a 10703( 2770) 5359 (1358) Atoms : protein, ions, water 1275 / 1 / / 27 Nr test set reflections a R work /R free (%) 0.186/0.205 (0.181/0.234) 0.244/0.263 (28.1/32.1) r.m.s.d.bonds (Å) / angles ( ) / / 1.36 B-wilson / B-average (Å 2 ) / / 55 Ramachandran: preferred/allowed (%) 97.5 / / 6.0 / 2.4 a numbers into parenthesis refer to the highest resolution bin. b Friedel pairs not merged
9 Supplemental Table S3. Interaction between the -N-terminal domain and the TssC1 peptide. (A) Analysis of the interaction surface. (B) Residues of TssC1 peptide interacting with the -N-terminal domain. (C) Residues of the -N-terminal domain interacting with the TssC1 peptide. ASA : accessible surface area. BSA : buried surface area. Analysis performed with PISA.! A! B" Peptide 1 ClpV-Nt Interface Å % % Total surface Å Peptide 1 ASA BSA ASP VAL TYR SER LEU PHE GLU LYS ILE ASN LEU ! C" -Nt ASA BSA SER ALA LEU ARG LEU HIS ALA LEU GLU ALA SER GLN ILE ARG PRO GLN LEU
Supplementary Table 1. Cystometric parameters in sham-operated wild type and Trpv4 -/- rats during saline infusion and
 WT sham Trpv4 -/- sham Saline 10µM GSK1016709A P value Saline 10µM GSK1016709A P value Number 10 10 8 8 Intercontractile interval (sec) 143 (102 155) 98.4 (71.4 148) 0.01 96 (92 121) 109 (95 123) 0.3 Voided
WT sham Trpv4 -/- sham Saline 10µM GSK1016709A P value Saline 10µM GSK1016709A P value Number 10 10 8 8 Intercontractile interval (sec) 143 (102 155) 98.4 (71.4 148) 0.01 96 (92 121) 109 (95 123) 0.3 Voided
Seven gene deletions in seven days: Fast generation of Escherichia coli strains tolerant to acetate and osmotic stress Supplementary Figure S1.
 Seven gene deletions in seven days: Fast generation of Escherichia coli strains tolerant to acetate and osmotic stress Sheila I. Jensen 1*, Rebecca M. Lennen 1, Markus J. Herrgård 1, Alex T. Nielsen 1*
Seven gene deletions in seven days: Fast generation of Escherichia coli strains tolerant to acetate and osmotic stress Sheila I. Jensen 1*, Rebecca M. Lennen 1, Markus J. Herrgård 1, Alex T. Nielsen 1*
Nan Wang, Qingming Dong, Jingjing Li, Rohit K. Jangra, Meiyun Fan, Allan R. Brasier, Stanley M. Lemon, Lawrence M. Pfeffer, Kui Li
 Supplemental Material IRF3-dependent and NF- B-independent viral induction of the zinc-finger antiviral protein Nan Wang, Qingming Dong, Jingjing Li, Rohit K. Jangra, Meiyun Fan, Allan R. Brasier, Stanley
Supplemental Material IRF3-dependent and NF- B-independent viral induction of the zinc-finger antiviral protein Nan Wang, Qingming Dong, Jingjing Li, Rohit K. Jangra, Meiyun Fan, Allan R. Brasier, Stanley
Supporting Information
 Supporting Information Plasmid Construction DNA cloning was carried out according to standard protocols. The sequence of all plasmids was confirmed by DNA sequencing and amplified using the E. coli strain
Supporting Information Plasmid Construction DNA cloning was carried out according to standard protocols. The sequence of all plasmids was confirmed by DNA sequencing and amplified using the E. coli strain
Modular Optimization of Hemicellulose-utilizing Pathway in. Corynebacterium glutamicum for Consolidated Bioprocessing of
 [Supplementary materials] Modular Optimization of Hemicellulose-utilizing Pathway in Corynebacterium glutamicum for Consolidated Bioprocessing of Hemicellulosic Biomass Sung Sun Yim 1, Jae Woong Choi 1,
[Supplementary materials] Modular Optimization of Hemicellulose-utilizing Pathway in Corynebacterium glutamicum for Consolidated Bioprocessing of Hemicellulosic Biomass Sung Sun Yim 1, Jae Woong Choi 1,
Manuscript Title: Identification of a thermostable fungal lytic polysaccharide monooxygenase and
 1 2 3 4 5 Journal name: Applied Microbiology and Biotechnology Manuscript Title: Identification of a thermostable fungal lytic polysaccharide monooxygenase and evaluation of its effect on lignocellulosic
1 2 3 4 5 Journal name: Applied Microbiology and Biotechnology Manuscript Title: Identification of a thermostable fungal lytic polysaccharide monooxygenase and evaluation of its effect on lignocellulosic
Supplemental Information. Investigation of Penicillin Binding Protein. (PBP)-like Peptide Cyclase and Hydrolase
 Cell Chemical Biology, Volume upplemental Information Investigation of Penicillin Binding Protein (PBP)-like Peptide Cyclase and ydrolase in urugamide on-ribosomal Peptide Biosynthesis Yongjun Zhou, Xiao
Cell Chemical Biology, Volume upplemental Information Investigation of Penicillin Binding Protein (PBP)-like Peptide Cyclase and ydrolase in urugamide on-ribosomal Peptide Biosynthesis Yongjun Zhou, Xiao
A cell-based screening system for RNA Polymerase I inhibitors
 Electronic Supplementary Material (ESI) for MedChemComm. This journal is The Royal Society of Chemistry 2019 Supporting Information A cell-based screening system for RNA Polymerase I inhibitors Xiao Tan,
Electronic Supplementary Material (ESI) for MedChemComm. This journal is The Royal Society of Chemistry 2019 Supporting Information A cell-based screening system for RNA Polymerase I inhibitors Xiao Tan,
Supplementary materials to: Whole-mount single molecule FISH method for zebrafish embryo
 Supplementary materials to: Whole-mount single molecule FISH method for zebrafish embryo Yuma Oka and Thomas N. Sato Supplementary Figure S1. Whole-mount smfish with and without the methanol pretreatment.
Supplementary materials to: Whole-mount single molecule FISH method for zebrafish embryo Yuma Oka and Thomas N. Sato Supplementary Figure S1. Whole-mount smfish with and without the methanol pretreatment.
Suppl. Materials. Polyhydroxyalkanoate (PHA) Granules Have no Phospholipids. Germany
 Suppl. Materials Polyhydroxyalkanoate (PHA) Granules Have no Phospholipids Stephanie Bresan 1, Anna Sznajder 1, Waldemar Hauf 2, Karl Forchhammer 2, Daniel Pfeiffer 3 and Dieter Jendrossek 1 1 Institute
Suppl. Materials Polyhydroxyalkanoate (PHA) Granules Have no Phospholipids Stephanie Bresan 1, Anna Sznajder 1, Waldemar Hauf 2, Karl Forchhammer 2, Daniel Pfeiffer 3 and Dieter Jendrossek 1 1 Institute
A fehérjék hierarchikus szerkezete
 Fehérjék felosztása A fehérjék hierarchikus szerkezete Smeller László Semmelweis Egyetem Biofizikai és Sugárbiológiai Intézet Biológiai funkció alapján Enzimek (pl.: tripszin, citokróm-c ) Transzportfehérjék
Fehérjék felosztása A fehérjék hierarchikus szerkezete Smeller László Semmelweis Egyetem Biofizikai és Sugárbiológiai Intézet Biológiai funkció alapján Enzimek (pl.: tripszin, citokróm-c ) Transzportfehérjék
Markerless Escherichia coli rrn Deletion Strains for Genetic Determination of Ribosomal Binding Sites
 Markerless Escherichia coli rrn Deletion Strains for Genetic Determination of Ribosomal Binding Sites Selwyn Quan *, Ole Skovgaard, Robert E. McLaughlin, Ed T. Buurman,1, and Catherine L. Squires * Department
Markerless Escherichia coli rrn Deletion Strains for Genetic Determination of Ribosomal Binding Sites Selwyn Quan *, Ole Skovgaard, Robert E. McLaughlin, Ed T. Buurman,1, and Catherine L. Squires * Department
kpis(ppk20) kpis(ppk46)
 Table S1. C. elegans strains used in this study Strains carrying transgenes Strain Transgene Genotype Reference IT213 tcer-1 prom:tcer-1orf:gfp:tcer-1 3'utr unc-119(ed3) III; kpis(ppk6) This study IT283
Table S1. C. elegans strains used in this study Strains carrying transgenes Strain Transgene Genotype Reference IT213 tcer-1 prom:tcer-1orf:gfp:tcer-1 3'utr unc-119(ed3) III; kpis(ppk6) This study IT283
Expression analysis of PIN genes in root tips and nodules of Lotus japonicus
 Article Expression analysis of PIN genes in root tips and nodules of Lotus japonicus Izabela Sańko-Sawczenko 1, Dominika Dmitruk 1, Barbara Łotocka 1, Elżbieta Różańska 1 and Weronika Czarnocka 1, * 1
Article Expression analysis of PIN genes in root tips and nodules of Lotus japonicus Izabela Sańko-Sawczenko 1, Dominika Dmitruk 1, Barbara Łotocka 1, Elżbieta Różańska 1 and Weronika Czarnocka 1, * 1
Supporting Information
 Supporting Information Paired design of dcas9 as a systematic platform for the detection of featured nucleic acid sequences in pathogenic strains Yihao Zhang 1,2,8, Long Qian 4,8, Weijia Wei 1,3,7,8, Yu
Supporting Information Paired design of dcas9 as a systematic platform for the detection of featured nucleic acid sequences in pathogenic strains Yihao Zhang 1,2,8, Long Qian 4,8, Weijia Wei 1,3,7,8, Yu
CLUSTALW Multiple Sequence Alignment
 Version 3.2 CLUSTALW Multiple Sequence Alignment Selected Sequences) FETA_GORGO FETA_HORSE FETA_HUMAN FETA_MOUSE FETA_PANTR FETA_RAT Import Alignments) Return Help Report Bugs Fasta label *) Workbench
Version 3.2 CLUSTALW Multiple Sequence Alignment Selected Sequences) FETA_GORGO FETA_HORSE FETA_HUMAN FETA_MOUSE FETA_PANTR FETA_RAT Import Alignments) Return Help Report Bugs Fasta label *) Workbench
Supplemental Table S1. Overview of MYB transcription factor genes analyzed for expression in red and pink tomato fruit.
 Supplemental Table S1. Overview of MYB transcription factor genes analyzed for expression in red and pink tomato fruit. MYB Primer pairs TC AtMYB Forward Reverse TC199266 MYB12 AGGCTCTTGGAGGTCGTTACC CAACTCTTTCCGCATCTCAATAATC
Supplemental Table S1. Overview of MYB transcription factor genes analyzed for expression in red and pink tomato fruit. MYB Primer pairs TC AtMYB Forward Reverse TC199266 MYB12 AGGCTCTTGGAGGTCGTTACC CAACTCTTTCCGCATCTCAATAATC
University of Bristol - Explore Bristol Research
 Al-Salihi, S. A. A., Scott, T. A., Bailey, A. M., & Foster, G. D. (2017). Improved vectors for Agrobacterium mediated genetic manipulation of Hypholoma spp. and other homobasidiomycetes. Journal of Microbiological
Al-Salihi, S. A. A., Scott, T. A., Bailey, A. M., & Foster, G. D. (2017). Improved vectors for Agrobacterium mediated genetic manipulation of Hypholoma spp. and other homobasidiomycetes. Journal of Microbiological
pjnc-pgluc Codon-optimized Gaussia luciferase (pgluc) Vector backbone: JM83 pjnc-pgluc is resistant to ampicillin and neomycin High or low copy:
 pjnc-pgluc Gene/Insert name: Codon-optimized Gaussia luciferase (pgluc) Vector backbone: pcmv-jnc Vector type: Mammalian cells Backbone size w/o insert (bp): 5375 Bacterial resistance: Ampicillin and neomycin
pjnc-pgluc Gene/Insert name: Codon-optimized Gaussia luciferase (pgluc) Vector backbone: pcmv-jnc Vector type: Mammalian cells Backbone size w/o insert (bp): 5375 Bacterial resistance: Ampicillin and neomycin
Bioinformatika 2 5.. előad
 5.. előad adás Prof. Poppe László BME Szerves Kémia és Technológia Tsz. Bioinformatika proteomika Előadás és gyakorlat 2009. 03. 21. Fehérje térszerkezet t megjelenítése A fehérjék meglehetősen összetett
5.. előad adás Prof. Poppe László BME Szerves Kémia és Technológia Tsz. Bioinformatika proteomika Előadás és gyakorlat 2009. 03. 21. Fehérje térszerkezet t megjelenítése A fehérjék meglehetősen összetett
The role of aristolochene synthase in diphosphate activation
 Supporting information for The role of aristolochene synthase in diphosphate activation Juan A. Faraldos, Verónica González and Rudolf K. Allemann * School of Chemistry, Cardiff University, Main Building,
Supporting information for The role of aristolochene synthase in diphosphate activation Juan A. Faraldos, Verónica González and Rudolf K. Allemann * School of Chemistry, Cardiff University, Main Building,
Trinucleotide Repeat Diseases: CRISPR Cas9 PacBio no PCR Sequencing MFMER slide-1
 Trinucleotide Repeat Diseases: CRISPR Cas9 PacBio no PCR Sequencing 2015 MFMER slide-1 Fuch s Eye Disease TCF 4 gene Fuchs occurs in about 4% of the US population. Leads to deteriorating vision without
Trinucleotide Repeat Diseases: CRISPR Cas9 PacBio no PCR Sequencing 2015 MFMER slide-1 Fuch s Eye Disease TCF 4 gene Fuchs occurs in about 4% of the US population. Leads to deteriorating vision without
Supplementary Figure 1
 Supplementary Figure 1 Plot of delta-afe of sequence variants detected between resistant and susceptible pool over the genome sequence of the WB42 line of B. vulgaris ssp. maritima. The delta-afe values
Supplementary Figure 1 Plot of delta-afe of sequence variants detected between resistant and susceptible pool over the genome sequence of the WB42 line of B. vulgaris ssp. maritima. The delta-afe values
Forensic SNP Genotyping using Nanopore MinION Sequencing
 Forensic SNP Genotyping using Nanopore MinION Sequencing AUTHORS Senne Cornelis 1, Yannick Gansemans 1, Lieselot Deleye 1, Dieter Deforce 1,#,Filip Van Nieuwerburgh 1,#,* 1 Laboratory of Pharmaceutical
Forensic SNP Genotyping using Nanopore MinION Sequencing AUTHORS Senne Cornelis 1, Yannick Gansemans 1, Lieselot Deleye 1, Dieter Deforce 1,#,Filip Van Nieuwerburgh 1,#,* 1 Laboratory of Pharmaceutical
Supporting Information
 Supporting Information Cell-free GFP simulations Cell-free simulations of degfp production were consistent with experimental measurements (Fig. S1). Dual emmission GFP was produced under a P70a promoter
Supporting Information Cell-free GFP simulations Cell-free simulations of degfp production were consistent with experimental measurements (Fig. S1). Dual emmission GFP was produced under a P70a promoter
PIAL1 (At1g08910) ORF sequence
 PIAL1 (At1g08910) ORF sequence ATGGTTATTCCGGCGACTTCTAGGTTTGGGTTTCGTGCTGAATTCAACACCAAGGAGTTTCAAGCTTCCTGCATCTCTCT! CGCTAATGAAATCGATGCGGCTATTGGGAGAAATGAAGTTCCAGGGAATATTCAAGAGCTCGCTTTAATCCTCAATAATG! TGTGCCGACGTAAATGTGATGATTATCAAACCAGGGCGGTGGTAATGGCGCTGATGATCTCAGTCAAGAGTGCTTGTCAG!
PIAL1 (At1g08910) ORF sequence ATGGTTATTCCGGCGACTTCTAGGTTTGGGTTTCGTGCTGAATTCAACACCAAGGAGTTTCAAGCTTCCTGCATCTCTCT! CGCTAATGAAATCGATGCGGCTATTGGGAGAAATGAAGTTCCAGGGAATATTCAAGAGCTCGCTTTAATCCTCAATAATG! TGTGCCGACGTAAATGTGATGATTATCAAACCAGGGCGGTGGTAATGGCGCTGATGATCTCAGTCAAGAGTGCTTGTCAG!
Excel vagy Given-When-Then? Vagy mindkettő?
 TESZT & TEA BUDAPEST AGILE MEETUP Pénzügyi számítások automatizált agilis tesztelése: Excel vagy Given-When-Then? Vagy mindkettő? NAGY GÁSPÁR TechTalk developer coach Budapest, 2014 február 6. SpecFlow
TESZT & TEA BUDAPEST AGILE MEETUP Pénzügyi számítások automatizált agilis tesztelése: Excel vagy Given-When-Then? Vagy mindkettő? NAGY GÁSPÁR TechTalk developer coach Budapest, 2014 február 6. SpecFlow
University of Bristol - Explore Bristol Research
 Tan, B. G., Wellesley, F. C., Savery, N. J., & Szczelkun, M. D. (2016). Length heterogeneity at conserved sequence block 2 in human mitochondrial DNA acts as a rheostat for RNA polymerase POLRMT activity.
Tan, B. G., Wellesley, F. C., Savery, N. J., & Szczelkun, M. D. (2016). Length heterogeneity at conserved sequence block 2 in human mitochondrial DNA acts as a rheostat for RNA polymerase POLRMT activity.
Mapping Sequencing Reads to a Reference Genome
 Mapping Sequencing Reads to a Reference Genome High Throughput Sequencing RN Example applications: Sequencing a genome (DN) Sequencing a transcriptome and gene expression studies (RN) ChIP (chromatin immunoprecipitation)
Mapping Sequencing Reads to a Reference Genome High Throughput Sequencing RN Example applications: Sequencing a genome (DN) Sequencing a transcriptome and gene expression studies (RN) ChIP (chromatin immunoprecipitation)
pleics-06 Amp pleics-13 Amp/Zeo N-His 10/ 3 x
 Protease Sequence primers Vector Name AR Tag/Flag cleavage Tag and linker pleics-01 Amp N-His 6 TEV Extra SM His6-SSGVDLGT-TEV site-1 T7 Promoter pleics-01-seq-r pleics-02 Amp N-GST TEV Extra SM GST-SS-TEV
Protease Sequence primers Vector Name AR Tag/Flag cleavage Tag and linker pleics-01 Amp N-His 6 TEV Extra SM His6-SSGVDLGT-TEV site-1 T7 Promoter pleics-01-seq-r pleics-02 Amp N-GST TEV Extra SM GST-SS-TEV
A fehérjék hierarchikus szerkezete
 Fehérjék felosztása A fehérjék hierarchikus szerkezete Smeller László Semmelweis Egyetem Biofizikai és Sugárbiológiai Intézet Biológiai funkció alapján Enzimek (pl.: tripszin, citokróm-c ) Transzportfehérjék
Fehérjék felosztása A fehérjék hierarchikus szerkezete Smeller László Semmelweis Egyetem Biofizikai és Sugárbiológiai Intézet Biológiai funkció alapján Enzimek (pl.: tripszin, citokróm-c ) Transzportfehérjék
tccattaattcgacagaccagagttaaataatccttgtatgccattgtgatcacatctacagttcagattttgtatttca
 Figure Legends for Supplementary Materials: Fig. 1. Nucleotide sequence of the zebrafish lumican (zlum) gene. Exons are indicated by uppercase letters, and introns are indicated by lowercase letters. The
Figure Legends for Supplementary Materials: Fig. 1. Nucleotide sequence of the zebrafish lumican (zlum) gene. Exons are indicated by uppercase letters, and introns are indicated by lowercase letters. The
MELLÉKLET / ANNEX. EU MEGFELELŐSÉGI NYILATKOZAT-hoz for EU DECLARATION OF CONFORMITY
 TRACON BUDAPEST KFT. TRACON BUDAPEST LTD. 2120 DUNAKESZI, PALLAG U. 23. TEL.: (27) 540-000, FAX: (27) 540-005 WWW.TRACON.HU EU MEGFELELŐSÉGI NYILATKOZAT, a 79/1997. (XII. 31.) IKIM számú és a 374/2012.
TRACON BUDAPEST KFT. TRACON BUDAPEST LTD. 2120 DUNAKESZI, PALLAG U. 23. TEL.: (27) 540-000, FAX: (27) 540-005 WWW.TRACON.HU EU MEGFELELŐSÉGI NYILATKOZAT, a 79/1997. (XII. 31.) IKIM számú és a 374/2012.
(11) Lajstromszám: E 008 257 (13) T2 EURÓPAI SZABADALOM SZÖVEGÉNEK FORDÍTÁSA
 !HU00000827T2! (19) HU (11) Lajstromszám: E 008 27 (13) T2 MAGYAR KÖZTÁRSASÁG Szellemi Tulajdon Nemzeti Hivatala EURÓPAI SZABADALOM SZÖVEGÉNEK FORDÍTÁSA (21) Magyar ügyszám: E 0 727848 (22) A bejelentés
!HU00000827T2! (19) HU (11) Lajstromszám: E 008 27 (13) T2 MAGYAR KÖZTÁRSASÁG Szellemi Tulajdon Nemzeti Hivatala EURÓPAI SZABADALOM SZÖVEGÉNEK FORDÍTÁSA (21) Magyar ügyszám: E 0 727848 (22) A bejelentés
SOLiD Technology. library preparation & Sequencing Chemistry (sequencing by ligation!) Imaging and analysis. Application specific sample preparation
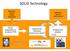 SOLiD Technology Application specific sample preparation Application specific data analysis library preparation & emulsion PCR Sequencing Chemistry (sequencing by ligation!) Imaging and analysis SOLiD
SOLiD Technology Application specific sample preparation Application specific data analysis library preparation & emulsion PCR Sequencing Chemistry (sequencing by ligation!) Imaging and analysis SOLiD
Nemzeti Akkreditáló Testület. SZŰKÍTETT RÉSZLETEZŐ OKIRAT (2) a NAT-1-1560/2012 nyilvántartási számú akkreditált státuszhoz
 Nemzeti Akkreditáló Testület SZŰKÍTETT RÉSZLETEZŐ OKIRAT (2) a NAT-1-1560/2012 nyilvántartási számú akkreditált státuszhoz A Bonafarm-Bábolna Takarmány Kft. Vizsgálólaboratórium (2942 Nagyigmánd, Burgert
Nemzeti Akkreditáló Testület SZŰKÍTETT RÉSZLETEZŐ OKIRAT (2) a NAT-1-1560/2012 nyilvántartási számú akkreditált státuszhoz A Bonafarm-Bábolna Takarmány Kft. Vizsgálólaboratórium (2942 Nagyigmánd, Burgert
Architecture of the Trypanosome RNA Editing Accessory Complex, MRB1
 Architecture of the Trypanosome RNA Editing Accessory Complex, MRB1 Michelle L. Ammerman, Kurtis M. Downey, Hassan Hashimi, John C. Fisk, Danielle L. Tomasello, Drahomíra Faktorová, Lucie Hanzálková, Tony
Architecture of the Trypanosome RNA Editing Accessory Complex, MRB1 Michelle L. Ammerman, Kurtis M. Downey, Hassan Hashimi, John C. Fisk, Danielle L. Tomasello, Drahomíra Faktorová, Lucie Hanzálková, Tony
Limitations and challenges of genetic barcode quantification
 Limitations and challenges of genetic barcode quantification Lars hielecke, im ranyossy, ndreas Dahl, Rajiv iwari, Ingo Roeder, Hartmut eiger, Boris Fehse, Ingmar lauche and Kerstin ornils SUPPLEMENRY
Limitations and challenges of genetic barcode quantification Lars hielecke, im ranyossy, ndreas Dahl, Rajiv iwari, Ingo Roeder, Hartmut eiger, Boris Fehse, Ingmar lauche and Kerstin ornils SUPPLEMENRY
T-helper Type 2 driven Inflammation Defines Major Subphenotypes of Asthma
 T-helper Type 2 driven Inflammation Defines Major Subphenotypes of Asthma Prescott G. Woodruff, M.D., M.P.H., Barmak Modrek, Ph.D., David F. Choy, B.S., Guiquan Jia, M.D., Alexander R. Abbas, Ph.D., Almut
T-helper Type 2 driven Inflammation Defines Major Subphenotypes of Asthma Prescott G. Woodruff, M.D., M.P.H., Barmak Modrek, Ph.D., David F. Choy, B.S., Guiquan Jia, M.D., Alexander R. Abbas, Ph.D., Almut
Using the CW-Net in a user defined IP network
 Using the CW-Net in a user defined IP network Data transmission and device control through IP platform CW-Net Basically, CableWorld's CW-Net operates in the 10.123.13.xxx IP address range. User Defined
Using the CW-Net in a user defined IP network Data transmission and device control through IP platform CW-Net Basically, CableWorld's CW-Net operates in the 10.123.13.xxx IP address range. User Defined
INFORMATIKA EMELT SZINT%
 Szövegszerkesztés, prezentáció, grafika, weblapkészítés 1. A fényképezés története Táblázatkezelés 2. Maradékos összeadás Adatbázis-kezelés 3. Érettségi Algoritmizálás, adatmodellezés 4. Fehérje Maximális
Szövegszerkesztés, prezentáció, grafika, weblapkészítés 1. A fényképezés története Táblázatkezelés 2. Maradékos összeadás Adatbázis-kezelés 3. Érettségi Algoritmizálás, adatmodellezés 4. Fehérje Maximális
Bioinformatics: Blending. Biology and Computer Science
 Bioinformatics: Blending Biology and Computer Science MDNMSITNTPTSNDACLSIVHSLMCHRQ GGESETFAKRAIESLVKKLKEKKDELDSL ITAITTNGAHPSKCVTIQRTLDGRLQVAG RKGFPHVIYARLWRWPDLHKNELKHVK YCQYAFDLKCDSVCVNPYHYERVVSPGI DLSGLTLQSNAPSSMMVKDEYVHDFEG
Bioinformatics: Blending Biology and Computer Science MDNMSITNTPTSNDACLSIVHSLMCHRQ GGESETFAKRAIESLVKKLKEKKDELDSL ITAITTNGAHPSKCVTIQRTLDGRLQVAG RKGFPHVIYARLWRWPDLHKNELKHVK YCQYAFDLKCDSVCVNPYHYERVVSPGI DLSGLTLQSNAPSSMMVKDEYVHDFEG
Flowering time. Col C24 Cvi C24xCol C24xCvi ColxCvi
 Flowering time Rosette leaf number 50 45 40 35 30 25 20 15 10 5 0 Col C24 Cvi C24xCol C24xCvi ColxCvi Figure S1. Flowering time in three F 1 hybrids and their parental lines as measured by leaf number
Flowering time Rosette leaf number 50 45 40 35 30 25 20 15 10 5 0 Col C24 Cvi C24xCol C24xCvi ColxCvi Figure S1. Flowering time in three F 1 hybrids and their parental lines as measured by leaf number
3. Sejtalkotó molekulák III.
 3. Sejtalkotó molekulák III. Fehérjék, fehérjeszintézis (transzkripció, transzláció, posztszintetikus módosítások). Enzimműködés 3.1 Fehérjék A genetikai információ egyik fő manifesztálódása Számos funkció
3. Sejtalkotó molekulák III. Fehérjék, fehérjeszintézis (transzkripció, transzláció, posztszintetikus módosítások). Enzimműködés 3.1 Fehérjék A genetikai információ egyik fő manifesztálódása Számos funkció
Construction of a cube given with its centre and a sideline
 Transformation of a plane of projection Construction of a cube given with its centre and a sideline Exercise. Given the center O and a sideline e of a cube, where e is a vertical line. Construct the projections
Transformation of a plane of projection Construction of a cube given with its centre and a sideline Exercise. Given the center O and a sideline e of a cube, where e is a vertical line. Construct the projections
MEGFELELŐSÉGI NYILATKOZAT DECLARATION OF CONFORMITY
 TRACON BUDAPEST KFT. TRACON BUDAPEST LTD. 2120 DUNAKESZI, PALLAG U. 23. TEL.: (27) 540-000, FAX: (27) 540-005 WWW.TRACON.HU MEGFELELŐSÉGI NYILATKOZAT a 79/1997. (XII.31.) IKIM rendelet, valamint az MSZ
TRACON BUDAPEST KFT. TRACON BUDAPEST LTD. 2120 DUNAKESZI, PALLAG U. 23. TEL.: (27) 540-000, FAX: (27) 540-005 WWW.TRACON.HU MEGFELELŐSÉGI NYILATKOZAT a 79/1997. (XII.31.) IKIM rendelet, valamint az MSZ
(11) Lajstromszám: E 008 220 (13) T2 EURÓPAI SZABADALOM SZÖVEGÉNEK FORDÍTÁSA
 !HU000008220T2! (19) HU (11) Lajstromszám: E 008 220 (13) T2 MAGYAR KÖZTÁRSASÁG Magyar Szabadalmi Hivatal EURÓPAI SZABADALOM SZÖVEGÉNEK FORDÍTÁSA (21) Magyar ügyszám: E 06 829727 (22) A bejelentés napja:
!HU000008220T2! (19) HU (11) Lajstromszám: E 008 220 (13) T2 MAGYAR KÖZTÁRSASÁG Magyar Szabadalmi Hivatal EURÓPAI SZABADALOM SZÖVEGÉNEK FORDÍTÁSA (21) Magyar ügyszám: E 06 829727 (22) A bejelentés napja:
Involvement of ER Stress in Dysmyelination of Pelizaeus-Merzbacher Disease with PLP1 Missense Mutations Shown by ipsc-derived Oligodendrocytes
 Stem Cell Reports, Volume 2 Supplemental Information Involvement of ER Stress in Dysmyelination of Pelizaeus-Merzbacher Disease with PLP1 Missense Mutations Shown by ipsc-derived Oligodendrocytes Yuko
Stem Cell Reports, Volume 2 Supplemental Information Involvement of ER Stress in Dysmyelination of Pelizaeus-Merzbacher Disease with PLP1 Missense Mutations Shown by ipsc-derived Oligodendrocytes Yuko
FÖLDRAJZ ANGOL NYELVEN
 Földrajz angol nyelven középszint 0821 ÉRETTSÉGI VIZSGA 2009. május 14. FÖLDRAJZ ANGOL NYELVEN KÖZÉPSZINTŰ ÍRÁSBELI ÉRETTSÉGI VIZSGA JAVÍTÁSI-ÉRTÉKELÉSI ÚTMUTATÓ OKTATÁSI ÉS KULTURÁLIS MINISZTÉRIUM Paper
Földrajz angol nyelven középszint 0821 ÉRETTSÉGI VIZSGA 2009. május 14. FÖLDRAJZ ANGOL NYELVEN KÖZÉPSZINTŰ ÍRÁSBELI ÉRETTSÉGI VIZSGA JAVÍTÁSI-ÉRTÉKELÉSI ÚTMUTATÓ OKTATÁSI ÉS KULTURÁLIS MINISZTÉRIUM Paper
Mock Orc2. NPE sperm pre-rc replication
 Time (min): HSS + M13 0 30 50 70 Orc2 HSS NPE sperm pre-rc repliction c % Repliction 60 40 20 0 HSS + sp NPE Orc2 Fig. S1: M13 ssdna-induced destruction of does not require ORC. S1: M13 ssdna ws incuted
Time (min): HSS + M13 0 30 50 70 Orc2 HSS NPE sperm pre-rc repliction c % Repliction 60 40 20 0 HSS + sp NPE Orc2 Fig. S1: M13 ssdna-induced destruction of does not require ORC. S1: M13 ssdna ws incuted
BEVEZETÉS Az objektum fogalma
 BEVEZETÉS Az objektum fogalma Program (1) Adat (2) Objektum Kiadványszerkesztés Word Táblázatkezelés Excel CAD AutoCad Adatbáziskezelés Access 1 Program (1) Adat (2) Objektum Adatmodell (2) A valós világ
BEVEZETÉS Az objektum fogalma Program (1) Adat (2) Objektum Kiadványszerkesztés Word Táblázatkezelés Excel CAD AutoCad Adatbáziskezelés Access 1 Program (1) Adat (2) Objektum Adatmodell (2) A valós világ
Adatbázis másolás Slony-I segítségével
 Adatbázis másolás Slony-I segítségével Akár a magas elérhetõség érdekében, akár mentésként vagy leállás nélküli verziófrissítés miatt van szükségünk másolatkészítésre, ez a rugalmas eszköz mindent szinkronban
Adatbázis másolás Slony-I segítségével Akár a magas elérhetõség érdekében, akár mentésként vagy leállás nélküli verziófrissítés miatt van szükségünk másolatkészítésre, ez a rugalmas eszköz mindent szinkronban
TELJESÍTMÉNY NYILATKOZAT 0832-CPD-1651
 E-mail: info@fulleon.co.uk Web: www.cooperfulleon.co m TELJESÍTMÉNY NYILATKOZAT 0832-CPD-1651 Termék azonosító kód: ROLP/SV és ROLP/SV/WP Típus, adagszám vagy gyári szám, illetve bármilyen más elem, amely
E-mail: info@fulleon.co.uk Web: www.cooperfulleon.co m TELJESÍTMÉNY NYILATKOZAT 0832-CPD-1651 Termék azonosító kód: ROLP/SV és ROLP/SV/WP Típus, adagszám vagy gyári szám, illetve bármilyen más elem, amely
A évi fizikai Nobel-díj
 A 2012. évi fizikai Nobel-díj "for ground-breaking experimental methods that enable measuring and manipulation of individual quantum systems" Serge Haroche David Wineland Ecole Normale Superieure, Párizs
A 2012. évi fizikai Nobel-díj "for ground-breaking experimental methods that enable measuring and manipulation of individual quantum systems" Serge Haroche David Wineland Ecole Normale Superieure, Párizs
2. Local communities involved in landscape architecture in Óbuda
 Év Tájépítésze pályázat - Wallner Krisztina 2. Közösségi tervezés Óbudán Óbuda jelmondata: Közösséget építünk, ennek megfelelően a formálódó helyi közösségeket bevonva fejlesztik a közterületeket. Békásmegyer-Ófaluban
Év Tájépítésze pályázat - Wallner Krisztina 2. Közösségi tervezés Óbudán Óbuda jelmondata: Közösséget építünk, ennek megfelelően a formálódó helyi közösségeket bevonva fejlesztik a közterületeket. Békásmegyer-Ófaluban
1. Gyakorlat: Telepítés: Windows Server 2008 R2 Enterprise, Core, Windows 7
 1. Gyakorlat: Telepítés: Windows Server 2008 R2 Enterprise, Core, Windows 7 1.1. Új virtuális gép és Windows Server 2008 R2 Enterprise alap lemez létrehozása 1.2. A differenciális lemezek és a két új virtuális
1. Gyakorlat: Telepítés: Windows Server 2008 R2 Enterprise, Core, Windows 7 1.1. Új virtuális gép és Windows Server 2008 R2 Enterprise alap lemez létrehozása 1.2. A differenciális lemezek és a két új virtuális
KN-CP50. MANUAL (p. 2) Digital compass. ANLEITUNG (s. 4) Digitaler Kompass. GEBRUIKSAANWIJZING (p. 10) Digitaal kompas
 KN-CP50 MANUAL (p. ) Digital compass ANLEITUNG (s. 4) Digitaler Kompass MODE D EMPLOI (p. 7) Boussole numérique GEBRUIKSAANWIJZING (p. 0) Digitaal kompas MANUALE (p. ) Bussola digitale MANUAL DE USO (p.
KN-CP50 MANUAL (p. ) Digital compass ANLEITUNG (s. 4) Digitaler Kompass MODE D EMPLOI (p. 7) Boussole numérique GEBRUIKSAANWIJZING (p. 0) Digitaal kompas MANUALE (p. ) Bussola digitale MANUAL DE USO (p.
Ellenőrző lista. 2. Hálózati útvonal beállítások, kapcsolatok, névfeloldások ellenőrzése: WebEC és BKPR URL-k kliensről történő ellenőrzése.
 Ellenőrző lista 1. HW/SW rendszer követelmények meglétének ellenőrzése: A telepítési segédlet által megjelölt elemek meglétének, helyes üzemének ellenőrzése. 2. Hálózati útvonal beállítások, kapcsolatok,
Ellenőrző lista 1. HW/SW rendszer követelmények meglétének ellenőrzése: A telepítési segédlet által megjelölt elemek meglétének, helyes üzemének ellenőrzése. 2. Hálózati útvonal beállítások, kapcsolatok,
Számítógéppel irányított rendszerek elmélete. Gyakorlat - Mintavételezés, DT-LTI rendszermodellek
 Számítógéppel irányított rendszerek elmélete Gyakorlat - Mintavételezés, DT-LTI rendszermodellek Hangos Katalin Villamosmérnöki és Információs Rendszerek Tanszék e-mail: hangos.katalin@virt.uni-pannon.hu
Számítógéppel irányított rendszerek elmélete Gyakorlat - Mintavételezés, DT-LTI rendszermodellek Hangos Katalin Villamosmérnöki és Információs Rendszerek Tanszék e-mail: hangos.katalin@virt.uni-pannon.hu
AZ IS30 BAKTERIÁLIS INSZERCIÓS ELEM CÉLSZEKVENCIA VÁLASZTÁSÁNAK MOLEKULÁRIS TÉNYEZŐI DOKTORI ÉRTEKEZÉS TÉZISEI SZABÓ MÓNIKA
 AZ IS30 BAKTERIÁLIS INSZERCIÓS ELEM CÉLSZEKVENCIA VÁLASZTÁSÁNAK MOLEKULÁRIS TÉNYEZŐI DOKTORI ÉRTEKEZÉS TÉZISEI SZABÓ MÓNIKA Gödöllő 2007. 1 A Doktori Iskola megnevezése: Szent István Egyetem Biológia Tudományi
AZ IS30 BAKTERIÁLIS INSZERCIÓS ELEM CÉLSZEKVENCIA VÁLASZTÁSÁNAK MOLEKULÁRIS TÉNYEZŐI DOKTORI ÉRTEKEZÉS TÉZISEI SZABÓ MÓNIKA Gödöllő 2007. 1 A Doktori Iskola megnevezése: Szent István Egyetem Biológia Tudományi
4. Gyakorlat: Csoportházirend beállítások
 4. Gyakorlat: Csoportházirend beállítások 4.1. A Default Domain Policy jelszóra vonatkozó beállításai 4.2. Parancsikon, mappa és hálózati meghajtó megjelenítése csoport házirend segítségével 4.3. Alkalmazások
4. Gyakorlat: Csoportházirend beállítások 4.1. A Default Domain Policy jelszóra vonatkozó beállításai 4.2. Parancsikon, mappa és hálózati meghajtó megjelenítése csoport házirend segítségével 4.3. Alkalmazások
Érzékenység vs dwell time
 Érzékenység vs dwell time analyte peak area [cps] 3.000.000 2.500.000 2.000.000 1.500.000 1.000.000 500.000 0 0 20 40 60 80 100 dwell time [ms] 25ms dwell time alatt számottevő a csökkenés (API 2000 LC/MS/MS
Érzékenység vs dwell time analyte peak area [cps] 3.000.000 2.500.000 2.000.000 1.500.000 1.000.000 500.000 0 0 20 40 60 80 100 dwell time [ms] 25ms dwell time alatt számottevő a csökkenés (API 2000 LC/MS/MS
A fehérjék hierarchikus szerkezete. Szerkezeti hierarchia. A fehérjék építőkövei az aminosavak. Fehérjék felosztása
 Fehérjék felosztása A fehérjék hierarchikus szerkezete Smeller László Semmelweis Egyetem Biofizikai és Sugárbiológiai Intézet Biológiai funkció alapján Enzimek (pl.: tripszin, citokróm-c ) Transzportfehérjék
Fehérjék felosztása A fehérjék hierarchikus szerkezete Smeller László Semmelweis Egyetem Biofizikai és Sugárbiológiai Intézet Biológiai funkció alapján Enzimek (pl.: tripszin, citokróm-c ) Transzportfehérjék
WCF, Entity Framework, ASP.NET, WPF 1. WCF service-t (adatbázissal Entity Framework) 2. ASP.NET kliens 3. WPF kliens
 WCF, Entity Framework, ASP.NET, WPF 1. WCF service-t (adatbázissal Entity Framework) 2. ASP.NET kliens 3. WPF kliens Hozzunk létre egy ASP.NET Empty Web Site projektet! A projekt neve legyen WCFAPP1. Ez
WCF, Entity Framework, ASP.NET, WPF 1. WCF service-t (adatbázissal Entity Framework) 2. ASP.NET kliens 3. WPF kliens Hozzunk létre egy ASP.NET Empty Web Site projektet! A projekt neve legyen WCFAPP1. Ez
Tutorial 1 The Central Dogma of molecular biology
 oday DN RN rotein utorial 1 he entral Dogma of molecular biology Information flow in genetics:» ranscription» ranslation» Making sense of genomic information Information content in DN - Information content
oday DN RN rotein utorial 1 he entral Dogma of molecular biology Information flow in genetics:» ranscription» ranslation» Making sense of genomic information Information content in DN - Information content
- Supplementary Data
 1 Open Access Asian Australas. J. Anim. Sci. Vol. 29, No. 3 : 321-326 March 2016 http://dx.doi.org/10.5713/ajas.15.0331 www.ajas.info pissn 1011-2367 eissn 1976-5517 Analysis of Swine Leukocyte Antigen
1 Open Access Asian Australas. J. Anim. Sci. Vol. 29, No. 3 : 321-326 March 2016 http://dx.doi.org/10.5713/ajas.15.0331 www.ajas.info pissn 1011-2367 eissn 1976-5517 Analysis of Swine Leukocyte Antigen
FÖLDRAJZ ANGOL NYELVEN GEOGRAPHY
 Földrajz angol nyelven középszint 0612 É RETTSÉGI VIZSGA 2006. október 25. FÖLDRAJZ ANGOL NYELVEN GEOGRAPHY KÖZÉPSZINTŰ ÍRÁSBELI ÉRETTSÉGI VIZSGA INTERMEDIATE LEVEL WRITTEN EXAM JAVÍTÁSI-ÉRTÉKELÉSI ÚTMUTATÓ
Földrajz angol nyelven középszint 0612 É RETTSÉGI VIZSGA 2006. október 25. FÖLDRAJZ ANGOL NYELVEN GEOGRAPHY KÖZÉPSZINTŰ ÍRÁSBELI ÉRETTSÉGI VIZSGA INTERMEDIATE LEVEL WRITTEN EXAM JAVÍTÁSI-ÉRTÉKELÉSI ÚTMUTATÓ
A magyar racka juh tejének beltartalmi változása a laktáció alatt
 A magyar racka juh tejének beltartalmi változása a laktáció alatt Nagy László Komlósi István Debreceni Egyetem Agrártudományi Centrum, Mezőgazdaságtudományi Kar, Állattenyésztés- és Takarmányozástani Tanszék,
A magyar racka juh tejének beltartalmi változása a laktáció alatt Nagy László Komlósi István Debreceni Egyetem Agrártudományi Centrum, Mezőgazdaságtudományi Kar, Állattenyésztés- és Takarmányozástani Tanszék,
Mai témák. Fehérjék dinamikájának jelentősége. Számítógépes modellezés jelentősége
 Mai témák Fehérjék szerkezetének predikciója, szerkezeti adatok felhasználása adatbázisok segítségével, a számítógépes molekuladinamikai modellezés alapjai Hegedűs Tamás tamas@hegelab.org Bevezetés szimulációk
Mai témák Fehérjék szerkezetének predikciója, szerkezeti adatok felhasználása adatbázisok segítségével, a számítógépes molekuladinamikai modellezés alapjai Hegedűs Tamás tamas@hegelab.org Bevezetés szimulációk
The beet R locus encodes a new cytochrome P450 required for red. betalain production.
 The beet R locus encodes a new cytochrome P450 required for red betalain production. Gregory J. Hatlestad, Rasika M. Sunnadeniya, Neda A. Akhavan, Antonio Gonzalez, Irwin L. Goldman, J. Mitchell McGrath,
The beet R locus encodes a new cytochrome P450 required for red betalain production. Gregory J. Hatlestad, Rasika M. Sunnadeniya, Neda A. Akhavan, Antonio Gonzalez, Irwin L. Goldman, J. Mitchell McGrath,
Performance Modeling of Intelligent Car Parking Systems
 Performance Modeling of Intelligent Car Parking Systems Károly Farkas Gábor Horváth András Mészáros Miklós Telek Technical University of Budapest, Hungary EPEW 2014, Florence, Italy Outline Intelligent
Performance Modeling of Intelligent Car Parking Systems Károly Farkas Gábor Horváth András Mészáros Miklós Telek Technical University of Budapest, Hungary EPEW 2014, Florence, Italy Outline Intelligent
Biológiai molekulák számítógépes szimulációja Balog Erika
 Bológa molekulák számíógépes szmulácóa Balog Eka Semmelwes Egyeem, Bofzka és Sugábológa Inéze SZEKVENCIA ALA THR SER THR LYS LYS LEU HSD LYS GLU PRO ALA ILE LEU LYS ALA ILE ASP ASP THR TYR VAL LYS PRO
Bológa molekulák számíógépes szmulácóa Balog Eka Semmelwes Egyeem, Bofzka és Sugábológa Inéze SZEKVENCIA ALA THR SER THR LYS LYS LEU HSD LYS GLU PRO ALA ILE LEU LYS ALA ILE ASP ASP THR TYR VAL LYS PRO
16F628A megszakítás kezelése
 16F628A megszakítás kezelése A 'megszakítás' azt jelenti, hogy a program normális, szekvenciális futása valamilyen külső hatás miatt átmenetileg felfüggesztődik, és a vezérlést egy külön rutin, a megszakításkezelő
16F628A megszakítás kezelése A 'megszakítás' azt jelenti, hogy a program normális, szekvenciális futása valamilyen külső hatás miatt átmenetileg felfüggesztődik, és a vezérlést egy külön rutin, a megszakításkezelő
HASZNÁLATI ÚTMUTATÓ. Az LCD kijelző mutatja a folyamat jellemzőit: írási sebesség, készenlét %-ban, hátralevő adatmennyiség, hátralevő idő
 HASZNÁLATI ÚTMUTATÓ Másolás Másolás mester DVD-ről vagy a beépített HDD-ről. 1. Copy Helyezze el a mester DVD-t a CD-ROM meghajtóban, az írható üres lemezeket az írókban. Ezután nyomja meg az ENT gombot
HASZNÁLATI ÚTMUTATÓ Másolás Másolás mester DVD-ről vagy a beépített HDD-ről. 1. Copy Helyezze el a mester DVD-t a CD-ROM meghajtóban, az írható üres lemezeket az írókban. Ezután nyomja meg az ENT gombot
TELJESÍTMÉNY NYILATKOZAT 0333-CPD
 E-mail: info@fulleon.co.uk TELJESÍTMÉNY NYILATKOZAT 0333-CPD-075441 Termék azonosító kód: Típus, adagszám vagy gyári szám, illetve bármilyen más elem, amely lehetővé teszi az építési termékek azonosítását
E-mail: info@fulleon.co.uk TELJESÍTMÉNY NYILATKOZAT 0333-CPD-075441 Termék azonosító kód: Típus, adagszám vagy gyári szám, illetve bármilyen más elem, amely lehetővé teszi az építési termékek azonosítását
supplementary information
 DOI: 10.1038/nc1870 lk6 lk6 lk3 lk3 lk6 lk6 lk3 lk3 lk6 lk6 lk3 lk3 lk6 lk6 lk3 lk3 lk6 lk6 lk3 lk3 lk6 lk6 lk3 lk3 lk6 lk6 lk3 lk3 lk6 lk6 lk3 lk3 lk6 lk6 lk3 lk3 MELRVRTGNEKFWTLILLGV----SWIYP-S-AHANVLDTMLQRSSGKVTAGRKETNGGS.D.YR--SCLRWLCVL.I.L----LGLVE-E-NL...S..L.N...EE-.R..--...RQL------L.I.VV.T..----CLLLTLCSGAGQNP.HV..GTGV.LDS--RR--PGD.KST--------F.I.VF..LVAAVLLS.-LAEGGQNP.Y..HGTGV.PGSDPRRAQA.D
DOI: 10.1038/nc1870 lk6 lk6 lk3 lk3 lk6 lk6 lk3 lk3 lk6 lk6 lk3 lk3 lk6 lk6 lk3 lk3 lk6 lk6 lk3 lk3 lk6 lk6 lk3 lk3 lk6 lk6 lk3 lk3 lk6 lk6 lk3 lk3 lk6 lk6 lk3 lk3 MELRVRTGNEKFWTLILLGV----SWIYP-S-AHANVLDTMLQRSSGKVTAGRKETNGGS.D.YR--SCLRWLCVL.I.L----LGLVE-E-NL...S..L.N...EE-.R..--...RQL------L.I.VV.T..----CLLLTLCSGAGQNP.HV..GTGV.LDS--RR--PGD.KST--------F.I.VF..LVAAVLLS.-LAEGGQNP.Y..HGTGV.PGSDPRRAQA.D
Horizontal gene transfer drives adaptive colonization of apple trees by the fungal pathogen Valsa mali. Zhiyuan Yin, Baitao Zhu, Hao Feng, Lili Huang*
 Supplementary Information Horizontal gene transfer drives adaptive colonization of apple trees by the fungal pathogen Valsa mali Zhiyuan Yin, Baitao Zhu, Hao Feng, Lili Huang* State Key Laboratory of Crop
Supplementary Information Horizontal gene transfer drives adaptive colonization of apple trees by the fungal pathogen Valsa mali Zhiyuan Yin, Baitao Zhu, Hao Feng, Lili Huang* State Key Laboratory of Crop
Bagi Nyílászáró Gyártó és Kereskedelmi Kft.
 Bagi Nyílászáró Gyártó és Kereskedelmi Kft. 2191 BAG Jókai utca 13. Adószám: HU13390727-2-13 Cégjegyzék:13-09-101221 Telefon:+36-70-550-8452 Fax:+36-28-408-088 E-mail: bagifa@invitel.hu ksj.manufacturing@gmail.com
Bagi Nyílászáró Gyártó és Kereskedelmi Kft. 2191 BAG Jókai utca 13. Adószám: HU13390727-2-13 Cégjegyzék:13-09-101221 Telefon:+36-70-550-8452 Fax:+36-28-408-088 E-mail: bagifa@invitel.hu ksj.manufacturing@gmail.com
Széchenyi István Egyetem www.sze.hu/~herno
 Oldal: 1/6 A feladat során megismerkedünk a C# és a LabVIEW összekapcsolásának egy lehetőségével, pontosabban nagyon egyszerű C#- ban írt kódból fordítunk DLL-t, amit meghívunk LabVIEW-ból. Az eljárás
Oldal: 1/6 A feladat során megismerkedünk a C# és a LabVIEW összekapcsolásának egy lehetőségével, pontosabban nagyon egyszerű C#- ban írt kódból fordítunk DLL-t, amit meghívunk LabVIEW-ból. Az eljárás
A fehérjék szerkezete és az azt meghatározó kölcsönhatások
 A fehérjék szerkezete és az azt meghatározó kölcsönhatások 1. A fehérjék szerepe az élõlényekben 2. A fehérjék szerkezetének szintjei 3. A fehérjék konformációs stabilitásáért felelõs kölcsönhatások 4.
A fehérjék szerkezete és az azt meghatározó kölcsönhatások 1. A fehérjék szerepe az élõlényekben 2. A fehérjék szerkezetének szintjei 3. A fehérjék konformációs stabilitásáért felelõs kölcsönhatások 4.
 9. Előadás Fehérjék Előzmények Peptidkémia Analitikai kémia Protein kémia 1901 E.Fischer : Gly-Gly 1923 F. Pregl : Mikroanalitika 1952 Stein and Moore : Aminosav analizis 1932 Bergman és Zervas : Benziloxikarbonil
9. Előadás Fehérjék Előzmények Peptidkémia Analitikai kémia Protein kémia 1901 E.Fischer : Gly-Gly 1923 F. Pregl : Mikroanalitika 1952 Stein and Moore : Aminosav analizis 1932 Bergman és Zervas : Benziloxikarbonil
Phenotype. Genotype. It is like any other experiment! What is a bioinformatics experiment? Remember the Goal. Infectious Disease Paradigm
 It is like any other experiment! What is a bioinformatics experiment? You need to know your data/input sources You need to understand your methods and their assumptions You need a plan to get from point
It is like any other experiment! What is a bioinformatics experiment? You need to know your data/input sources You need to understand your methods and their assumptions You need a plan to get from point
Planetary Nebulae, PN PNe PN
 2 2 e-mail: masaaki@oao.nao.ac.jp 719 0232 3037 5 Subaru Telescope, National Astronomical Observatory of Japan, 650 North A ohoku Place, Hilo, Hawaii 96720, U.S.A. e-mail: tajitsu@subaru.naoj.org 397 0101
2 2 e-mail: masaaki@oao.nao.ac.jp 719 0232 3037 5 Subaru Telescope, National Astronomical Observatory of Japan, 650 North A ohoku Place, Hilo, Hawaii 96720, U.S.A. e-mail: tajitsu@subaru.naoj.org 397 0101
practices Mosaic and timed mowing Mosaic and timed mowing Mosaic and timed mowing 10 m wide fallow strips (4 parcels)
 Name of treatment (Action C5 in HUKM10004 by KMNPD and MME) Goal of the treatment Description of treatment and management practices Treated area Maximum No. of sub-groups ha pcs. Type1. Extensive mosaic
Name of treatment (Action C5 in HUKM10004 by KMNPD and MME) Goal of the treatment Description of treatment and management practices Treated area Maximum No. of sub-groups ha pcs. Type1. Extensive mosaic
ANNEX V / V, MELLÉKLET
 ANNEX V / V, MELLÉKLET VETERINARY CERTIFICATE EMBRYOS OF DOMESTIC ANIMALS OF THE BOVINE SPECIES FOR IMPORTS COLLECTED OR PRODUCED BEFORE 1 st JANUARY 2006 ÁLLAT-EGÉSZSÉGÜGYI BIZONYÍTVÁNY 2006. JANUÁR 1-JE
ANNEX V / V, MELLÉKLET VETERINARY CERTIFICATE EMBRYOS OF DOMESTIC ANIMALS OF THE BOVINE SPECIES FOR IMPORTS COLLECTED OR PRODUCED BEFORE 1 st JANUARY 2006 ÁLLAT-EGÉSZSÉGÜGYI BIZONYÍTVÁNY 2006. JANUÁR 1-JE
Első lépések. File/New. A mentés helyét érdemes módosítani! Pl. Dokumentumok. Fájlnév: pl. Proba
 Első lépések File/New A mentés helyét érdemes módosítani! Pl. Dokumentumok Fájlnév: pl. Proba (megj. ékezetes karaktereket nem használhatunk a fájlnévben) 1 Konvejor pálya elkészítése System/New Rendszer
Első lépések File/New A mentés helyét érdemes módosítani! Pl. Dokumentumok Fájlnév: pl. Proba (megj. ékezetes karaktereket nem használhatunk a fájlnévben) 1 Konvejor pálya elkészítése System/New Rendszer
Glyma10g15850 aagggatccattctggaaccatatcttgctgtg ttgggtacccttggatgcaggatgacacg AtMKK6, AT5g56580
 Supplemental table 1. Soybean MAPK, MAPKK, and MAPKKK genes and primers for VIGS cloning Gene_ID Forward Primer Reverse Primer Arabidopsis Ortholog Glyma04g03210 aagggatcctgcagcaagaaccagttcat ttgggtaccctaatggatgcgcattaggataaag
Supplemental table 1. Soybean MAPK, MAPKK, and MAPKKK genes and primers for VIGS cloning Gene_ID Forward Primer Reverse Primer Arabidopsis Ortholog Glyma04g03210 aagggatcctgcagcaagaaccagttcat ttgggtaccctaatggatgcgcattaggataaag
Baldwin St S Ashburn Rd Baldwin St N Anderson St Thickson Rd N Exhibit 'A' to Amendment to the Whitby Official Plan Exhibit 1 Deferral #2 Modification
 Baldwin St S Ashburn Rd Baldwin St N Anderson St Thickson Rd N Exhibit 'A' to Amendment to the Whitby Official Plan Exhibit 1 Modification #3 Brawley Rd W Brawley Rd E U Add: Community Central Area Add:
Baldwin St S Ashburn Rd Baldwin St N Anderson St Thickson Rd N Exhibit 'A' to Amendment to the Whitby Official Plan Exhibit 1 Modification #3 Brawley Rd W Brawley Rd E U Add: Community Central Area Add:
Hasznos és kártevő rovarok monitorozása innovatív szenzorokkal (LIFE13 ENV/HU/001092)
 Hasznos és kártevő rovarok monitorozása innovatív szenzorokkal (LIFE13 ENV/HU/001092) www.zoolog.hu Dr. Dombos Miklós Tudományos főmunkatárs MTA ATK TAKI Innovative Real-time Monitoring and Pest control
Hasznos és kártevő rovarok monitorozása innovatív szenzorokkal (LIFE13 ENV/HU/001092) www.zoolog.hu Dr. Dombos Miklós Tudományos főmunkatárs MTA ATK TAKI Innovative Real-time Monitoring and Pest control
PHP II. WEB technológiák. Tóth Zsolt. Miskolci Egyetem. Tóth Zsolt (Miskolci Egyetem) PHP II. 2014 1 / 19
 PHP II. WEB technológiák Tóth Zsolt Miskolci Egyetem 2014 Tóth Zsolt (Miskolci Egyetem) PHP II. 2014 1 / 19 Tartalomjegyzék Objektum Orientált Programozás 1 Objektum Orientált Programozás Öröklődés 2 Fájlkezelés
PHP II. WEB technológiák Tóth Zsolt Miskolci Egyetem 2014 Tóth Zsolt (Miskolci Egyetem) PHP II. 2014 1 / 19 Tartalomjegyzék Objektum Orientált Programozás 1 Objektum Orientált Programozás Öröklődés 2 Fájlkezelés
Az Ön kézikönyve GUDE W 520 http://hu.yourpdfguides.com/dref/2446099
 Elolvashatja az ajánlásokat a felhasználói kézikönyv, a műszaki vezető, illetve a telepítési útmutató GUDE W 520. Megtalálja a választ minden kérdésre az GUDE W 520 a felhasználói kézikönyv (információk,
Elolvashatja az ajánlásokat a felhasználói kézikönyv, a műszaki vezető, illetve a telepítési útmutató GUDE W 520. Megtalálja a választ minden kérdésre az GUDE W 520 a felhasználói kézikönyv (információk,
SZOFTVEREK A SORBANÁLLÁSI ELMÉLET OKTATÁSÁBAN
 SZOFTVEREK A SORBANÁLLÁSI ELMÉLET OKTATÁSÁBAN Almási Béla, almasi@math.klte.hu Sztrik János, jsztrik@math.klte.hu KLTE Matematikai és Informatikai Intézet Abstract This paper gives a short review on software
SZOFTVEREK A SORBANÁLLÁSI ELMÉLET OKTATÁSÁBAN Almási Béla, almasi@math.klte.hu Sztrik János, jsztrik@math.klte.hu KLTE Matematikai és Informatikai Intézet Abstract This paper gives a short review on software
TELJESÍTMÉNY NYILATKOZAT 0333-CPD
 E-mail: info@ fu leon.co.uk TELJESÍTMÉNY NYILATKOZAT 0333-CPD-075444 Termék azonosító kód: Típus, adagszám vagy gyári szám, illetve bármilyen más elem, amely lehetővé teszi az építési termékek azonosítását
E-mail: info@ fu leon.co.uk TELJESÍTMÉNY NYILATKOZAT 0333-CPD-075444 Termék azonosító kód: Típus, adagszám vagy gyári szám, illetve bármilyen más elem, amely lehetővé teszi az építési termékek azonosítását
Klaszterezés, 2. rész
 Klaszterezés, 2. rész Csima Judit BME, VIK, Számítástudományi és Információelméleti Tanszék 208. április 6. Csima Judit Klaszterezés, 2. rész / 29 Hierarchikus klaszterezés egymásba ágyazott klasztereket
Klaszterezés, 2. rész Csima Judit BME, VIK, Számítástudományi és Információelméleti Tanszék 208. április 6. Csima Judit Klaszterezés, 2. rész / 29 Hierarchikus klaszterezés egymásba ágyazott klasztereket
IKR Agrár Kft. biztonsági elemzése Füzesabony Területi Központ
 2016. IKR Agrár Kft. biztonsági elemzése Füzesabony Területi Központ 5. számú melléklet Kockázat csökkentő intézkedés tervezete AGEL-CBI Kft. Verzió: 1.0 2016.04.30. IKR Agrár Kft. biztonsági elemzése
2016. IKR Agrár Kft. biztonsági elemzése Füzesabony Területi Központ 5. számú melléklet Kockázat csökkentő intézkedés tervezete AGEL-CBI Kft. Verzió: 1.0 2016.04.30. IKR Agrár Kft. biztonsági elemzése
AZ ERDÕ NÖVEKEDÉSÉNEK VIZSGÁLATA TÉRINFORMATIKAI ÉS FOTOGRAMMETRIAI MÓDSZEREKKEL KARSZTOS MINTATERÜLETEN
 Tájökológiai Lapok 5 (2): 287 293. (2007) 287 AZ ERDÕ NÖVEKEDÉSÉNEK VIZSGÁLATA TÉRINFORMATIKAI ÉS FOTOGRAMMETRIAI MÓDSZEREKKEL KARSZTOS MINTATERÜLETEN ZBORAY Zoltán Honvédelmi Minisztérium Térképészeti
Tájökológiai Lapok 5 (2): 287 293. (2007) 287 AZ ERDÕ NÖVEKEDÉSÉNEK VIZSGÁLATA TÉRINFORMATIKAI ÉS FOTOGRAMMETRIAI MÓDSZEREKKEL KARSZTOS MINTATERÜLETEN ZBORAY Zoltán Honvédelmi Minisztérium Térképészeti
Bioinformatika és genomanalízis az orvostudományban. Biológiai adatbázisok. Cserző Miklós 2018
 Bioinformatika és genomanalízis az orvostudományban Biológiai adatbázisok Cserző Miklós 2018 A mai előadás Mi az adatbázis A biológia kapcsolata az adatbázisokkal Az adatbázisok típusai Adatbázis formátumok,
Bioinformatika és genomanalízis az orvostudományban Biológiai adatbázisok Cserző Miklós 2018 A mai előadás Mi az adatbázis A biológia kapcsolata az adatbázisokkal Az adatbázisok típusai Adatbázis formátumok,
Számla Hiteles másolat 2/2.példány 1.lap
 Számla Hiteles másolat 2/2.példány 1.lap 1111 Fizetési mód/payment method Atutalás Teljesítés kelt/fulfillment date 2000-07-28 Számla kelt/invoice date 2000-07-28 Fizetési határidõ/payment due 2000-07-28
Számla Hiteles másolat 2/2.példány 1.lap 1111 Fizetési mód/payment method Atutalás Teljesítés kelt/fulfillment date 2000-07-28 Számla kelt/invoice date 2000-07-28 Fizetési határidõ/payment due 2000-07-28
BKI13ATEX0030/1 EK-Típus Vizsgálati Tanúsítvány/ EC-Type Examination Certificate 1. kiegészítés / Amendment 1 MSZ EN 60079-31:2014
 (1) EK-TípusVizsgálati Tanúsítvány (2) A potenciálisan robbanásveszélyes környezetben történő alkalmazásra szánt berendezések, védelmi rendszerek 94/9/EK Direktíva / Equipment or Protective Systems Intended
(1) EK-TípusVizsgálati Tanúsítvány (2) A potenciálisan robbanásveszélyes környezetben történő alkalmazásra szánt berendezések, védelmi rendszerek 94/9/EK Direktíva / Equipment or Protective Systems Intended
LED UTCAI LÁMPATESTEK STREET LIGHTING
 LED UTCAI LÁMPATESTEK STREET LIGHTING LED Utcai lámpatestek Street Lights 1. Öntött ADC12 alumínium ház NOBEL porszórt festéssel 3. Edzett optikai lencsék irányított sugárzási szöggel 5. Meanwell meghajtó
LED UTCAI LÁMPATESTEK STREET LIGHTING LED Utcai lámpatestek Street Lights 1. Öntött ADC12 alumínium ház NOBEL porszórt festéssel 3. Edzett optikai lencsék irányított sugárzási szöggel 5. Meanwell meghajtó
