Supplementary Table 2. Summary of putative S-nitrosylated cysteines under nitrosative stress in C. reinhardtii.
|
|
|
- Ernő Magyar
- 6 évvel ezelőtt
- Látták:
Átírás
1 Supplementary Table 2. Summary of putative S-nitrosylated cysteines under nitrosative stress in C. reinhardtii. Accession number: accession number in UniprotKB protein database. RefSeq accession number: accession number in NCBI protein database. Cysteine number: total number of cysteines in the full-length sequence. Cysteine position: experimental S-nitrosylated cysteine found by SNOSID method. S-NO: Cysteine found as S-nitrosylated in A. thaliana (Fares et al. (2011) Proteomics investigation of endogeneous S-nitrosylation in Arabidopsis. Bioch. Biophys. Res. Commun. 416, ). S-SG: Cysteine found as glutathionylated in C. reinhardtii (Zaffagnini et al. (2012) Glutathionylation in the photosynthetic model organism Chlamydomonas reinhardtii : a proteomic survey. Mol Cell Proteomics. 11, M ). This table is available as an Excel file in the ProteomeXchange database with identifier PXD000569
2 Accession number Description Cysteine number Cysteine position S-NO sites flanking sequences S-NO S-SG P protein 3 8 MAVDREECVYMAKLAEQAERYDE A8ILN4 1-hydroxy-2-methyl-2-(E)-butenyl 4-diphosphate synthase IRVSLTEDPEFEMDPCKRLAGLGERAWGSGL A8ICB3 20S proteasome beta subunit B, type beta SGVLNDLGSGSNVDLCVITKDGVEYLRNYEY A8IHB3 26S proteasome regulatory subunit VMAKDELSGADIKAVCTEAGLLALRERRMRV A8IV67 26S proteasome regulatory subunit ARLCPNSTGADIRSVCTEAGMYAIRARRKTV A8J6Q7 3-deoxy-D-arabino-heptulosonate 7-phosphate synthetase CEEIARMPPLIFAGECRTLQSRLAKASTGDA A8JEV9 4-hydroxy-3-methylbut-2-enyl diphosphate reductase LTDFKEKGVQMVDTTCPWVAKVWNSVDTHTR Q8SAQ6 6-phosphogluconate dehydrogenase, decarboxylating ALKSERVAASKSFTSCVTPGPVAGVDKAQLI 338 QLINDVRAALYASKICSYAQGMNIIKAKSVE A8IAT4 acetohydroxy acid isomeroreductase IGLRPDSPSWAEAEACGFSKTDGTLGEVFEQ 466 NPYMHARGVAFMVDNCSYTARLGSRKWAPRF A8IX80 acetohydroxyacid dehydratase TLYKYIKNVSSASTGCVTDS A8JCR6 acetolactate synthase, small subunit FRARVVDVGDETLSLCVTGDPGKLTAMIKVM A8JCQ8 acetyl CoA synthetase AIRSLMSFGDQWVSQCDTSSLKLLGSVGEPI A8J933 acetylornithine aminotransferase AKRLVENSFADKAFFCNTGTEANEGAIKFAR A8HMQ1 aconitate hydratase KNGWPTELKAGLIGSCTNSSYEDMARAASVA X 466 FDKIGGTVLSNScGPCIGQWKRTDVPKGEAN P53498 actin 5 12 MADEGEVSALVCDNGSGMVKAGFAGDD A8IJ60 active subunit of chloroplast ClpP peptidase IMYHKANLNRIMADYCQQPLSKIEEDTDRDR Q6UKY4 acyl-carrier protein EADKIASVGDAINYICSNPMAK A8HS14 ADP-glucose pyrophosphorylase large subunit PIGGAYRLIDVPMSNCINSGISKIYILTQFN X
3 Q9LLL6 ADP-glucose pyrophosphorylase small subunit PLGANYRLIDIPVSNCLNSNVTKIYCLTQFN 133 IPVSNCLNSNVTKIYCLTQFNSASLNRHLSQ 237 KHRNSGAAITIAALPCAEKEASAFGLMKIDE 390 IYTMSRFLPPSKVMDCDVNMSIIGDGcVIKA A8J3Y6 AIR synthase-related protein AAVSLRDAMLDLGVACDGGKDSLSMAAAAGG 1098 RDVWEETSFVLERLQCAEEcVAQEQAGLKTA A8JFJ2 alanine-glyoxylate transaminase 6 47 ASEEVVAVDNAARKGCDGVLEVPSRLLMGPG A8JH72 alanine-glyoxylate transaminase YLPAVYKMVREAGGVCIADEVQTGFGRTGGH A8J4D3 alpha-amylase WNKFGGRLAWDASAICSNNPSFGGRGNPKQG 235 LNYNQDAHRQRTVNWCDSTGGTSAAFDFTTK A8HRR9 alpha-cop GDFVRIGADASGLLICPSQQTR A8IU73 alternative oxidase NICDDEMEHVKTMKACQDETVSQDIITRRED A8J173 aspartate semialdehyde dehydrogenase MAVTPLDRVANVKRMCVSTYQAASGAGAAAM A8I8X8 ATP citrate lyase, subunit B FKIGDTAGTLDNIVACKLHRPGSVGFVSKSG 389 FGGAIDDAARYFKAACDAGQSPSEFVEGLKK P06541 ATP synthase CF1 beta subunit 2 63 TIRAKNSAGTEMAVTCEVQQLLGDNCVRAVS 73 EMAVTCEVQQLLGDNCVRAVSMNPTEGLMRG A8IXF1 ATP-sulfurylase VRSTLPANQDVLAFQCRNPIHKAHYELFIRA P38482 beta subunit of mitochondrial ATP synthase MNVIGEPVDEQGPIECSEVWSIHREAPEFTE P04690 beta tubulin MREIVHIQGGQCGNQIGAKFWEVVSDEH X 354 SYFVEWIPNNVKSSVCDIPPKGLKMSATFIG X A8J1S7 bifunctional aspartate kinase/homoserine dehydrogenase NFLSSHYIPNRVIVDCTASDAPASKYMTWMQ A8I7T8 binding protein AVKAKIDARNQLETYCYNMKSTVEDKMKDKI
4 A8IPQ9 calcium-dependent protein kinase TATQILQHEWMRENGCAADQPIQLEVLSRMK A8I542 calcium-transporting ATPase, endoplasmic reticulum-type SVAVNKGSDPVADPNCELQSKECMLFAGTAI A8IED9 centromere/microtubule binding protein SCEAGDPKVTGNLIVCIDRATRLVKAQQAAG Q695H0 cgmp-dependent protein kinase LANMTDPEFKELLKACEQTDYNPGEVIAVAN P00877 Chain A, Crystal Structure Of Rubisco From Chlamydomonas Reinhar VERDKLNKYGRGLLGCTIKPKLGLSAKNYGR X 247 TGEVKGHYLNATAGTCEEMMKRAVXAKELGV 427 GNAPGAAANRVALEACTQARNEGRDLAREGG X X 459 VIRSACKWSPELAAACEVWKEIKFEFDTIDK P12113 chloroplast ATP synthase gamma chain GVNQRVRQEDVDSPLCAVRPVKSVLLVVLTG 152 RYRELTAMGVKVNLVCVGRKGAQYFARRKQY Q66YD3 chloroplast DnaJ-like protein VRKTTGVRKGTTPSTCPQCQGTGQVVSAVRT 220 TTGVRKGTTPSTCPQCQGTGQVVSAVRTPLG 243 SAVRTPLGTFQQVSLCPRCEGSGQISQPCTK A8IA39 chloroplast elongation factor G LEIIVDRLRREFKVECEVGAPQVNYREGISR Q1G2Y1 chloroplast polyribonucleotide phosphorylase THGSALFTRGETQSICVATLGSANDALRSDT Q6RBZ1 circadian RNA-binding protein CHLAMY 1 subunit C QVVQRFGVGSTEVLECPKTMVGRIIGKGGET Q6RBZ0 circadian RNA-binding protein CHLAMY 1 subunit C NITDVHVLNKGNAPGCAFVTYERWAHAEAAM A8I4S9 clathrin heavy chain FERLFAAGQYKEAAECAAESPQGQLRTRDVM A8J5T5 COP-II coat subunit CGGHTDQAVSYWARNCTGASGTTTEVLQNVI Q9FYV4 copper chaperone 2 13 MSTEVVLKVDMMCNGCVGAVQRVLGKLD 16 MSTEVVLKVDMMCNGCVGAVQRVLGKLDGVD Q4U0V8 coproporphyrinogen III oxidase 7 94 WERDASNPNAGYGITCVLEDGKVLEKAAANI Q9S7V1 coproporphyrinogen III oxidase HQPKAGSPEAELLDACRNPRVWV
5 Q84X73 CR061 protein VPGGIIMPDKATLSLCAIEDGEYKHDKIEFW A8J355 cystathionine gamma-synthase ILAISRLCHAKGAFVCVDSTFATPINTRALE A8J7T7 cysteine endopeptidase GYIRLKMGSTDAEGLCGIAMAPSYPVKTGPN X Q9FQ96 cytochrome c CHSMQYLHWRQLVGVCYTEEEAKALAAETEV A8HQA8 DEAD-box RNA helicase VLAPTRELAVQIQQECQRFGASSRIKNTVVY A8ITV1 decapping enzyme complex catalytic component GFPRGKVNEGETEATCAIREVLEETGYDIRS A8IU49 dehydrogenase PRKSGSILRTQVAEVCVAALTEPAAANKVVE A8J8B3 diaminopimelate epimerase LTMRIFNSDGSEPEMCGNGIRCLAKFVSDID A8HPL8 dihydrolipoamide dehydrogenase GLKVAVIEGHDIGGTCVNRGCVPSKALLAAS A8J1T4 dihydrolipoyl dehydrogenase 8 65 YVAAIKAAQLGLSVACVEGRGALGGTCLNVG 76 LSVACVEGRGALGGTCLNVGCIPSKALLNSS 81 VEGRGALGGTCLNVGCIPSKALLNSSHMYME A8IDM1 DNA topoisomerase I AGHEKDEDEADTVGcCTLKVENIEIMGDNKV A8JI07 dual function alcohol dehydrogenase / acetaldehyde dehydrogenase FASEFIYNKYKHTKTCGVIEHDPAGGIQKVA 668 RVYEVPELGKKATMVCIPTTSGTGSEVTPFS A8IQV3 EF-hand and p25-alpha domain containg protein AQLKLTAAQAASDQGCDVSATATDAGRQTCP A8JHX9 elongation factor TAALRITDGALVVVDCVEGVcVQTETVLRQA 372 YEGPLDDTYATAIRNCDPNGPLMVYISKMIP X 474 LDQYITKTATITKEGCDDAFPMKAMKFSVSP 728 RRVIYAAELTAQPRLCEPVYLVEIQAPEQAL A8ISZ1 elongation factor EF QITRALPDIVPAVSGCMNDSKQSVKDVATET A8J280 elongation factor-like protein QSARCGENLRLRLSGCEETDISPGFVLSSVK A8JH98 enolase 8 23 EKHGLEKKVEEVLNLCVKEKPEEPLSFMAKA X
6 A8HYM2 eukaryotic initiation factor 6 42 KALERPPEYVLKYYGCELGAQTNFDKASGTS A8I232 eukaryotic initiation factor KMSKGEVLMLNIGSMCTGARVLAVKGDLAKL A8ILL5 eukaryotic initiation factor IKSAIRAAEATSTEECPVKMKLVAPPLYVLT A8IRV6 eukaryotic initiation factor GRLGVRELKMLESRACNAISWSPQGRIIVLA A8IW20 eukaryotic initiation factor GSDKPLQFRQILLNTCQDEFEGAAEAREALV A8JBN7 eukaryotic initiation factor 5 78 AQSGTGKSSMISVSTCQLVDVQQRECQVLIL A8JDV9 F1F0 ATP synthase gamma subunit GKQNITVAVSSDRGLCGGLNSNIAKYTRALL A8JHB4 ferredoxin-dependent glutamate synthase KQALTALGCMEHRGACSADDDSGDGAGLMTQ 901 SIDQVEPAAAIMERFCTGGMSLGAISRETHE 1344 KVKLTLTGSGGQSFGCFcVKGLEVKLVGEAN A8J6Y8 ferredoxin-nadp reductase 8 83 RSVEKITGPKATGETCHIIIETEGKIPFWEG 143 SSRYGDDFDGQTASLCVRRAVYVDPETGKED 165 VDPETGKEDPAKKGLCSNFLCDATPGTEISM 170 GKEDPAKKGLCSNFLCDATPGTEISMTGPTG Q84X68 flagella membrane glycoprotein 1B IIAAANSTAAKAISGCVLSADGKALLVTLGA 1199 VLEVKAGTTAKGLASCSLAGTTLTVKLLGNN 1397 PFTSTVNGATLTKATCDTIVEVLNGGDATKP 2180 IKMQLPLTSNLGTVNCATTGIKVRVDDATDK 2810 SATGTITTTPATVDDCNKIVTFTPAKTLAAT 2828 IVTFTPAKTLAATSPCSVAGTTLTVNLDKAT Q6JIY9 flagellar adenylate kinase RALDQAAQFESSIMPCKTVLFFDCPEEEMEK A8HW56 flagellar associated protein GDTVLLKGKKRKDTVCIVLSDDTVDENKIRM 737 ENPDAMMEDEPDPVPCITKAHFEEAMKYARR A8I495 flagellar associated protein 8 54 NLLTKVGVPAENFPFCTIDPNNARVNVPDDR 184 LKSRGLKKEQLEELECAGRILEHLNKEIDVR
7 A8I748 flagellar associated protein TVKLPVDFPAAVASGCAKTEGLLASDVSCDT A8I7W0 flagellar associated protein CLAVASAAPLRNSADCVNVDLLKVTGVNVFP A8JG73 flagellar associated protein 1 85 GKMLLSAGDKTVALLCNVPKELQESYPHVKA A8IXD1 flagellar associated protein, adenosine kinase-like protein KANGCRPRVVVFTQGCDPTIVAVGGRVSRYP A8IKQ0 fructose-1,6-bisphosphatase MDDPQKMMEQCVMNVCQPGSRLKCAGYCLYS A8JCY4 fructose-1,6-bisphosphate aldolase RFAKWRAVVKIGEAGCPSTTAVLENAHGLAR Q42690 fructose-1,6-bisphosphate aldolase 6 58 SKGRGILAMDESNATCGKRLDSIGVENTEEN X 142 DKGLVPLSNTNGESWCMGLDGLDKRCAEYYK 256 EGILLKPAMVTPGADCKNKAGPAKVAEYTLK X A8JBY6 gamma tubulin interacting protein LDKGAGDVSEVQLNSCLRQAFTATNVVGGSS A8J3F9 GDP-D-mannose pyrophosphorylase GWDSKVGAWSRLENHCVLGEDVTVKDELYLN A8HNE8 geranylgeranyl reductase 9 93 RKLRVAVIGGGPSGACAAETLAKGGVETFLL 437 VFYRSNPAREAFVELCEDSYVQKMTFDSYLY A8JHE3 global transcription factor QEAEAAVKAGQYAMICLSDRGYGPNRAPVPS 1242 ITLGCIMMRKCHTNTCPVGVATQDPELRAKF A8IWA6 glutamate synthase, NADH-dependent QEAEAAVKAGQYAMICLSDRGYGPNRAPVPS 1237 ITLGCIMMRKCHTNTCPVGVATQDPELRAKF 1973 GKKVVVIGGGDTGTDCIATSLRHGATSIVNL Q39566 glutamate-1-semialdehyde aminotransferase ALKAQIDKGTSFGAPCELENVLAKMVIDRVP 287 LCFDEVMTGFRIAKGCAQEHFGITPDLTTMG E9N731 glutamyl-trna reductase binding protein AAAVEMSNLKSGVNSCSLMVQAATQPARAVG A8I7Q4 glutaredoxin, CGFS type 4 72 KASIAKDKGTPDSPQCGFSRMACVVLNAYGV A8IYH1 glutaredoxin, CPYC type 3 71 IAGKYTVVELENRADCDAMQDALLDITGGRS A8JFT3 glyceraldehyde 3-phosphate dehydrogenase APVKDTPAVLNVVVGCNDDKYDPAVDHIVTA
8 311 TDYVNDARSSIVDAACTQVIDGTMVKIYAWY A8JHS0 glyceraldehyde 3-phosphate dehydrogenase, minor splicing variant DEDVVSSDFVTDPASCTVDAKAGIMLSPTFV A8IYT1 glyceraldehyde 3-phosphate dehydrogenase, nonphosphorylating ISNPSTRQTAYQVQACTQDEVNKMFESAKVA A8IXZ1 glycerate kinase GQKTALVLGISAPQGCGKTTIVEQLELLFNW A8IVM9 glycine cleavage system, P protein KFLVSSKcHPQTIAVCQTRAEGLGLEAVVVD A8IWJ3 glycine cleavage system, T protein DGKVVGEITSGAFSPCLKKNIAMGYVDKDFA A8IZS5 glycine-rich RNA-binding protein 2 60 GFGFVTLEANAAKSACSECDGTEFMGRTIRV 63 FVTLEANAAKSACSECDGTEFMGRTIRVNEA B2XYE5 guanine nucleotide-binding protein beta subunit-like KYTIGEPEGHTEWVSCVRFSPMTTNPIIVSG A8JEU4 heat shock protein 70A EVEEFEHHLKELEGVCNPIITRLYQGGAGAG Q39603 heat shock protein 70B KHIDTQLTRAKFEEMCNDLLERCKVPVQQAL A8J3P5 heat shock protein 70E APKKKKVKKTDVPVQCVGAcGYSKSQLDDFF 560 KKVKKTDVPVQCVGACGYSKSQLDDFFEKEC A8J1U1 heat shock protein 90A EKKRKEELASQFEPLCRLMKDILGDKVEKVM Q66T67 heat shock protein 90C RVEKVTVSNRLLDSPCALVTSKFGWSANMER A8JE98 heterogeneous nuclear ribonucleoprotein 1 29 MAGGVQGQIGPDGFPCVRLRGLPFDVMEGDI A8I3J7 homoserine dehydrogenase AAEADAAGGVVRYVGCVDAENGNASVTLRTY A8I826 homoserine kinase MMAVKEAAKAAGAYGCTISGAGPTAVAIVDD A8J841 hydroxymethylpyrimidine phosphate synthase KAEDLTFRQAPTIADCFPDSEKMYKEVKYDD 218 RAIIPANKcHLELEPCVIGRNFMTKVNSNFG 581 PAKTAHFCSMCGPKFCSMNITQELRQMVQAE A8J3Y3 hypothetical protein CHLREDRAFT_ GGAAAAAAARDDNPPCNTLFVGNLSDSVDEN
9 A8JGP5 Ribulose bisphosphate carboxylase small chain 4 64 AYVSNESAIRFGSVSCLYYDNRYWTMWKLPM Q84X75 CR051 protein 3 54 LTKTTAREYSGRTITCNAVAPGFIASDMTAA A8II85 hypothetical protein CHLREDRAFT_ ARAKQQRITPDVQTGCIScASGDGK A8IS20 hypothetical protein CHLREDRAFT_ LEGWRAAGLDVQGIVCDVSDRAQRGELAAKV A8JBL6 hypothetical protein CHLREDRAFT_ DIPLKQKIFADLEKACRPDAILSTNTSTIDI Q84X77 CR027 protein QRSPPRPAGGSSAGGCGcGSSGGGGGGCKSG A8I720 hypothetical protein CHLREDRAFT_ NMRRLDSLRHRVRVLCPSVEANEITTEAEAR A8IH45 hypothetical protein CHLREDRAFT_ YVLKGAQRTSGSTQSCCPAGNIAATLPSGRR 268 VLKGAQRTSGSTQSCCPAGNIAATLPSGRRL A8ISA5 hypothetical protein CHLREDRAFT_ SPMFKKGWKKTIIDMCYSPPVDGAKAVIHAA A8JIU3 hypothetical protein CHLREDRAFT_ AKRIESITWHLKDRGCDNFDVVEKATTSPAS A8IRL5 hypothetical protein CHLREDRAFT_ SAAATEGASNAVPADCVSPEGRMLVFTLADS A8HVL0 hypothetical protein CHLREDRAFT_ WEVTARDFADKADEVCVQSVDDIGKVYKKVQ A8IJD8 hypothetical protein CHLREDRAFT_ STVSLARSTVPGEDVCIGAGTSIGEGSRVVQ A8INI0 hypothetical protein CHLREDRAFT_ ADLDLGYNEIKDDGACALAQALKANPEGAPR A8IT30 hypothetical protein CHLREDRAFT_ AELAKEHEARAAGGECGGGTGEAEcPRVRLV A8J654 hypothetical protein CHLREDRAFT_ AIDAVLARSTGPTGDCYSFERGN A8J7Z6 hypothetical protein CHLREDRAFT_ IPFDATVRISLDTNLCMIKENPEDGPSCAAT A8J725 hypothetical protein CHLREDRAFT_ QELIPLVKFMGNTGGCEPQAWSKDPPLVYIN A8J3W1 hypothetical protein CHLREDRAFT_ APMLTDEEQAAEDAACAKVIDTVFNAAKPAS
10 A8J538 hypothetical protein CHLREDRAFT_ RTALADTPANRFVNDCTNLVQEQRYEEYVTR A8J5B8 hypothetical protein CHLREDRAFT_ STIKAAELEAVGSAVCAKVHTVTHPGDLWLK X 248 TALEAFHHMALDHKSCLGIVDNTGKLIGSVS X Q6PLP7 hypothetical protein CHLREDRAFT_ WQPCKADGSGFETEGCGELTCDGKTADHGNL 639 ADGSGFETEGCGELTCDGKTADHGNLRRKGT A8HWI6 hypothetical protein CHLREDRAFT_ IGSDLRFGVQLQTMTCDTPKVYKIHDKLYLG A8JAG1 hypothetical protein CHLREDRAFT_ KVALLEKETFPRDKYCGDAVCTPAIRILEDM 107 EKETFPRDKYCGDAVCTPAIRILEDMGVLQE 216 VTSATGTVVRGRVLVCADGATSRLATKLGYC 231 CADGATSRLATKLGYCTEAPKGICSRAFIEG A8JAP7 Inosine-5'-monophosphate dehydrogenase SKLYELKNTRGFTSVCVTDTGALGGKLLGVV 326 AGADGLRVGMGSGSICTTQEVCAVGRGQATA 332 RVGMGSGSICTTQEVCAVGRGQATAVYHVAR A8JAQ6 hypothetical protein CHLREDRAFT_ KTLEEVVHEAGAASGCPKAKGNLLYTVASKY A8JFZ2 hypothetical protein CHLREDRAFT_ ADLYGGIGDVSATGDCAGDAKALCKDVNSGD A8JJD2 hypothetical protein CHLREDRAFT_ GLLNRSRDIMRQGATCFPDDQVEAKKALARW A8IZ88 hypothetical protein CHLREDRAFT_ KALIEAMRRGASVAECVQVIESAGGSK A8JG58 hypothetical protein CHLREDRAFT_ VKAKLEAGQTATVSNCDGQTFEIKAGMVEIK A8ISA8 hypothetical protein CHLREDRAFT_ AAGSGGAAATSSGGDCRVLIYGGYSGEAVEG A8J742 hypothetical protein CHLREDRAFT_ TVIRQKQAIRDELQSCDATRESIRAEMRSIR A8INX1 inactive subunit of chloroplast ClpP complex MKFTGMNQDQIEQATCRDHFMTPEQAKLEGL Q93Y52 inorganic pyrophosphatase WKVIAISADDPKAALCNDVEDVEKHFPGEIQ A8IXE2 intraflagellar transport particle protein SKKTQAGGTDVFAVACTDGSVKILSRTGRVE
11 Q39577 isocitrate lyase NAYELMKALIEAGASCVHFEDQLASAKKCGH 247 DKPFMTGERTAEGFYCVRGGIDAAIARGLAY 301 AAIHAQYPGKLLAYNCSPSFNWKKLSADEIS A8JG03 isopropylmalate dehydratase, large subunit GSAIEGMNMDERMTICNMVVEAGGKNGVIAP 402 TQKVWADVYTMPVPGCDGKTAAEIFEESGCI 444 GGPRDTFARMNEPEVCVSTTNRNFPGRMGHK X A8HYG8 isopropylmalate dehydratase, small subunit MKTEYPIIIGGQNFGCGSSREHAPVALGAAG A8HXS9 isopropylmalate synthase ANSLAGAQAGARQIECTINGIGERAGNASLE A8JG56 L-ascorbate peroxidase EAAIKEVVTTEDGPLCVRLVLNDAADWDPVT A8IT07 LCIB-like gene RIARRLAYEKINPLDCSLVDVTKAAERVISA A2SY49 light-harvesting chlorophyll-a/b binding protein Lhcb GAEFYRNTETDPEKRCYPGGVFDPLKLASED A2SY45 light-harvesting chlorophyll-a/b binding protein LhcbM2b 1 76 LELIHARWAMLGALGCITPELLAKNGIPFGE Q75NZ1 low-co2 inducible protein LCIC LMAARSLADVPPGDICAGQRGSVLQEIPYGY A8J7C8 lysyl-trna synthetase, LysRS NSDLDAEGYAQVHNECKRGDIVGVEGFPGKS 502 NMTERFELFINKREVCNAYTELNDPLKQREL A8IMZ5 magnesium chelatase subunit I QGEVNYDFRVKISQICSDLNVDGIRGDIVTN P93106 malate dehydrogenase PVKVTGYTGPEELGACLKGADLIVIPAGVPR A8IL08 membrane AAA-metalloprotease QEVPETGVVFDDVAGCDGAKLELQEVVDFLK A8J3U9 mitochondrial ATP synthase subunit 5, OSCP subunit LAYEELMLAHKKEVHCTVVTAQPLDDAERAV A8HTX7 mitochondrial F1F0 ATP synthase associated 31.2 kda protein AAFLEDVKKVQWFDACVAENPAVGPKVTA Q96550 mitochondrial F1F0 ATP synthase, alpha subunit ARVYGLKSVQAGELVCFDSGVKGMALNLQAD 527 TKGYLDKVPVNQITACEDVILKHVDPRLFKI
12 A8J236 molybdopterin biosynthesis protein GPAPRDVTPEATEAVCSRMMPGYGEQMRAIS Q6VPQ2 MPBQ/MSBQ methyltransferase DPNLKVVDVGGGTGFCTLGVVKTVKPENVTL A8J980 mrna export protein 6 90 ASAWKNDGSGIFLAGCDKAVRLWDLASNQAV A8JGS3 MYG1/GAMM1-like protein YCVYQDDRDKSYRVQCVSVGPGSFENRRSLP A8HWC2 NAD malic enzyme VMGLPPAALGQQRIVCVGAGSAGMGVARMIA A8JCP5 NAD(P) transhydrogenase VEALAKRRATAIGMDCIPRTISRAQMFDSLS Q6V9A9 NADH:ubiquinone oxidoreductase 11 kda subunit 2 53 EMKKANPHFPILIRECAGTEASLTARYDFGA A8IHL3 NADH:ubiquinone oxidoreductase ND9 subunit HLLSPRWNNRIRVKVCVDEVTAVPTLCKVFN 195 RVKVCVDEVTAVPTLCKVFNTANWFERETWD P93104 NADP-malate dehydrogenase 3 74 ESVSRNCKVLVVGNPCNTNALIAMENAPNIP A8J544 nucleolar protein, component of C/D snornps SRYLANKCSIASRIDCFMDGNTNVFGEKMRE A8I9L3 nucleoredoxin MSDKPADVICGDVCRRVTPEEAAAR Q39580 outer dynein arm light chain KNADMSEEMQADAVDCATQALEKYNIEKDIA B1B601 Parkin-co-regulated gene product 3 35 AGKAPPAANLSGTGSCFDTTSLSPARAGAHK A8HUU9 peptidyl-prolyl cis-trans isomerase, cyclophilin-type GSRSGETAFDVMVADCGELPAGAARAAAPRQ A8IVQ7 peptidyl-prolyl cis-trans isomerase, cyclophilin-type 8 95 FMIQGGDFVKGDGTGCVSIYGSRFPDENFIA A8JD64 peptidyl-prolyl cis-trans isomerase, cyclophilin-type GSSSGKTRATIAIADCGQLA A8HN08 PfkB-type carbohydrate kinase 9 30 EKLRTEKLETQGGGNCANALTAAARLGLYPT A8J0N7 phosphoenolpyruvate carboxykinase, splice variant 9 52 RFTNNTKQDAAVPAPCDMQFVLDSKFTRESG
13 A8J8Z2 phosphoglucomutase WKFFGNLMDAGKcSVCGEESFGTGGDHIREK 439 KQYGRNFFSRYDYEECASADADKMVAHLRDV Q94KV1 phosphoglyceromutase VEELEAVLAELRGKGCDIAIASGGGRMQVTM P19824 phosphoribulokinase 5 47 DKDKTVVIGLAADSGCGKSTFMRRMTSIFGG X Q39615 photosystem I reaction center subunit II, 20 kda MRQGPNLLKFGKKEQCLALTTQLRNKFKLTP B2X290 photosystem I subunit VII [Chlamydomonas moewusii] 9 11 MAHVVKIYDTCIGCTQCVRACPLDVL 14 MAHVVKIYDTCIGCTQCVRACPLDVL 17 AHVVKIYDTCIGCTQCVRACPLDVLEMPV 54 MASAPRTEDCVGCKRCETACPTDFLSVRVYL 58 PRTEDCVGCKRCETACPTDFLSVRVYLGSES Q6J213 phytoene desaturase SRSPLLSVYADMSTTCKEYYDTEKSMLELVF Q93Z22 plasma membrane-type proton ATPase ARMSAVEEMAGMDILCSDKTGTLTLNKLSID 516 LFDPPRHDTKDTIEYCHGQGIEVKMVTGDHL A8I8Z4 plastid ribosomal protein L KPPGAKGVYWKSMYVCTTMGPSLRIGVSALQ A8JAL6 plastid ribosomal protein L EKRKGRGYAAGQGGTCGFGNRGQKARSGPSV A8J3Z3 plastid ribosomal protein L KEGIHPQWFEEAKVICNGVEVMTVGGTKATY A8IYS1 plastid ribosomal protein L IADAIYQQTGRNVSDCEITVPEIKSVGTYEC Q70DX8 plastid ribosomal protein S DDGAYVEIGAKAIAFCPVTEcSFARLKTPLE A8J8M5 plastid ribosomal protein S ctaiigngnglvgvgcqagrevatavkralv A8JCR1 PP2A-twin subunit DKTVKLWKVYEKKVSCLSNFNLEGRNNGGTA A8HN94 predicted protein 4 80 SLTLTHKGAEVVAQLCTKSLGGLTENDFIVA A8HNF0 predicted protein AGAARVIAVDGSAGACDSARRIVRANGLDDR
14 A8HP72 predicted protein 6 76 KTQVKRALETYVKTDCPSTTAPSSTVWRCMS A8HPV5 predicted protein 1 55 APLDAARMTVDAAGTCAEAARGITQYVPKPK A8HRV2 predicted protein TPMTEFRLESVPDMNCDALDAKTPKGEVLIR A8IBN3 predicted protein YIPETGRGIQGATSHCLGQNFSKMFNITYED A8IFH0 predicted protein TRLAAIEDMAGMNMLCSDKTGTLTLNKMAIQ A8IK94 predicted protein YTYKSAREALEDDPECSNPETQALARFSGAE A8ITK6 predicted protein DVFTVAGARGQNLVECPNCKADMIFDEYKRV A8ITU2 predicted protein 7 98 DLPKGYQISQYDEPICSGGRLEVEVDGVMKR A8IV25 predicted protein QALPQFQKGLGTPGLCAAEGEWVEPKKDAGK A8IVG4 predicted protein RDMVTAGQVEEARALCVAQVDEYVGKIASDF A8IXZ2 predicted protein LVRFATDNESIGAVICKRADQLQASCVVMAK A8IYA3 predicted protein 2 90 TSEMWHPNVYDDGKVCISILHSPGDDPSGYE A8IYA5 predicted protein KAADKAREAAELRAGCEAAVAADQPADVAKL A8IYX9 predicted protein 1 30 LTGVTRVEVQDTSGGCGAMYRIAIQAADFKG X A8IYZ6 predicted protein 3 29 KPANFLVDRAWKVKVCDFGLASNSKAQAGAG A8IZZ6 predicted protein 4 42 NGGRILYPGSDLPTACPVTEDKEWRASHDHA A8J0B3 predicted protein 8 63 ASVSFEQPDEASLVSCRFIVPHHVTKPGQQL 285 PLYEGSGDIQAKVVICNADGSPAVWEPGTTN A8J1C4 predicted protein 3 83 DELVLACRDKLSSGGCSPDGLSKVVSALPLL A8J2L0 predicted protein AGEGADGEAPAGDFACGDCDRTFASQQGLRR
15 117 GADGEAPAGDFACGDCDRTFASQQGLRRHLQ A8JD33 predicted protein EFLIKGAPGSTAELLCNASAGDPVSVSPVMG A8JDL8 predicted protein 6 52 SMHGLPTDRPKIERLCADIQSQHGVRVAYSE A8JDU0 predicted protein GSSSTYAITHYFNKACPSQWSAVAGSPATK A8JDV3 predicted protein LDLLEEVPYAELKALCAEAGLSAAGKKAELQ A8JF87 predicted protein ATVTKTGAGVDVALICDGSGAGRSGKDKKDV A8JFA1 predicted protein 4 93 VYGRQMGGVPEESEGCAVcKVDTGLGGQPCY A8JGW7 predicted protein PGVDVHDEAAVKDKLCADIAAAGDGDDASAA A8JH69 predicted protein SEVLFLETNSERYQLCNVSISDDPKGKDLFT A8JHA6 predicted protein 4 97 NLEEYEAAKEAFEAGCALAPDNTFKTWIRKC A8JHB7 predicted protein AQYLFQRRGPLATTGCDHGAFVRTSSSLSQP A8JHF3 predicted protein AVRAVRHRALEQAKLCNTGGPAGELKRMEQQ A8JHJ9 predicted protein TNMARMNGANGVSFGCVNTAAPVDPKEEPKKE A8JJI9 predicted protein 1 25 DLENVSRSAALINQSCHVRVLDIRKFLDGIY Q84X70 CR084 protein GVEITQAALDLGAEECSKRTTDAMRDAHKKS A8HXC5 prephenate dehydratase AQIVARQGLQGVGAICSRRAAELYGLDVLEE A8HPV3 presequence protease LKELQETPDSPEALACIPALKLSDIPKTITK A8IS98 PRLI-interacting factor L QLEAEPQPNQPAPGACPGQAARSVYLSIAES A8HXI4 protein phosphatase 2A regulatory subunit 9 24 LPSMVAYSLFKRLTGCEAGEEEGAAVPRDTF Q8LKK4 protofilament ribbon protein of flagellar microtubules 4 17 SGGAPPIPKLPGYTVCLPQSLSDKGFKKGQT
16 A8HWA8 pyridine nucleotide binding protein PAEFEAAIGSANKIVCAVGAAEDQALNFSAP A8IW49 pyruvate phosphate dikinase DAAEARKFGAEGIGLCRTEHMFFASEERIAT VERGRAARPDLKIGICGEQGGEPRTVAFLHE Q27YU3 radial spoke protein GSGGATLTESQVTGLCQQAGIADAVVAKVME Q39586 RecName: Full=5-methyltetrahydropteroyltriglutamate--homocystein PILIGPVTFVSLSRGCELPLDQAVARLLPTY Q9SW75 RecName: Full=60S ribosomal protein L10a 4 72 VPRPRMRVRAGDVKHCEQAGAIGVDAKGVED P12853 RecName: Full=Oxygen-evolving enhancer protein 1, chloroplastic; S 3 74 GLTYLQVKGSGIANTCPVLESGTTNLKELKA 98 NLKELKAGSYKLENFCIEPTSFTVKEESQFK Q6R2V6 RecName: Full=Phosphoenolpyruvate carboxylase 2; Short=PEP car GVRRDRATREPNPNSCDAVFARLITEGVDPE P41758 RecName: Full=Phosphoglycerate kinase, chloroplastic; Flags: Precu TVSIANTLAGLTPKGCITIIGGGDSVAAVEQ X P23489 RecName: Full=Ribulose bisphosphate carboxylase/oxygenase activa TRYREASDIIKKGRMCSLFINDLDAGAGRMG 289 EKYYWNPTREDRIGVCMGIFQEDNVQRREVE P46284 RecName: Full=Sedoheptulose-1,7-bisphosphatase, chloroplastic; Al CDGKAVSALDIPILVCDQRTQICYGSIGEVR 362 ALDIPILVCDQRTQICYGSIGEVRRFEEYMY P09205 RecName: Full=Tubulin alpha-2 chain TVVPGVDLAKVQRAVCMISNSTAIGEIFSRL A8J597 ribosomal protein L12, component of cytosolic 80S ribosome and MPPKFDPSDVVEVCVRVTGGEVGAASSLA 142 ANGCKEILGTAVSVGCKVDGKDPRDVISAID A8IKZ2 ribosomal protein L18, component of cytosolic 80S ribosome and FTETARARITKAGGECLTFDQLALQRPTGKD A8HN50 ribosomal protein L18a, component of cytosolic 80S ribosome and RKLRRVKKANGQIIACNEIFERKPTLAKNYG A8JI94 ribosomal protein L22, component of cytosolic 80S ribosome and AKGAAKKKALVFTIDCSKPVEDKIMDISSFE A8J9W0 ribosomal protein L23, component of cytosolic 80S ribosome and KFRMALGLPVAAVMNCADNTGAKNLYVISVV 57 VVGWGSRLNKLPAAACGDMVMASVKKGKPDL
17 A8J239 ribosomal protein L23a, component of cytosolic 80S ribosome and VNTLIRPDGQKKAYVCLTADYDALDVANKIG A8ICT1 ribosomal protein L30, component of cytosolic 80S ribosome and GVHHYGGNNVDLGTACGKYYRVSVLAITDPG Q8GUQ9 ribosomal protein L38, component of cytosolic 80S ribosome and TTKFKVRCSKYLYTLCVADSDKADKLKQSLP A8IVK1 ribosomal protein L8, component of cytosolic 80S ribosome and 60S GNVKPLGDMPEGTIVCNVEEKAGDRGALARC A8JHC3 ribosomal protein S11, component of cytosolic 80S ribosome and PCFRVREGDSVVIGQCRPLSKTVRFNVLRVT A8IGY1 ribosomal protein S13, component of cytosolic 80S ribosome and PSWCKTTGAEVQEMICKFARKGMTPSQIGIV A8HVP2 ribosomal protein S18, component of cytosolic 80S ribosome and VCKKAEVDLRKRAGECSADELERMMGIVANP A8HME4 ribosomal protein S2, component of cytosolic 80S ribosome and 40S ARTPKKVLQMAGIEDCYTCSRGSTKTLGNFV 211 PKKVLQMAGIEDCYTCSRGSTKTLGNFVRAT A8IS22 ribosomal protein S26, component of cytosolic 80S ribosome and RNIVDASALRDMQEACVVDNYALPKIYRKVY A8HVK4 ribosomal protein S27a, component of cytosolic 80S ribosome and 4 5 YKVDDSGKVQRLRKQCPNcGPGTFMATHFDR A8HS48 ribosomal protein S3a, component of cytosolic 80S ribosome and AKFIPESIGKDIEKSCQGIYPLQNTFIRKVK A8J2I5 ribosomal protein S5, component of cytosolic 80S ribosome and 40S GAREAAFRNIKTIAECLADELVNAAKGSSNR X X A8J1G8 ribosomal protein S6, component of cytosolic 80S ribosome and 40S YGRRKGERRRKSVRGCIVSPDLAVLNLVIVK X A8IB25 ribosomal protein Sa, component of cytosolic 80S ribosome and 40S 5 18 LSQKEQDIQMMLAAQCHLGTKNCHYQMERYM A8I9I9 rieske ferredoxin 5 KTALFTAEVKDKCVICPAHGTAFDVKTGEVK A8IBR0 RNA binding rrna processing protein VVVRSAGDKRNVLVGCGPGDVAGAVTKEADA A8IXE0 S-Adenosyl homocysteine hydrolase LEIDLAEAEMPGLMACRSEFGPAQPFKGAKI X A8HYU5 S-Adenosylmethionine synthetase 9 23 LYTSESVNEGHPDKICDQVSDAVLDACLEQD 34 PDKICDQVSDAVLDACLEQDPDSKVACETCC 76 TTKAKVDYEAIVRKVCKEIGFTSEDVGLDAD
18 93 EIGFTSEDVGLDADKCKVLVHIEEQSPDIGQ A8JFZ0 serine glyoxylate aminotransferase VHNETTTGVTSDIAGCRKAMDAAGHPALLLV A8JFK4 serine hydroxymethyltransferase 2 7 DLRPEGVTGSKMEKACDLCHITLNKNAVVGD A8J4R9 serine hydroxymethyltransferase SENFTSKAVMQALGSCMTNKYSEGRPNARYY 341 NTEEFRTYQKQVVANCSALCGRLQQHGYKIV A8JHT4 serine/threonine protein kinase LAKGSAETGMVEAYMCAKVKRNPLVASSCAE A8I848 seryl trna synthetase 2 49 MPGLQRYGEISSASNCTDYQSRRLNIRYSGG A8IRT6 seryl-trna synthetase EAWFPASATFRELVSCSNcLDYQSRRLDVRL A8HNU4 signal peptide peptidase-like protein KATSRQVADATADAACNAYDRIADQEALQRE A8I9S6 spliceosome component, nuclear pre-mrna splicing factor 9 3 MFCSISGTVPEQGVVSTK A8J4I2 SR protein factor GGPGGPGGREMRcYECGEIGHIARDCRNMRG A8IJ34 sterol-c24-methyltransferase FKENTFDGAYAIEATCHAPKLEQVYGEIYRV B2XYH3 striated fiber assemblin 1 24 DIEHLATSLVGKVNECVAAIDEERNGRVQEQ A8JIJ1 subunit of exon junction complex DECDKMLEKLDMRADCQEIFKLTPHEKQVMM A8HQF0 subunit of retromer complex GSEHRDALTAAATAGCMRLVARREQCRALCA A8JH66 subunit of the signal recognition particle KVKPRDSFEVAFNRSCGLVEAGQLASAEGEL A8HP06 succinate dehydrogenase subunit A LRAAVGLSELGFNTACITKLFPTRSHTVAAQ 260 GGYGRAYFSATSAHTCTGDGNAMAARAGIPL A8IQ09 succinate-coa ligase beta chain, minor isoform AKQIKALYTLFDKSDCTMVEVNPLAEGLDGS A8II42 T-complex protein 1, gamma subunit EIKESDIGTGAGLFECVKIGDEFFTFIVDCK 454 VGVAMEVIPRTLAQNCGANVIRTLTKLRAKH
19 A8J7J2 T-complex protein, epsilon subunit KKRQLEEKNPYLGIDCNDTGTNDMREQNVFE 520 TVMGKKQQLFLATQVCKMILKIDDVIKPSDY A8IYP6 thioredoxin h FLKVDVDELADVAGECGVKAMPTFIGYFNGE Q84XS0 thioredoxin o 4 67 SAKLADVAGSGSLMICDFTAKWCGPCRMIAP A8HZZ1 threonine synthase MCKELGLVDKMPRLVCAQAQNANPLYQAFSK 472 QSKVAYHSKQVPGMTCQFANPPVQVREDLGA A8J1M9 thylakoid lumenal 17.4 kda protein VWEDALIGSQDVGKLCENPTLTGESRAQVGC A8IAN1 transketolase TTIALSRQNMPNIPNCSVEGVAKGAYTIHDT C0LL59 translation elongation factor-like protein DFIKNMISGAAQADVCLLMVPADGNFTTAIQ A8ISB6 transmembrane protein of CMA family YKFSGSGTNTTLTGSCLLSKDFTNQDYCNYA Q5S7Y5 triose phosphate isomerase DKLRIIYGGSVSDTNCKDLSKQEDIDGFLVG X 273 LVGGASLKGAAFVTICNAAGPKAKP O64991 tryptophan synthase beta AETGAGQHGVATATICARLGLKCIVYMGAKD A2PZB5 UDP-glucose 4-epimerase like protein HVSAVVKTLATPDLGCTPINLGTGKGTSVLE A2PZC3 UDP-glucose dehydrogenase IGPKFLNASVGFGGSCFQKDILNLCYVCESV A2PZC2 UDP-Glucose:protein transglucosylase GRYDDMWAGWCTKVICDHLGVGCKTGLPYVW A8IW47 vacuolar ATP synthase subunit E CCGGVVLISGDGRINCSNTLDDRLKIAYQAN X X A8I164 vacuolar ATP synthase, subunit A IAIDSGDVFIPRGVACPSLDIVKQWEFQPKG 304 MTLPDGREESIMKRTCLVANTSNMPVAAREA A8J1K0 vacuolar proton translocating ATPase subunit A NDFGEPITNTIDPRDCQMVYEGVLKMPPDSA A8I647 zeta-carotene desaturase DYIDSMEGATLSGRQCAYSILNATPGIQKSL
Supplementary Table 2. Gossypol-induced Decrease of Levels of Reduced Cysteine-containing Peptides
 Supplementary Table 2. Gossypol-induced Decrease of Levels of Reduced Cysteine-containing Peptides Accession Description Sequences of Cys-containing peptides IPI00021263 14-3-3 protein zeta/delta DICNDVLSLLEK
Supplementary Table 2. Gossypol-induced Decrease of Levels of Reduced Cysteine-containing Peptides Accession Description Sequences of Cys-containing peptides IPI00021263 14-3-3 protein zeta/delta DICNDVLSLLEK
Architecture of the Trypanosome RNA Editing Accessory Complex, MRB1
 Architecture of the Trypanosome RNA Editing Accessory Complex, MRB1 Michelle L. Ammerman, Kurtis M. Downey, Hassan Hashimi, John C. Fisk, Danielle L. Tomasello, Drahomíra Faktorová, Lucie Hanzálková, Tony
Architecture of the Trypanosome RNA Editing Accessory Complex, MRB1 Michelle L. Ammerman, Kurtis M. Downey, Hassan Hashimi, John C. Fisk, Danielle L. Tomasello, Drahomíra Faktorová, Lucie Hanzálková, Tony
Glyma10g15850 aagggatccattctggaaccatatcttgctgtg ttgggtacccttggatgcaggatgacacg AtMKK6, AT5g56580
 Supplemental table 1. Soybean MAPK, MAPKK, and MAPKKK genes and primers for VIGS cloning Gene_ID Forward Primer Reverse Primer Arabidopsis Ortholog Glyma04g03210 aagggatcctgcagcaagaaccagttcat ttgggtaccctaatggatgcgcattaggataaag
Supplemental table 1. Soybean MAPK, MAPKK, and MAPKKK genes and primers for VIGS cloning Gene_ID Forward Primer Reverse Primer Arabidopsis Ortholog Glyma04g03210 aagggatcctgcagcaagaaccagttcat ttgggtaccctaatggatgcgcattaggataaag
Genetikai panel kialakítása a hazai tejhasznú szarvasmarha állományok hasznos élettartamának növelésére
 Genetikai panel kialakítása a hazai tejhasznú szarvasmarha állományok hasznos élettartamának növelésére Dr. Czeglédi Levente Dr. Béri Béla Kutatás-fejlesztés támogatása a megújuló energiaforrások és agrár
Genetikai panel kialakítása a hazai tejhasznú szarvasmarha állományok hasznos élettartamának növelésére Dr. Czeglédi Levente Dr. Béri Béla Kutatás-fejlesztés támogatása a megújuló energiaforrások és agrár
Nan Wang, Qingming Dong, Jingjing Li, Rohit K. Jangra, Meiyun Fan, Allan R. Brasier, Stanley M. Lemon, Lawrence M. Pfeffer, Kui Li
 Supplemental Material IRF3-dependent and NF- B-independent viral induction of the zinc-finger antiviral protein Nan Wang, Qingming Dong, Jingjing Li, Rohit K. Jangra, Meiyun Fan, Allan R. Brasier, Stanley
Supplemental Material IRF3-dependent and NF- B-independent viral induction of the zinc-finger antiviral protein Nan Wang, Qingming Dong, Jingjing Li, Rohit K. Jangra, Meiyun Fan, Allan R. Brasier, Stanley
kpis(ppk20) kpis(ppk46)
 Table S1. C. elegans strains used in this study Strains carrying transgenes Strain Transgene Genotype Reference IT213 tcer-1 prom:tcer-1orf:gfp:tcer-1 3'utr unc-119(ed3) III; kpis(ppk6) This study IT283
Table S1. C. elegans strains used in this study Strains carrying transgenes Strain Transgene Genotype Reference IT213 tcer-1 prom:tcer-1orf:gfp:tcer-1 3'utr unc-119(ed3) III; kpis(ppk6) This study IT283
Horizontal gene transfer drives adaptive colonization of apple trees by the fungal pathogen Valsa mali. Zhiyuan Yin, Baitao Zhu, Hao Feng, Lili Huang*
 Supplementary Information Horizontal gene transfer drives adaptive colonization of apple trees by the fungal pathogen Valsa mali Zhiyuan Yin, Baitao Zhu, Hao Feng, Lili Huang* State Key Laboratory of Crop
Supplementary Information Horizontal gene transfer drives adaptive colonization of apple trees by the fungal pathogen Valsa mali Zhiyuan Yin, Baitao Zhu, Hao Feng, Lili Huang* State Key Laboratory of Crop
SUPPLEMENTARY INFORMATION
 doi:10.1038/nature14095 Supplementary Table 1: MAGE oligonucleotides. Name Sequence Strategy acpp.high.1.ny t*g*cttaacgcccagctgttcgccgataattttcttaacgcgttcctagatagtgctcatactcttaaatttcctatcaaaactcgctttc
doi:10.1038/nature14095 Supplementary Table 1: MAGE oligonucleotides. Name Sequence Strategy acpp.high.1.ny t*g*cttaacgcccagctgttcgccgataattttcttaacgcgttcctagatagtgctcatactcttaaatttcctatcaaaactcgctttc
Akt1 Akt kinase activity Creb signaling CCTTACAGCCCTCAAGTACTCATTC GGCGTACTCCATGACAAAGCA Arc Actin binding
 Additional File 1 (Table S1): Description and primer sequences of 80 candidate genes tested as potential synaptic plasticity formation genes in qrt-pcr array. Abbreviations: LTP: long-term potentiation,
Additional File 1 (Table S1): Description and primer sequences of 80 candidate genes tested as potential synaptic plasticity formation genes in qrt-pcr array. Abbreviations: LTP: long-term potentiation,
Supporting Information
 Supporting Information Cell-free GFP simulations Cell-free simulations of degfp production were consistent with experimental measurements (Fig. S1). Dual emmission GFP was produced under a P70a promoter
Supporting Information Cell-free GFP simulations Cell-free simulations of degfp production were consistent with experimental measurements (Fig. S1). Dual emmission GFP was produced under a P70a promoter
Az izomváltozások szabályozása a smoothelin-like 1 fehérje által. Tudományos hétfő előadássorozat. Lontay Beáta Orvosi Vegytani Intézet
 Az izomváltozások szabályozása a smoothelin-like 1 fehérje által Tudományos hétfő előadássorozat Lontay Beáta Orvosi Vegytani Intézet Kutatási témák: 1. A protein foszfatázok és kinázok szerepe a neurotranszmitter
Az izomváltozások szabályozása a smoothelin-like 1 fehérje által Tudományos hétfő előadássorozat Lontay Beáta Orvosi Vegytani Intézet Kutatási témák: 1. A protein foszfatázok és kinázok szerepe a neurotranszmitter
Diabéteszes redox változások hatása a stresszfehérjékre
 Semmelweis Egyetem Molekuláris Orvostudományok Tudományági Doktori Iskola Pathobiokémia Program Doktori (Ph.D.) értekezés Diabéteszes redox változások hatása a stresszfehérjékre dr. Nardai Gábor Témavezeto:
Semmelweis Egyetem Molekuláris Orvostudományok Tudományági Doktori Iskola Pathobiokémia Program Doktori (Ph.D.) értekezés Diabéteszes redox változások hatása a stresszfehérjékre dr. Nardai Gábor Témavezeto:
MMTV proviral insertion sites and associated genes
 Supplementary Table 1 (Theodorou et al.) : MMTV proviral insertion sites and associated genes Tumor Nr Gene ENSMUSG Sequence 11 1110012J17Rik ENSMUSG00000052105 GGCCGCTCTCCAATCTGAAGGAGTNAGGACAGGTG CTCGGCCAGNGAGAACCTGTACTTGGACGCTTTGT
Supplementary Table 1 (Theodorou et al.) : MMTV proviral insertion sites and associated genes Tumor Nr Gene ENSMUSG Sequence 11 1110012J17Rik ENSMUSG00000052105 GGCCGCTCTCCAATCTGAAGGAGTNAGGACAGGTG CTCGGCCAGNGAGAACCTGTACTTGGACGCTTTGT
13. RNS szintézis és splicing
 13. RNS szintézis és splicing 1 Visszatekintés: Az RNS típusai és szerkezete Hírvivő RNS = mrns (messenger RNA = mrna) : fehérjeszintézis pre-mrns érett mrns (intronok kivágódnak = splicing) Transzfer
13. RNS szintézis és splicing 1 Visszatekintés: Az RNS típusai és szerkezete Hírvivő RNS = mrns (messenger RNA = mrna) : fehérjeszintézis pre-mrns érett mrns (intronok kivágódnak = splicing) Transzfer
Modular Optimization of Hemicellulose-utilizing Pathway in. Corynebacterium glutamicum for Consolidated Bioprocessing of
 [Supplementary materials] Modular Optimization of Hemicellulose-utilizing Pathway in Corynebacterium glutamicum for Consolidated Bioprocessing of Hemicellulosic Biomass Sung Sun Yim 1, Jae Woong Choi 1,
[Supplementary materials] Modular Optimization of Hemicellulose-utilizing Pathway in Corynebacterium glutamicum for Consolidated Bioprocessing of Hemicellulosic Biomass Sung Sun Yim 1, Jae Woong Choi 1,
Pro- és antioxidáns hatások szerepe az endoplazmás retikulum eredetű stresszben és apoptózisban
 Pro- és antioxidáns hatások szerepe az endoplazmás retikulum eredetű stresszben és apoptózisban Az endoplazmás retikulum (ER) számos környezeti és metabolikus hatás szenzora. Mindazon tényezők, melyek
Pro- és antioxidáns hatások szerepe az endoplazmás retikulum eredetű stresszben és apoptózisban Az endoplazmás retikulum (ER) számos környezeti és metabolikus hatás szenzora. Mindazon tényezők, melyek
Supplementary Table 1. Cystometric parameters in sham-operated wild type and Trpv4 -/- rats during saline infusion and
 WT sham Trpv4 -/- sham Saline 10µM GSK1016709A P value Saline 10µM GSK1016709A P value Number 10 10 8 8 Intercontractile interval (sec) 143 (102 155) 98.4 (71.4 148) 0.01 96 (92 121) 109 (95 123) 0.3 Voided
WT sham Trpv4 -/- sham Saline 10µM GSK1016709A P value Saline 10µM GSK1016709A P value Number 10 10 8 8 Intercontractile interval (sec) 143 (102 155) 98.4 (71.4 148) 0.01 96 (92 121) 109 (95 123) 0.3 Voided
T-helper Type 2 driven Inflammation Defines Major Subphenotypes of Asthma
 T-helper Type 2 driven Inflammation Defines Major Subphenotypes of Asthma Prescott G. Woodruff, M.D., M.P.H., Barmak Modrek, Ph.D., David F. Choy, B.S., Guiquan Jia, M.D., Alexander R. Abbas, Ph.D., Almut
T-helper Type 2 driven Inflammation Defines Major Subphenotypes of Asthma Prescott G. Woodruff, M.D., M.P.H., Barmak Modrek, Ph.D., David F. Choy, B.S., Guiquan Jia, M.D., Alexander R. Abbas, Ph.D., Almut
Trinucleotide Repeat Diseases: CRISPR Cas9 PacBio no PCR Sequencing MFMER slide-1
 Trinucleotide Repeat Diseases: CRISPR Cas9 PacBio no PCR Sequencing 2015 MFMER slide-1 Fuch s Eye Disease TCF 4 gene Fuchs occurs in about 4% of the US population. Leads to deteriorating vision without
Trinucleotide Repeat Diseases: CRISPR Cas9 PacBio no PCR Sequencing 2015 MFMER slide-1 Fuch s Eye Disease TCF 4 gene Fuchs occurs in about 4% of the US population. Leads to deteriorating vision without
A fotoszintézis molekuláris biofizikája (Vass Imre, 2000) 39
 A fotoszintézis molekuláris biofizikája (Vass Imre, 2000) 39 6. A citokróm b 6 f komplex A két fotokémiai rendszer közötti elektrontranszportot a citokróm b 6 f komplex közvetíti. Funkciója a kétszeresen
A fotoszintézis molekuláris biofizikája (Vass Imre, 2000) 39 6. A citokróm b 6 f komplex A két fotokémiai rendszer közötti elektrontranszportot a citokróm b 6 f komplex közvetíti. Funkciója a kétszeresen
Supplemental Information. Investigation of Penicillin Binding Protein. (PBP)-like Peptide Cyclase and Hydrolase
 Cell Chemical Biology, Volume upplemental Information Investigation of Penicillin Binding Protein (PBP)-like Peptide Cyclase and ydrolase in urugamide on-ribosomal Peptide Biosynthesis Yongjun Zhou, Xiao
Cell Chemical Biology, Volume upplemental Information Investigation of Penicillin Binding Protein (PBP)-like Peptide Cyclase and ydrolase in urugamide on-ribosomal Peptide Biosynthesis Yongjun Zhou, Xiao
Supplementary materials to: Whole-mount single molecule FISH method for zebrafish embryo
 Supplementary materials to: Whole-mount single molecule FISH method for zebrafish embryo Yuma Oka and Thomas N. Sato Supplementary Figure S1. Whole-mount smfish with and without the methanol pretreatment.
Supplementary materials to: Whole-mount single molecule FISH method for zebrafish embryo Yuma Oka and Thomas N. Sato Supplementary Figure S1. Whole-mount smfish with and without the methanol pretreatment.
Supplemental Information. RNase H1-Dependent Antisense Oligonucleotides. Are Robustly Active in Directing RNA Cleavage
 YMTHE, Volume 25 Supplemental Information RNase H1-Dependent Antisense Oligonucleotides Are Robustly Active in Directing RNA Cleavage in Both the Cytoplasm and the Nucleus Xue-Hai Liang, Hong Sun, Joshua
YMTHE, Volume 25 Supplemental Information RNase H1-Dependent Antisense Oligonucleotides Are Robustly Active in Directing RNA Cleavage in Both the Cytoplasm and the Nucleus Xue-Hai Liang, Hong Sun, Joshua
A tömegspektrometria kvalitatív és kvantitatív proteomikai alkalmazása. Szájli Emília
 A tömegspektrometria kvalitatív és kvantitatív proteomikai alkalmazása Ph.D. értekezés tézisei Szájli Emília Témavezető: Dr. Medzihradszky-Fölkl Katalin Kémia Doktori Iskola Szegedi Tudományegyetem Magyar
A tömegspektrometria kvalitatív és kvantitatív proteomikai alkalmazása Ph.D. értekezés tézisei Szájli Emília Témavezető: Dr. Medzihradszky-Fölkl Katalin Kémia Doktori Iskola Szegedi Tudományegyetem Magyar
Receptor Tyrosine-Kinases
 Receptor Tyrosine-Kinases MAPkinase pathway PI3Kinase Protein Kinase B pathway PI3K/PK-B pathway Phosphatidyl-inositol-bisphosphate...(PI(4,5)P 2...) Phosphatidyl-inositol-3-kinase (PI3K) Protein kinase
Receptor Tyrosine-Kinases MAPkinase pathway PI3Kinase Protein Kinase B pathway PI3K/PK-B pathway Phosphatidyl-inositol-bisphosphate...(PI(4,5)P 2...) Phosphatidyl-inositol-3-kinase (PI3K) Protein kinase
Barangolások a peptidek és fehérjék világában
 Barangolások a peptidek és fehérjék világában Orbán Erika Kutató-fejlesztő, Biotechnológiai analitikai osztály, Richter Gedeon Vegyészeti Gyár Nyrt. Magyar Tudomány Ünnepe, Kémiai Tudományok Osztálya és
Barangolások a peptidek és fehérjék világában Orbán Erika Kutató-fejlesztő, Biotechnológiai analitikai osztály, Richter Gedeon Vegyészeti Gyár Nyrt. Magyar Tudomány Ünnepe, Kémiai Tudományok Osztálya és
A KAR-2, egy antimitotikus ágens egyedi farmakológiájának atomi és molekuláris alapjai
 A KAR-2, egy antimitotikus ágens egyedi farmakológiájának atomi és molekuláris alapjai A doktori értekezés tézisei Horváth István Eötvös Loránd Tudományegyetem Biológia Doktori Iskola (A Doktori Iskola
A KAR-2, egy antimitotikus ágens egyedi farmakológiájának atomi és molekuláris alapjai A doktori értekezés tézisei Horváth István Eötvös Loránd Tudományegyetem Biológia Doktori Iskola (A Doktori Iskola
Átlagtermés és rekordtermés 8 növénykultúrában
 Átlagtermés és rekordtermés 8 növénykultúrában Növény Rekord termés * Átlag termés * Veszteség típusa Biotikus Abiotikus Abiotikus stressz okozta kiesés (% a rekord terméshez képest) Kukorica 19300 4600
Átlagtermés és rekordtermés 8 növénykultúrában Növény Rekord termés * Átlag termés * Veszteség típusa Biotikus Abiotikus Abiotikus stressz okozta kiesés (% a rekord terméshez képest) Kukorica 19300 4600
Nukleotid anyagcsere
 Nukleotid anyagcsere Nukleotidok szerepe DNS és RNS prekurzorai Energiaközvetítők () Regulációs molekulák (camp, cgmp, ADP) Szénhidrát és lipidanyagcsre intermedierei (UDP-galaktóz Koenzimkomponensek (NAD,
Nukleotid anyagcsere Nukleotidok szerepe DNS és RNS prekurzorai Energiaközvetítők () Regulációs molekulák (camp, cgmp, ADP) Szénhidrát és lipidanyagcsre intermedierei (UDP-galaktóz Koenzimkomponensek (NAD,
Crash Course in Omics Terminology, Concepts & Data Types
 Crash Course in Omics Terminology, Concepts & Data Types 14 th Annual EuPathDB Workshop Jessie Kissinger June 2, 2019 FungiDB MicrosporidiaDB AmoebaDB PlasmoDB ToxoDB CryptoDB PiroplasmaDB TrichDB GiardiaDB
Crash Course in Omics Terminology, Concepts & Data Types 14 th Annual EuPathDB Workshop Jessie Kissinger June 2, 2019 FungiDB MicrosporidiaDB AmoebaDB PlasmoDB ToxoDB CryptoDB PiroplasmaDB TrichDB GiardiaDB
2011. október 11. Szabad János
 2011. október 11 Szabad János szabad@mdbio.szote.u-szeged.hu Egy állatsejt szervez dése - Export a sejtmagból a citoplazmába - Import a citoplazmából a sejtmagba - Import a sejtszervecskékbe - A szekréciós
2011. október 11 Szabad János szabad@mdbio.szote.u-szeged.hu Egy állatsejt szervez dése - Export a sejtmagból a citoplazmába - Import a citoplazmából a sejtmagba - Import a sejtszervecskékbe - A szekréciós
Új generációs sporttudományi képzés és tartalomfejlesztés, hazai és nemzetközi hálózatfejlesztés és társadalmasítás a Szegedi Tudományegyetemen
 Új generációs sporttudományi képzés és tartalomfejlesztés, hazai Pályázati azonosító: TÁMOP-4.1.2.E-15/1/Konv-2015-0002 3. alprojekt Sportélettani képzés és tartalomfejlesztés Pilot 1: Dinamikus változások
Új generációs sporttudományi képzés és tartalomfejlesztés, hazai Pályázati azonosító: TÁMOP-4.1.2.E-15/1/Konv-2015-0002 3. alprojekt Sportélettani képzés és tartalomfejlesztés Pilot 1: Dinamikus változások
tccattaattcgacagaccagagttaaataatccttgtatgccattgtgatcacatctacagttcagattttgtatttca
 Figure Legends for Supplementary Materials: Fig. 1. Nucleotide sequence of the zebrafish lumican (zlum) gene. Exons are indicated by uppercase letters, and introns are indicated by lowercase letters. The
Figure Legends for Supplementary Materials: Fig. 1. Nucleotide sequence of the zebrafish lumican (zlum) gene. Exons are indicated by uppercase letters, and introns are indicated by lowercase letters. The
ZÁRÓJELENTÉS. 1. A kutatási eredmények rövid összefoglalása
 ZÁRÓJELENTÉS a T038365 OTKA nyilvántartási számú Arabidopsis SCF-SnRK ubiquitin-ligáz komplexek szerkezetének vizsgálata és az alegységek funkcionális analízise című projektről 1. A kutatási eredmények
ZÁRÓJELENTÉS a T038365 OTKA nyilvántartási számú Arabidopsis SCF-SnRK ubiquitin-ligáz komplexek szerkezetének vizsgálata és az alegységek funkcionális analízise című projektről 1. A kutatási eredmények
(11) Lajstromszám: E 005 557 (13) T2 EURÓPAI SZABADALOM SZÖVEGÉNEK FORDÍTÁSA
 !HU000007T2! (19) HU (11) Lajstromszám: E 00 7 (13) T2 MAGYAR KÖZTÁRSASÁG Magyar Szabadalmi Hivatal EURÓPAI SZABADALOM SZÖVEGÉNEK FORDÍTÁSA (21) Magyar ügyszám: E 03 026690 (22) A bejelentés napja: 03.
!HU000007T2! (19) HU (11) Lajstromszám: E 00 7 (13) T2 MAGYAR KÖZTÁRSASÁG Magyar Szabadalmi Hivatal EURÓPAI SZABADALOM SZÖVEGÉNEK FORDÍTÁSA (21) Magyar ügyszám: E 03 026690 (22) A bejelentés napja: 03.
Table S1. Top and Bottom 10 most up- and down-regulated statistically significant transcripts (H 2 O 2 vs. untreated zebrafish larvae)
 Table S1. Top and Bottom 10 most up- and down-regulated statistically significant transcripts (H 2 O 2 vs. untreated zebrafish larvae) Rank Symbol logfc P- value Description 1 hsp70l 2.6 0.030 heat shock
Table S1. Top and Bottom 10 most up- and down-regulated statistically significant transcripts (H 2 O 2 vs. untreated zebrafish larvae) Rank Symbol logfc P- value Description 1 hsp70l 2.6 0.030 heat shock
Stresszfehérjék (hősokk fehérjék)
 Stresszfehérjék (hősokk fehérjék) Ama különféle fehérjék összefoglaló elnevezése, amelyeket az élő sejtek a megemelkedett hőmérsékletre (a hősokkra) válaszul fokozott mértékben termelnek. A hősokk-fehérjék
Stresszfehérjék (hősokk fehérjék) Ama különféle fehérjék összefoglaló elnevezése, amelyeket az élő sejtek a megemelkedett hőmérsékletre (a hősokkra) válaszul fokozott mértékben termelnek. A hősokk-fehérjék
Expression analysis of PIN genes in root tips and nodules of Lotus japonicus
 Article Expression analysis of PIN genes in root tips and nodules of Lotus japonicus Izabela Sańko-Sawczenko 1, Dominika Dmitruk 1, Barbara Łotocka 1, Elżbieta Różańska 1 and Weronika Czarnocka 1, * 1
Article Expression analysis of PIN genes in root tips and nodules of Lotus japonicus Izabela Sańko-Sawczenko 1, Dominika Dmitruk 1, Barbara Łotocka 1, Elżbieta Różańska 1 and Weronika Czarnocka 1, * 1
SOLiD Technology. library preparation & Sequencing Chemistry (sequencing by ligation!) Imaging and analysis. Application specific sample preparation
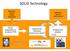 SOLiD Technology Application specific sample preparation Application specific data analysis library preparation & emulsion PCR Sequencing Chemistry (sequencing by ligation!) Imaging and analysis SOLiD
SOLiD Technology Application specific sample preparation Application specific data analysis library preparation & emulsion PCR Sequencing Chemistry (sequencing by ligation!) Imaging and analysis SOLiD
A doktori értekezés tézisei. A növényi NRP fehérjék lehetséges szerepe a hiszton defoszforiláció szabályozásában, és a hőstressz válaszban.
 A doktori értekezés tézisei A növényi NRP fehérjék lehetséges szerepe a hiszton defoszforiláció szabályozásában, és a hőstressz válaszban. Bíró Judit Témavezető: Dr. Fehér Attila Magyar Tudományos Akadémia
A doktori értekezés tézisei A növényi NRP fehérjék lehetséges szerepe a hiszton defoszforiláció szabályozásában, és a hőstressz válaszban. Bíró Judit Témavezető: Dr. Fehér Attila Magyar Tudományos Akadémia
Manuscript Title: Identification of a thermostable fungal lytic polysaccharide monooxygenase and
 1 2 3 4 5 Journal name: Applied Microbiology and Biotechnology Manuscript Title: Identification of a thermostable fungal lytic polysaccharide monooxygenase and evaluation of its effect on lignocellulosic
1 2 3 4 5 Journal name: Applied Microbiology and Biotechnology Manuscript Title: Identification of a thermostable fungal lytic polysaccharide monooxygenase and evaluation of its effect on lignocellulosic
Beszámoló a K 72569 OTKA Project keretében végzett munkáról. Szinopszis:
 Beszámoló a K 72569 OTKA Project keretében végzett munkáról Szinopszis: A 2008. április 1. és 2011. december 31. között végzett munka része volt az munkatársaimmal több, mint húsz éve végzett elméleti
Beszámoló a K 72569 OTKA Project keretében végzett munkáról Szinopszis: A 2008. április 1. és 2011. december 31. között végzett munka része volt az munkatársaimmal több, mint húsz éve végzett elméleti
University of Bristol - Explore Bristol Research
 Al-Salihi, S. A. A., Scott, T. A., Bailey, A. M., & Foster, G. D. (2017). Improved vectors for Agrobacterium mediated genetic manipulation of Hypholoma spp. and other homobasidiomycetes. Journal of Microbiological
Al-Salihi, S. A. A., Scott, T. A., Bailey, A. M., & Foster, G. D. (2017). Improved vectors for Agrobacterium mediated genetic manipulation of Hypholoma spp. and other homobasidiomycetes. Journal of Microbiological
Crash Course in Omics Terminology, Concepts & Data Types
 Crash Course in Omics Terminology, Concepts & Data Types 12 th Annual EuPathDB Workshop: 2017 Jessie Kissinger June 18, 2017 FungiDB MicrosporidiaDB AmoebaDB PlasmoDB ToxoDB CryptoDB PiroplasmaDB TrichDB
Crash Course in Omics Terminology, Concepts & Data Types 12 th Annual EuPathDB Workshop: 2017 Jessie Kissinger June 18, 2017 FungiDB MicrosporidiaDB AmoebaDB PlasmoDB ToxoDB CryptoDB PiroplasmaDB TrichDB
Phenotype. Genotype. It is like any other experiment! What is a bioinformatics experiment? Remember the Goal. Infectious Disease Paradigm
 It is like any other experiment! What is a bioinformatics experiment? You need to know your data/input sources You need to understand your methods and their assumptions You need a plan to get from point
It is like any other experiment! What is a bioinformatics experiment? You need to know your data/input sources You need to understand your methods and their assumptions You need a plan to get from point
(AD) β (A ) (2) ACDP ACDP ACDP APP. APP-C99 γ (3) A 40 A 42 A A A A 42/A 40. in vivo A A A A
 (AD) β (A) A AAPP APP-C99 γ A A40 A42 A AA42/A40 γ A A γ AD A A A A APP APP APP A A A A A A (1)AD A42 A AD AD (2) ACDP AACDP ACDP A A ACDP (3) (1)A in vivo A APP Gray and Whittaker 1962; Perez-Otano et
(AD) β (A) A AAPP APP-C99 γ A A40 A42 A AA42/A40 γ A A γ AD A A A A APP APP APP A A A A A A (1)AD A42 A AD AD (2) ACDP AACDP ACDP A A ACDP (3) (1)A in vivo A APP Gray and Whittaker 1962; Perez-Otano et
Supplemental Table S1. Overview of MYB transcription factor genes analyzed for expression in red and pink tomato fruit.
 Supplemental Table S1. Overview of MYB transcription factor genes analyzed for expression in red and pink tomato fruit. MYB Primer pairs TC AtMYB Forward Reverse TC199266 MYB12 AGGCTCTTGGAGGTCGTTACC CAACTCTTTCCGCATCTCAATAATC
Supplemental Table S1. Overview of MYB transcription factor genes analyzed for expression in red and pink tomato fruit. MYB Primer pairs TC AtMYB Forward Reverse TC199266 MYB12 AGGCTCTTGGAGGTCGTTACC CAACTCTTTCCGCATCTCAATAATC
Mágikus lövedék; hogyan állítsunk az irányzékon
 Mágikus lövedék; hogyan állítsunk az irányzékon Mező Gábor ELTE TTK, Kémiai Intézet ELTE-MTA Peptidkémiai Kutatócsoport Chemotaxis Workshop 2018. 12. 07. Egy kis kitérő az operák világába Carl Maria von
Mágikus lövedék; hogyan állítsunk az irányzékon Mező Gábor ELTE TTK, Kémiai Intézet ELTE-MTA Peptidkémiai Kutatócsoport Chemotaxis Workshop 2018. 12. 07. Egy kis kitérő az operák világába Carl Maria von
Markerless Escherichia coli rrn Deletion Strains for Genetic Determination of Ribosomal Binding Sites
 Markerless Escherichia coli rrn Deletion Strains for Genetic Determination of Ribosomal Binding Sites Selwyn Quan *, Ole Skovgaard, Robert E. McLaughlin, Ed T. Buurman,1, and Catherine L. Squires * Department
Markerless Escherichia coli rrn Deletion Strains for Genetic Determination of Ribosomal Binding Sites Selwyn Quan *, Ole Skovgaard, Robert E. McLaughlin, Ed T. Buurman,1, and Catherine L. Squires * Department
Supporting Information
 Supporting Information Paired design of dcas9 as a systematic platform for the detection of featured nucleic acid sequences in pathogenic strains Yihao Zhang 1,2,8, Long Qian 4,8, Weijia Wei 1,3,7,8, Yu
Supporting Information Paired design of dcas9 as a systematic platform for the detection of featured nucleic acid sequences in pathogenic strains Yihao Zhang 1,2,8, Long Qian 4,8, Weijia Wei 1,3,7,8, Yu
TRANSZPORTFOLYAMATOK 1b. Fehérjék. 1b. FEHÉRJÉK TRANSZPORTJA A MEMBRÁNONOKBA ÉS A SEJTSZERVECSKÉK BELSEJÉBE ÁLTALÁNOS
 1b. FEHÉRJÉK TRANSZPORTJA A MEMBRÁNONOKBA ÉS A SEJTSZERVECSKÉK BELSEJÉBE ÁLTALÁNOS DIA 1 Fő fehérje transzport útvonalak Egy tipikus emlős sejt közel 10,000 féle fehérjét tartalmaz (a test pedig összesen
1b. FEHÉRJÉK TRANSZPORTJA A MEMBRÁNONOKBA ÉS A SEJTSZERVECSKÉK BELSEJÉBE ÁLTALÁNOS DIA 1 Fő fehérje transzport útvonalak Egy tipikus emlős sejt közel 10,000 féle fehérjét tartalmaz (a test pedig összesen
Crash Course in Omics Terminology, Concepts & Data Types
 Crash Course in Omics Terminology, Concepts & Data Types 13 th Annual EuPathDB Workshop Jessie Kissinger June 17th, 2018 FungiDB MicrosporidiaDB AmoebaDB PlasmoDB ToxoDB CryptoDB PiroplasmaDB TrichDB GiardiaDB
Crash Course in Omics Terminology, Concepts & Data Types 13 th Annual EuPathDB Workshop Jessie Kissinger June 17th, 2018 FungiDB MicrosporidiaDB AmoebaDB PlasmoDB ToxoDB CryptoDB PiroplasmaDB TrichDB GiardiaDB
AGRÁRIUM vs BIOÜZEMANYAGOK. VERSENY VAGY PARTNERSÉG?
 A Szegedi Tudományegyetem quadruplehelix modell alapú gazdasági- és társadalmi pozicionálása, a tudástranszfer gyakorlatának kialakítása Hódmezővásárhely-Szeged kiemelt növekedési zónában c. projekt keretében
A Szegedi Tudományegyetem quadruplehelix modell alapú gazdasági- és társadalmi pozicionálása, a tudástranszfer gyakorlatának kialakítása Hódmezővásárhely-Szeged kiemelt növekedési zónában c. projekt keretében
University of Bristol - Explore Bristol Research
 Tan, B. G., Wellesley, F. C., Savery, N. J., & Szczelkun, M. D. (2016). Length heterogeneity at conserved sequence block 2 in human mitochondrial DNA acts as a rheostat for RNA polymerase POLRMT activity.
Tan, B. G., Wellesley, F. C., Savery, N. J., & Szczelkun, M. D. (2016). Length heterogeneity at conserved sequence block 2 in human mitochondrial DNA acts as a rheostat for RNA polymerase POLRMT activity.
Dr. Csala Miklós. Tudományos Publikációk Jegyzéke
 Dr. Csala Miklós Tudományos Publikációk Jegyzéke A közlemények száma kategóriánként közlemények egyszerzős első- / utolsószerzős társszerzős összesen könyvfejezet - 3 1 4 tankönyvfejezet, egyetemi jegyzet
Dr. Csala Miklós Tudományos Publikációk Jegyzéke A közlemények száma kategóriánként közlemények egyszerzős első- / utolsószerzős társszerzős összesen könyvfejezet - 3 1 4 tankönyvfejezet, egyetemi jegyzet
Biológus MSc. Molekuláris biológiai alapismeretek
 Biológus MSc Molekuláris biológiai alapismeretek A nukleotidok építőkövei A nukleotidok szerkezete Nukleotid = N-tartalmú szerves bázis + pentóz + foszfát N-glikozidos kötés 5 1 4 2 3 (Foszfát)észter-kötés
Biológus MSc Molekuláris biológiai alapismeretek A nukleotidok építőkövei A nukleotidok szerkezete Nukleotid = N-tartalmú szerves bázis + pentóz + foszfát N-glikozidos kötés 5 1 4 2 3 (Foszfát)észter-kötés
7. előadás: A plazma mebrán szerkezete és funkciója. Anyagtranszport a plazma membránon keresztül.
 7. előadás: A plazma mebrán szerkezete és funkciója. Anyagtranszport a plazma membránon keresztül. A plazma membrán határolja el az élő sejteket a környezetüktől Szelektív permeabilitást mutat, így lehetővé
7. előadás: A plazma mebrán szerkezete és funkciója. Anyagtranszport a plazma membránon keresztül. A plazma membrán határolja el az élő sejteket a környezetüktől Szelektív permeabilitást mutat, így lehetővé
Biotranszformáció. Csala Miklós. Semmelweis Egyetem Orvosi Vegytani, Molekuláris Biológiai és Patobiokémiai Intézet
 Biotranszformáció Csala Miklós Semmelweis Egyetem rvosi Vegytani, Molekuláris Biológiai és Patobiokémiai Intézet direkt bilirubin hem (porfirin) X koleszterin X epesavak piruvát acil-koa citoplazma piruvát
Biotranszformáció Csala Miklós Semmelweis Egyetem rvosi Vegytani, Molekuláris Biológiai és Patobiokémiai Intézet direkt bilirubin hem (porfirin) X koleszterin X epesavak piruvát acil-koa citoplazma piruvát
pjnc-pgluc Codon-optimized Gaussia luciferase (pgluc) Vector backbone: JM83 pjnc-pgluc is resistant to ampicillin and neomycin High or low copy:
 pjnc-pgluc Gene/Insert name: Codon-optimized Gaussia luciferase (pgluc) Vector backbone: pcmv-jnc Vector type: Mammalian cells Backbone size w/o insert (bp): 5375 Bacterial resistance: Ampicillin and neomycin
pjnc-pgluc Gene/Insert name: Codon-optimized Gaussia luciferase (pgluc) Vector backbone: pcmv-jnc Vector type: Mammalian cells Backbone size w/o insert (bp): 5375 Bacterial resistance: Ampicillin and neomycin
A MITOKONDRIÁLIS CITOKRÓM C POSZTTRANSZLÁCIÓS ÉRÉSE. Doktori (Ph.D.) értekezés tézisei. Tenger Katalin
 A MITOKONDRIÁLIS CITOKRÓM C POSZTTRANSZLÁCIÓS ÉRÉSE Doktori (Ph.D.) értekezés tézisei Tenger Katalin Témavezető: Dr. Zimányi László Konzulens: Dr. Rákhely Gábor Biológia Doktori Iskola Szegedi Tudományegyetem
A MITOKONDRIÁLIS CITOKRÓM C POSZTTRANSZLÁCIÓS ÉRÉSE Doktori (Ph.D.) értekezés tézisei Tenger Katalin Témavezető: Dr. Zimányi László Konzulens: Dr. Rákhely Gábor Biológia Doktori Iskola Szegedi Tudományegyetem
BEVEZETÉS Az objektum fogalma
 BEVEZETÉS Az objektum fogalma Program (1) Adat (2) Objektum Kiadványszerkesztés Word Táblázatkezelés Excel CAD AutoCad Adatbáziskezelés Access 1 Program (1) Adat (2) Objektum Adatmodell (2) A valós világ
BEVEZETÉS Az objektum fogalma Program (1) Adat (2) Objektum Kiadványszerkesztés Word Táblázatkezelés Excel CAD AutoCad Adatbáziskezelés Access 1 Program (1) Adat (2) Objektum Adatmodell (2) A valós világ
Genome 373: Hidden Markov Models I. Doug Fowler
 Genome 373: Hidden Markov Models I Doug Fowler Review From Gene Prediction I transcriptional start site G open reading frame transcriptional termination site promoter 5 untranslated region 3 untranslated
Genome 373: Hidden Markov Models I Doug Fowler Review From Gene Prediction I transcriptional start site G open reading frame transcriptional termination site promoter 5 untranslated region 3 untranslated
A humán tripszinogén 4 expressziója és eloszlási mintázata az emberi agyban
 A humán tripszinogén 4 expressziója és eloszlási mintázata az emberi agyban Doktori (PhD) értekezés Siklódi Erika Rozália Biológia Doktori Iskola Iskolavezető: Prof. Erdei Anna, tanszékvezető egyetemi
A humán tripszinogén 4 expressziója és eloszlási mintázata az emberi agyban Doktori (PhD) értekezés Siklódi Erika Rozália Biológia Doktori Iskola Iskolavezető: Prof. Erdei Anna, tanszékvezető egyetemi
Biokatalitikus Baeyer-Villiger oxidációk Doktori (PhD) értekezés tézisei. Muskotál Adél. Dr. Vonderviszt Ferenc
 Biokatalitikus Baeyer-Villiger oxidációk Doktori (PhD) értekezés tézisei Készítette: Muskotál Adél Környezettudományok Doktori Iskola Témavezető: Dr. Vonderviszt Ferenc egyetemi tanár Pannon Egyetem Műszaki
Biokatalitikus Baeyer-Villiger oxidációk Doktori (PhD) értekezés tézisei Készítette: Muskotál Adél Környezettudományok Doktori Iskola Témavezető: Dr. Vonderviszt Ferenc egyetemi tanár Pannon Egyetem Műszaki
HUMAN IMMUNODEFICIENCY VIRUS (HIV) ÉS AIDS
 HUMAN IMMUNODEFICIENCY VIRUS (HIV) ÉS AIDS Dr. Mohamed Mahdi MD. MPH. Department of Infectology and Pediatric Immunology University of Debrecen 2010 Történelmi tények a HIV-ről 1981: Első megjelenés San
HUMAN IMMUNODEFICIENCY VIRUS (HIV) ÉS AIDS Dr. Mohamed Mahdi MD. MPH. Department of Infectology and Pediatric Immunology University of Debrecen 2010 Történelmi tények a HIV-ről 1981: Első megjelenés San
Extracelluláris vezikulák szerepe autoimmun betegségekben. Pállinger Éva
 Extracelluláris vezikulák szerepe autoimmun betegségekben Pállinger Éva Az extracelluláris vezikulák osztályozása György-Szabó et al. Cell Mol Life Sci 2011. Exoszómák képződése Mikrovezikulák képződése
Extracelluláris vezikulák szerepe autoimmun betegségekben Pállinger Éva Az extracelluláris vezikulák osztályozása György-Szabó et al. Cell Mol Life Sci 2011. Exoszómák képződése Mikrovezikulák képződése
Computational Neuroscience
 Computational Neuroscience Zoltán Somogyvári senior research fellow KFKI Research Institute for Particle and Nuclear Physics Supporting materials: http://www.kfki.hu/~soma/bscs/ BSCS 2010 Lengyel Máté:
Computational Neuroscience Zoltán Somogyvári senior research fellow KFKI Research Institute for Particle and Nuclear Physics Supporting materials: http://www.kfki.hu/~soma/bscs/ BSCS 2010 Lengyel Máté:
Fehérjeglikoziláció az endoplazmás retikulumban mint lehetséges daganatellenes támadáspont
 Fehérjeglikoziláció az endoplazmás retikulumban mint lehetséges daganatellenes támadáspont Doktori tézisek Dr. Konta Laura Éva Semmelweis Egyetem Molekuláris Orvostudományok Tudományági Doktori Iskola
Fehérjeglikoziláció az endoplazmás retikulumban mint lehetséges daganatellenes támadáspont Doktori tézisek Dr. Konta Laura Éva Semmelweis Egyetem Molekuláris Orvostudományok Tudományági Doktori Iskola
Az Oxidatív stressz hatása a PIBF receptor alegységek összeszerelődésére.
 Újabban világossá vált, hogy a Progesterone-induced blocking factor (PIBF) amely a progesteron számos immunológiai hatását közvetíti, nem csupán a lymphocytákban és terhességgel asszociált szövetekben,
Újabban világossá vált, hogy a Progesterone-induced blocking factor (PIBF) amely a progesteron számos immunológiai hatását közvetíti, nem csupán a lymphocytákban és terhességgel asszociált szövetekben,
Szakmai önéletrajz. Tanulmányok: Tudományos minısítés:
 Dr. Benyó Zoltán, Egyetemi Tanári Pályázat 2009, Szakmai Önéletrajz, 1. oldal Szakmai önéletrajz Név: Dr. Benyó Zoltán Születési hely, idı: Budapest, 1967. június 15. Családi állapot: nıs, 2 gyermek (Barnabás,
Dr. Benyó Zoltán, Egyetemi Tanári Pályázat 2009, Szakmai Önéletrajz, 1. oldal Szakmai önéletrajz Név: Dr. Benyó Zoltán Születési hely, idı: Budapest, 1967. június 15. Családi állapot: nıs, 2 gyermek (Barnabás,
Mai témák. Fehérjék dinamikájának jelentősége. Számítógépes modellezés jelentősége
 Mai témák Fehérjék szerkezetének predikciója, szerkezeti adatok felhasználása adatbázisok segítségével, a számítógépes molekuladinamikai modellezés alapjai Hegedűs Tamás tamas@hegelab.org Bevezetés szimulációk
Mai témák Fehérjék szerkezetének predikciója, szerkezeti adatok felhasználása adatbázisok segítségével, a számítógépes molekuladinamikai modellezés alapjai Hegedűs Tamás tamas@hegelab.org Bevezetés szimulációk
Szénhidrátanyagcsere. net
 Szénhidrátanyagcsere net Glukogén prekurzorok belépése a glukoneogenezisbe glikogén glukóz-1p glukóz-6p G6P-áz glukóz F1,6bP-áz fruktóz-1,6biszp fruktóz-6p glicerinaldehid-3p + dioh-aceton-p 1,3-biszfoszfo-glicerát
Szénhidrátanyagcsere net Glukogén prekurzorok belépése a glukoneogenezisbe glikogén glukóz-1p glukóz-6p G6P-áz glukóz F1,6bP-áz fruktóz-1,6biszp fruktóz-6p glicerinaldehid-3p + dioh-aceton-p 1,3-biszfoszfo-glicerát
STEREOCHEMISTRY COOH C* H CH 3 COOH H 3 C COOH COOH CH 2 OH O C
 * STEEEMISTY D-glyceraldehyde D-glicerinaldehid 2 * 2 L-glyceraldehyde -lactic acid L-glicerinaldehid -tejsav * 3 S-lactic acid S-tejsav 3 * meso-tartaric acid mezo-borkősav * * * * D-erythrose D-eritróz
* STEEEMISTY D-glyceraldehyde D-glicerinaldehid 2 * 2 L-glyceraldehyde -lactic acid L-glicerinaldehid -tejsav * 3 S-lactic acid S-tejsav 3 * meso-tartaric acid mezo-borkősav * * * * D-erythrose D-eritróz
A cell-based screening system for RNA Polymerase I inhibitors
 Electronic Supplementary Material (ESI) for MedChemComm. This journal is The Royal Society of Chemistry 2019 Supporting Information A cell-based screening system for RNA Polymerase I inhibitors Xiao Tan,
Electronic Supplementary Material (ESI) for MedChemComm. This journal is The Royal Society of Chemistry 2019 Supporting Information A cell-based screening system for RNA Polymerase I inhibitors Xiao Tan,
PIAL1 (At1g08910) ORF sequence
 PIAL1 (At1g08910) ORF sequence ATGGTTATTCCGGCGACTTCTAGGTTTGGGTTTCGTGCTGAATTCAACACCAAGGAGTTTCAAGCTTCCTGCATCTCTCT! CGCTAATGAAATCGATGCGGCTATTGGGAGAAATGAAGTTCCAGGGAATATTCAAGAGCTCGCTTTAATCCTCAATAATG! TGTGCCGACGTAAATGTGATGATTATCAAACCAGGGCGGTGGTAATGGCGCTGATGATCTCAGTCAAGAGTGCTTGTCAG!
PIAL1 (At1g08910) ORF sequence ATGGTTATTCCGGCGACTTCTAGGTTTGGGTTTCGTGCTGAATTCAACACCAAGGAGTTTCAAGCTTCCTGCATCTCTCT! CGCTAATGAAATCGATGCGGCTATTGGGAGAAATGAAGTTCCAGGGAATATTCAAGAGCTCGCTTTAATCCTCAATAATG! TGTGCCGACGTAAATGTGATGATTATCAAACCAGGGCGGTGGTAATGGCGCTGATGATCTCAGTCAAGAGTGCTTGTCAG!
 ü É Í ü ü ü Í ü ű ü ü ü ű ü ű ű ű ü ü ü ű ü Í ü ű ü ü ü Ű Í É É Á Ő Á Ó Á Á Á Á É Á Á Á Á É Á Í Á Á Í Í ű Á É É Á Á Ö Í Á Á Á Á Á É Á Á Ó ű Í ü ü ü ű ű ü ü ű ü Á ü ű ü Í Í Í ü Í Í ű ű ü ü ü ü ű ü ű ü ü
ü É Í ü ü ü Í ü ű ü ü ü ű ü ű ű ű ü ü ü ű ü Í ü ű ü ü ü Ű Í É É Á Ő Á Ó Á Á Á Á É Á Á Á Á É Á Í Á Á Í Í ű Á É É Á Á Ö Í Á Á Á Á Á É Á Á Ó ű Í ü ü ü ű ű ü ü ű ü Á ü ű ü Í Í Í ü Í Í ű ű ü ü ü ü ű ü ű ü ü
 Í Á Á É ö ö ö ö ö ű ü ö ű ű ű ö ö ö ü ö ü í ü í í í ü í ü Á ü ö ö ü ö ü ö ö ü ö í ö ö ü ö ü í ö ü ű ö ü ö ü í ö í ö ű ű ö ö ú ö ü ö ű ű ű í ö ű í ű ö ű ü ö í ű í í ö í ö ö Ó Í ö ű ű ű ű í í ű ű í í Ü ö
Í Á Á É ö ö ö ö ö ű ü ö ű ű ű ö ö ö ü ö ü í ü í í í ü í ü Á ü ö ö ü ö ü ö ö ü ö í ö ö ü ö ü í ö ü ű ö ü ö ü í ö í ö ű ű ö ö ú ö ü ö ű ű ű í ö ű í ű ö ű ü ö í ű í í ö í ö ö Ó Í ö ű ű ű ű í í ű ű í í Ü ö
 Ű Í ó Ü Ö Á Á Ó Ö Ü Ü Ü Ü Á Í Ü Á Á Ü Ü Ü Ü Ü Ü Ö Ü Í Ü Ü Ü Ü Ü Ü Ü Ü Ü Ü Á Í Ü Í Í Á Í Í Ü Í Í Ü Á Ü Ü Ü Ü Ü Ü Ü Ü Ő Ö Á ÁÍ Á Ü Ü Á Í Ü Í Á Ü Á Í ó Í Í Ü Ü ő Í Ü Ű Ü Ü Ü Ü Í Ü Ü Ü Ü Ü Ü Ü Í Ü Á Ü Ö Á
Ű Í ó Ü Ö Á Á Ó Ö Ü Ü Ü Ü Á Í Ü Á Á Ü Ü Ü Ü Ü Ü Ö Ü Í Ü Ü Ü Ü Ü Ü Ü Ü Ü Ü Á Í Ü Í Í Á Í Í Ü Í Í Ü Á Ü Ü Ü Ü Ü Ü Ü Ü Ő Ö Á ÁÍ Á Ü Ü Á Í Ü Í Á Ü Á Í ó Í Í Ü Ü ő Í Ü Ű Ü Ü Ü Ü Í Ü Ü Ü Ü Ü Ü Ü Í Ü Á Ü Ö Á
 ű í ú ü Á ü ü ü ü ü É É É Ü í ü Á í í ű í ú É É É Ü Í í í í Á í í Á í Á Í É Ő Ú ú Ú í í í íí í ú í í Í í Í Í É í í Í Í í ú í ü Ó í Í ú Í Í ű í ű í í í Í É Ü ű í ü ű í ú É É É Ü ű í í í í ü í Í í Ú Í í
ű í ú ü Á ü ü ü ü ü É É É Ü í ü Á í í ű í ú É É É Ü Í í í í Á í í Á í Á Í É Ő Ú ú Ú í í í íí í ú í í Í í Í Í É í í Í Í í ú í ü Ó í Í ú Í Í ű í ű í í í Í É Ü ű í ü ű í ú É É É Ü ű í í í í ü í Í í Ú Í í
AGE SUN «YEUX» ELÉRHETŐ: 2009 JÚNIUS
 AGE SUN «YEUX» ELÉRHETŐ: 2009 JÚNIUS AGE SUN YEUX Segít megakadályozni a szemkörnyéki területek öregedését iért szükséges specifikus fényvédelem a szemkörnyéki területre? A hámréteg különbözik az arcbőrétől
AGE SUN «YEUX» ELÉRHETŐ: 2009 JÚNIUS AGE SUN YEUX Segít megakadályozni a szemkörnyéki területek öregedését iért szükséges specifikus fényvédelem a szemkörnyéki területre? A hámréteg különbözik az arcbőrétől
A TRICHODERMA REESEI GOMBA ÉS S GENETIKÁJA
 A TRICHODERMA REESEI GOMBA CELLULÁZ Z ENZIM TERMELÉSÉNEK BIOKÉMI MIÁJA ÉS S GENETIKÁJA KARAFFA LEVENTE Debreceni Egyetem Természettudom szettudományi és s Technológiai Kar Genetikai és s Alkalmazott Mikrobiológiai
A TRICHODERMA REESEI GOMBA CELLULÁZ Z ENZIM TERMELÉSÉNEK BIOKÉMI MIÁJA ÉS S GENETIKÁJA KARAFFA LEVENTE Debreceni Egyetem Természettudom szettudományi és s Technológiai Kar Genetikai és s Alkalmazott Mikrobiológiai
Suppl. Materials. Polyhydroxyalkanoate (PHA) Granules Have no Phospholipids. Germany
 Suppl. Materials Polyhydroxyalkanoate (PHA) Granules Have no Phospholipids Stephanie Bresan 1, Anna Sznajder 1, Waldemar Hauf 2, Karl Forchhammer 2, Daniel Pfeiffer 3 and Dieter Jendrossek 1 1 Institute
Suppl. Materials Polyhydroxyalkanoate (PHA) Granules Have no Phospholipids Stephanie Bresan 1, Anna Sznajder 1, Waldemar Hauf 2, Karl Forchhammer 2, Daniel Pfeiffer 3 and Dieter Jendrossek 1 1 Institute
MELLÉKLET / ANNEX. EU MEGFELELŐSÉGI NYILATKOZAT-hoz for EU DECLARATION OF CONFORMITY
 TRACON BUDAPEST KFT. TRACON BUDAPEST LTD. 2120 DUNAKESZI, PALLAG U. 23. TEL.: (27) 540-000, FAX: (27) 540-005 WWW.TRACON.HU EU MEGFELELŐSÉGI NYILATKOZAT, a 79/1997. (XII. 31.) IKIM számú és a 374/2012.
TRACON BUDAPEST KFT. TRACON BUDAPEST LTD. 2120 DUNAKESZI, PALLAG U. 23. TEL.: (27) 540-000, FAX: (27) 540-005 WWW.TRACON.HU EU MEGFELELŐSÉGI NYILATKOZAT, a 79/1997. (XII. 31.) IKIM számú és a 374/2012.
Vezikuláris transzport
 Molekuláris Sejtbiológia Vezikuláris transzport Dr. habil KŐHIDAI László Semmelweis Egyetem, Genetikai, Sejt- és Immunbiológiai Intézet 2005. november 3. Intracelluláris vezikul uláris transzport Kommunikáció
Molekuláris Sejtbiológia Vezikuláris transzport Dr. habil KŐHIDAI László Semmelweis Egyetem, Genetikai, Sejt- és Immunbiológiai Intézet 2005. november 3. Intracelluláris vezikul uláris transzport Kommunikáció
Az internet ökoszisztémája és evolúciója. Gyakorlat 4
 Az internet ökoszisztémája és evolúciója Gyakorlat 4 Tartományok közti útválasztás konfigurálása: alapok Emlékeztető: interfészkonfiguráció R1 R2 link konfigurációja R1 routeren root@openwrt:/# vtysh OpenWrt#
Az internet ökoszisztémája és evolúciója Gyakorlat 4 Tartományok közti útválasztás konfigurálása: alapok Emlékeztető: interfészkonfiguráció R1 R2 link konfigurációja R1 routeren root@openwrt:/# vtysh OpenWrt#
1. likmliiklexpokoflob. nmaihafqaholaflinnofwetbopfcuflitibfdobftercufdorpjbtjbjbmatmaikmliikletisuloikof KOFn`FtIHOFtIHomBlOfnBlinkbF:
 tilin\kirhafqabfcr HOlAFtIbFdObFTERtPFjAIkLOB timbhotoftijaililefkwerxebxpvbpe12/4/2014n`flob. ymaipfcuflitibfdobfterxpvbddpjbnofholaftjbxhorpkbfmbtimnublef. tihn`btofxhrlitimbxhrhikapeleftkaxpokoftkai:
tilin\kirhafqabfcr HOlAFtIbFdObFTERtPFjAIkLOB timbhotoftijaililefkwerxebxpvbpe12/4/2014n`flob. ymaipfcuflitibfdobfterxpvbddpjbnofholaftjbxhorpkbfmbtimnublef. tihn`btofxhrlitimbxhrhikapeleftkaxpokoftkai:
Apoptózis. 1. Bevezetés 2. Külső jelút 3. Belső jelút
 Jelutak Apoptózis 1. Bevezetés 2. Külső jelút 3. Belső jelút Apoptózis Sejtmag 1. Kondenzálódó sejtmag apoptózis autofágia nekrózis Lefűződések Összezsugorodás Fragmentálódó sejtmag Apoptotikus test Fagocita
Jelutak Apoptózis 1. Bevezetés 2. Külső jelút 3. Belső jelút Apoptózis Sejtmag 1. Kondenzálódó sejtmag apoptózis autofágia nekrózis Lefűződések Összezsugorodás Fragmentálódó sejtmag Apoptotikus test Fagocita
Transzláció. Szintetikus folyamatok Energiájának 90%-a
 Transzláció Transzláció Fehérje bioszintézis a genetikai információ kifejeződése Szükséges: mrns: trns: ~40 Riboszóma: 4 rrns + ~ 70 protein 20 Aminosav aktiváló enzim ~12 egyéb enzim Szintetikus folyamatok
Transzláció Transzláció Fehérje bioszintézis a genetikai információ kifejeződése Szükséges: mrns: trns: ~40 Riboszóma: 4 rrns + ~ 70 protein 20 Aminosav aktiváló enzim ~12 egyéb enzim Szintetikus folyamatok
A fehérjék hierarchikus szerkezete
 Fehérjék felosztása A fehérjék hierarchikus szerkezete Smeller László Semmelweis Egyetem Biofizikai és Sugárbiológiai Intézet Biológiai funkció alapján Enzimek (pl.: tripszin, citokróm-c ) Transzportfehérjék
Fehérjék felosztása A fehérjék hierarchikus szerkezete Smeller László Semmelweis Egyetem Biofizikai és Sugárbiológiai Intézet Biológiai funkció alapján Enzimek (pl.: tripszin, citokróm-c ) Transzportfehérjék
Fiatal Biotechnológusok Országos Konferenciája SZERDA
 Fiatal Biotechnológusok Országos Konferenciája 2018 2018.03.28. - SZERDA 9:00-10:00 REGISZTRÁCIÓ, POSZTEREK ELHELYEZÉSE 10:00-10:15 MEGNYITÓ Fehér Attila, Tamás László 10:15-10:45 PLENÁRIS ELŐADÁS - PLE1
Fiatal Biotechnológusok Országos Konferenciája 2018 2018.03.28. - SZERDA 9:00-10:00 REGISZTRÁCIÓ, POSZTEREK ELHELYEZÉSE 10:00-10:15 MEGNYITÓ Fehér Attila, Tamás László 10:15-10:45 PLENÁRIS ELŐADÁS - PLE1
Membrántranszport. Gyógyszerész előadás Dr. Barkó Szilvia
 Membrántranszport Gyógyszerész előadás 2017.04.10 Dr. Barkó Szilvia Sejt membránok A sejtmembrán funkciói Védelem Kommunikáció Molekulák importja és exportja Sejtmozgás Általános szerkezet Lipid kettősréteg
Membrántranszport Gyógyszerész előadás 2017.04.10 Dr. Barkó Szilvia Sejt membránok A sejtmembrán funkciói Védelem Kommunikáció Molekulák importja és exportja Sejtmozgás Általános szerkezet Lipid kettősréteg
Jelutak. Apoptózis. Apoptózis Bevezetés 2. Külső jelút 3. Belső jelút. apoptózis autofágia nekrózis. Sejtmag. Kondenzálódó sejtmag
 Jelutak Apoptózis 1. Bevezetés 2. Külső jelút 3. Belső jelút Apoptózis Sejtmag Kondenzálódó sejtmag 1. autofágia nekrózis Lefűződések Összezsugorodás Fragmentálódó sejtmag Apoptotikus test Fagocita bekebelezi
Jelutak Apoptózis 1. Bevezetés 2. Külső jelút 3. Belső jelút Apoptózis Sejtmag Kondenzálódó sejtmag 1. autofágia nekrózis Lefűződések Összezsugorodás Fragmentálódó sejtmag Apoptotikus test Fagocita bekebelezi
Informatika 1. Informatika el adás. Kovács Kristóf, Pálovics Róbert. Budapesti M szaki Egyetem november 13.
 Informatika 1 9. el adás Kovács Kristóf, Pálovics Róbert Budapesti M szaki Egyetem 2013. november 13. CSS HTML formázasára, elhelyezésére szolgál Cél az újrafelhasználhatóság és könny módosítás CSS kód
Informatika 1 9. el adás Kovács Kristóf, Pálovics Róbert Budapesti M szaki Egyetem 2013. november 13. CSS HTML formázasára, elhelyezésére szolgál Cél az újrafelhasználhatóság és könny módosítás CSS kód
A posztszinapszis és a PSD (posztszinaptikus. szinaptikus plaszticitásban játszott szerepük
 A posztszinapszis és a PSD (posztszinaptikus denzitás), valamint a szinaptikus plaszticitásban játszott szerepük A posztszinapszis szimmetrikus (Gray II): variábilis, nagy vezikulák; ált. gátló aszimmetrikus
A posztszinapszis és a PSD (posztszinaptikus denzitás), valamint a szinaptikus plaszticitásban játszott szerepük A posztszinapszis szimmetrikus (Gray II): variábilis, nagy vezikulák; ált. gátló aszimmetrikus
Autoan'testek vizsgáló módszerei, HLA 'pizálás. Immunológiai és Biotechnológiai Intézet PTE- ÁOK
 Autoan'testek vizsgáló módszerei, HLA 'pizálás Immunológiai és Biotechnológiai Intézet PTE- ÁOK Természetes autoan'testek Pathológiás autoan'testek Természetes autoan'testek IgM Alacsony affinitás Alacsony
Autoan'testek vizsgáló módszerei, HLA 'pizálás Immunológiai és Biotechnológiai Intézet PTE- ÁOK Természetes autoan'testek Pathológiás autoan'testek Természetes autoan'testek IgM Alacsony affinitás Alacsony
Immunológia alapjai előadás MHC. szerkezete és genetikája, és az immunológiai felismerésben játszott szerepe. Antigén bemutatás.
 Immunológia alapjai 5-6. előadás MHC szerkezete és genetikája, és az immunológiai felismerésben játszott szerepe. Antigén bemutatás. Antigén felismerés Az ellenanyagok és a B sejt receptorok natív formában
Immunológia alapjai 5-6. előadás MHC szerkezete és genetikája, és az immunológiai felismerésben játszott szerepe. Antigén bemutatás. Antigén felismerés Az ellenanyagok és a B sejt receptorok natív formában
Immunológia alapjai. 23-24. előadás. Immunológiai tolerancia. Fiziológiás és patológiás autoimmunitás.
 Immunológia alapjai 23-24. előadás Immunológiai tolerancia. Fiziológiás és patológiás autoimmunitás. Tolerált bőr graftok MHC (H2) azonos egereken TOLERANCIA & AUTOIMMUNITÁS Toleranciáról beszélünk, ha
Immunológia alapjai 23-24. előadás Immunológiai tolerancia. Fiziológiás és patológiás autoimmunitás. Tolerált bőr graftok MHC (H2) azonos egereken TOLERANCIA & AUTOIMMUNITÁS Toleranciáról beszélünk, ha
List of publications.
 1 List of publications. 1. Venetianer,P., Ullmann,A. and Straub,F.B.: Die Rolle der Ribonukleinsaure bei der Pankreasamylasesynthese. Acta Physiol. Acad. Sci. Hung. Suppl. XVI. (1959) 58. 2. Straub,F.B.,
1 List of publications. 1. Venetianer,P., Ullmann,A. and Straub,F.B.: Die Rolle der Ribonukleinsaure bei der Pankreasamylasesynthese. Acta Physiol. Acad. Sci. Hung. Suppl. XVI. (1959) 58. 2. Straub,F.B.,
V48 Prezentáció I Presentation irodaház I office building I Budapest XIII., Váci út 48. / e-f 23/07/2012
 irodaház I office building I Office Building, Area table 1 Office related areas Office Terrace Storage Car Parks 2 (in m²) (in m²) (in m²) (in No of pl) 2nd Basement level - - 211 59 1st Basement level
irodaház I office building I Office Building, Area table 1 Office related areas Office Terrace Storage Car Parks 2 (in m²) (in m²) (in m²) (in No of pl) 2nd Basement level - - 211 59 1st Basement level
Supplementary Material. Promoter deletion analysis reveals root-specific expression of the alkenal reductase gene (OS-AER1) in Oryza sativa
 10.1071/FP18237_AC CSIRO 2019 Supplementary Material: Functional Plant Biology, 2019, 46(4), 376 391. Supplementary Material Promoter deletion analysis reveals root-specific expression of the alkenal reductase
10.1071/FP18237_AC CSIRO 2019 Supplementary Material: Functional Plant Biology, 2019, 46(4), 376 391. Supplementary Material Promoter deletion analysis reveals root-specific expression of the alkenal reductase
