Supplemental Information. Investigation of Penicillin Binding Protein. (PBP)-like Peptide Cyclase and Hydrolase
|
|
|
- Imre Faragó
- 6 évvel ezelőtt
- Látták:
Átírás
1 Cell Chemical Biology, Volume upplemental Information Investigation of Penicillin Binding Protein (PBP)-like Peptide Cyclase and ydrolase in urugamide on-ribosomal Peptide Biosynthesis Yongjun Zhou, Xiao Lin, Chunmin Xu, Yaoyao hen, hu-ping Wang, ongze Liao, Lei Li, ai Deng, and ou-wen Lin
2 upplementary Information Table of Contents Tables and Figures Table. Protein Analysis of sur BGCs Derived from LW0 and J0 Table. The ligonucleotide Primers Used in the tudy Table. The Plasmids Generated in the tudy Table. R-M Data of the Compounds in the tudy Figure. D-PAGE Gel of the Purified Proteins Figure. ure Active ite Prediction Figure. Protein equence Alignment of the ure omologues Data. QTF-M/M Analysis of the Compounds Generated in ure Assays Data. QTF-M/M Analysis of the ynthesized Peptidyl-AC ubstrates Data. and C MR pectra of the ynthesized Peptidyl-AC ubstrates
3 Table. Protein Analysis of sur BGCs Derived from LW0 and J0 (related to Figure ) Gene ize (a.a.) Predicted function Identity (%) Gene id in J0 urr GntR family DA-binding transcriptional regulator 00 XR orf Membrane protein 00 XR ure Beta-lactamase 00 XR0 ura RP (A-T, C-A-T-E, C-A-T-E, C-A-T) 99 XR9 urb RP (A-T, C-A-T-E, C-A-T, C-A-T) 00 XR urc 9 RP (C-A-T, C-A-T-E, C-A-T, C-A-T-E, C-A-T, C-A-T-E) 00 XR urd RP (C-A-T, C-A-T-E, C-A-T-E) 99 XR orf Drug resistance transporter 99 XR orf 0 TetR family transporter regulator 00 XR Table. The ligonucleotide Primers Used in the tudy (related to Figure and "ligonucleotides" in KEY REURCE TABLE) Primers equence Description In-frame deletion of sure gene lacd-l- GACTGATCAAGGCGAATACTTCAACTCCACGGTGCTCCTG 9 bp product, bp lacd-l-a lacd-r- lacd-r-a TAGTCGGAGTAGCTCTCGACGAACTGCATGACCAGTTC TGGTCATGCAGTTCGTCGAGAGCTACTCCGACTACCGCCTC TC AAAAGATCCGTCGACCTGCAGGCATGCAGCGTGTCGAGGG TGAC overlapping (L) with dei side of py bp product, bp L with indiii side of py, bp L with left fragment Identification of sure strain or complementation of sure strain with intact sure gene lac- ATATACATATGGGTGCCGAGGGGGCGGAAC bp for mutant, lac-a ATCGACCGCTCATCCTGGAACATCGTG 9 bp for wide type Complementation of sure strain with site-muated (A) sure gene The mutated gene was generated by way of overlapping PCR. MsurE- TTGTGGGCACAATCGTGCCGGTTG 0 bp MsurE-A TCACCGCGCCGAAGGGGAAACC MsurE- GTTTCCCCTTCGGCGCGGTGACCAAGTTCC bp MsurE-A TTCACACAGGAAACAGCTATGACATGATTACGAATTCGATATCTC Protein expression of ure with a -terminal is tag based on pet9a (dei, XhoI)
4 PsurE- PsurE-A ATATACATATGCATCACCACCATCATCATGGTGCCGAGGGG GCGGAAC AATATCTCGAGTCAGAGCCGGTGCATGGCCCGGCCGCCGT Protein expression of ure with A site mutation MurE- A-A A- MurE-A AATTTTGTTTAACTTTAAGAAGGAGATATACATATGCATCACCACCATC AGGAACTTGGTCACCGCGCCGAAGGGGAAACCCGTC TTCCCCTTCGGCGCGGTGACCAAGTTCCTCACCGCCGAAC TTTGTTAGCAGCCGGATCTCAGTGGTGGTGGTGGTGGTGCTC Protein expression of ure with KA site mutation MurE- KA-A KA- MurE-A see above TGAGGAACGCGGTCACCGAGCCGAAGG CTTCGGCTCGGTGACCGCGTTCCTCACCGCCGAACTGGTC see above Protein expression of ure with YA site mutation MurE- YA-A YA- MurE-A see above TGTTGGACGCGGAGAAGGCGAGGCCCGGCGGGA CTCGCCTTCTCCGCGTCCAACACCGGGTACTGCCTG see above Protein expression of ure with A site mutation MurE- A-A A- MurE-A see above AGGCAGTACCCGGTCGCGGAGTAGGAGAAGGCGAGG CTACTCCGCGACCGGGTACTGCCTGCTGGGCGCGGT see above 9 bp product with dei and XhoI sites Table. The Plasmids Generated in the tudy (related to Figure and "Plasmid Constructions" in the section of TAR METD) Plasmids Descriptions In-frame deletion of sure gene in sur BGC prj Two PCR products were amplified from the genomic DA of LW0 with the primers lacd-l- and -A for the left fragment, and lacd-r- and -A for the right fragment, respectively (Table ). The two PCR products were assembled with the vector fragment of py (dei, indiii) by the way of Gibson assembly. Complementation of sure strain prj9 prj A PCR fragment of sure amplified from the genomic DA of LW0 with the primers, lac- and-a (Table ), was digested with dei and EcoRV and then ligated into pib9 (dei, EcoRV) by T ligase. Two PCR products amplified from prj9, respectively with the pairs of primers MsurE- and MsurE-A, and MsurE- and MsurE-A (Table ), were assembled into one fragment. The fragment was then introduced into pib9 (dei, EcoRV) by the way of Gibson assembly.
5 Protein expression of ure with -terminal is tag in E. coli BL(DE) plys. prj Wide type ure The PCR product of sure amplified from prj9 with the primers, PsurE- and -A (Table ), was digested with dei and XhoI, followed by ligation into the corresponding site of pet9a prj0 ite mutation (KA) The two PCR products (see primers in Table ) of prj ite mutation (A) sure containing the target mutation site at overlapping region were introduced into pet9a prj ite mutation (YA) (dei, XhoI) by using Gibson assembly. prj ite mutation (A) prj9 ite mutation (A) The synthesized DA fragment of sure with target prj ite mutation (DA) mutation was introduced into pet9a (dei, XhoI). prj ite mutation (D) Table. R-M Data of the Compounds in the tudy (related to Figure and Table ) Products Formula calc. m/z det. m/z Error (ppm) The compounds produced in enzymatic assays or by strain LW0 C ~ C C C C C C 9... C The substrates used in enzymatic assay 0 C C C C C C 9..0.
6 D-PAGE gel of ure protein purification The concentration of imidazole in elution (mm) FT pellet. kd kd kd kd kd Figure. D-PAGE Gel of the Purified Proteins (related to Figure ) Putative key residues in the active site key residues in the active site key residues in the active site Tyr is Asp Lys er Asn Predicted ure overall structure ADP overall structure DAP overall structure Figure. ure Active ite Prediction (related to Figure and Figure ) tructural and Active ite Comparison among ure, ADP (PDB id YP) and DAP (PDB id EI_A). The proposed active sites and structure of ure is predicted based on the site-mutation and the comparative analysis.
7 () cmcpbp () MPFDAWQERFDALRTEVPGAALAVFVDG-DLELAGVLRGTGVAVTTDVFQGVAKVYTATLVMQLVDAGELRLDTRVADVL Pab () EQUECEMDVGKLEFIVEKMAERKVPGIIIIKDGDVVYAKGFGYRVEARLPTPETIYGIGITKFTALAIMKLVEEGGLLDDPVEKFV ADP () MKTRQITCALALLIAGLLYTTQTLIVKAEPTQVVQTTQRDRVKQAVRDTLQLGFPGILAKTEGG-KTWYAAGVALKKPMKTDFRFRIGVTKTFTATVVLQLAEERLLDDIEKWL DAP () MKFDTALEAFVRIPQYKGPGGVVAVVKDGEVVLQAWGFADLRTRTPMTLDTRMPICVKQFTCAVLLDAVGE-PELLDDALEAYL MppK () MDELTLTELAARRVPGAQLAVIEG-ERFLVTGVCDTAGAPVERTAFPVGLTKPFTAALAMILVADGDVDLDEPLRGQL DsaJ () MLGIIAEEKVGAPFLPLDRLGCLGLGKEGVVGAQLAVYRG-TLRTWEFGEEEYGGGRPVRRDAFPYGVTKALTAAVLQLVDGDLDLRPVRTWL Ulm () MGDYADPADCGAGDGAPVG--LDLDRLARDCDVVGGQLALQG-TLTTWEFGTEEAGGRPVVGAFPYGVTKAFTATAVLQLAGDGDLDLDRPVRELL ure () VGAEGAERDAVGALFEELVRERVTGAQLAVYRDG-ALEYATGLAVRTGEPVTPRTGFPFGVTKFLTAELVMQFVCDGDLDLDDPLAGLL Consensus () D L A L L EKVPGAQLAV DG L WAFGV DLATG PVT DTAFPIGVTK FTA LVLQLV DGDLDLDDPV WL xxk K () cmcpbp (90) PGFA VADAEVARTVTIGRLLTGIAGDFTLDTGRG-DDCLARFVDACADVG QDCPPDTVIYCTGYAILGRIVEVLTGQWDDALRDRLFTPLGLQMTLP-- Pab (9) IKLR PFGEPVTVLLTGIPLGYAEAFIDGMVGGDWLPVTPEETIAFARDMEKWAVAKPGERFFYLTGYVLLGKIIEKVGVYEEYIKKKILEPLGMRYFFK-- ADP() PGVIQ GGYDDKQITIRQLLTGIAEYTRKFDLMDTKKYRAEELVKMG-IMP------PDFAPGKWYTGYVLLGILIETVTGYAEEIERIIEPLELTFLPG- DAP (9) DKFED ER-PAVRDLCQGLRDYWALVLCGADPEGVFLPAQAQLLRRLKT------TFEPGYYCGFRILADLIEATGRTLVDILERIFAPAGMKRAELI-- MppK () PEFG AGELVT---LRQLLTGLPDVPEGDEAGGGDRARWVARYCRTAD LTAPGTVFYIGYVVVGRLIEAVTGMWQEAIAILLEPLGTRPAFVVG-- DsaJ(0) DDRG AAGPGLGATLRQLLTAGLP----DDDGDATLRRWLTGFLATGPADAP------ALWPAPGFYVGYGIAGRVVEAATGLTWWQAVRDYLLAPLGTGIGLLPGAP Ulm(0) PEAEAEAEIEVEAGGTGARADGGPALAATLRQLLTAGLP----DDDERAPLRRWLTGFLALP----V------GPWPAPGFYVGYGIAGRVVEAVTGLTWEAVRDFLLPLGTAITVLP--- ure (9) PDLG RAAGPALGTATVRQLLTAGVVDI--EYDEMRGPYRRFAAACARQP ALFPPGLAFYTGYCLLGAVIEAAGMDWWTAMDCLLRPLGIEPAFLDP- Consensus() PD G AAG A TLRQLLTGLP D DDG A RWL A LG A FPPG FYTGY ILGRLIEAVTGLW EAIRDRLL PLGL AFL Yx Y () cmcpbp(9) EEALRFRVAMLGELGTDPEPAPVWDMLPRAGPYGRVLITAADVVRFARMLDDGVAPDGTRVLAAAALMRQQVAGCLDTWFMATGWGGWALYDWD-GVPGYGDGAGGQFYLRVVPGGVVAALL Pab(0) --EEVEKDKDVAMGYILDK-EGRLVPQPFPYGITADGGLLVLDLAKYLKMYIERDEIV------KEYIEKMETYIKVPWEIFGGEGYGYGLIIYPFLGEKLVGGVGMYTGYIGYIPEKKIGVAVL ADP() VIPGTKARGYIQLDGAEPKDVTYYPMGAGDMITADDLKFFYLLGGKLLKE QQLKQMLTTVPTGEAALGRYGLGIYETKLPGVIWGGGIPGFVTFAGGTLGGKTLAV DAP(9) DTALFDECTG-----YEGDTVRGFLPATRIQWMGDAGICALDMIAWEQFIDATRDDEG LYRRLGPQTFKDGVAAPYGFGLLETG-GKRLTGGGALRGWRCQRWCADERLTIAM MppK(9) -APATRPVATGAVQAVRDRVVPIPDQDLPEVEMPGALALAEDLVGFARLYFAGCPDPQPLDRATADDMCFDQLAIAIGPYG--MADGWGLGWARFDDG-AADVYGGTGDGTCLRFDPAGAVALT DsaJ() EPVRAVAGTTTTPGG-DRVVVRCADAGFTPAGGLAGAADLVRFAQLLDDPGDLDRDAVADETLREMARPVAGAEPFG--LADGWGLGLGFGPA-EDRWLGDGTLDGTTCLRIPKQGTVVALT Ulm() TDPGLPAGGLAGAADLVRLGRLLDEPGDPDLARLADPDALREMARPTAGADPFG--LADGWGPGLGRFGPA-GRWLGDGTLDGATCLRIPGRGTVVALT ure(00) QGGAARPVAEGALRAG-GERAERVDMALLAAAGGLVGATDLVTAARPLADRKTFAQDLLPEDAVLAMRTCVPDAEPFG--LADGWGLGLMRGTG-DGAWYGDGAVGGACLRIPDRLALALT Consensus() D V P PAGGLLGA DLVRFARLLDD D D L D L M R VAG PFG LADGWGLGLA F G G RLYGDGTL G TCLRI PG GL VALT (0) cmcpbp() TGGVATFFTDLFRELLGELAGVRMPEVFAPPAEPRPIDVAPLAGTYE--REGGAP Pab() EG----YPPYIAMYALALLLGKPEKELPFIYRERILKKVEGRYMGYKGTIKFEVKVDGDVVYLRALGRAFTYTIPLFPEVLEEDFIKCYTLGRKMYAEFYIKDKVDLIFERYRLIK ADP() LLAEPDPFKILLAEFK DAP() FFEGGAEVAFKLMIALGV-EVRVEADAWFGWLDDETGLVLLEDAGGRMKARFGTPEMMDVVAEARAVTTIRRDGETIELVRAELRLMKRVKGEAKDIIGRYDELDADLLLVE MppK(9) AATGAQLWDALVPRLRAMGLAVGDRPAPEPPTTP-PPVPDDCPGRYT--GDTEFVVQPGADGGLLLFGGAPELLCPDLRFTMRELG-GARPGRFVTDPATGRIGYLQITGRLAPRR DsaJ() TAGQGLWEAVLRELRAGLPVGVYVVPPPG-TAGFADCAGTYR--GDLAVTVLDDR-ILLELPGARDAADLVFRPGD--AGVFLGRFLKDATTGDVRVLQYGRTLLRQP---VAAQP Ulm() TPTGQALWDAVVDALRDADIDVGVRPAPPPAIA-AAAFADCTGTYR--GDLAVTVGIDGP-YLVLELPGGARELAQPLARTFR-----GAGFLGRFVTDARDAVALQYGRTLLREAGERRAR ure(0) ATAGPKLWEALVARLPEAGLDVGYALPVPDAP-LAPDAGLGTYA--GDLELMVTDAAGDLFLTREYDYRLLEDDLFVARGEPGALPITGRFVREPAGPVALLQYGGRAMRL Consensus(0) G LWDALV L AGL VG PPA A AD GTY GDL V DA LLL AA DL F R AA GRFV D G V LQY GR L R DG D Figure. Protein equence Alignment of the ure omologues (related to Figure and Figure ) The putative activities are labeled. The site mutations generated in ure are displayed with the AA sequence number. Data. QTF-M/M Analysis of the Compounds Generated in ure Assays (related
8 to Figure ).. The M fracture spectrum of the compound generated by ure with compound 0 as substrate (a) and the one isolated from the fermentation extract of LW0 (b). The above scheme is the predicted fragments of the M/M analysis. Chemical Formula: C 9 Exact Mass: 9. Chemical Formula: C 9 Exact Mass:. compound 9 Chemical Formula: C0 Exact Mass:.0 Chemical Formula: C Exact Mass: 9.9 Chemical Formula: C Exact Mass: 99.0 Chemical Formula: C Exact Mass:.09 0 Chemical Formula: C9 Exact Mass:.09 Chemical Formula: C9 Exact Mass:.9 Chemical Formula: C Exact Mass:. Chemical Formula: C0 Exact Mass:.00 Chemical Formula: C Exact Mass: 9. Chemical Formula: C Exact Mass:. Chemical Formula: C Exact Mass:.9 a) 0 9 b).. The M fracture spectrum of compound produced by J0 (a) or the one generated in ure assay with compound as substrate (b). The above scheme is the predicted fragments of the M/M analysis.
9 Chemical Formula: C Exact Mass: 0. Chemical Formula: C Exact Mass: 9.9 Chemical Formula: C Exact Mass:.9 compound Chemical Formula: C Exact Mass: 9.90 Chemical Formula: C 9 Exact Mass:. Chemical Formula: C Exact Mass: 99.9 Chemical Formula: C Exact Mass: 9. Chemical Formula: C Exact Mass: 00.0 a) b).. The M fracture spectrum of compound generated in ure assay with compound 0 as substrate. The above scheme is the predicted fragments of the M/M analysis. Exact Mass: 90. compound Chemical Formula: C99 Chemical Formula: C Exact Mass: 99.0 Chemical Formula: C Exact Mass:. Chemical Formula: C Exact Mass: 9.9 Chemical Formula: C9 Exact Mass:.9 Chemical Formula: C Exact Mass: 9. Chemical Formula: C Exact Mass:... The M fracture spectrum of compound 9 produced in ure assay with compound as substrate. The above scheme is the predicted fragments of the M/M analysis.
10 Chemical Formula: C09 Exact Mass: 9. compound 9 Chemical Formula: C9 Exact Mass:. Chemical Formula: C Exact Mass:.9 Chemical Formula: C9 Exact Mass: 0. Chemical Formula: C9 Exact Mass: 0. Chemical Formula: C0 Exact Mass: Chemical Formula: C Exact Mass: 9.0 Chemical Formula: C Exact Mass: 9.9 Chemical Formula: C0 Exact Mass:. Chemical Formula: C Exact Mass: 9. Chemical Formula: C Exact Mass:.09 Chemical Formula: C0 Exact Mass: The M fracture spectrum of compound 0 produced in ure assay with compound as substrate. The above scheme is the predicted fragments of the M/M analysis. Chemical Formula: C99 Exact Mass: 9.0 compound 0 Chemical Formula: C Exact Mass: 99.0 Chemical Formula: C Exact Mass: 0.0 Chemical Formula: C Exact Mass:. Chemical Formula: C0 Exact Mass:. Chemical Formula: C Exact Mass: 9.9 Chemical Formula: C9 Exact Mass:.9 9 Chemical Formula: C09 Exact Mass:.0 Chemical Formula: C Exact Mass: The M fracture spectrum of compound produced in ure assay with compound as substrate. The above scheme is the predicted fragments of the M/M analysis.
11 Chemical Formula: C9 Exact Mass:. compound Chemical Formula: C9 Exact Mass:.9 Chemical Formula: C0 Exact Mass:.00 Chemical Formula: C Exact Mass:.9 9 Chemical Formula: C Exact Mass:. Chemical Formula: C Exact Mass: 0. Chemical Formula: C Exact Mass: 9.9 Chemical Formula: C Exact Mass:. Chemical Formula: C Exact Mass: The M fracture spectrum of compound produced in ure assay with compound as substrate. The above scheme is the predicted fragments of the M/M analysis. Chemical Formula: C Exact Mass: 0. compound Chemical Formula: C Exact Mass: 99.0 Chemical Formula: C099 Exact Mass:.9 Chemical Formula: C0 Exact Mass: Chemical Formula: C9 Exact Mass: 9.0 Chemical Formula: C0 Exact Mass: 0. Chemical Formula: C0 Exact Mass: 9. Chemical Formula: C Exact Mass: 0. Chemical Formula: C Exact Mass:. 9
12 Data. QTF-M/M Analysis of the ynthesized Peptidyl-AC ubstrates (related to Figure ).. The M fracture spectrum of compound compounds 0 (a) and (b). The above scheme is the predicted fragments of the M/M analysis. Chemical Formula: C909 Exact Mass: 0. Chemical Formula: C9 Exact Mass: 9. compound 0 or Chemical Formula: C Exact Mass:. Chemical Formula: C Exact Mass: 99.0 Chemical Formula: C Exact Mass:. Chemical Formula: C Exact Mass:.9 Chemical Formula: C Exact Mass: 9.9 Chemical Formula: C9 Exact Mass: Chemical Formula: C09 Exact Mass:.0 Chemical Formula: C Exact Mass: 9. Chemical Formula: C Exact Mass:. Chemical Formula: C9 Exact Mass:. a) 0 9 b).. The M fracture spectrum of compound compounds. The above scheme is the predicted fragments of the M/M analysis.
13 Chemical Formula: C90 9 Exact Mass: 0.9 Chemical Formula: C09 Exact Mass: 9. compound Chemical Formula: C Exact Mass:. Chemical Formula: C Exact Mass: 99.0 Chemical Formula: C0 Exact Mass:. Chemical Formula: C Exact Mass:. Chemical Formula: C Exact Mass: Chemical Formula: C Exact Mass:. Chemical Formula: C9 Exact Mass:.9 Chemical Formula: C Exact Mass: 9. Chemical Formula: C Exact Mass: The M fracture spectrum of compound compounds. The above scheme is the predicted fragments of the M/M analysis. Chemical Formula: C90 9 Exact Mass: 99. Chemical Formula: C9 Exact Mass: 0. compound Chemical Formula: C Exact Mass: 99.0 Chemical Formula: C Exact Mass: 9. Chemical Formula: C Exact Mass:. Chemical Formula: C Exact Mass: 9. Chemical Formula: C Exact Mass: 9.9 Chemical Formula: C9 Exact Mass: Chemical Formula: C Exact Mass: 9. Chemical Formula: C Exact Mass:. Chemical Formula: C9 Exact Mass: The M fracture spectrum of compound compounds. The above scheme is the
14 predicted fragments of the M/M analysis. Chemical Formula: C09 Chemical Formula: C9 Chemical Formula: C9 Chemical Formula: C09 Exact Mass: 9.00 Exact Mass:. Exact Mass:. Exact Mass: 0.00 compound Chemical Formula: C Exact Mass:. 9 0 Chemical Formula: C0 Chemical Formula: C9 Chemical Formula: C Chemical Formula: C Exact Mass: 0. Exact Mass: 9.0 Exact Mass: 9. Exact Mass: 9.9 Chemical Formula: C0 Exact Mass: 0.0 Chemical Formula: C Exact Mass: The M fracture spectrum of compound compounds. The above scheme is the predicted fragments of the M/M analysis. Chemical Formula: C9 Exact Mass:. compound Chemical Formula: C099 Exact Mass:.9 Chemical Formula: C Exact Mass: 9. Chemical Formula: C Exact Mass: 0. Chemical Formula: C Exact Mass: 9.90 Chemical Formula: C0 Exact Mass:.9 Chemical Formula: C9900 Exact Mass: 9.9 Chemical Formula: C 9 Exact Mass: 9. Chemical Formula: C0 Exact Mass: Chemical Formula: C Exact Mass:
15 Data. and C MR pectra of the ynthesized Peptidyl-AC ubstrates (related to Figure ).. - (a) and C- (b) MR spectrum of 0 a)
16 b).. - (a) and C- (b) MR spectrum of a)
17 b).. - (a) and C- (b) MR spectrum of a) b)
18 .. - (a) and C- (b) MR spectrum of a) b)
19 .. - (a) and C- (b) MR spectrum of a) b)
20 .. - (a) and C- (b) MR spectrum of a) b)
21
Supporting Information
 Supporting Information Plasmid Construction DNA cloning was carried out according to standard protocols. The sequence of all plasmids was confirmed by DNA sequencing and amplified using the E. coli strain
Supporting Information Plasmid Construction DNA cloning was carried out according to standard protocols. The sequence of all plasmids was confirmed by DNA sequencing and amplified using the E. coli strain
Modular Optimization of Hemicellulose-utilizing Pathway in. Corynebacterium glutamicum for Consolidated Bioprocessing of
 [Supplementary materials] Modular Optimization of Hemicellulose-utilizing Pathway in Corynebacterium glutamicum for Consolidated Bioprocessing of Hemicellulosic Biomass Sung Sun Yim 1, Jae Woong Choi 1,
[Supplementary materials] Modular Optimization of Hemicellulose-utilizing Pathway in Corynebacterium glutamicum for Consolidated Bioprocessing of Hemicellulosic Biomass Sung Sun Yim 1, Jae Woong Choi 1,
Manuscript Title: Identification of a thermostable fungal lytic polysaccharide monooxygenase and
 1 2 3 4 5 Journal name: Applied Microbiology and Biotechnology Manuscript Title: Identification of a thermostable fungal lytic polysaccharide monooxygenase and evaluation of its effect on lignocellulosic
1 2 3 4 5 Journal name: Applied Microbiology and Biotechnology Manuscript Title: Identification of a thermostable fungal lytic polysaccharide monooxygenase and evaluation of its effect on lignocellulosic
Horizontal gene transfer drives adaptive colonization of apple trees by the fungal pathogen Valsa mali. Zhiyuan Yin, Baitao Zhu, Hao Feng, Lili Huang*
 Supplementary Information Horizontal gene transfer drives adaptive colonization of apple trees by the fungal pathogen Valsa mali Zhiyuan Yin, Baitao Zhu, Hao Feng, Lili Huang* State Key Laboratory of Crop
Supplementary Information Horizontal gene transfer drives adaptive colonization of apple trees by the fungal pathogen Valsa mali Zhiyuan Yin, Baitao Zhu, Hao Feng, Lili Huang* State Key Laboratory of Crop
Nan Wang, Qingming Dong, Jingjing Li, Rohit K. Jangra, Meiyun Fan, Allan R. Brasier, Stanley M. Lemon, Lawrence M. Pfeffer, Kui Li
 Supplemental Material IRF3-dependent and NF- B-independent viral induction of the zinc-finger antiviral protein Nan Wang, Qingming Dong, Jingjing Li, Rohit K. Jangra, Meiyun Fan, Allan R. Brasier, Stanley
Supplemental Material IRF3-dependent and NF- B-independent viral induction of the zinc-finger antiviral protein Nan Wang, Qingming Dong, Jingjing Li, Rohit K. Jangra, Meiyun Fan, Allan R. Brasier, Stanley
Supplementary Table 1. Cystometric parameters in sham-operated wild type and Trpv4 -/- rats during saline infusion and
 WT sham Trpv4 -/- sham Saline 10µM GSK1016709A P value Saline 10µM GSK1016709A P value Number 10 10 8 8 Intercontractile interval (sec) 143 (102 155) 98.4 (71.4 148) 0.01 96 (92 121) 109 (95 123) 0.3 Voided
WT sham Trpv4 -/- sham Saline 10µM GSK1016709A P value Saline 10µM GSK1016709A P value Number 10 10 8 8 Intercontractile interval (sec) 143 (102 155) 98.4 (71.4 148) 0.01 96 (92 121) 109 (95 123) 0.3 Voided
A cell-based screening system for RNA Polymerase I inhibitors
 Electronic Supplementary Material (ESI) for MedChemComm. This journal is The Royal Society of Chemistry 2019 Supporting Information A cell-based screening system for RNA Polymerase I inhibitors Xiao Tan,
Electronic Supplementary Material (ESI) for MedChemComm. This journal is The Royal Society of Chemistry 2019 Supporting Information A cell-based screening system for RNA Polymerase I inhibitors Xiao Tan,
Supporting Information
 Supporting Information Paired design of dcas9 as a systematic platform for the detection of featured nucleic acid sequences in pathogenic strains Yihao Zhang 1,2,8, Long Qian 4,8, Weijia Wei 1,3,7,8, Yu
Supporting Information Paired design of dcas9 as a systematic platform for the detection of featured nucleic acid sequences in pathogenic strains Yihao Zhang 1,2,8, Long Qian 4,8, Weijia Wei 1,3,7,8, Yu
Supporting Information
 Supporting Information Cell-free GFP simulations Cell-free simulations of degfp production were consistent with experimental measurements (Fig. S1). Dual emmission GFP was produced under a P70a promoter
Supporting Information Cell-free GFP simulations Cell-free simulations of degfp production were consistent with experimental measurements (Fig. S1). Dual emmission GFP was produced under a P70a promoter
Expression analysis of PIN genes in root tips and nodules of Lotus japonicus
 Article Expression analysis of PIN genes in root tips and nodules of Lotus japonicus Izabela Sańko-Sawczenko 1, Dominika Dmitruk 1, Barbara Łotocka 1, Elżbieta Różańska 1 and Weronika Czarnocka 1, * 1
Article Expression analysis of PIN genes in root tips and nodules of Lotus japonicus Izabela Sańko-Sawczenko 1, Dominika Dmitruk 1, Barbara Łotocka 1, Elżbieta Różańska 1 and Weronika Czarnocka 1, * 1
Involvement of ER Stress in Dysmyelination of Pelizaeus-Merzbacher Disease with PLP1 Missense Mutations Shown by ipsc-derived Oligodendrocytes
 Stem Cell Reports, Volume 2 Supplemental Information Involvement of ER Stress in Dysmyelination of Pelizaeus-Merzbacher Disease with PLP1 Missense Mutations Shown by ipsc-derived Oligodendrocytes Yuko
Stem Cell Reports, Volume 2 Supplemental Information Involvement of ER Stress in Dysmyelination of Pelizaeus-Merzbacher Disease with PLP1 Missense Mutations Shown by ipsc-derived Oligodendrocytes Yuko
University of Bristol - Explore Bristol Research
 Al-Salihi, S. A. A., Scott, T. A., Bailey, A. M., & Foster, G. D. (2017). Improved vectors for Agrobacterium mediated genetic manipulation of Hypholoma spp. and other homobasidiomycetes. Journal of Microbiological
Al-Salihi, S. A. A., Scott, T. A., Bailey, A. M., & Foster, G. D. (2017). Improved vectors for Agrobacterium mediated genetic manipulation of Hypholoma spp. and other homobasidiomycetes. Journal of Microbiological
Phenotype. Genotype. It is like any other experiment! What is a bioinformatics experiment? Remember the Goal. Infectious Disease Paradigm
 It is like any other experiment! What is a bioinformatics experiment? You need to know your data/input sources You need to understand your methods and their assumptions You need a plan to get from point
It is like any other experiment! What is a bioinformatics experiment? You need to know your data/input sources You need to understand your methods and their assumptions You need a plan to get from point
Markerless Escherichia coli rrn Deletion Strains for Genetic Determination of Ribosomal Binding Sites
 Markerless Escherichia coli rrn Deletion Strains for Genetic Determination of Ribosomal Binding Sites Selwyn Quan *, Ole Skovgaard, Robert E. McLaughlin, Ed T. Buurman,1, and Catherine L. Squires * Department
Markerless Escherichia coli rrn Deletion Strains for Genetic Determination of Ribosomal Binding Sites Selwyn Quan *, Ole Skovgaard, Robert E. McLaughlin, Ed T. Buurman,1, and Catherine L. Squires * Department
Suppl. Materials. Polyhydroxyalkanoate (PHA) Granules Have no Phospholipids. Germany
 Suppl. Materials Polyhydroxyalkanoate (PHA) Granules Have no Phospholipids Stephanie Bresan 1, Anna Sznajder 1, Waldemar Hauf 2, Karl Forchhammer 2, Daniel Pfeiffer 3 and Dieter Jendrossek 1 1 Institute
Suppl. Materials Polyhydroxyalkanoate (PHA) Granules Have no Phospholipids Stephanie Bresan 1, Anna Sznajder 1, Waldemar Hauf 2, Karl Forchhammer 2, Daniel Pfeiffer 3 and Dieter Jendrossek 1 1 Institute
SUPPLEMENTAL DATA. Structure and specificity of the Type VI secretion system ClpV-TssC interaction in enteroaggregative Escherichia coli.
 SUPPLEMENTAL DATA Structure and specificity of the Type VI secretion system ClpV-TssC interaction in enteroaggregative Escherichia coli. B. Douzi, Y.R. Brunet, S. Spinelli, V. Lensi, P. Legrand, S. Blangy,
SUPPLEMENTAL DATA Structure and specificity of the Type VI secretion system ClpV-TssC interaction in enteroaggregative Escherichia coli. B. Douzi, Y.R. Brunet, S. Spinelli, V. Lensi, P. Legrand, S. Blangy,
Seven gene deletions in seven days: Fast generation of Escherichia coli strains tolerant to acetate and osmotic stress Supplementary Figure S1.
 Seven gene deletions in seven days: Fast generation of Escherichia coli strains tolerant to acetate and osmotic stress Sheila I. Jensen 1*, Rebecca M. Lennen 1, Markus J. Herrgård 1, Alex T. Nielsen 1*
Seven gene deletions in seven days: Fast generation of Escherichia coli strains tolerant to acetate and osmotic stress Sheila I. Jensen 1*, Rebecca M. Lennen 1, Markus J. Herrgård 1, Alex T. Nielsen 1*
The role of aristolochene synthase in diphosphate activation
 Supporting information for The role of aristolochene synthase in diphosphate activation Juan A. Faraldos, Verónica González and Rudolf K. Allemann * School of Chemistry, Cardiff University, Main Building,
Supporting information for The role of aristolochene synthase in diphosphate activation Juan A. Faraldos, Verónica González and Rudolf K. Allemann * School of Chemistry, Cardiff University, Main Building,
Trinucleotide Repeat Diseases: CRISPR Cas9 PacBio no PCR Sequencing MFMER slide-1
 Trinucleotide Repeat Diseases: CRISPR Cas9 PacBio no PCR Sequencing 2015 MFMER slide-1 Fuch s Eye Disease TCF 4 gene Fuchs occurs in about 4% of the US population. Leads to deteriorating vision without
Trinucleotide Repeat Diseases: CRISPR Cas9 PacBio no PCR Sequencing 2015 MFMER slide-1 Fuch s Eye Disease TCF 4 gene Fuchs occurs in about 4% of the US population. Leads to deteriorating vision without
pjnc-pgluc Codon-optimized Gaussia luciferase (pgluc) Vector backbone: JM83 pjnc-pgluc is resistant to ampicillin and neomycin High or low copy:
 pjnc-pgluc Gene/Insert name: Codon-optimized Gaussia luciferase (pgluc) Vector backbone: pcmv-jnc Vector type: Mammalian cells Backbone size w/o insert (bp): 5375 Bacterial resistance: Ampicillin and neomycin
pjnc-pgluc Gene/Insert name: Codon-optimized Gaussia luciferase (pgluc) Vector backbone: pcmv-jnc Vector type: Mammalian cells Backbone size w/o insert (bp): 5375 Bacterial resistance: Ampicillin and neomycin
Architecture of the Trypanosome RNA Editing Accessory Complex, MRB1
 Architecture of the Trypanosome RNA Editing Accessory Complex, MRB1 Michelle L. Ammerman, Kurtis M. Downey, Hassan Hashimi, John C. Fisk, Danielle L. Tomasello, Drahomíra Faktorová, Lucie Hanzálková, Tony
Architecture of the Trypanosome RNA Editing Accessory Complex, MRB1 Michelle L. Ammerman, Kurtis M. Downey, Hassan Hashimi, John C. Fisk, Danielle L. Tomasello, Drahomíra Faktorová, Lucie Hanzálková, Tony
Flowering time. Col C24 Cvi C24xCol C24xCvi ColxCvi
 Flowering time Rosette leaf number 50 45 40 35 30 25 20 15 10 5 0 Col C24 Cvi C24xCol C24xCvi ColxCvi Figure S1. Flowering time in three F 1 hybrids and their parental lines as measured by leaf number
Flowering time Rosette leaf number 50 45 40 35 30 25 20 15 10 5 0 Col C24 Cvi C24xCol C24xCvi ColxCvi Figure S1. Flowering time in three F 1 hybrids and their parental lines as measured by leaf number
Glyma10g15850 aagggatccattctggaaccatatcttgctgtg ttgggtacccttggatgcaggatgacacg AtMKK6, AT5g56580
 Supplemental table 1. Soybean MAPK, MAPKK, and MAPKKK genes and primers for VIGS cloning Gene_ID Forward Primer Reverse Primer Arabidopsis Ortholog Glyma04g03210 aagggatcctgcagcaagaaccagttcat ttgggtaccctaatggatgcgcattaggataaag
Supplemental table 1. Soybean MAPK, MAPKK, and MAPKKK genes and primers for VIGS cloning Gene_ID Forward Primer Reverse Primer Arabidopsis Ortholog Glyma04g03210 aagggatcctgcagcaagaaccagttcat ttgggtaccctaatggatgcgcattaggataaag
Forensic SNP Genotyping using Nanopore MinION Sequencing
 Forensic SNP Genotyping using Nanopore MinION Sequencing AUTHORS Senne Cornelis 1, Yannick Gansemans 1, Lieselot Deleye 1, Dieter Deforce 1,#,Filip Van Nieuwerburgh 1,#,* 1 Laboratory of Pharmaceutical
Forensic SNP Genotyping using Nanopore MinION Sequencing AUTHORS Senne Cornelis 1, Yannick Gansemans 1, Lieselot Deleye 1, Dieter Deforce 1,#,Filip Van Nieuwerburgh 1,#,* 1 Laboratory of Pharmaceutical
The beet R locus encodes a new cytochrome P450 required for red. betalain production.
 The beet R locus encodes a new cytochrome P450 required for red betalain production. Gregory J. Hatlestad, Rasika M. Sunnadeniya, Neda A. Akhavan, Antonio Gonzalez, Irwin L. Goldman, J. Mitchell McGrath,
The beet R locus encodes a new cytochrome P450 required for red betalain production. Gregory J. Hatlestad, Rasika M. Sunnadeniya, Neda A. Akhavan, Antonio Gonzalez, Irwin L. Goldman, J. Mitchell McGrath,
SOLiD Technology. library preparation & Sequencing Chemistry (sequencing by ligation!) Imaging and analysis. Application specific sample preparation
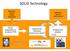 SOLiD Technology Application specific sample preparation Application specific data analysis library preparation & emulsion PCR Sequencing Chemistry (sequencing by ligation!) Imaging and analysis SOLiD
SOLiD Technology Application specific sample preparation Application specific data analysis library preparation & emulsion PCR Sequencing Chemistry (sequencing by ligation!) Imaging and analysis SOLiD
CLUSTALW Multiple Sequence Alignment
 Version 3.2 CLUSTALW Multiple Sequence Alignment Selected Sequences) FETA_GORGO FETA_HORSE FETA_HUMAN FETA_MOUSE FETA_PANTR FETA_RAT Import Alignments) Return Help Report Bugs Fasta label *) Workbench
Version 3.2 CLUSTALW Multiple Sequence Alignment Selected Sequences) FETA_GORGO FETA_HORSE FETA_HUMAN FETA_MOUSE FETA_PANTR FETA_RAT Import Alignments) Return Help Report Bugs Fasta label *) Workbench
University of Bristol - Explore Bristol Research
 Tan, B. G., Wellesley, F. C., Savery, N. J., & Szczelkun, M. D. (2016). Length heterogeneity at conserved sequence block 2 in human mitochondrial DNA acts as a rheostat for RNA polymerase POLRMT activity.
Tan, B. G., Wellesley, F. C., Savery, N. J., & Szczelkun, M. D. (2016). Length heterogeneity at conserved sequence block 2 in human mitochondrial DNA acts as a rheostat for RNA polymerase POLRMT activity.
MELLÉKLET / ANNEX. EU MEGFELELŐSÉGI NYILATKOZAT-hoz for EU DECLARATION OF CONFORMITY
 TRACON BUDAPEST KFT. TRACON BUDAPEST LTD. 2120 DUNAKESZI, PALLAG U. 23. TEL.: (27) 540-000, FAX: (27) 540-005 WWW.TRACON.HU EU MEGFELELŐSÉGI NYILATKOZAT, a 79/1997. (XII. 31.) IKIM számú és a 374/2012.
TRACON BUDAPEST KFT. TRACON BUDAPEST LTD. 2120 DUNAKESZI, PALLAG U. 23. TEL.: (27) 540-000, FAX: (27) 540-005 WWW.TRACON.HU EU MEGFELELŐSÉGI NYILATKOZAT, a 79/1997. (XII. 31.) IKIM számú és a 374/2012.
Mapping Sequencing Reads to a Reference Genome
 Mapping Sequencing Reads to a Reference Genome High Throughput Sequencing RN Example applications: Sequencing a genome (DN) Sequencing a transcriptome and gene expression studies (RN) ChIP (chromatin immunoprecipitation)
Mapping Sequencing Reads to a Reference Genome High Throughput Sequencing RN Example applications: Sequencing a genome (DN) Sequencing a transcriptome and gene expression studies (RN) ChIP (chromatin immunoprecipitation)
T-helper Type 2 driven Inflammation Defines Major Subphenotypes of Asthma
 T-helper Type 2 driven Inflammation Defines Major Subphenotypes of Asthma Prescott G. Woodruff, M.D., M.P.H., Barmak Modrek, Ph.D., David F. Choy, B.S., Guiquan Jia, M.D., Alexander R. Abbas, Ph.D., Almut
T-helper Type 2 driven Inflammation Defines Major Subphenotypes of Asthma Prescott G. Woodruff, M.D., M.P.H., Barmak Modrek, Ph.D., David F. Choy, B.S., Guiquan Jia, M.D., Alexander R. Abbas, Ph.D., Almut
Supplemental Table S1. Overview of MYB transcription factor genes analyzed for expression in red and pink tomato fruit.
 Supplemental Table S1. Overview of MYB transcription factor genes analyzed for expression in red and pink tomato fruit. MYB Primer pairs TC AtMYB Forward Reverse TC199266 MYB12 AGGCTCTTGGAGGTCGTTACC CAACTCTTTCCGCATCTCAATAATC
Supplemental Table S1. Overview of MYB transcription factor genes analyzed for expression in red and pink tomato fruit. MYB Primer pairs TC AtMYB Forward Reverse TC199266 MYB12 AGGCTCTTGGAGGTCGTTACC CAACTCTTTCCGCATCTCAATAATC
Supplementary Figure 1
 Supplementary Figure 1 Plot of delta-afe of sequence variants detected between resistant and susceptible pool over the genome sequence of the WB42 line of B. vulgaris ssp. maritima. The delta-afe values
Supplementary Figure 1 Plot of delta-afe of sequence variants detected between resistant and susceptible pool over the genome sequence of the WB42 line of B. vulgaris ssp. maritima. The delta-afe values
Oxacillin MIC µ g ml borderline oxacillin-resistant Staphylococcus aureus. oxacillin Staphylococcus aureus MRSA
 oxacillin Staphylococcus aureus MRSA 8 17 11 1 Oxacillin MIC 1248 µ gml borderline oxacillin-resistant Staphylococcus aureus BORSA79 NCCLS CLSIM100-S142004 cefoxitin disk oxacillin cefoxitin methicillin-resistant
oxacillin Staphylococcus aureus MRSA 8 17 11 1 Oxacillin MIC 1248 µ gml borderline oxacillin-resistant Staphylococcus aureus BORSA79 NCCLS CLSIM100-S142004 cefoxitin disk oxacillin cefoxitin methicillin-resistant
Bioinformatics: Blending. Biology and Computer Science
 Bioinformatics: Blending Biology and Computer Science MDNMSITNTPTSNDACLSIVHSLMCHRQ GGESETFAKRAIESLVKKLKEKKDELDSL ITAITTNGAHPSKCVTIQRTLDGRLQVAG RKGFPHVIYARLWRWPDLHKNELKHVK YCQYAFDLKCDSVCVNPYHYERVVSPGI DLSGLTLQSNAPSSMMVKDEYVHDFEG
Bioinformatics: Blending Biology and Computer Science MDNMSITNTPTSNDACLSIVHSLMCHRQ GGESETFAKRAIESLVKKLKEKKDELDSL ITAITTNGAHPSKCVTIQRTLDGRLQVAG RKGFPHVIYARLWRWPDLHKNELKHVK YCQYAFDLKCDSVCVNPYHYERVVSPGI DLSGLTLQSNAPSSMMVKDEYVHDFEG
Tutorial 1 The Central Dogma of molecular biology
 oday DN RN rotein utorial 1 he entral Dogma of molecular biology Information flow in genetics:» ranscription» ranslation» Making sense of genomic information Information content in DN - Information content
oday DN RN rotein utorial 1 he entral Dogma of molecular biology Information flow in genetics:» ranscription» ranslation» Making sense of genomic information Information content in DN - Information content
T Á J É K O Z T A T Ó. A 1108INT számú nyomtatvány a http://www.nav.gov.hu webcímen a Letöltések Nyomtatványkitöltő programok fülön érhető el.
 T Á J É K O Z T A T Ó A 1108INT számú nyomtatvány a http://www.nav.gov.hu webcímen a Letöltések Nyomtatványkitöltő programok fülön érhető el. A Nyomtatványkitöltő programok fület választva a megjelenő
T Á J É K O Z T A T Ó A 1108INT számú nyomtatvány a http://www.nav.gov.hu webcímen a Letöltések Nyomtatványkitöltő programok fülön érhető el. A Nyomtatványkitöltő programok fület választva a megjelenő
kpis(ppk20) kpis(ppk46)
 Table S1. C. elegans strains used in this study Strains carrying transgenes Strain Transgene Genotype Reference IT213 tcer-1 prom:tcer-1orf:gfp:tcer-1 3'utr unc-119(ed3) III; kpis(ppk6) This study IT283
Table S1. C. elegans strains used in this study Strains carrying transgenes Strain Transgene Genotype Reference IT213 tcer-1 prom:tcer-1orf:gfp:tcer-1 3'utr unc-119(ed3) III; kpis(ppk6) This study IT283
4 vana, vanb, vanc1, vanc2
 77 1) 2) 2) 3) 4) 5) 1) 2) 3) 4) 5) 16 12 7 17 4 8 2003 1 2004 7 3 Enterococcus 5 Enterococcus faecalis 1 Enterococcus avium 1 Enterococcus faecium 2 5 PCR E. faecium 1 E-test MIC 256 mg/ml vana MRSA MRSA
77 1) 2) 2) 3) 4) 5) 1) 2) 3) 4) 5) 16 12 7 17 4 8 2003 1 2004 7 3 Enterococcus 5 Enterococcus faecalis 1 Enterococcus avium 1 Enterococcus faecium 2 5 PCR E. faecium 1 E-test MIC 256 mg/ml vana MRSA MRSA
Animal welfare, etológia és tartástechnológia
 Animal welfare, etológia és tartástechnológia Animal welfare, ethology and housing systems Volume 4 Issue 2 Különszám Gödöllı 2008 468 EGYEDAZONOSÍTÁS ÉS SZÁRMAZÁSELLENİRZÉS HIPERPOLIMORF MIKROSZATELLITA
Animal welfare, etológia és tartástechnológia Animal welfare, ethology and housing systems Volume 4 Issue 2 Különszám Gödöllı 2008 468 EGYEDAZONOSÍTÁS ÉS SZÁRMAZÁSELLENİRZÉS HIPERPOLIMORF MIKROSZATELLITA
tccattaattcgacagaccagagttaaataatccttgtatgccattgtgatcacatctacagttcagattttgtatttca
 Figure Legends for Supplementary Materials: Fig. 1. Nucleotide sequence of the zebrafish lumican (zlum) gene. Exons are indicated by uppercase letters, and introns are indicated by lowercase letters. The
Figure Legends for Supplementary Materials: Fig. 1. Nucleotide sequence of the zebrafish lumican (zlum) gene. Exons are indicated by uppercase letters, and introns are indicated by lowercase letters. The
Correlation & Linear Regression in SPSS
 Petra Petrovics Correlation & Linear Regression in SPSS 4 th seminar Types of dependence association between two nominal data mixed between a nominal and a ratio data correlation among ratio data Correlation
Petra Petrovics Correlation & Linear Regression in SPSS 4 th seminar Types of dependence association between two nominal data mixed between a nominal and a ratio data correlation among ratio data Correlation
Eredeti gyógyszerkutatás. ELTE TTK vegyészhallgatók számára Dr Arányi Péter 2009 március, 4.ea. Szerkezet optimalizálás (I.)
 Eredeti gyógyszerkutatás ELTE TTK vegyészhallgatók számára Dr Arányi Péter 2009 március, 4.ea. Szerkezet optimalizálás (I.) 1 How do we proceed? New target Internal Literature Validation Selected Target
Eredeti gyógyszerkutatás ELTE TTK vegyészhallgatók számára Dr Arányi Péter 2009 március, 4.ea. Szerkezet optimalizálás (I.) 1 How do we proceed? New target Internal Literature Validation Selected Target
A rosszindulatú daganatos halálozás változása 1975 és 2001 között Magyarországon
 A rosszindulatú daganatos halálozás változása és között Eredeti közlemény Gaudi István 1,2, Kásler Miklós 2 1 MTA Számítástechnikai és Automatizálási Kutató Intézete, Budapest 2 Országos Onkológiai Intézet,
A rosszindulatú daganatos halálozás változása és között Eredeti közlemény Gaudi István 1,2, Kásler Miklós 2 1 MTA Számítástechnikai és Automatizálási Kutató Intézete, Budapest 2 Országos Onkológiai Intézet,
MEGFELELŐSÉGI NYILATKOZAT DECLARATION OF CONFORMITY
 TRACON BUDAPEST KFT. TRACON BUDAPEST LTD. 2120 DUNAKESZI, PALLAG U. 23. TEL.: (27) 540-000, FAX: (27) 540-005 WWW.TRACON.HU MEGFELELŐSÉGI NYILATKOZAT a 79/1997. (XII.31.) IKIM rendelet, valamint az MSZ
TRACON BUDAPEST KFT. TRACON BUDAPEST LTD. 2120 DUNAKESZI, PALLAG U. 23. TEL.: (27) 540-000, FAX: (27) 540-005 WWW.TRACON.HU MEGFELELŐSÉGI NYILATKOZAT a 79/1997. (XII.31.) IKIM rendelet, valamint az MSZ
(A F) H&E staining of the duodenum in the indicated genotypes at E16.5 (A, D), E18.5
 Fig. S1. Brg1 ablation leads to abnormal villous structure in the duodenum in the perinatal period (A F) H&E staining of the duodenum in the indicated genotypes at E16.5 (A, D), E18.5 (B, E), and P0.5
Fig. S1. Brg1 ablation leads to abnormal villous structure in the duodenum in the perinatal period (A F) H&E staining of the duodenum in the indicated genotypes at E16.5 (A, D), E18.5 (B, E), and P0.5
AZ ERDÕ NÖVEKEDÉSÉNEK VIZSGÁLATA TÉRINFORMATIKAI ÉS FOTOGRAMMETRIAI MÓDSZEREKKEL KARSZTOS MINTATERÜLETEN
 Tájökológiai Lapok 5 (2): 287 293. (2007) 287 AZ ERDÕ NÖVEKEDÉSÉNEK VIZSGÁLATA TÉRINFORMATIKAI ÉS FOTOGRAMMETRIAI MÓDSZEREKKEL KARSZTOS MINTATERÜLETEN ZBORAY Zoltán Honvédelmi Minisztérium Térképészeti
Tájökológiai Lapok 5 (2): 287 293. (2007) 287 AZ ERDÕ NÖVEKEDÉSÉNEK VIZSGÁLATA TÉRINFORMATIKAI ÉS FOTOGRAMMETRIAI MÓDSZEREKKEL KARSZTOS MINTATERÜLETEN ZBORAY Zoltán Honvédelmi Minisztérium Térképészeti
Adattípusok. Max. 2GByte
 Adattípusok Típus Méret Megjegyzés Konstans BIT 1 bit TRUE/FALSE SMALLINT 2 byte -123 INTEGER 4 byte -123 COUNTER 4 byte Automatikus 123 REAL 4 byte -12.34E-2 FLOAT 8 byte -12.34E-2 CURRENCY / MONEY 8
Adattípusok Típus Méret Megjegyzés Konstans BIT 1 bit TRUE/FALSE SMALLINT 2 byte -123 INTEGER 4 byte -123 COUNTER 4 byte Automatikus 123 REAL 4 byte -12.34E-2 FLOAT 8 byte -12.34E-2 CURRENCY / MONEY 8
Supplemental Information. RNase H1-Dependent Antisense Oligonucleotides. Are Robustly Active in Directing RNA Cleavage
 YMTHE, Volume 25 Supplemental Information RNase H1-Dependent Antisense Oligonucleotides Are Robustly Active in Directing RNA Cleavage in Both the Cytoplasm and the Nucleus Xue-Hai Liang, Hong Sun, Joshua
YMTHE, Volume 25 Supplemental Information RNase H1-Dependent Antisense Oligonucleotides Are Robustly Active in Directing RNA Cleavage in Both the Cytoplasm and the Nucleus Xue-Hai Liang, Hong Sun, Joshua
DI-, TETRA- ÉS HEXAPLOID TRITICUM FAJOK GENOMJAINAK ELEMZÉSE ÉS AZOK ÖSSZEHASONLÍTÁSA FLUORESZCENS IN SITU HIBRIDIZÁCIÓVAL
 Hagyomány és haladás a növénynemesítésben DI-, TETRA- ÉS HEXAPLOID TRITICUM FAJOK GENOMJAINAK ELEMZÉSE ÉS AZOK ÖSSZEHASONLÍTÁSA FLUORESZCENS IN SITU HIBRIDIZÁCIÓVAL VARGA MÓNIKA, MOLNÁR ISTVÁN ÉS KOVÁCS
Hagyomány és haladás a növénynemesítésben DI-, TETRA- ÉS HEXAPLOID TRITICUM FAJOK GENOMJAINAK ELEMZÉSE ÉS AZOK ÖSSZEHASONLÍTÁSA FLUORESZCENS IN SITU HIBRIDIZÁCIÓVAL VARGA MÓNIKA, MOLNÁR ISTVÁN ÉS KOVÁCS
Adattípusok. Max. 2GByte
 Adattípusok Típus Méret Megjegyzés Konstans BIT 1 bit TRUE/FALSE TINIINT 1 byte 12 SMALLINT 2 byte -123 INTEGER 4 byte -123 COUNTER 4 byte Automatikus 123 REAL 4 byte -12.34E-2 FLOAT 8 byte -12.34E-2 CURRENCY
Adattípusok Típus Méret Megjegyzés Konstans BIT 1 bit TRUE/FALSE TINIINT 1 byte 12 SMALLINT 2 byte -123 INTEGER 4 byte -123 COUNTER 4 byte Automatikus 123 REAL 4 byte -12.34E-2 FLOAT 8 byte -12.34E-2 CURRENCY
vancomycin CFSL cefepime CFPM cefozopran CZOP 4 cephem cefpirome CPR cefoselis Inhibitory Concentration FIC index
 vancomycin cephem 1a 2 1 1 2 a : 15 4 15 15 9 5 1999 2 methicillin MRSA12 vancomycinvcm 4 cephem cefpiromecprcefoseliscfslcefepimecfpmcefozopranczop VCM CPR 12 77 75.5CFSL 82.4CFPM 81 79.4 CZOP 76 74.5
vancomycin cephem 1a 2 1 1 2 a : 15 4 15 15 9 5 1999 2 methicillin MRSA12 vancomycinvcm 4 cephem cefpiromecprcefoseliscfslcefepimecfpmcefozopranczop VCM CPR 12 77 75.5CFSL 82.4CFPM 81 79.4 CZOP 76 74.5
Supplementary Figure 1
 Supplementary Figure 1 Barcode analysis for the identification of mirnas that break B cell tolerance. (a) Barcode identification method. Genomic DNA samples from purified splenic B cells that escaped tolerance
Supplementary Figure 1 Barcode analysis for the identification of mirnas that break B cell tolerance. (a) Barcode identification method. Genomic DNA samples from purified splenic B cells that escaped tolerance
3. Nemzetközi talajinformációs rendszerek
 Magyar Tudományos Akadémia Agrártudományi Kutatóközpont Talajtani és Agrokémiai Intézet Környezetinformatikai Osztály Pásztor László: Térbeli Talajinformációs Rendszerek/ Bevezetés a digitális talajtérképezésbe
Magyar Tudományos Akadémia Agrártudományi Kutatóközpont Talajtani és Agrokémiai Intézet Környezetinformatikai Osztály Pásztor László: Térbeli Talajinformációs Rendszerek/ Bevezetés a digitális talajtérképezésbe
DECLARATION OF PERFORMANCE No. GST REV 1.03 According to Construction Products Regulation EU No. 305/2011
 DECLARATION OF PERFORMANCE No. According to Construction Products Regulation EU No. 305/2011 This declaration is available in the following languages: English Declaration of Performance Page 2-3 Hungarian
DECLARATION OF PERFORMANCE No. According to Construction Products Regulation EU No. 305/2011 This declaration is available in the following languages: English Declaration of Performance Page 2-3 Hungarian
Genome 373: Hidden Markov Models I. Doug Fowler
 Genome 373: Hidden Markov Models I Doug Fowler Review From Gene Prediction I transcriptional start site G open reading frame transcriptional termination site promoter 5 untranslated region 3 untranslated
Genome 373: Hidden Markov Models I Doug Fowler Review From Gene Prediction I transcriptional start site G open reading frame transcriptional termination site promoter 5 untranslated region 3 untranslated
Miskolci Egyetem Gazdaságtudományi Kar Üzleti Információgazdálkodási és Módszertani Intézet. Correlation & Linear. Petra Petrovics.
 Correlation & Linear Regression in SPSS Petra Petrovics PhD Student Types of dependence association between two nominal data mixed between a nominal and a ratio data correlation among ratio data Exercise
Correlation & Linear Regression in SPSS Petra Petrovics PhD Student Types of dependence association between two nominal data mixed between a nominal and a ratio data correlation among ratio data Exercise
BEVEZETÉS Az objektum fogalma
 BEVEZETÉS Az objektum fogalma Program (1) Adat (2) Objektum Kiadványszerkesztés Word Táblázatkezelés Excel CAD AutoCad Adatbáziskezelés Access 1 Program (1) Adat (2) Objektum Adatmodell (2) A valós világ
BEVEZETÉS Az objektum fogalma Program (1) Adat (2) Objektum Kiadványszerkesztés Word Táblázatkezelés Excel CAD AutoCad Adatbáziskezelés Access 1 Program (1) Adat (2) Objektum Adatmodell (2) A valós világ
Supplementary materials to: Whole-mount single molecule FISH method for zebrafish embryo
 Supplementary materials to: Whole-mount single molecule FISH method for zebrafish embryo Yuma Oka and Thomas N. Sato Supplementary Figure S1. Whole-mount smfish with and without the methanol pretreatment.
Supplementary materials to: Whole-mount single molecule FISH method for zebrafish embryo Yuma Oka and Thomas N. Sato Supplementary Figure S1. Whole-mount smfish with and without the methanol pretreatment.
Construction of a cube given with its centre and a sideline
 Transformation of a plane of projection Construction of a cube given with its centre and a sideline Exercise. Given the center O and a sideline e of a cube, where e is a vertical line. Construct the projections
Transformation of a plane of projection Construction of a cube given with its centre and a sideline Exercise. Given the center O and a sideline e of a cube, where e is a vertical line. Construct the projections
AZ ANABOLIKUS ÉS SZEKVENCIÁLIS KEZELÉS HELYE AZ OSTEOPOROSIS TERÁPIÁJÁBAN
 AZ ANABOLIKUS ÉS SZEKVENCIÁLIS KEZELÉS HELYE AZ OSTEOPOROSIS TERÁPIÁJÁBAN SEMMELWEIS EGYETEM Általános Orvostudományi Kar Semmelweis Egyetem Anabolikus kezelések teriparatid abaloparatid romosozumab Szekvenciális
AZ ANABOLIKUS ÉS SZEKVENCIÁLIS KEZELÉS HELYE AZ OSTEOPOROSIS TERÁPIÁJÁBAN SEMMELWEIS EGYETEM Általános Orvostudományi Kar Semmelweis Egyetem Anabolikus kezelések teriparatid abaloparatid romosozumab Szekvenciális
 ö ó ö ó ő ó ő ö ó ó ö ó ó ö ő Ü ö ő ó Á Ő Á Á Ó ó ó Ú Ú Í ó Ó Ó ő ó Ó ő ó ő óú Ú Á ő ó ö ő ő ő Ü ó ó ő ó Ú Á Á ó Á ő óú ó ő ű Ú ó Í ő ó Á ö Á ó Á ó Á Í ó ő ó ű ű Ő ö Í ő Ö ő ő ő ő ö Ü ó Á Á Á Ő Ő ő
ö ó ö ó ő ó ő ö ó ó ö ó ó ö ő Ü ö ő ó Á Ő Á Á Ó ó ó Ú Ú Í ó Ó Ó ő ó Ó ő ó ő óú Ú Á ő ó ö ő ő ő Ü ó ó ő ó Ú Á Á ó Á ő óú ó ő ű Ú ó Í ő ó Á ö Á ó Á ó Á Í ó ő ó ű ű Ő ö Í ő Ö ő ő ő ő ö Ü ó Á Á Á Ő Ő ő
escience Regionális Egyetemi sközpont
 escience Regionális Egyetemi Tudásk sközpont ELTE Innováci ciós s Nap Dr. Papp Gábor egyetemi tanár, igazgató Misszió Az escience Regionális Egyetemi Tudásközpont egy olyan kutatásszervezési és kutatásmenedzselési
escience Regionális Egyetemi Tudásk sközpont ELTE Innováci ciós s Nap Dr. Papp Gábor egyetemi tanár, igazgató Misszió Az escience Regionális Egyetemi Tudásközpont egy olyan kutatásszervezési és kutatásmenedzselési
Dr. Ottó Szabolcs Országos Onkológiai Intézet
 Nemzeti Kutatási és Fejlesztési Program 1. Főirány: Életminőség javítása Nemzeti Onkológiai Kutatás-Fejlesztési Konzorcium a daganatos halálozás csökkentésére 1/48/2001. Részjelentés: 200. November 0.-2004.
Nemzeti Kutatási és Fejlesztési Program 1. Főirány: Életminőség javítása Nemzeti Onkológiai Kutatás-Fejlesztési Konzorcium a daganatos halálozás csökkentésére 1/48/2001. Részjelentés: 200. November 0.-2004.
MELLÉKLET / ANNEX. EU MEGFELELŐSÉGI NYILATKOZAT-hoz for EU DECLARATION OF CONFORMITY
 TRACON BUDAPEST KFT. TRACON BUDAPEST LTD. 2120 DUNAKESZI, PALLAG U. 23. TEL.: (27) 540-000, FAX: (27) 540-005 WWW.TRACON.HU EU MEGFELELŐSÉGI NYILATKOZAT a 79/1997. (XII. 31.) IKIM számú és a 374/2012.
TRACON BUDAPEST KFT. TRACON BUDAPEST LTD. 2120 DUNAKESZI, PALLAG U. 23. TEL.: (27) 540-000, FAX: (27) 540-005 WWW.TRACON.HU EU MEGFELELŐSÉGI NYILATKOZAT a 79/1997. (XII. 31.) IKIM számú és a 374/2012.
A fehérjék hierarchikus szerkezete
 Fehérjék felosztása A fehérjék hierarchikus szerkezete Smeller László Semmelweis Egyetem Biofizikai és Sugárbiológiai Intézet Biológiai funkció alapján Enzimek (pl.: tripszin, citokróm-c ) Transzportfehérjék
Fehérjék felosztása A fehérjék hierarchikus szerkezete Smeller László Semmelweis Egyetem Biofizikai és Sugárbiológiai Intézet Biológiai funkció alapján Enzimek (pl.: tripszin, citokróm-c ) Transzportfehérjék
FATERMÉSI FOK MEGHATÁROZÁSA AZ EGÉSZÁLLOMÁNY ÁTLAGNÖVEDÉKE ALAPJÁN
 4. évfolyam 2. szám 2 0 1 4 101 107. oldal FATERMÉSI FOK MEGHATÁROZÁSA AZ EGÉSZÁLLOMÁNY ÁTLAGNÖVEDÉKE ALAPJÁN Veperdi Gábor Nyugat-magyarországi Egyetem, Erdômérnöki Kar Kivonat A fatermési fok meghatározása
4. évfolyam 2. szám 2 0 1 4 101 107. oldal FATERMÉSI FOK MEGHATÁROZÁSA AZ EGÉSZÁLLOMÁNY ÁTLAGNÖVEDÉKE ALAPJÁN Veperdi Gábor Nyugat-magyarországi Egyetem, Erdômérnöki Kar Kivonat A fatermési fok meghatározása
First experiences with Gd fuel assemblies in. Tamás Parkó, Botond Beliczai AER Symposium 2009.09.21 25.
 First experiences with Gd fuel assemblies in the Paks NPP Tams Parkó, Botond Beliczai AER Symposium 2009.09.21 25. Introduction From 2006 we increased the heat power of our units by 8% For reaching this
First experiences with Gd fuel assemblies in the Paks NPP Tams Parkó, Botond Beliczai AER Symposium 2009.09.21 25. Introduction From 2006 we increased the heat power of our units by 8% For reaching this
PIAL1 (At1g08910) ORF sequence
 PIAL1 (At1g08910) ORF sequence ATGGTTATTCCGGCGACTTCTAGGTTTGGGTTTCGTGCTGAATTCAACACCAAGGAGTTTCAAGCTTCCTGCATCTCTCT! CGCTAATGAAATCGATGCGGCTATTGGGAGAAATGAAGTTCCAGGGAATATTCAAGAGCTCGCTTTAATCCTCAATAATG! TGTGCCGACGTAAATGTGATGATTATCAAACCAGGGCGGTGGTAATGGCGCTGATGATCTCAGTCAAGAGTGCTTGTCAG!
PIAL1 (At1g08910) ORF sequence ATGGTTATTCCGGCGACTTCTAGGTTTGGGTTTCGTGCTGAATTCAACACCAAGGAGTTTCAAGCTTCCTGCATCTCTCT! CGCTAATGAAATCGATGCGGCTATTGGGAGAAATGAAGTTCCAGGGAATATTCAAGAGCTCGCTTTAATCCTCAATAATG! TGTGCCGACGTAAATGTGATGATTATCAAACCAGGGCGGTGGTAATGGCGCTGATGATCTCAGTCAAGAGTGCTTGTCAG!
Supplementary: A strategy to optimize the generation of stable chromobody cell lines for
 Supplementary: A strategy to optimize the generation of stable chromobody cell lines for visualization and quantification of endogenous proteins in living cells Bettina-Maria Keller 1, Julia Maier 1, Melissa
Supplementary: A strategy to optimize the generation of stable chromobody cell lines for visualization and quantification of endogenous proteins in living cells Bettina-Maria Keller 1, Julia Maier 1, Melissa
- Supplementary Data
 1 Open Access Asian Australas. J. Anim. Sci. Vol. 29, No. 3 : 321-326 March 2016 http://dx.doi.org/10.5713/ajas.15.0331 www.ajas.info pissn 1011-2367 eissn 1976-5517 Analysis of Swine Leukocyte Antigen
1 Open Access Asian Australas. J. Anim. Sci. Vol. 29, No. 3 : 321-326 March 2016 http://dx.doi.org/10.5713/ajas.15.0331 www.ajas.info pissn 1011-2367 eissn 1976-5517 Analysis of Swine Leukocyte Antigen
SZTE-ELTE PROTEOMIKAI INNOVÁCIÓ: KONCEPCIÓ, EREDMÉNYEK, JÖVŐKÉP
 SZTE-ELTE PROTEOMIKAI INNOVÁCIÓ: KONCEPCIÓ, EREDMÉNYEK, JÖVŐKÉP Minden elméletet úgy is meg lehet alkotni, hogy az általa sugallt kísérletek csak magát az elméletet erősítsék meg Konrad Lorenz A SEJTMŰKÖDÉS
SZTE-ELTE PROTEOMIKAI INNOVÁCIÓ: KONCEPCIÓ, EREDMÉNYEK, JÖVŐKÉP Minden elméletet úgy is meg lehet alkotni, hogy az általa sugallt kísérletek csak magát az elméletet erősítsék meg Konrad Lorenz A SEJTMŰKÖDÉS
Construction work for the oil and gas industry
 Construction work for the oil and gas industry Info Version 3 Url http://com.mercell.com/permalink/36701750.aspx External tender id 130943-2013 Tender type Contract Award Document type Contract award Procurement
Construction work for the oil and gas industry Info Version 3 Url http://com.mercell.com/permalink/36701750.aspx External tender id 130943-2013 Tender type Contract Award Document type Contract award Procurement
Új temékek az UD- GenoMed Kft. kínálatában!
 Új temékek az UD- GenoMed Kft. kínálatában! Szolgáltatásaink: Medical Genomic Technologies Kft. Betegtoborzás és biobanking Bioinformatika o Adatelemzés/adatbányászás o Integrált adatbázis készítés Sejtvonal
Új temékek az UD- GenoMed Kft. kínálatában! Szolgáltatásaink: Medical Genomic Technologies Kft. Betegtoborzás és biobanking Bioinformatika o Adatelemzés/adatbányászás o Integrált adatbázis készítés Sejtvonal
On The Number Of Slim Semimodular Lattices
 On The Number Of Slim Semimodular Lattices Gábor Czédli, Tamás Dékány, László Ozsvárt, Nóra Szakács, Balázs Udvari Bolyai Institute, University of Szeged Conference on Universal Algebra and Lattice Theory
On The Number Of Slim Semimodular Lattices Gábor Czédli, Tamás Dékány, László Ozsvárt, Nóra Szakács, Balázs Udvari Bolyai Institute, University of Szeged Conference on Universal Algebra and Lattice Theory
EEA, Eionet and Country visits. Bernt Röndell - SES
 EEA, Eionet and Country visits Bernt Röndell - SES Európai Környezetvédelmi Ügynökség Küldetésünk Annak elősegítése, hogy az EU és a tagállamok a szükséges információk alapján hozhassák meg a környezet
EEA, Eionet and Country visits Bernt Röndell - SES Európai Környezetvédelmi Ügynökség Küldetésünk Annak elősegítése, hogy az EU és a tagállamok a szükséges információk alapján hozhassák meg a környezet
Átlagtermés és rekordtermés 8 növénykultúrában
 Átlagtermés és rekordtermés 8 növénykultúrában Növény Rekord termés * Átlag termés * Veszteség típusa Biotikus Abiotikus Abiotikus stressz okozta kiesés (% a rekord terméshez képest) Kukorica 19300 4600
Átlagtermés és rekordtermés 8 növénykultúrában Növény Rekord termés * Átlag termés * Veszteség típusa Biotikus Abiotikus Abiotikus stressz okozta kiesés (% a rekord terméshez képest) Kukorica 19300 4600
Biokatalitikus Baeyer-Villiger oxidációk Doktori (PhD) értekezés tézisei. Muskotál Adél. Dr. Vonderviszt Ferenc
 Biokatalitikus Baeyer-Villiger oxidációk Doktori (PhD) értekezés tézisei Készítette: Muskotál Adél Környezettudományok Doktori Iskola Témavezető: Dr. Vonderviszt Ferenc egyetemi tanár Pannon Egyetem Műszaki
Biokatalitikus Baeyer-Villiger oxidációk Doktori (PhD) értekezés tézisei Készítette: Muskotál Adél Környezettudományok Doktori Iskola Témavezető: Dr. Vonderviszt Ferenc egyetemi tanár Pannon Egyetem Műszaki
Statistical Dependence
 Statistical Dependence Petra Petrovics Statistical Dependence Deinition: Statistical dependence exists when the value o some variable is dependent upon or aected by the value o some other variable. Independent
Statistical Dependence Petra Petrovics Statistical Dependence Deinition: Statistical dependence exists when the value o some variable is dependent upon or aected by the value o some other variable. Independent
A BÜKKI KARSZTVÍZSZINT ÉSZLELŐ RENDSZER KERETÉBEN GYŰJTÖTT HIDROMETEOROLÓGIAI ADATOK ELEMZÉSE
 KARSZTFEJLŐDÉS XIX. Szombathely, 2014. pp. 137-146. A BÜKKI KARSZTVÍZSZINT ÉSZLELŐ RENDSZER KERETÉBEN GYŰJTÖTT HIDROMETEOROLÓGIAI ADATOK ELEMZÉSE ANALYSIS OF HYDROMETEOROLIGYCAL DATA OF BÜKK WATER LEVEL
KARSZTFEJLŐDÉS XIX. Szombathely, 2014. pp. 137-146. A BÜKKI KARSZTVÍZSZINT ÉSZLELŐ RENDSZER KERETÉBEN GYŰJTÖTT HIDROMETEOROLÓGIAI ADATOK ELEMZÉSE ANALYSIS OF HYDROMETEOROLIGYCAL DATA OF BÜKK WATER LEVEL
1 Copyright 2012, Oracle and/or its affiliates. All rights reserved. Insert Information Protection Policy Classification from Slide 13
 1 Copyright 2012, Oracle and/or its affiliates. All rights reserved. Insert Information Protection Policy Classification from Slide 13 Tippek és trükkök - Másfél év üzemeltetési tapasztalatai Exadata környezetben
1 Copyright 2012, Oracle and/or its affiliates. All rights reserved. Insert Information Protection Policy Classification from Slide 13 Tippek és trükkök - Másfél év üzemeltetési tapasztalatai Exadata környezetben
 Ö Á Í Í ű ű ú ű ű ű ű ú ú ú ú ű ű ű ű ű ű ű ű ű ú ű ú ú ú ű ú Á ú ű ű Ó ú ű ű ű ú Ó ú ű ú É ú ú ú ű ű ú ű ú Ú Á ú É ú Ó ú ú ú ú ű ű ű ú É Á É É ű ű Í ú ú Ó Í ű Í ű ű ú ű ű ű É ű ú Á ű ű ú Í ű Á ű ú ú É
Ö Á Í Í ű ű ú ű ű ű ű ú ú ú ú ű ű ű ű ű ű ű ű ű ú ű ú ú ú ű ú Á ú ű ű Ó ú ű ű ű ú Ó ú ű ú É ú ú ú ű ű ú ű ú Ú Á ú É ú Ó ú ú ú ú ű ű ű ú É Á É É ű ű Í ú ú Ó Í ű Í ű ű ú ű ű ű É ű ú Á ű ű ú Í ű Á ű ú ú É
 ö ö ö ö ö ö ö ű ű ö ö ö ö ö Ő ö Ó Ú ö Ö ö ö ö ö Ö Ő ö ö Í Ó Ó Ő ö ö ö ö ö Ő Ő Ó Ő É ö Ú ö ö Ő ö ö ö ö ö ö ö Ő ö Ő É ö Ő ö ö Ő ö ö ö Ó ű ö ö ö Ő ö ö ö Í Ő Ó Í ö ö ö ö Ő Ő Ő Ő Í Ó Ő Ő Í Ő ö ö ö ö ö Ő Ő ö
ö ö ö ö ö ö ö ű ű ö ö ö ö ö Ő ö Ó Ú ö Ö ö ö ö ö Ö Ő ö ö Í Ó Ó Ő ö ö ö ö ö Ő Ő Ó Ő É ö Ú ö ö Ő ö ö ö ö ö ö ö Ő ö Ő É ö Ő ö ö Ő ö ö ö Ó ű ö ö ö Ő ö ö ö Í Ő Ó Í ö ö ö ö Ő Ő Ő Ő Í Ó Ő Ő Í Ő ö ö ö ö ö Ő Ő ö
 Ú ű ü ü Ü ű É É Ö Ö Á ü ü ü ű É ú Á Ö Ü ü ü ű É Á É Ű ű Ü Ü ű ü ű ü ű ü Ü ü ü Ű Á Á Á ű ú ű Á Ó Ó É Á Ó Á Ó ű ü ü ű ű ü ú ú ü ü ü ű ü ű Ü ű ü ü ú ü Ö ü ú ú ü ü ü ü ű ú ü Ó ü Ó Ó ü ü Ó ü ü Ó ű ű ú ű ű ü
Ú ű ü ü Ü ű É É Ö Ö Á ü ü ü ű É ú Á Ö Ü ü ü ű É Á É Ű ű Ü Ü ű ü ű ü ű ü Ü ü ü Ű Á Á Á ű ú ű Á Ó Ó É Á Ó Á Ó ű ü ü ű ű ü ú ú ü ü ü ű ü ű Ü ű ü ü ú ü Ö ü ú ú ü ü ü ü ű ú ü Ó ü Ó Ó ü ü Ó ü ü Ó ű ű ú ű ű ü
 ä ú á á á á á ú á á á ĺ ę ą ą ú á á á á ĺ á ĺ ĺ á á á ö í ů á á á í ł ü ü á á ĺź ĺ á á ó Źá ó á ű ö á á ó í á á ó á ä ü ú á á á á á ü ĺí ü ö áĺ ü á í á ó á ö á á á ó ü á ö á ĺ Ż Ż á í ö Á ź í á á á á ö
ä ú á á á á á ú á á á ĺ ę ą ą ú á á á á ĺ á ĺ ĺ á á á ö í ů á á á í ł ü ü á á ĺź ĺ á á ó Źá ó á ű ö á á ó í á á ó á ä ü ú á á á á á ü ĺí ü ö áĺ ü á í á ó á ö á á á ó ü á ö á ĺ Ż Ż á í ö Á ź í á á á á ö
Széchenyi István Egyetem www.sze.hu/~herno
 Oldal: 1/6 A feladat során megismerkedünk a C# és a LabVIEW összekapcsolásának egy lehetőségével, pontosabban nagyon egyszerű C#- ban írt kódból fordítunk DLL-t, amit meghívunk LabVIEW-ból. Az eljárás
Oldal: 1/6 A feladat során megismerkedünk a C# és a LabVIEW összekapcsolásának egy lehetőségével, pontosabban nagyon egyszerű C#- ban írt kódból fordítunk DLL-t, amit meghívunk LabVIEW-ból. Az eljárás
KockaKobak Országos Matematikaverseny 8. osztály
 KockaKobak Országos Matematikaverseny 8. osztály 2012. november 12. Feladatok: PÉCSI ISTVÁN, középiskolai tanár SZÉP JÁNOS, középiskolai tanár Lektorok: LADÁNYI ANDREA, középiskolai tanár TÓTH JÁNOS, középiskolai
KockaKobak Országos Matematikaverseny 8. osztály 2012. november 12. Feladatok: PÉCSI ISTVÁN, középiskolai tanár SZÉP JÁNOS, középiskolai tanár Lektorok: LADÁNYI ANDREA, középiskolai tanár TÓTH JÁNOS, középiskolai
Téli kedvezményes ajánlataink
 Téli kedvezményes ajánlataink Ajánlataink 2017 január 31-ig érvényesek! BIOBASE műszerek a NucleotestBio Kft. kínálatában! Steril box-ok, lamináris box-ok: Hűtők, fagyasztók, ultramélyhűtők, autoklávok,
Téli kedvezményes ajánlataink Ajánlataink 2017 január 31-ig érvényesek! BIOBASE műszerek a NucleotestBio Kft. kínálatában! Steril box-ok, lamináris box-ok: Hűtők, fagyasztók, ultramélyhűtők, autoklávok,
 ő ľ ľü ľ ľ ü Ü Ü ľ ő ľ Ő ń ľü ľ íľ ő ő źů ő í í ü ö ü ľ ź ő ö ü ő ľő ő ö ü źů ź ź í ö ľ ź ő ľ ü ö ö ź ő đí ź ľ ő ö ű í í ö ü ö í í ú ü í ź ő ő í ú í ő Ó ő ü ú í í ú í ú ő ú ľ ő ü ő ü ű ő ő í ü ö ő í ą
ő ľ ľü ľ ľ ü Ü Ü ľ ő ľ Ő ń ľü ľ íľ ő ő źů ő í í ü ö ü ľ ź ő ö ü ő ľő ő ö ü źů ź ź í ö ľ ź ő ľ ü ö ö ź ő đí ź ľ ő ö ű í í ö ü ö í í ú ü í ź ő ő í ú í ő Ó ő ü ú í í ú í ú ő ú ľ ő ü ő ü ű ő ő í ü ö ő í ą
Correlation & Linear Regression in SPSS
 Correlation & Linear Regression in SPSS Types of dependence association between two nominal data mixed between a nominal and a ratio data correlation among ratio data Exercise 1 - Correlation File / Open
Correlation & Linear Regression in SPSS Types of dependence association between two nominal data mixed between a nominal and a ratio data correlation among ratio data Exercise 1 - Correlation File / Open
Limitations and challenges of genetic barcode quantification
 Limitations and challenges of genetic barcode quantification Lars hielecke, im ranyossy, ndreas Dahl, Rajiv iwari, Ingo Roeder, Hartmut eiger, Boris Fehse, Ingmar lauche and Kerstin ornils SUPPLEMENRY
Limitations and challenges of genetic barcode quantification Lars hielecke, im ranyossy, ndreas Dahl, Rajiv iwari, Ingo Roeder, Hartmut eiger, Boris Fehse, Ingmar lauche and Kerstin ornils SUPPLEMENRY
E-VRV ELOSZTÓ KIVITELI TERV
 0 meglévő épület klimatizálás Építés helye: 800 Tatabánya, Komáromi utca. 800 Tatabánya, Komáromi utca. Megrendelő: MARÉP KFT. 890 Tata, Egység u.. Pf.:. Azonosító: E-VRV Tervjel: E-C-0P7-EVRV Verzió:
0 meglévő épület klimatizálás Építés helye: 800 Tatabánya, Komáromi utca. 800 Tatabánya, Komáromi utca. Megrendelő: MARÉP KFT. 890 Tata, Egység u.. Pf.:. Azonosító: E-VRV Tervjel: E-C-0P7-EVRV Verzió:
PLATTÍROZOTT ALUMÍNIUM LEMEZEK KÖTÉSI VISZONYAINAK TECHNOLÓGIAI VIZSGÁLATA TECHNOLOGICAL INVESTIGATION OF PLATED ALUMINIUM SHEETS BONDING PROPERTIES
 Anyagmérnöki Tudományok, 37. kötet, 1. szám (2012), pp. 371 379. PLATTÍROZOTT ALUMÍNIUM LEMEZEK KÖTÉSI VISZONYAINAK TECHNOLÓGIAI VIZSGÁLATA TECHNOLOGICAL INVESTIGATION OF PLATED ALUMINIUM SHEETS BONDING
Anyagmérnöki Tudományok, 37. kötet, 1. szám (2012), pp. 371 379. PLATTÍROZOTT ALUMÍNIUM LEMEZEK KÖTÉSI VISZONYAINAK TECHNOLÓGIAI VIZSGÁLATA TECHNOLOGICAL INVESTIGATION OF PLATED ALUMINIUM SHEETS BONDING
discosnp demo - Peterlongo Pierre 1 DISCOSNP++: Live demo
 discosnp demo - Peterlongo Pierre 1 DISCOSNP++: Live demo Download and install discosnp demo - Peterlongo Pierre 3 Download web page: github.com/gatb/discosnp Chose latest release (2.2.10 today) discosnp
discosnp demo - Peterlongo Pierre 1 DISCOSNP++: Live demo Download and install discosnp demo - Peterlongo Pierre 3 Download web page: github.com/gatb/discosnp Chose latest release (2.2.10 today) discosnp
TELJESÍTMÉNY NYILATKOZAT 0832-CPD-1651
 E-mail: info@fulleon.co.uk Web: www.cooperfulleon.co m TELJESÍTMÉNY NYILATKOZAT 0832-CPD-1651 Termék azonosító kód: ROLP/SV és ROLP/SV/WP Típus, adagszám vagy gyári szám, illetve bármilyen más elem, amely
E-mail: info@fulleon.co.uk Web: www.cooperfulleon.co m TELJESÍTMÉNY NYILATKOZAT 0832-CPD-1651 Termék azonosító kód: ROLP/SV és ROLP/SV/WP Típus, adagszám vagy gyári szám, illetve bármilyen más elem, amely
Lignocellulóz bontó enzimek előállítása és felhasználása
 Lignocellulóz bontó enzimek előállítása és felhasználása Ph.D. ÉRTEKEZÉS TÉZISEI Juhász Tamás Témavezető: Dr. Réczey Istvánné 2005 Mezőgazdasági Kémiai Technológia Tanszék Budapesti Műszaki és Gazdaságtudományi
Lignocellulóz bontó enzimek előállítása és felhasználása Ph.D. ÉRTEKEZÉS TÉZISEI Juhász Tamás Témavezető: Dr. Réczey Istvánné 2005 Mezőgazdasági Kémiai Technológia Tanszék Budapesti Műszaki és Gazdaságtudományi
8. Gyakorlat SQL. DDL (Data Definition Language) adatdefiníciós nyelv utasításai:
 8. Gyakorlat SQL SQL: Structured Query Language; a relációs adatbáziskezelők szabványos, strukturált lekérdező nyelve SQL szabványok: SQL86, SQL89, SQL92, SQL99, SQL3 Az SQL utasításokat mindig pontosvessző
8. Gyakorlat SQL SQL: Structured Query Language; a relációs adatbáziskezelők szabványos, strukturált lekérdező nyelve SQL szabványok: SQL86, SQL89, SQL92, SQL99, SQL3 Az SQL utasításokat mindig pontosvessző
Salt-Enabled Visual Detection of DNA
 Electronic Supplementary Material (ESI) for ChemComm. This journal is The Royal Society of Chemistry 2014 Supporting Information for Salt-Enabled Visual Detection of DNA Xilong Wang, Xun Chen, Sha Sun,
Electronic Supplementary Material (ESI) for ChemComm. This journal is The Royal Society of Chemistry 2014 Supporting Information for Salt-Enabled Visual Detection of DNA Xilong Wang, Xun Chen, Sha Sun,
 í í ö ő í í í Ö ö í ő í í í í í í Í Ó í ö ő ú ö ú í í ő ő í ö ő í ő í í í ö í í ő ü í ü ő ö í ü ö ö í ö ü ö ő ö ö í í í í ö ő ő ú ö í ő ö ö í ő ö í ő í ü ő í ü ö í í ö í í í ö í ő ö í ő ő ü ö í ő í ö ő
í í ö ő í í í Ö ö í ő í í í í í í Í Ó í ö ő ú ö ú í í ő ő í ö ő í ő í í í ö í í ő ü í ü ő ö í ü ö ö í ö ü ö ő ö ö í í í í ö ő ő ú ö í ő ö ö í ő ö í ő í ü ő í ü ö í í ö í í í ö í ő ö í ő ő ü ö í ő í ö ő
 Í í É Á ö ü Ó Ü ö ü Ü í őú Ü í Í ő í Ó ú í ú í ö í ő ö ö Í í í ú ú ö ő ö ő ö ö ö í í ö ö ö ő ö í ö ő ö í ő ö í ÍÍ ö ő ü í ő ö Ü Ü ö í ő ü ü Ü í Ü Ü ö Ü Ü Ü í ö ő ű ő Ú ő ő ö ő í ö ü ő ö í ű Í Ú ö Ú ü ö
Í í É Á ö ü Ó Ü ö ü Ü í őú Ü í Í ő í Ó ú í ú í ö í ő ö ö Í í í ú ú ö ő ö ő ö ö ö í í ö ö ö ő ö í ö ő ö í ő ö í ÍÍ ö ő ü í ő ö Ü Ü ö í ő ü ü Ü í Ü Ü ö Ü Ü Ü í ö ő ű ő Ú ő ő ö ő í ö ü ő ö í ű Í Ú ö Ú ü ö
