Phoma fajok filogenetikai vizsgálata
|
|
|
- Mariska Nagy
- 9 évvel ezelőtt
- Látták:
Átírás
1 Phoma fajok filogenetikai vizsgálata Irinyi László Kövics György Sándor Erzsébet Debreceni Egyetem Agrártudományi Centrum, Mezőgazdaságtudományi Kar, Növényvédelmi Tanszék, Debrecen ÖSSZEFOGLALÁS A Phoma genusba tartozó gombafajok többsége fitopatogén, opportunista parazita vagy szaprofiton életmódot folytat. Az egyes Phoma fajokat több standard táptalajon megfigyelhető morfológiai bélyeg alapján rendszerezik. Más gombacsoportoknál már használt, és többé-kevésbé megbízhatónak bizonyult molekuláris markerek szekvencia analízise a Phoma fajoknál napjainkig alig került alkalmazásra. Vizsgálatainkhoz olyan filogenetikai markert kerestünk, amely már megbízhatónak bizonyult mind a fajok közötti, mind a fajon belüli csoportok elkülönítésére más gombataxonoknál. Ezek közül választásunk az élővilágban erősen konzervatív, transzlációs elongációs faktorra (tef1) esett, melynek intron régiója az rdns-ben található (Internal Transcribed Spacer=ITS) szekvenciák mellett az egyik leggyakrabban alkalmazott alternatív filogenetikai marker. A tef1 szekvencia által kódolt fehérje (EF-1α) az evolúció során konzervatív, minden sejtben megtalálható, a fehérjeszintézisben kulcsfontosságú protein. Számos tanulmány támasztja alá, hogy a tef1 szekvencia rendelkezik azon tulajdonságokkal, amelyek alkalmassá teszik a fajon belüli és a fajok közötti filogenetikai kapcsolatok meghatározására. Vizsgálatunk során 11 Phoma faj 12 izolátumából izoláltunk genomi DNS-t, majd két specifikus primerrel (EF1-728F és EF1-986R) amplifikáltuk a tef1 gén nagy intronját tartalmazó szakaszát, és meghatároztuk azok szekvenciáját. A fajok filogenetikai elemzését tef1 szekvenciák parsimony-típusú elemzésével végeztük a PAUP*4.0b program alkalmazásával. A tef1 szekvenciák alapján az egyes Phoma fajok jól elkülöníthetők egymástól, ami bizonyítja, hogy a tef1 szekvencia a Phoma fajoknál is alkalmas molekuláris alapon történő filogenetikai rendszerezésre. Az elemzés során kapott filogenetikai törzsfa azonban nem minden esetben mutatott egyezést a morfológia bélyegeken alapuló Phoma taxonokkal. A tef1 szekvencia alapján a Phoma fajok egyértelműen elkülönülnek a közeli rokon Ascochyta fajoktól is. Kulcsszavak: Phoma, ITS régió, transzlációs elongációs faktor, filogenetika SUMMARY The cosmopolitan Phoma genus contains mainly phytopathogenic, opportunistic parasites, and saprophyte fungal species. Up to now, the characterization of Phoma species and other taxa of Phoma has been determined on the basis of morphology on standardized media, and gene sequence analysis was only used as a confirmative or distinctive complement. In this study, we tried to find molecular markers which can be used as phylogenetics markers in the molecular based classification in the Phoma genus. We employed a part of the translation elongation factor 1 subunit alpha (EF-1α=tef1) containing both introns and exons and ITS region containing the internal transcribed spacer regions 1 and 2 and the 5.8S rdna, as potential genetic markers to infer phylogenetic relationships among different Phoma taxa. Twelve different Phoma species sequences were analysed together with the closely related Ascochyta ones. The constructed phylogenetic trees, based on tef1 and ITS sequences, do not support the traditional Phoma sections based on morphological characterization. However, we managed to distinguish between the Phoma strains and Ascochyta species by comparing their tef1 sequences through parsimony analysis. We proved that a tef1 can be a useful phylogenetic marker to resolve phylogenetic relationships at species level in Phoma genus. Both parsimony sequence analyses confirmed that the Phyllosticta sojicola species is identical to the Phoma exigua var. exigua species as Kövics et al. (1999) claimed. However, the evolutionary distance by ITS sequences within Phoma species is too small to get well based consequences for the phylogenetic relationships of Phoma genus. Further investigations would be necessary to clarify whether the tef1 and ITS sequences as phylogenetic molecular markers are well suited for the classification of Phoma species. Keywords: Phoma, ITS sequences, translation elongation factor, phylogenetics BEVEZETÉS A Coelomycetes osztályba tartozó Phoma genus világszerte elterjedt, többségében fitopatogén, opportunista parazita vagy szaprofiton életmódot folytató fajokat foglal magába. Napjainkig mintegy 2000 Phoma fajt azonosítottak világszerte (Boerema et al., 2004). A tradicionális és a molekuláris mikológiában több út is lehetséges az egyes gombafajok identifikálására, kezdve a morfológiai bélyegekkel, az élettani és biokémiai funkciókon át egészen a legmodernebbnek tekintett nukleinsav szekvenciák összehasonlító elemzéséig. A gombataxonómiában jelenleg 3 elfogadott faj fogalom van elterjedve: a morfológiai, a biológiai, és a filogenetikai. A morfológiai faj fogalom az egyes gombafajok morfológiai megjelenését, bélyegeit veszi alapul, és ennek alapján rendszerezi őket. A biológiai faj fogalom szerint azon populációk tartoznak egy fajba, amelyek egymással szexuális úton szaporodási közösséget alkotnak (Mayr, 1940). A biológia faj fogalom azonban csak korlátozva használható a gombák taxonjaira, mivel a gombák mintegy 20%-a nem rendelkezik meiospórával, azaz szexuális úton szaporodni nem képesek (Reynolds és Taylor, 1993). Ennek ellenére nagyon sok aszexuálisan szaporodó gombafaj, beleértve a Phoma genus tagjait is, meglepően nagy genetikai variabilitással rendelkezik (Khon, 1995; Talhinhas et al., 2002). 100
2 A legújabb, filogenetikai faj fogalom bizonyos nukleinsav szekvenciák változatosságát (DNS polimorfizmus) alapul véve próbálja meghatározni a különbségeket vagy azonosságokat az izolátumok között. Harrington és Rizzo (1999) szerint filogenetikai szempontból azonos fajnak tekintendő a populáció azon legkisebb egysége, amely bizonyíthatóan egy közös őstől származik, és valamilyen egyedi, jól diagnosztizálható fenotípusos jelleggel rendelkezik. A gombafajok többségének rendszerezése egészen napjainkig a régebbi, tradicionálisnak tekintett morfológiai alapon, esetenként a biológiai faj kategória alapján történt. A morfológiai alapú faj meghatározásnak azonban gyakori gyengesége, hogy a morfológiai alapon egy fajba sorolt izolátumokról a genetikai vizsgálatok alapján bebizonyosodik, hogy valójában nem egy fajba tartoznak. Mivel a Phoma fajok többsége nem szaporodik ivaros úton, csak ivartalanul, ezért a biológiai faj fogalom nem alkalmazható erre a genusra. A morfológia faj fogalom viszont, a fentebb említett okok miatt nem ad mindig megbízható eredményt. A molekuláris biológia rohamos fejlődésének köszönhetően elérhetővé válik a taxonómusok számára a filogenetikai összefüggések feltárása. Taylor et al. (2000) szerint a filogenetikai módszerek hamarosan széles körben elterjedtté és népszerűvé válnak a gombataxonómiával foglalkozó mikológusok körében, mivel egyaránt alkalmazható ivarosan és ivartalanul szaporodó fajokra is. Napjainkig a Phoma fajok rendszerezése a többi gombacsoporthoz hasonlóan nagyrészt morfológiai, fenotípusos és fiziológai vizsgálatokon alapult. Ennek a munkának az összefoglalásaként a közelmúltban jelent meg egy monográfia Phoma Identification Manual címmel (Boerema et al., 2004), amelyben a szerzők összegzik a Phoma fajok morfológiai szempontok alapján, több mint 40 év kutatási eredményei nyomán tisztázott rendszerét. A 90-es évek közepén a molekuláris biológia és a biokémia fejlődésének köszönhetően izoenzimeket (fehérje polimorfizmus) próbáltak meg molekuláris markerként használni a Phoma genusban, hogy elkülönítsék a morfológiailag azonos megjelenésű, de feltételezhetően eltérő fajhoz tartozó izolátumokat (Kövics és Gruyter, 1995). A későbbiek során azonban a fehérje polimorfizmus profil meghatározásokat egyre inkább felváltották a DNS polimorfizmus vizsgálatok a filogenetikai céllal történő elemzések során. A DNS polimorfizmusnak, amely egy adott gén (marker) nukleinsav sorrendjének változatosságán alapszik, három nagy előnye van a fehérje polimorfizmussal szemben. Az első, hogy a szekvencia változás már az első szinten, a DNS szintjén kimutatható. A második előny, hogy DNS szinten sokkal nagyobb valószínűséggel történnek evolúciós változások, (amelyek nem biztos, hogy fehérje szinten is megmutatkoznak), és végül ehhez kapcsolódik a harmadik előny is, hogy ezen változások nincsenek szelekciós nyomásnak kitéve, hacsak nem befolyásolja jelentősen az egyed fenotípusát, így azok hosszabb távon képesek fennmaradni a genomban. Napjainkban a filogenetikai törzsfa készítésben az egyik leggyakrabban használt molekuláris biológiai módszer kiválasztott DNS szakaszok nukleinsav sorrendjének meghatározása, és ezeknek az összehasonlító elemzése. A filogenetikai célú szekvencia összehasonlítás nagyrészt olyan genom szakaszok vizsgálatán alapul, amelyek minden élőlényben előfordulnak, és meglehetősen konzervatívak maradnak az evolúció során, mint pl. a riboszómális géneket kódoló szekvenciák. Ezek közül nagyon sok tudományos munka alapszik az úgynevezett ITS (Internal Transcribed Spacer) régió vizsgálatán, annak köszönhetően, hogy ennek a régiónak a nukleinsav sorrendjén, amely nagy variabilitást mutat más régiókhoz képest, így alkalmas mind a fajon belüli, mind a fajok közötti filogenetikai kapcsolatok felderítésére. Lutzoni et al. (2004) szerint a gombák körében végzett filogenetikai vizsgálatok 83,9%-a a riboszómális géneket kódoló tandem szekvenciák vizsgálatán alapult. A molekuláris biológiában újabb és újabb géneket (markereket) találnak, amelyek tulajdonságaikból adódóan alkalmasak lehetnek filogenetikai kapcsolatok elemzésére. Vizsgálatunkban az ITS régió mellet a más élőlény csoportok mellett a gombáknál is eredményesen használt transzlációs elongációs faktort kódoló gén (tef1) nagy intronját tartalmazó szakaszát használtuk a Phoma fajok közötti filogenetikai kapcsolatok vizsgálatára. Filogenetikai kapcsolatok feltárására az egyik leggyakrabban használt molekuláris markerek a riboszómális géneket kódoló szekvenciák (rdns) (Avise, 2004). Ezek közül is különösen azok, amelyek gyakorlatilag azonos szekvenciájú tandem másolatként vannak jelen a genomban. Minden egyes másolat rendelkezik kódoló és nem kódoló régiókkal. A kódoló régiókhoz tartoznak a 18S, 5.8S, és 28S alegységeket kódoló gének (Gerbi, 1985), amelyek sokkal kevésbé változékonyak, mint a nem kódoló szakaszok. Ez utóbbiak lehetnek átírtak, mint az Internal Transcribed Spacer 1 és 2, valamint olyan régiók, amelyek nem íródnak át (Intergenic Spacer 1 és 2). Mivel a kódoló régiók evolúciósan erősen konzerváltak, az évmilliók alatt alig változott szakaszok, ezért főleg nagyobb taxonok rokonsági viszonyainak megállapítására használják (Hillis és Dixon, 1991). A két ITS régiót, evolúciós mércével mérve viszonylag gyorsan változó tulajdonságaiból adódóan, többségében alacsonyabb szintű rendszertani kapcsolatok vizsgálatánál használják, mint például közel rokon fajok vagy populációk egymástól való elkülönítésére gombáknál, növényeknél és állatoknál egyaránt (Baldwin, 1992; Schlötterer et al., 1994; Mai és Coleman, 1997; Weekers et al., 2001; Oliverio et al., 2002; Chen et al., 2000, 2002). Vizsgálatunkhoz az rdns régió egy olyan szakaszát választottuk, amely tartalmazza a ITS 1 és 2, valamint az 5,8 rdns egy részét (1. ábra). 101
3 1. ábra: az ITS régió sematikus vázlata, valamint a PCR reakcióban használt primerek helyzete Forrás: White et al., 1990 Figure 1: Schematic structure of ITS region in Phoma spp. and location of primers for phylogenetic analysis A translation elongation factor 1 subunit alpha (EF-1α) fehérje a sejten belül a citoszólban található. Eukariótáknál és Archaea baktériumoknál a fehérjeszintézis folyamatában elsődleges funkciója, hogy katalizálja a GTP függő aminoacil-trns komplex felbomlását (Moldave, 1985). Az EF-1α fehérje, és így a fehérjét kódoló gén is erősen konzervált az élővilágban, ami alkalmassá teszi a filogenetikai vizsgálatokra (Roger et al., 1999). A fehérjét kódoló tef1 gén minden élő szervezetben megtalálható és az ITS szekvenciákkal szemben nagy előnye, hogy a gén csak egy kópiában van jelen a genomban (Baldauf és Doolittle, 1997). A fajok közötti és fajon belüli rendszertani kapcsolatok felderítésére egyaránt alkalmas, mint azt Druzhinina és Kubicek (2005) is bizonyították Trichoderma fajoknál, illetve Roger et al. (1999) egyéb fajoknál (pl. Mucor racemosus, Podospora anserina). Egyetlen hátránya, hogy a fehérjét kódoló tef1 gén rövidebb, mint más filogenetikai markerként használt gének. Mintegy 2 kb hosszúságú, intront és exont egyaránt tartalmazó szakasz. Filogenetikai vizsgálatunkhoz a tef1 gén nagy intronját tartalmazó fragmentjét választottuk (2. ábra). Vizsgálatainkban arra a kérdésre kerestük a választ, hogy az ITS régió valamint a EF-1α fehérjét kódoló tef1 gén alkalmas-e rendszertani vizsgálatokra a Phoma genusban? 2. ábra: tef1 gén sematikus vázlata, valamint a PCR reakcióban használt primerek helyzete Forrás: Druzhinina és Kubicek, 2005 Figure 2: Schematic structure of tef1 gene in Phoma spp. and location of primers for phylogenetic analyses (Druzhinina and Kubicek, 2005) ANYAG ÉS MÓDSZER Vizsgálatainkban a Debreceni Egyetem Növényvédelmi Tanszékén található törzsgyűjteményből 11 Phoma faj 12 izolátumát vizsgáltunk (1. táblázat). Minden egyes fajt morfológiai és élettani jellegzetességeik alapján azonosítottunk a Boerema et al. (2004) által közreadott Phoma monográfia alapján. Az izolátumokat 50 ml folyékony maláta tápoldatban tenyésztettük 48 órán keresztül, 100 ml-es Erlenmayer lombikban, sötétben, rázatva (125 rpm). A sejteket dörzsmozsár segítségével, folyékony nitrogén jelenlétében tártuk fel, majd genomi DNS-t izoláltunk E.Z.N.A. Fungal DNA Isolation Kit (Omega Bio-tek Inc., USA) alkalmazásával, a gyártó utasításai szerint. A ITS fragment felszaporításához az SR6R és LR1 primerpárt (White et al., 1990), míg a tef1 fragment amplifikálásához az EF1-728F és EF1-986R primerpárt használtuk (Druzhinina és Kubicek, 2005). A tisztított PCR termékek szekvenálását az MWG Biotech, Germany végezte. A szekvenciákat a ClustalX ( Thompson et al., 1997) program felhasználásával rendeztük össze, majd a GeneDoc (Nicholas et al., 1997) program segítségével manuálisan finomítottuk az illesztést, ahol szükséges volt. Ezt követően a filogenetikai analízist a PAUP*4.0b (Swofford, 2002) program segítségével végeztük, parsimony-típusú analízist végezve, heurisztikus keresést alkalmazva, ami a felrajzolható törzsfák közül megkeresi a legkevesebb bázisváltozást tükrözőt. A törzsfák készítésében külső csoportként további fajok tef1 (2. táblázat) és ITS (3. táblázat) szekvenciáit is bevontuk. A törzsfa készítéséhez a TreeView programot használtuk. A felrajzolt törzsfát bootstrap analízissel (1000 ismétlés) ellenőriztük. 102
4 A kísérletbe bevont Phoma fajok listája 1. táblázat Fajnév(1) Izolátum kód(2) Saját(3) Eredeti(4) Gazdanövény(5) Phoma eupyrena D/058 CBS Phaseolus vulgaris Phoma destructiva D/033? Lycopersicon esculentum Phoma pinodella D/035 D/035 Glycine max Phoma foveata D/048 PD 76/1021 Chenopodium quinoa Phoma herbarum D/143 MTCC 2319? Phoma exigua var. exigua D/075 D/075 Glycine max Phoma exigua var. exigua D/077 D/077 Glycine max Phoma exigua var. linicola D/071 P D 86/73 Linum usitatissimum Phoma glomerata D/034 D/034 Glycine max Phoma multirostrata D/044 P D 77/508 Ph ylodendron sp. Phoma plurivora D/072 PD 75/907 Medicago sativa Phyllosticta sojicola (=Phoma exigua var. exigua?) D/050 CBS Glycine max Table 1: Isolates of Phoma species species(1), isolate number(2), our collection(3), original(4), host of origin(5) EREDMÉNYEK Első lépésként a Phoma fajok morfológiai azonosítását végeztük el Boerema et al. (2004) monográfiája alapján. A mikroszkópi- és telepjellemzők alapján kapott eredmények azt mutatták, hogy a törzsgyűjteményünkben szereplő fajok valóban megegyeznek az 1. táblázatban szereplő fajokkal. Transzlációs elongációs faktor A genomi DNS izolációját követően a PCR reakcióban a felhasznált primerekkel (EF1-728F és EF1-986R) egy bp nagyságú, a tef1 gén nagy intronját tartalmazó szakasz amplifikálódott mind a 12 izolátum esetében. A PCR reakcióban melléktermék nem képződött, amely a primerek nagyfokú specifikusságát bizonyítja. A PCR reakció után az felszaporodott fragmentek szekvenálási eredményei alapján az elemzést parsimony-típusú analízissel, a PAUP*4.0b program (bootstrap=1000) alkalmazásával végeztük el. A filogenetikai törzsfa készítésében további Ascochyta és Phoma fajok tef1 szekvenciáit is bevontuk (2. táblázat), valamint külső csoportként a Phoma fajoktól két távolabbi Ascomycota faj, a Claviceps sorghi és a Leptosphaerulina trifolii tef1 szekvenciáit használtuk. A tef1 szekvencia elemzésével kapott törzsfa alapján (3. ábra) a vizsgált Phoma fajok egyértelműen elkülönültek a közel rokon Ascochyta nemzetség fajaitól. Mivel a Phoma és Ascochyta fajok elkülönítése morfológiai alapon gyakran nem könnyű feladat az in vivo különböző sejtszámú konídiummal rendelkező Phoma (pseudo-ascochyta) fajok esetében, ez a molekuláris bélyeg további segítséget nyújthat a hovatartozás egyértelmű megállapításához. 2. táblázat A tef1 fragmentek alapján készült filogenetikai törzsfakészítésbe külső csoportként bevont fajok listája, valamint a tef1 szekvenciájuknak hozzáférési száma Fajnév(1) Izolátum Hozzáférési kód(2) szám(3) Leptosphaer ulina trifolii WAC 6693 AY Ascochyta pisi AP2 DQ teleomorf: Didymella lentis anamorf: Ascochyta lentis SAT AL AY (Kaiser et al., 1997) Ascochyta fabae f. sp. viciae (= Ascochyta fabae) AV11 DQ teleomorf: Didymella lentis anamorf: Ascochyta lentis AL1 DQ teleomorf: Didymella fabae anamorf: Ascochyta fabae AF1 DQ (Kaiser és mtsai, 1997) Claviceps sorghi? AY Claviceps sorghi? AY Phoma pinodella CBS AY Phoma pinodella WAC 7978 AY Forrás: Table 2: Species involved in the phylogenetic analyses and the accession number of their tef1 fragments species(1), isolate number(2), accession number(3) A Phoma fajok egy része (P. plurivora, P. herbarum, P. desrtuctiva, P. glomerata) egyértelműen elkülöníthetők a tef1 szekvencia segítségével a többi vizsgált Phoma fajtól. Más részük fajcsoportot alkot a tef1 szekvenciák alapján. Nem különíthetőek el egymástól a Phoma foveta és Phoma multirostrata, valamint a Phoma pinodella és Phoma eupyrena fajok sem. A több izolátummal is képviselt fajok mind egy csoportba kerültek az elemzés során, ami megerősíti a tef1 szekvencia alkalmasságát a Phoma fajok elkülönítésére. 103
5 Az eredetileg Phyllosticta sojicola-ként deponált izolátum (CBS ) a tef1 szekvencia alapján a Phoma exigua var. exigua csoportba került, ami alátámasztja Kövics et al. (1999) feltételezését, miszerint a Phyllosticta sojicola megegyezik a Phoma exigua var. exigua fajjal. Az egyes csoportok nem mutatnak egyezést a morfológia bélyegeken alapuló Phoma taxonokkal (Boerema et al., 2004) (3. ábra). 3. ábra: A tef1 szekvenciák parsimony elemzése alapján készített filogenetikai törzsfa Megjegyzés: A vonalakra írt számok az elágazás valószínűségét jelölik, százalékban, amelyet 1000 ismétlésben elvégzett bootstrap analízis alapján kaptunk. Az 50%-nál kisebb valószínűségű elágazások értékeit nem tüntettük fel. Jobb oldali oszlopok: a morfológiai tenyészbélyegeken alapuló faji besorolás Phoma genuson belüli szekcióit jelzik.(1) Figure 3: Phylogenetic relationships of Phoma strains inferred by the parsimony analysis of ITS sequences. The numbers above the lines represent the bootstrap (bootstrap=1000) values. The columns on the right side represent the Phoma section based on morphological characterization.(1) 104
6 A DNS szekvencia analízise a Phoma-k esetében eddig csak kisebb csoportok elkülönítő vizsgálatára korlátozódott. Az ITS szekvenciákat használták fel a Phoma lingam teleomorf alakjának (Leptosphaeria maculans-leptosphaeria biglobosa fajkomplexének) vizsgálatára (Mendes et al., 2003), illetve a Phoma tracheiphila izolátumok elkülönítésére (Balmas et al., 2005). Vizsgálataink alapján a transzkripciós elongációs faktor 1 (tef1) nagy intron régióját tartalmazó DNS szakasz megfelelően használható a Phoma fajok elkülönítésére a közeli rokon Ascochyta fajoktól. A tef1 szekvencia vizsgálata alapján az összes Phoma egyetlen, a közeli rokon Ascochyta genus megvizsgált fajaitól elkülönülő csoportot alkot. Ez a Phoma genus monofiletikus eredetét bizonyítja. A tef1 szekvencia az analizált 11 Phoma faj 14 izolátumának összehasonlító elemzése alapján alkalmasnak látszik a Phoma fajok egy részének egymástól való elkülönítésére. A tef1 szekvenciák alapján egymástól el nem különíthető fajok esetében (Phoma pinodella és Phoma eupyrena) további variábilis szekvenciák vizsgálata szükséges. ITS fragment A PCR reakciót követően egy 0,6kb nagyságú fragment szaporodott fel mindenegyes mintában, amely tartalmazta az ITS1, ITS2, valamint az 5,8S régiókat. Az ITS fragment filogenetikai analíziséhez további Phoma, Ascochyta, Leptosphaeria és Didymell a fajokat i s bevontunk, amelyeknek az ITS szekvenciáit a NCBI által üzemeltettet GenBank-ból töltöttük le (3. táblázat). Az ITS szekvenciák alapján készült filogenetikai törzsfa a 4. ábrán látható. A különbség Phoma és Ascochyta, Leptosphaeria és Didymella fajok között nem volt jelentős, mindössze csak 23 bázishelyet tekintett informatívnak a program a parsimony elemzés során. Valamint bootstrap elemzést követően csak 3 klaszternek volt 80%-nál nagyobb a valószínűsége. Az ITS szekvencia parsimony elemzése alapján is a Phyllosticta sojicola a Phoma exigua csoportba került csakúgy, mint a tef1 szekvencia alapján. A Phoma foveta és Phoma multirostrata a tef1 szekvenciához hasonlón az ITS szekvencia alapján is egy csoportba került. A továbbiakban az ITS szekvenciát egyéb módszerrel szükséges elemezni, mint például a MEGA vagy maximum likelihood módszer, amely a parsimony analízisnél megbízhatóbb törzsfát eredményezhet. 3. táblázat Az ITS fragmentek alapján készült filogenetikai törzsfakészítésbe bevont fajok listája, valamint az ITS szekvenciájuknak hozzáférési száma Fajnév(1) Izolátum Hozzáférési kód(2) szám(3) Phoma exigua var. heteromorpha? AY Phoma exigua CSL AY Phoma exigua var. populi CBS AF Phoma exigua var. linicola CBS AF Phoma exigua? AY P homa herbarum? DQ Phoma herbarum ATCC AY Phoma pinodella VPRI DQ Phoma pinodella VPRI DQ Phoma pinodella CBS AY Phoma pinodella WAC 7978 AY Phoma glomerata? AF Phoma glomerata? AY Phoma glomerata? AY Phoma eupyrena G r61 AJ Leptosphaerulina trifolii WAC 6693 AY Ascochyta sp. Georgia6 DQ Ascochyta pisi AP1 DQ Ascochyta lentis MU AL1 AY Didymella lentis AL1 DQ Didymella fabae AF1 DQ Forrás: Table 3: Species involved in the phylogenetic analyses and the accession number of their ITS fragments species(1), isolation number(2), accession number(3) ÖSSZEGZÉS A tef1 szekvencia, a többi fonalas gombához (Druzhinina és Kubicek, 2005) hasonlóan alkalmasnak látszik a Phoma fajok filogenetikai elemzésére. Az ITS szekvenciák parsimony módszerrel történő filogenetikai elemzése nem ad megbízható eredményt, amelynek egyik oka az lehet, hogy a szekvencia kevés variábilis bázishelyet tartalmaz. Ez egyben azt is jelentheti, hogy az evolúciós távolság az ITS szekvencia alapján az egyes Phoma fajok között túl kicsi ahhoz, hogy rokonsági kapcsolatokat állapítsunk meg a Phoma nemzetségben. Más nemzetségekhez hasonlóan további szekvenciák bevonása szükséges, mint pl. az actin vagy tubulin a filogenetikai elemzésekbe Mindkét szekvencia elemzés megerősítette azt a korábbi feltételezést (Kövics et al., 1999), hogy a Phyllosticta sojicola azonos a Phoma exigua var. exigua fajjal. 105
7 4. ábra: Az ITS szekvenciák parsimony elemzése alapján készített filogenetikai törzsfa Megjegyzés: A vonalakra írt számok az elágazás valószínűségét jelölik, százalékban, amelyet 1000 ismétlésben elvégzett bootstrap analízis alapján kaptunk. Az 50%-nál kisebb valószínűségű elágazások értékeit nem tüntettük fel. Jobb oldali oszlopok: a morfológiai tenyészbélyegeken alapuló faji besorolás Phoma genuson belüli szekcióit jelzik.(1) Figure 4: Phylogenetic relationships of Phoma strains inferred by the parsimony analysis of ITS sequences The numbers above the lines represent the bootstrap (bootstrap=1000) values. The columns on the right side represent the Phoma section based on morphological characterization.(1) IRODALOM Avise, J.C. (2004): Molecular markers, natural history, and evolution. 2nd ed. Underland, MA: Sinauer Associates Baldauf, S.L.-Doolittle, W.F. (1997): Origin and evolution of slime molds (Mycetozoa). Proc. Natl. Acad. Sci. USA Baldwin, B.G. (1992): Phylogenetic utility of the internal transcribed spacers of nuclear ribosomal DNA in plants: an example from the Composite. Mol. Phylogenet. Evol Balmas, V.-Scherm, B.-Ghignone, S.-Salem, A.O.M.-Cacciola, S.O.-Migheli, Q. (2005): Characterisation of Phoma tracheiphila by RAPD-PCR, microsatellite-primed PCR and ITS rdna sequencing and development of species primers for in planta PCR detection. European Journal of Plant Pathology Boerema, G.H.-Gruyter, J. de-noordeloos, M.E.-Hamers, M.E.C. (2004): Phoma identification manual. CABI Publishing. CAB International Wallingford, Oxfordshire, UK Chen, C.A.-Wallace, C.C.-Wolstenholme, J. (2002): Analysis of mitochondrial 12S RNA gene supports a two-clade hypothesis of the evolutionary history of scleractinian corals. Mol. Phylogenet. Evol Chen, C.A.-Yu, J.K.-Wei, N.W. (2000): Strategies for amplification by polymerase chain reaction of the complete sequence of nuclear large subunit ribosomal RNA-encoding gene in corals. Mar. Biotechnol Druzhinina, I.-Kubicek, C.P. (2005): Species concepts and biodiversity in Trichoderma and Hypocrea: from aggregate species to species cluster? J. Zhejiang Univ. Sci. 6B (2)
8 Gerbi, S.A. (1985): Evolution of ribosomal DNA. pp In: Molecular evolutionary genetics. Macintyre, R.J. (Ed.) Plenum, New York Harrington, T.C.-Rizzo, D.M. (1999): Defining species in the fungi. pp In: Structure and Dynamics of Fungal Populations. Worral, J.J. (Ed.) Kluwer Academic, Dordrecht Hillis, D.M.-Dixon, M.T. (1991): Ribosomal DNA: molecular evolution and phylogenetic inference. Q. Rev. Biol Kaiser, W.J.-Wang, B.C.-Rogers, J.D. (1997): Ascochyta fabae and A. lentis: Host specificity, teleomorphs (Didymella), Hybrid analysis, and taxonomic status. Plant Dis Khon, L.M. (1995): The clonal dynamic in wild and agricultural plant-pathogen populations. Canadian Journal of Botany 73. (Suppl. 1): S Kövics, G.J.-Gruyter, J. de (1995): Comparable esterase isozyme analysis of some Phoma species occur on soybean. (A szóján előforduló néhány Phoma faj észteráz izoenzim mintázatainak összehasonlító vizsgálata.) Proceedings of Debrecen Agricultural University (DATE Tudományos Közleményei) Kövics, G.J.-Gruyter, J. de-aa, H.A. van der (1999): Phoma sojicola comb. nov. and other hyaline-spored coelomycetes pathogenic on soybean. Mycol. Res Lutzoni, F.-Kauff, F.-Cox, C.J.-McLaughlin, D.-Celio, G.- Dentinger, B.-Padamsee, M.-Hibbett, D.-James, T.Y.-Baloch, E.-Grube, M.-Reeb, V.-Hofstetter, V.-Schoch, C.-Arnold, A.E.-Miadlikowska, J.-Spatafora, J.-Johnson, D.-Hambleton, S.-Crockett, M.-Shoemaker, R.-Sung, G.H.-Lucking, R.- Lumbsch, T.-O Donnell, K.-Binder, M.-Diederich, P.-Ertz, D.-Gueidan, C.-Hansen, K.-Harris, R.C.-Hosaka, K.-Lim, Y.W.-Matheny, B.-Nishida, H.-Pfister, D.-Rogers, J.- Rossman, A.-Schmitt, I.-Sipman, H.-Stone, J.-Sugiyama, J.- Yahr, R.-Vilgalys, R. (2004): Assembling the fungal tree of life: progress classi.cation and evolution of subcellular traits. Am. J. Bot Mai, J.C.-Coleman, A.W. (1997): The internal transcribed spacer 2 exhibits a common secondary structure in green algae and flowering plants. J. Mol. Evol Mayr, E. (1940): Systematics and the origin of species from a viewpoint of a zoologist. Columbia University Press, New York Mendes-Pereira, E.-Balesdent, M.H.-Brun, H.-Rouxel, T. (2003): Molecular phylogeny of the Leptosphaeria maculans-l. biglobosa species complex. Mycol. Res Moldave, K. (1985): Eukaryotic protein synthesis. Annu. Rev. Biochem Nicholas, K.B.-Nicholas, H.B. Jr.-Deerfield, D.W. II. (1997): GeneDoc: Analysis and Visualization of Genetic Variation, Embnew. news Oliverio, M.-Cervelli, M.-Mariottini, P. (2002): ITS2 rrna evolution and its congruence with the phylogeny of muricid neogastropods (Caenogastropoda, Muricoidea). Mol. Phylogenet. Evol Reynolds, D.R.-Taylor, J.W. (1993): The fungal holomorph: An overview. pp In: The fungal holomorph: mitotic, meiotic and pleomorphic speciation in fungal systematics. Reynolds, D.R.-Taylor, J.W. (Eds.) CAB International, Wallingford, UK Roger, A.J.-Sandblom, O.-Doolittle, W.F.-Philippe, H. (1999): An evaluation of elongation factor 1α as a phylogenetic marker for eukaryots. Mol. Biol. Evol Schlötterer, C.-Hauser, M.-Haeseler, A.-von Tautz, D. (1994): Comparative evolutionary analysis of rdna ITS regions in Drosophila. Mol. Biol. Evol Swofford, D.L. (2002): PAUP: Phylogenetic Analysis Using Parsimony (and other methods). Version 4b10. Sinauer Associates, Sunderland, MA Talhinhas, P.-Sreenivasaprasad, S.-Neves-Martins, J.-Oliveira, H. (2002): Genetic and morphological characterization of Colletotrichum acutatum causing anthracnose of lupins. Phytopathology, Taylor, J.W.-Jacobson, D.J.-Kroken, S.-Kasuga, T.-Geiser, D.M.- Hibbett, D.S.-Fisher, M.C. (2000): Phylogenetic species recognition and species concepts in fungi. Fungal Genet. Biol Thompson, J.D.-Gibson, T.J.-Plewniak, F.-Jeanmougin, F.- Higgins, D.G. (1997): The ClustalX windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools. Nucleic Acids Research, Weekers, P.H.H.-Jonckheere, F.J. de-dumont, H.J. (2001): Phylogenetic relationships inferred from ribosomal ITS sequences and biogeographic patterns in representative of the genus Calopteryx (Insecta: Odonata) of the West Mediterranean and adjacent west European zone. Mol. Phylogenet. Evol White, T.J.-Bruns, T.-Lee, S.-Taylor, J. (1990): Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics. pp In: PCR protocols. A guide to methods and applications. Innis, M.A.-Gelfand, D.H.-Sninsky, J.J.-White, T.J. (Eds.) Academic Press, New York 107
Genetikai markerek alkalmazása a szelídgesztenye kéregrák (Cryphonectria parasitica) populációinak jellemzésére
 Genetikai markerek alkalmazása a szelídgesztenye kéregrák (Cryphonectria parasitica) populációinak jellemzésére Görcsös Gábor Tarcali Gábor Irinyi László Radócz László Debreceni Egyetem Agrár- és Gazdálkodástudományok
Genetikai markerek alkalmazása a szelídgesztenye kéregrák (Cryphonectria parasitica) populációinak jellemzésére Görcsös Gábor Tarcali Gábor Irinyi László Radócz László Debreceni Egyetem Agrár- és Gazdálkodástudományok
SZÓJÁN EL FORDULÓ PHOMA-SZER GOMBÁK FILOGENETIKAI VIZSGÁLATA
 SZÓJÁN EL FORDULÓ PHOMA-SZER GOMBÁK FILOGENETIKAI VIZSGÁLATA Irinyi László Kövics György Sándor Erzsébet Debreceni Egyetem, Agrár-és M szaki Tudományok Centruma, Növényvédelmi Tanszék, Debrecen Összefoglalás
SZÓJÁN EL FORDULÓ PHOMA-SZER GOMBÁK FILOGENETIKAI VIZSGÁLATA Irinyi László Kövics György Sándor Erzsébet Debreceni Egyetem, Agrár-és M szaki Tudományok Centruma, Növényvédelmi Tanszék, Debrecen Összefoglalás
Bakteriális identifikáció 16S rrns gén szekvencia alapján
 Bakteriális identifikáció 16S rrns gén szekvencia alapján MOHR ANITA SIPOS RITA, SZÁNTÓ-EGÉSZ RÉKA, MICSINAI ADRIENN 2100 Gödöllő, Szent-Györgyi Albert út 4. info@biomi.hu, www.biomi.hu TÖRZS AZONOSÍTÁS
Bakteriális identifikáció 16S rrns gén szekvencia alapján MOHR ANITA SIPOS RITA, SZÁNTÓ-EGÉSZ RÉKA, MICSINAI ADRIENN 2100 Gödöllő, Szent-Györgyi Albert út 4. info@biomi.hu, www.biomi.hu TÖRZS AZONOSÍTÁS
AZ EGRI BORVIDÉK BOTRYTIS CINEREA POPULÁCIÓINAK GENETIKAI JELLEMZÉSE
 AZ EGRI BORVIDÉK BOTRYTIS CINEREA POPULÁCIÓINAK GENETIKAI JELLEMZÉSE Sándor Erzsébet 1 Váczy Kálmán 2 Kövics György János 1 Karaffa Levente 3 1 Debreceni Egyetem, Agrártudományi Centrum, Mez gazdaságtudományi
AZ EGRI BORVIDÉK BOTRYTIS CINEREA POPULÁCIÓINAK GENETIKAI JELLEMZÉSE Sándor Erzsébet 1 Váczy Kálmán 2 Kövics György János 1 Karaffa Levente 3 1 Debreceni Egyetem, Agrártudományi Centrum, Mez gazdaságtudományi
SOLiD Technology. library preparation & Sequencing Chemistry (sequencing by ligation!) Imaging and analysis. Application specific sample preparation
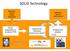 SOLiD Technology Application specific sample preparation Application specific data analysis library preparation & emulsion PCR Sequencing Chemistry (sequencing by ligation!) Imaging and analysis SOLiD
SOLiD Technology Application specific sample preparation Application specific data analysis library preparation & emulsion PCR Sequencing Chemistry (sequencing by ligation!) Imaging and analysis SOLiD
Szakmai zárójelentés OTKA azonosító: NN 75255
 ZÁRÓ SZAKMAI BESZÁMOLÓ A A MORTIERELLALES REND (FUNGI, ZYGOMYCETES) ÁTFOGÓ FILOGENETIKAI ÉS TAXONÓMIAI REVÍZIÓJA MORFOLÓGIAI, FIZIOLÓGIAI ÉS MOLEKULÁRIS ELJÁRÁSOKKAL CÍMŰ NEMZETKÖZI EGYÜTTMŰKÖDÉSI (NN)
ZÁRÓ SZAKMAI BESZÁMOLÓ A A MORTIERELLALES REND (FUNGI, ZYGOMYCETES) ÁTFOGÓ FILOGENETIKAI ÉS TAXONÓMIAI REVÍZIÓJA MORFOLÓGIAI, FIZIOLÓGIAI ÉS MOLEKULÁRIS ELJÁRÁSOKKAL CÍMŰ NEMZETKÖZI EGYÜTTMŰKÖDÉSI (NN)
Manuscript Title: Identification of a thermostable fungal lytic polysaccharide monooxygenase and
 1 2 3 4 5 Journal name: Applied Microbiology and Biotechnology Manuscript Title: Identification of a thermostable fungal lytic polysaccharide monooxygenase and evaluation of its effect on lignocellulosic
1 2 3 4 5 Journal name: Applied Microbiology and Biotechnology Manuscript Title: Identification of a thermostable fungal lytic polysaccharide monooxygenase and evaluation of its effect on lignocellulosic
Markerless Escherichia coli rrn Deletion Strains for Genetic Determination of Ribosomal Binding Sites
 Markerless Escherichia coli rrn Deletion Strains for Genetic Determination of Ribosomal Binding Sites Selwyn Quan *, Ole Skovgaard, Robert E. McLaughlin, Ed T. Buurman,1, and Catherine L. Squires * Department
Markerless Escherichia coli rrn Deletion Strains for Genetic Determination of Ribosomal Binding Sites Selwyn Quan *, Ole Skovgaard, Robert E. McLaughlin, Ed T. Buurman,1, and Catherine L. Squires * Department
Algaközösségek ökológiai, morfológiai és genetikai diverzitásának összehasonlítása szentély jellegű és emberi használatnak kitett élőhelykomplexekben
 Algaközösségek ökológiai, morfológiai és genetikai diverzitásának összehasonlítása szentély jellegű és emberi használatnak kitett élőhelykomplexekben Duleba Mónika Környezettudományi Doktori Iskola I.
Algaközösségek ökológiai, morfológiai és genetikai diverzitásának összehasonlítása szentély jellegű és emberi használatnak kitett élőhelykomplexekben Duleba Mónika Környezettudományi Doktori Iskola I.
Orvosi Genomtudomány 2014 Medical Genomics 2014. Április 8 Május 22 8th April 22nd May
 Orvosi Genomtudomány 2014 Medical Genomics 2014 Április 8 Május 22 8th April 22nd May Hét / 1st week (9. kalendariumi het) Takács László / Fehér Zsigmond Magyar kurzus Datum/ido Ápr. 8 Apr. 9 10:00 10:45
Orvosi Genomtudomány 2014 Medical Genomics 2014 Április 8 Május 22 8th April 22nd May Hét / 1st week (9. kalendariumi het) Takács László / Fehér Zsigmond Magyar kurzus Datum/ido Ápr. 8 Apr. 9 10:00 10:45
A jövedelem alakulásának vizsgálata az észak-alföldi régióban az 1997-99. évi adatok alapján
 A jövedelem alakulásának vizsgálata az észak-alföldi régióban az 1997-99. évi adatok alapján Rózsa Attila Debreceni Egyetem Agrártudományi Centrum, Agrárgazdasági és Vidékfejlesztési Intézet, Számviteli
A jövedelem alakulásának vizsgálata az észak-alföldi régióban az 1997-99. évi adatok alapján Rózsa Attila Debreceni Egyetem Agrártudományi Centrum, Agrárgazdasági és Vidékfejlesztési Intézet, Számviteli
A humán mitokondriális genom: Evolúció, mutációk, polimorfizmusok, populációs vonatkozások. Egyed Balázs ELTE Genetikai Tanszék
 A humán mitokondriális genom: Evolúció, mutációk, polimorfizmusok, populációs vonatkozások Egyed Balázs ELTE Genetikai Tanszék Endoszimbiotikus gén-transzfer (Timmis et al., 2004, Nat Rev Gen) Endoszimbiotikus
A humán mitokondriális genom: Evolúció, mutációk, polimorfizmusok, populációs vonatkozások Egyed Balázs ELTE Genetikai Tanszék Endoszimbiotikus gén-transzfer (Timmis et al., 2004, Nat Rev Gen) Endoszimbiotikus
MIKROSZKÓPIKUS GOMBÁK MIKOTOXIN-BONTÓ KÉPESSÉGÉNEK. Péteri Adrienn Zsanett DOKTORI ÉRTEKEZÉS TÉZISEI
 DOKTORI ÉRTEKEZÉS TÉZISEI MIKROSZKÓPIKUS GOMBÁK MIKOTOXIN-BONTÓ KÉPESSÉGÉNEK VIZSGÁLATA Péteri Adrienn Zsanett Témavezetők: Dr. Varga János, Egyetemi docens Dr. Vágvölgyi Csaba, Tanszékvezető egyetemi
DOKTORI ÉRTEKEZÉS TÉZISEI MIKROSZKÓPIKUS GOMBÁK MIKOTOXIN-BONTÓ KÉPESSÉGÉNEK VIZSGÁLATA Péteri Adrienn Zsanett Témavezetők: Dr. Varga János, Egyetemi docens Dr. Vágvölgyi Csaba, Tanszékvezető egyetemi
Flowering time. Col C24 Cvi C24xCol C24xCvi ColxCvi
 Flowering time Rosette leaf number 50 45 40 35 30 25 20 15 10 5 0 Col C24 Cvi C24xCol C24xCvi ColxCvi Figure S1. Flowering time in three F 1 hybrids and their parental lines as measured by leaf number
Flowering time Rosette leaf number 50 45 40 35 30 25 20 15 10 5 0 Col C24 Cvi C24xCol C24xCvi ColxCvi Figure S1. Flowering time in three F 1 hybrids and their parental lines as measured by leaf number
DI-, TETRA- ÉS HEXAPLOID TRITICUM FAJOK GENOMJAINAK ELEMZÉSE ÉS AZOK ÖSSZEHASONLÍTÁSA FLUORESZCENS IN SITU HIBRIDIZÁCIÓVAL
 Hagyomány és haladás a növénynemesítésben DI-, TETRA- ÉS HEXAPLOID TRITICUM FAJOK GENOMJAINAK ELEMZÉSE ÉS AZOK ÖSSZEHASONLÍTÁSA FLUORESZCENS IN SITU HIBRIDIZÁCIÓVAL VARGA MÓNIKA, MOLNÁR ISTVÁN ÉS KOVÁCS
Hagyomány és haladás a növénynemesítésben DI-, TETRA- ÉS HEXAPLOID TRITICUM FAJOK GENOMJAINAK ELEMZÉSE ÉS AZOK ÖSSZEHASONLÍTÁSA FLUORESZCENS IN SITU HIBRIDIZÁCIÓVAL VARGA MÓNIKA, MOLNÁR ISTVÁN ÉS KOVÁCS
Animal welfare, etológia és tartástechnológia
 Animal welfare, etológia és tartástechnológia Animal welfare, ethology and housing systems Volume 4 Issue 2 Különszám Gödöllı 2008 468 EGYEDAZONOSÍTÁS ÉS SZÁRMAZÁSELLENİRZÉS HIPERPOLIMORF MIKROSZATELLITA
Animal welfare, etológia és tartástechnológia Animal welfare, ethology and housing systems Volume 4 Issue 2 Különszám Gödöllı 2008 468 EGYEDAZONOSÍTÁS ÉS SZÁRMAZÁSELLENİRZÉS HIPERPOLIMORF MIKROSZATELLITA
Architecture of the Trypanosome RNA Editing Accessory Complex, MRB1
 Architecture of the Trypanosome RNA Editing Accessory Complex, MRB1 Michelle L. Ammerman, Kurtis M. Downey, Hassan Hashimi, John C. Fisk, Danielle L. Tomasello, Drahomíra Faktorová, Lucie Hanzálková, Tony
Architecture of the Trypanosome RNA Editing Accessory Complex, MRB1 Michelle L. Ammerman, Kurtis M. Downey, Hassan Hashimi, John C. Fisk, Danielle L. Tomasello, Drahomíra Faktorová, Lucie Hanzálková, Tony
A STRATÉGIAALKOTÁS FOLYAMATA
 BUDAPESTI CORVINUS EGYETEM VÁLLALATGAZDASÁGTAN INTÉZET VERSENYKÉPESSÉG KUTATÓ KÖZPONT Szabó Zsolt Roland: A STRATÉGIAALKOTÁS FOLYAMATA VERSENYBEN A VILÁGGAL 2004 2006 GAZDASÁGI VERSENYKÉPESSÉGÜNK VÁLLALATI
BUDAPESTI CORVINUS EGYETEM VÁLLALATGAZDASÁGTAN INTÉZET VERSENYKÉPESSÉG KUTATÓ KÖZPONT Szabó Zsolt Roland: A STRATÉGIAALKOTÁS FOLYAMATA VERSENYBEN A VILÁGGAL 2004 2006 GAZDASÁGI VERSENYKÉPESSÉGÜNK VÁLLALATI
Supporting Information
 Supporting Information Cell-free GFP simulations Cell-free simulations of degfp production were consistent with experimental measurements (Fig. S1). Dual emmission GFP was produced under a P70a promoter
Supporting Information Cell-free GFP simulations Cell-free simulations of degfp production were consistent with experimental measurements (Fig. S1). Dual emmission GFP was produced under a P70a promoter
Egy 10,3 kb méretű, lineáris, a mitokondriumban lokalizált DNS-plazmidot izoláltunk a
 Egy 10,3 kb méretű, lineáris, a mitokondriumban lokalizált DNS-plazmidot izoláltunk a Fusarium proliferatum (Gibberella intermedia) ITEM 2337-es törzséből, és a plazmidot pfp1- nek neveztük el. Proteináz
Egy 10,3 kb méretű, lineáris, a mitokondriumban lokalizált DNS-plazmidot izoláltunk a Fusarium proliferatum (Gibberella intermedia) ITEM 2337-es törzséből, és a plazmidot pfp1- nek neveztük el. Proteináz
COOPERATION IN THE CEREAL SECTOR OF THE SOUTH PLAINS REGIONS STRÉN, BERTALAN. Keywords: cooperation, competitiveness, cereal sector, region, market.
 COOPERATION IN THE CEREAL SECTOR OF THE SOUTH PLAINS REGIONS STRÉN, BERTALAN Keywords: cooperation, competitiveness, cereal sector, region, market. Using a questionnaire, we determined the nature and strength
COOPERATION IN THE CEREAL SECTOR OF THE SOUTH PLAINS REGIONS STRÉN, BERTALAN Keywords: cooperation, competitiveness, cereal sector, region, market. Using a questionnaire, we determined the nature and strength
Gyakorlati bioinformatika
 Gyakorlati bioinformatika Szekvenciaillesztés PhD kurzus 2. Szekvenciaillesztés Bagossi Péter Fajtái: - egyszer ill. többszörös illesztés - globális ill. lokális illesztés Alkalmazása: - adatbázisokban
Gyakorlati bioinformatika Szekvenciaillesztés PhD kurzus 2. Szekvenciaillesztés Bagossi Péter Fajtái: - egyszer ill. többszörös illesztés - globális ill. lokális illesztés Alkalmazása: - adatbázisokban
Molekuláris evolúció második gyakorlat
 Molekuláris evolúció második gyakorlat Szekvenciák illesztése (alignment készítés) Szekvenciák szerkesztése Programok: ClustalX (http://evolution.genetics.washington.edu/phylip/software.html) GeneDoc (http://www.psc.edu/biomed/genedoc/)
Molekuláris evolúció második gyakorlat Szekvenciák illesztése (alignment készítés) Szekvenciák szerkesztése Programok: ClustalX (http://evolution.genetics.washington.edu/phylip/software.html) GeneDoc (http://www.psc.edu/biomed/genedoc/)
Szent István Egyetem. Állatorvos-tudományi Doktori Iskola
 Szent István Egyetem Állatorvos-tudományi Doktori Iskola Madarak (Aves) és tolltetveik (Phthiraptera) gazda-parazita kapcsolatának evolúciós, ökológiai és faunisztikai vizsgálata PhD értekezés tézisei
Szent István Egyetem Állatorvos-tudományi Doktori Iskola Madarak (Aves) és tolltetveik (Phthiraptera) gazda-parazita kapcsolatának evolúciós, ökológiai és faunisztikai vizsgálata PhD értekezés tézisei
A magyar racka juh tejének beltartalmi változása a laktáció alatt
 A magyar racka juh tejének beltartalmi változása a laktáció alatt Nagy László Komlósi István Debreceni Egyetem Agrártudományi Centrum, Mezőgazdaságtudományi Kar, Állattenyésztés- és Takarmányozástani Tanszék,
A magyar racka juh tejének beltartalmi változása a laktáció alatt Nagy László Komlósi István Debreceni Egyetem Agrártudományi Centrum, Mezőgazdaságtudományi Kar, Állattenyésztés- és Takarmányozástani Tanszék,
Animal welfare, etológia és tartástechnológia
 Animal welfare, etológia és tartástechnológia Animal welfare, ethology and housing systems Volume 5 Issue 4 Különszám Gödöllı 2009 459 MORFOLÓGIAI ÉS GENETIKAI VIZSGÁLATOK MAGYARORSZÁGI TÖRPEHARCSÁKON
Animal welfare, etológia és tartástechnológia Animal welfare, ethology and housing systems Volume 5 Issue 4 Különszám Gödöllı 2009 459 MORFOLÓGIAI ÉS GENETIKAI VIZSGÁLATOK MAGYARORSZÁGI TÖRPEHARCSÁKON
SZAKMAI ZÁRÓJELENTÉS OTKA T Erdei fákon kórokozó Phytophthora fajok molekuláris azonosítása
 SZAKMAI ZÁRÓJELENTÉS OTKA T 049077 Erdei fákon kórokozó Phytophthora fajok molekuláris azonosítása Előzmények: Előző OTKA projektünk során (T-037352) a Phytophthora fajok erdei fákon való magyarországi
SZAKMAI ZÁRÓJELENTÉS OTKA T 049077 Erdei fákon kórokozó Phytophthora fajok molekuláris azonosítása Előzmények: Előző OTKA projektünk során (T-037352) a Phytophthora fajok erdei fákon való magyarországi
Kövics György publikációs listája 2009 Kövics György Növénykórtani vademecum: angol-magyar magyar-angol szakszókincs etimológiai és fogalmi
 örgy publikációs listája 2009 örgy Növénykórtani vademecum: angol-magyar magyar-angol szakszókincs etimológiai és fogalmi magyarázatokkal Debrecen: 2009. 470 p. 1 (ISBN:978 963 88096 0 5) Könyv/Kézikönyv/Tudományos
örgy publikációs listája 2009 örgy Növénykórtani vademecum: angol-magyar magyar-angol szakszókincs etimológiai és fogalmi magyarázatokkal Debrecen: 2009. 470 p. 1 (ISBN:978 963 88096 0 5) Könyv/Kézikönyv/Tudományos
University of Bristol - Explore Bristol Research
 Al-Salihi, S. A. A., Scott, T. A., Bailey, A. M., & Foster, G. D. (2017). Improved vectors for Agrobacterium mediated genetic manipulation of Hypholoma spp. and other homobasidiomycetes. Journal of Microbiological
Al-Salihi, S. A. A., Scott, T. A., Bailey, A. M., & Foster, G. D. (2017). Improved vectors for Agrobacterium mediated genetic manipulation of Hypholoma spp. and other homobasidiomycetes. Journal of Microbiological
Expression analysis of PIN genes in root tips and nodules of Lotus japonicus
 Article Expression analysis of PIN genes in root tips and nodules of Lotus japonicus Izabela Sańko-Sawczenko 1, Dominika Dmitruk 1, Barbara Łotocka 1, Elżbieta Różańska 1 and Weronika Czarnocka 1, * 1
Article Expression analysis of PIN genes in root tips and nodules of Lotus japonicus Izabela Sańko-Sawczenko 1, Dominika Dmitruk 1, Barbara Łotocka 1, Elżbieta Różańska 1 and Weronika Czarnocka 1, * 1
Construction of a cube given with its centre and a sideline
 Transformation of a plane of projection Construction of a cube given with its centre and a sideline Exercise. Given the center O and a sideline e of a cube, where e is a vertical line. Construct the projections
Transformation of a plane of projection Construction of a cube given with its centre and a sideline Exercise. Given the center O and a sideline e of a cube, where e is a vertical line. Construct the projections
Molekuláris biológiai eljárások alkalmazása a GMO analitikában és az élelmiszerbiztonság területén
 Molekuláris biológiai eljárások alkalmazása a GMO analitikában és az élelmiszerbiztonság területén Dr. Dallmann Klára A molekuláris biológia célja az élőlények és sejtek működésének molekuláris szintű
Molekuláris biológiai eljárások alkalmazása a GMO analitikában és az élelmiszerbiztonság területén Dr. Dallmann Klára A molekuláris biológia célja az élőlények és sejtek működésének molekuláris szintű
A Fusarium solani-val szembeni ellenállóképesség vizsgálata különböző zöldborsó fajtákon és nemesítési kombinációkon
 A Fusarium solani-val szembeni ellenállóképesség vizsgálata különböző zöldborsó fajtákon és nemesítési kombinációkon Mendlerné Drienyovszki Nóra 1 Mándi Lajosné 2 Debreceni Egyetem Agrártudományi Centrum,
A Fusarium solani-val szembeni ellenállóképesség vizsgálata különböző zöldborsó fajtákon és nemesítési kombinációkon Mendlerné Drienyovszki Nóra 1 Mándi Lajosné 2 Debreceni Egyetem Agrártudományi Centrum,
Evolúcióbiológia. Biológus B.Sc tavaszi félév
 Evolúcióbiológia Biológus B.Sc. 2011. tavaszi félév A biológiában minden csak az evolúció fényében válik érthetővé Theodosius Dobzhansky : Nothing in biology makes sense except in the light of evolution.
Evolúcióbiológia Biológus B.Sc. 2011. tavaszi félév A biológiában minden csak az evolúció fényében válik érthetővé Theodosius Dobzhansky : Nothing in biology makes sense except in the light of evolution.
Éter típusú üzemanyag-adalékok mikrobiális bontása: a Methylibium sp. T29 jelű, új MTBE-bontó törzs izolálása és jellemzése
 Éter típusú üzemanyag-adalékok mikrobiális bontása: a Methylibium sp. T29 jelű, új MTBE-bontó törzs izolálása és jellemzése Doktori értekezés tézisei Szabó Zsolt Témavezető: Dr. Bihari Zoltán vezető kutató
Éter típusú üzemanyag-adalékok mikrobiális bontása: a Methylibium sp. T29 jelű, új MTBE-bontó törzs izolálása és jellemzése Doktori értekezés tézisei Szabó Zsolt Témavezető: Dr. Bihari Zoltán vezető kutató
Bioinformatika és genomanalízis az orvostudományban. Biológiai adatbázisok. Cserző Miklós 2018
 Bioinformatika és genomanalízis az orvostudományban Biológiai adatbázisok Cserző Miklós 2018 A mai előadás Mi az adatbázis A biológia kapcsolata az adatbázisokkal Az adatbázisok típusai Adatbázis formátumok,
Bioinformatika és genomanalízis az orvostudományban Biológiai adatbázisok Cserző Miklós 2018 A mai előadás Mi az adatbázis A biológia kapcsolata az adatbázisokkal Az adatbázisok típusai Adatbázis formátumok,
Bioinformatics: Blending. Biology and Computer Science
 Bioinformatics: Blending Biology and Computer Science MDNMSITNTPTSNDACLSIVHSLMCHRQ GGESETFAKRAIESLVKKLKEKKDELDSL ITAITTNGAHPSKCVTIQRTLDGRLQVAG RKGFPHVIYARLWRWPDLHKNELKHVK YCQYAFDLKCDSVCVNPYHYERVVSPGI DLSGLTLQSNAPSSMMVKDEYVHDFEG
Bioinformatics: Blending Biology and Computer Science MDNMSITNTPTSNDACLSIVHSLMCHRQ GGESETFAKRAIESLVKKLKEKKDELDSL ITAITTNGAHPSKCVTIQRTLDGRLQVAG RKGFPHVIYARLWRWPDLHKNELKHVK YCQYAFDLKCDSVCVNPYHYERVVSPGI DLSGLTLQSNAPSSMMVKDEYVHDFEG
2011. január április 10. IPK Gatersleben (Németország) május 17. Kruppa Klaudia
 2011. január 10. 2011. április 10. IPK Gatersleben (Németország) Gatersleben (G-life) Country State District Town Administration Germany Saxony-Anhalt Salzlandkreis Seeland Basic statistics Area 16.00
2011. január 10. 2011. április 10. IPK Gatersleben (Németország) Gatersleben (G-life) Country State District Town Administration Germany Saxony-Anhalt Salzlandkreis Seeland Basic statistics Area 16.00
Hibridspecifikus tápanyag-és vízhasznosítás kukoricánál csernozjom talajon
 Hibridspecifikus tápanyag-és vízhasznosítás kukoricánál csernozjom talajon Karancsi Lajos Gábor Debreceni Egyetem Agrár és Gazdálkodástudományok Centruma Mezőgazdaság-, Élelmiszertudományi és Környezetgazdálkodási
Hibridspecifikus tápanyag-és vízhasznosítás kukoricánál csernozjom talajon Karancsi Lajos Gábor Debreceni Egyetem Agrár és Gazdálkodástudományok Centruma Mezőgazdaság-, Élelmiszertudományi és Környezetgazdálkodási
AZ ÁRPA SZÁRAZSÁGTŰRÉSÉNEK VIZSGÁLATA: QTL- ÉS ASSZOCIÁCIÓS ANALÍZIS, MARKER ALAPÚ SZELEKCIÓ, TILLING
 Hagyomány és haladás a növénynemesítésben AZ ÁRPA SZÁRAZSÁGTŰRÉSÉNEK VIZSGÁLATA: QTL- ÉS ASSZOCIÁCIÓS ANALÍZIS, MARKER ALAPÚ SZELEKCIÓ, BÁLINT ANDRÁS FERENC 1, SZIRA FRUZSINA 1, ANDREAS BÖRNER 2, KERSTIN
Hagyomány és haladás a növénynemesítésben AZ ÁRPA SZÁRAZSÁGTŰRÉSÉNEK VIZSGÁLATA: QTL- ÉS ASSZOCIÁCIÓS ANALÍZIS, MARKER ALAPÚ SZELEKCIÓ, BÁLINT ANDRÁS FERENC 1, SZIRA FRUZSINA 1, ANDREAS BÖRNER 2, KERSTIN
A Badacsonyi Borrégió élesztő mikrobiótájának azonosítása: terroir élesztők izolálása, karakterizálása és borászati hasznosítása
 Gerőcs Annamária 1 Farkas Tibor 2 Nemes-Barnás Katalin 3 Májer János 4 Szőke Barna 5 Olasz Ferenc 6 A Badacsonyi Borrégió élesztő mikrobiótájának azonosítása: terroir élesztők izolálása, karakterizálása
Gerőcs Annamária 1 Farkas Tibor 2 Nemes-Barnás Katalin 3 Májer János 4 Szőke Barna 5 Olasz Ferenc 6 A Badacsonyi Borrégió élesztő mikrobiótájának azonosítása: terroir élesztők izolálása, karakterizálása
BALATONI NÁDASOK KLONÁLIS DIVERZITÁSÁNAK VIZSGÁLATA RAPD-PCR MÓDSZERREL
 Bot. Közlem. 94(1 2): 133 140, 2007. BALATONI NÁDASOK KLONÁLIS DIVERZITÁSÁNAK VIZSGÁLATA RAPD-PCR MÓDSZERREL LUKÁCS VIKTÓRIA 1, HERODEK SÁNDOR 1, MAJOR ÁGNES 2 1 MTA Balatoni Limnológiai Kutatóintézet,
Bot. Közlem. 94(1 2): 133 140, 2007. BALATONI NÁDASOK KLONÁLIS DIVERZITÁSÁNAK VIZSGÁLATA RAPD-PCR MÓDSZERREL LUKÁCS VIKTÓRIA 1, HERODEK SÁNDOR 1, MAJOR ÁGNES 2 1 MTA Balatoni Limnológiai Kutatóintézet,
Supplementary Table 1. Cystometric parameters in sham-operated wild type and Trpv4 -/- rats during saline infusion and
 WT sham Trpv4 -/- sham Saline 10µM GSK1016709A P value Saline 10µM GSK1016709A P value Number 10 10 8 8 Intercontractile interval (sec) 143 (102 155) 98.4 (71.4 148) 0.01 96 (92 121) 109 (95 123) 0.3 Voided
WT sham Trpv4 -/- sham Saline 10µM GSK1016709A P value Saline 10µM GSK1016709A P value Number 10 10 8 8 Intercontractile interval (sec) 143 (102 155) 98.4 (71.4 148) 0.01 96 (92 121) 109 (95 123) 0.3 Voided
Magyar Emlőstani Évkönyv. Szerzői tájékoztató (2016. február 27.)
 Magyar Emlőstani Évkönyv Szerzői tájékoztató (2016. február 27.) A Magyar Emlőstani Évkönyv (Hungarian Yearbook of Mammalogy) a Milvus Csoport Madártani és Természetvédelmi Egyesület és a Bükki Emlőstani
Magyar Emlőstani Évkönyv Szerzői tájékoztató (2016. február 27.) A Magyar Emlőstani Évkönyv (Hungarian Yearbook of Mammalogy) a Milvus Csoport Madártani és Természetvédelmi Egyesület és a Bükki Emlőstani
FATERMÉSI FOK MEGHATÁROZÁSA AZ EGÉSZÁLLOMÁNY ÁTLAGNÖVEDÉKE ALAPJÁN
 4. évfolyam 2. szám 2 0 1 4 101 107. oldal FATERMÉSI FOK MEGHATÁROZÁSA AZ EGÉSZÁLLOMÁNY ÁTLAGNÖVEDÉKE ALAPJÁN Veperdi Gábor Nyugat-magyarországi Egyetem, Erdômérnöki Kar Kivonat A fatermési fok meghatározása
4. évfolyam 2. szám 2 0 1 4 101 107. oldal FATERMÉSI FOK MEGHATÁROZÁSA AZ EGÉSZÁLLOMÁNY ÁTLAGNÖVEDÉKE ALAPJÁN Veperdi Gábor Nyugat-magyarországi Egyetem, Erdômérnöki Kar Kivonat A fatermési fok meghatározása
Honlap szerkesztés Google Tudós alkalmazásával
 Dr. Mester Gyula Honlap szerkesztés Google Tudós alkalmazásával Összefoglaló: A közlemény tematikája honlap szerkesztés Google Tudós alkalmazásával. A bevezetés után a tudományos teljesítmény mérésének
Dr. Mester Gyula Honlap szerkesztés Google Tudós alkalmazásával Összefoglaló: A közlemény tematikája honlap szerkesztés Google Tudós alkalmazásával. A bevezetés után a tudományos teljesítmény mérésének
A cell-based screening system for RNA Polymerase I inhibitors
 Electronic Supplementary Material (ESI) for MedChemComm. This journal is The Royal Society of Chemistry 2019 Supporting Information A cell-based screening system for RNA Polymerase I inhibitors Xiao Tan,
Electronic Supplementary Material (ESI) for MedChemComm. This journal is The Royal Society of Chemistry 2019 Supporting Information A cell-based screening system for RNA Polymerase I inhibitors Xiao Tan,
Szent István Egyetem Állatorvos-tudományi Doktori Iskola. Sertés circovírusok járványtani vizsgálata
 Szent István Egyetem Állatorvos-tudományi Doktori Iskola Sertés circovírusok járványtani vizsgálata PhD dolgozat tézisei Készítette: Dr. Cságola Attila Témavezet : Dr. Tuboly Tamás 2009 Szent István Egyetem
Szent István Egyetem Állatorvos-tudományi Doktori Iskola Sertés circovírusok járványtani vizsgálata PhD dolgozat tézisei Készítette: Dr. Cságola Attila Témavezet : Dr. Tuboly Tamás 2009 Szent István Egyetem
Fiatal kutatói beszámoló
 Talajbiológiai- és biokémiai osztály Fiatal kutatói beszámoló Gazdag Orsolya tudományos segédmunkatárs Témavezető: Dr. Ködöböcz László 2013.12.03. Témacím: Különböző gazdálkodási módok hatása a talaj baktérium-közösségeinek
Talajbiológiai- és biokémiai osztály Fiatal kutatói beszámoló Gazdag Orsolya tudományos segédmunkatárs Témavezető: Dr. Ködöböcz László 2013.12.03. Témacím: Különböző gazdálkodási módok hatása a talaj baktérium-közösségeinek
A Multi Locus Sequence Typing (MLST) alkalmazhatósága az élelmiszermikrobiológiában
 A Multi Locus Sequence Typing (MLST) alkalmazhatósága az élelmiszermikrobiológiában Sipos Rita, Lukács Alena, Simon Janka, Szántó-Egész Réka, Micsinai Adrienn 2100 Gödöllő, Szent-Györgyi Albert út 4. info@biomi.hu,
A Multi Locus Sequence Typing (MLST) alkalmazhatósága az élelmiszermikrobiológiában Sipos Rita, Lukács Alena, Simon Janka, Szántó-Egész Réka, Micsinai Adrienn 2100 Gödöllő, Szent-Györgyi Albert út 4. info@biomi.hu,
CLUSTALW Multiple Sequence Alignment
 Version 3.2 CLUSTALW Multiple Sequence Alignment Selected Sequences) FETA_GORGO FETA_HORSE FETA_HUMAN FETA_MOUSE FETA_PANTR FETA_RAT Import Alignments) Return Help Report Bugs Fasta label *) Workbench
Version 3.2 CLUSTALW Multiple Sequence Alignment Selected Sequences) FETA_GORGO FETA_HORSE FETA_HUMAN FETA_MOUSE FETA_PANTR FETA_RAT Import Alignments) Return Help Report Bugs Fasta label *) Workbench
The beet R locus encodes a new cytochrome P450 required for red. betalain production.
 The beet R locus encodes a new cytochrome P450 required for red betalain production. Gregory J. Hatlestad, Rasika M. Sunnadeniya, Neda A. Akhavan, Antonio Gonzalez, Irwin L. Goldman, J. Mitchell McGrath,
The beet R locus encodes a new cytochrome P450 required for red betalain production. Gregory J. Hatlestad, Rasika M. Sunnadeniya, Neda A. Akhavan, Antonio Gonzalez, Irwin L. Goldman, J. Mitchell McGrath,
Supporting Information
 Supporting Information Paired design of dcas9 as a systematic platform for the detection of featured nucleic acid sequences in pathogenic strains Yihao Zhang 1,2,8, Long Qian 4,8, Weijia Wei 1,3,7,8, Yu
Supporting Information Paired design of dcas9 as a systematic platform for the detection of featured nucleic acid sequences in pathogenic strains Yihao Zhang 1,2,8, Long Qian 4,8, Weijia Wei 1,3,7,8, Yu
A rosszindulatú daganatos halálozás változása 1975 és 2001 között Magyarországon
 A rosszindulatú daganatos halálozás változása és között Eredeti közlemény Gaudi István 1,2, Kásler Miklós 2 1 MTA Számítástechnikai és Automatizálási Kutató Intézete, Budapest 2 Országos Onkológiai Intézet,
A rosszindulatú daganatos halálozás változása és között Eredeti közlemény Gaudi István 1,2, Kásler Miklós 2 1 MTA Számítástechnikai és Automatizálási Kutató Intézete, Budapest 2 Országos Onkológiai Intézet,
Conserved ortholog set (COS) markerek térképezése Aegilops kromoszómákon
 Conserved ortholog set (COS) markerek térképezése Aegilops kromoszómákon Rövid tanulmányút 2011. 01.03-03. 30., John Inn Centre, Dept. of Crop Genetics, Norwich Research Park, Norwich NR4 7UH, UK Supervisor:
Conserved ortholog set (COS) markerek térképezése Aegilops kromoszómákon Rövid tanulmányút 2011. 01.03-03. 30., John Inn Centre, Dept. of Crop Genetics, Norwich Research Park, Norwich NR4 7UH, UK Supervisor:
A KUKORICA STRESSZREZISZTENCIA KUTATÁSOK EREDMÉNYEIBŐL
 A martonvásári agrárkutatások hatodik évtizede A KUKORICA STRESSZREZISZTENCIA KUTATÁSOK EREDMÉNYEIBŐL MARTON L. CSABA, SZŐKE CSABA ÉS PINTÉR JÁNOS Kukoricanemesítési Osztály Bevezetés Hazai éghajlati viszonyaink
A martonvásári agrárkutatások hatodik évtizede A KUKORICA STRESSZREZISZTENCIA KUTATÁSOK EREDMÉNYEIBŐL MARTON L. CSABA, SZŐKE CSABA ÉS PINTÉR JÁNOS Kukoricanemesítési Osztály Bevezetés Hazai éghajlati viszonyaink
A sertések lágyék- és heresérv tünetegyüttesének genetikai háttere
 Nagy Szabolcs 1 Benedek Zsuzsanna 2 Polgár J. Péter 3 Magnus Andersson 4 A sertések lágyék- és heresérv tünetegyüttesének genetikai háttere Genetic background of scrotal and inguinal hernia in swine nagy.szabolcs@georgikon.hu
Nagy Szabolcs 1 Benedek Zsuzsanna 2 Polgár J. Péter 3 Magnus Andersson 4 A sertések lágyék- és heresérv tünetegyüttesének genetikai háttere Genetic background of scrotal and inguinal hernia in swine nagy.szabolcs@georgikon.hu
Két új stressz-patogén Botryosphaeria-faj előfordulása Magyarországon
 Két új stressz-patogén Botryosphaeria-faj előfordulása Magyarországon Láday Miklós és Vajna László MTA Növényvédelmi Kutatóintézete, 1525 Budapest, Pf. 102. Botryosphaeria fajokról általában Botryosphaeria-fajok
Két új stressz-patogén Botryosphaeria-faj előfordulása Magyarországon Láday Miklós és Vajna László MTA Növényvédelmi Kutatóintézete, 1525 Budapest, Pf. 102. Botryosphaeria fajokról általában Botryosphaeria-fajok
III. ATK Tudományos Nap
 Magyar Tudományos Akadémia Agrártudományi Kutatóközpont III. ATK Tudományos Nap Összefoglalók Szerkesztette JANDA TIBOR 2014. november 13. Martonvásár Kiadja a Magyar Tudományos Akadémia Agrártudományi
Magyar Tudományos Akadémia Agrártudományi Kutatóközpont III. ATK Tudományos Nap Összefoglalók Szerkesztette JANDA TIBOR 2014. november 13. Martonvásár Kiadja a Magyar Tudományos Akadémia Agrártudományi
A zsírszövet mellett az agyvelő lipidekben leggazdagabb szervünk. Pontosabban az agy igen gazdag hosszú szénláncú politelítetlen zsírsavakban
 BEVEZETÉS ÉS A KUTATÁS CÉLJA A zsírszövet mellett az agyvelő lipidekben leggazdagabb szervünk. Pontosabban az agy igen gazdag hosszú szénláncú politelítetlen zsírsavakban (LCPUFA), mint az arachidonsav
BEVEZETÉS ÉS A KUTATÁS CÉLJA A zsírszövet mellett az agyvelő lipidekben leggazdagabb szervünk. Pontosabban az agy igen gazdag hosszú szénláncú politelítetlen zsírsavakban (LCPUFA), mint az arachidonsav
Modular Optimization of Hemicellulose-utilizing Pathway in. Corynebacterium glutamicum for Consolidated Bioprocessing of
 [Supplementary materials] Modular Optimization of Hemicellulose-utilizing Pathway in Corynebacterium glutamicum for Consolidated Bioprocessing of Hemicellulosic Biomass Sung Sun Yim 1, Jae Woong Choi 1,
[Supplementary materials] Modular Optimization of Hemicellulose-utilizing Pathway in Corynebacterium glutamicum for Consolidated Bioprocessing of Hemicellulosic Biomass Sung Sun Yim 1, Jae Woong Choi 1,
Nagy Emese: Polimorfizmus és rokonsági körök vizsgálata kukoricában (Zea mays) Témavezetők: Cs. L. Marton G Gyulai
 Nagy Emese: Polimorfizmus és rokonsági körök vizsgálata kukoricában (Zea mays) Témavezetők: Cs. L. Marton G Gyulai BEVEZETÉS A molekuláris biológiai és genetikai módszerek gyors fejlődése egyre inkább
Nagy Emese: Polimorfizmus és rokonsági körök vizsgálata kukoricában (Zea mays) Témavezetők: Cs. L. Marton G Gyulai BEVEZETÉS A molekuláris biológiai és genetikai módszerek gyors fejlődése egyre inkább
Összefoglalás. Summary. Bevezetés
 A talaj kálium ellátottságának vizsgálata módosított Baker-Amacher és,1 M CaCl egyensúlyi kivonószerek alkalmazásával Berényi Sándor Szabó Emese Kremper Rita Loch Jakab Debreceni Egyetem Agrár és Műszaki
A talaj kálium ellátottságának vizsgálata módosított Baker-Amacher és,1 M CaCl egyensúlyi kivonószerek alkalmazásával Berényi Sándor Szabó Emese Kremper Rita Loch Jakab Debreceni Egyetem Agrár és Műszaki
Supplementary materials to: Whole-mount single molecule FISH method for zebrafish embryo
 Supplementary materials to: Whole-mount single molecule FISH method for zebrafish embryo Yuma Oka and Thomas N. Sato Supplementary Figure S1. Whole-mount smfish with and without the methanol pretreatment.
Supplementary materials to: Whole-mount single molecule FISH method for zebrafish embryo Yuma Oka and Thomas N. Sato Supplementary Figure S1. Whole-mount smfish with and without the methanol pretreatment.
A doktori értekezés tézisei. A növényi NRP fehérjék lehetséges szerepe a hiszton defoszforiláció szabályozásában, és a hőstressz válaszban.
 A doktori értekezés tézisei A növényi NRP fehérjék lehetséges szerepe a hiszton defoszforiláció szabályozásában, és a hőstressz válaszban. Bíró Judit Témavezető: Dr. Fehér Attila Magyar Tudományos Akadémia
A doktori értekezés tézisei A növényi NRP fehérjék lehetséges szerepe a hiszton defoszforiláció szabályozásában, és a hőstressz válaszban. Bíró Judit Témavezető: Dr. Fehér Attila Magyar Tudományos Akadémia
Mapping Sequencing Reads to a Reference Genome
 Mapping Sequencing Reads to a Reference Genome High Throughput Sequencing RN Example applications: Sequencing a genome (DN) Sequencing a transcriptome and gene expression studies (RN) ChIP (chromatin immunoprecipitation)
Mapping Sequencing Reads to a Reference Genome High Throughput Sequencing RN Example applications: Sequencing a genome (DN) Sequencing a transcriptome and gene expression studies (RN) ChIP (chromatin immunoprecipitation)
Molekuláris biológiai módszerek alkalmazása a biológiai környezeti kármentesítésben
 Molekuláris biológiai módszerek alkalmazása a biológiai környezeti kármentesítésben Dr. Kovács Tamás, Kovács Árpád László Kármentesítés Aktuális Kérdései 2013 Budapest, 2013.03.21-22 Bioremediáció során
Molekuláris biológiai módszerek alkalmazása a biológiai környezeti kármentesítésben Dr. Kovács Tamás, Kovács Árpád László Kármentesítés Aktuális Kérdései 2013 Budapest, 2013.03.21-22 Bioremediáció során
Correlation & Linear Regression in SPSS
 Petra Petrovics Correlation & Linear Regression in SPSS 4 th seminar Types of dependence association between two nominal data mixed between a nominal and a ratio data correlation among ratio data Correlation
Petra Petrovics Correlation & Linear Regression in SPSS 4 th seminar Types of dependence association between two nominal data mixed between a nominal and a ratio data correlation among ratio data Correlation
BÁLINT András Beszámoló az AGRISAFE által támogatott tanulmányútról 2008 november 2009 február. 1. Az IPK bemutatása 2.
 BÁLINT András Beszámoló az AGRISAFE által támogatott tanulmányútról 2008 november 2009 február 1. Az IPK bemutatása 2. A TILLING módszer Hol található az IPK? Gatersleben Általános adatok az IPK-ról Leibniz
BÁLINT András Beszámoló az AGRISAFE által támogatott tanulmányútról 2008 november 2009 február 1. Az IPK bemutatása 2. A TILLING módszer Hol található az IPK? Gatersleben Általános adatok az IPK-ról Leibniz
CELTIS FAJOK KOMPLEX ETIOLÓGIÁJÚ BETEGSÉGÉNEK DIAGNÓZISA (esettanulmány)
 NÖVÉNYVÉDELEM 46 (10), 2010 465 CELTIS FAJOK KOMPLEX ETIOLÓGIÁJÚ BETEGSÉGÉNEK DIAGNÓZISA (esettanulmány) Vajna László MTA Növényvédelmi Kutatóintézete, 1525 Budapest Pf. 102. Díszfák, erdei fafajok és
NÖVÉNYVÉDELEM 46 (10), 2010 465 CELTIS FAJOK KOMPLEX ETIOLÓGIÁJÚ BETEGSÉGÉNEK DIAGNÓZISA (esettanulmány) Vajna László MTA Növényvédelmi Kutatóintézete, 1525 Budapest Pf. 102. Díszfák, erdei fafajok és
Oszvald Mária. A búza tartalékfehérjék tulajdonságainak in vitro és in vivo vizsgálata rizs modell rendszerben
 Budapesti Műszaki és Gazdaságtudományi Egyetem Alkalmazott Biotechnológia és Élelmiszertudományi Tanszék PHD ÉRTEKEZÉS TÉZISEI Készítette: Oszvald Mária A búza tartalékfehérjék tulajdonságainak in vitro
Budapesti Műszaki és Gazdaságtudományi Egyetem Alkalmazott Biotechnológia és Élelmiszertudományi Tanszék PHD ÉRTEKEZÉS TÉZISEI Készítette: Oszvald Mária A búza tartalékfehérjék tulajdonságainak in vitro
VI. Magyar Földrajzi Konferencia 524-529
 Van Leeuwen Boudewijn Tobak Zalán Szatmári József 1 BELVÍZ OSZTÁLYOZÁS HAGYOMÁNYOS MÓDSZERREL ÉS MESTERSÉGES NEURÁLIS HÁLÓVAL BEVEZETÉS Magyarország, különösen pedig az Alföld váltakozva szenved aszályos
Van Leeuwen Boudewijn Tobak Zalán Szatmári József 1 BELVÍZ OSZTÁLYOZÁS HAGYOMÁNYOS MÓDSZERREL ÉS MESTERSÉGES NEURÁLIS HÁLÓVAL BEVEZETÉS Magyarország, különösen pedig az Alföld váltakozva szenved aszályos
avagy az ipari alkalmazhatóság kérdése biotechnológiai tárgyú szabadalmi bejelentéseknél Dr. Győrffy Béla, Egis Nyrt., Budapest
 Iparilag alkalmazható szekvenciák, avagy az ipari alkalmazhatóság kérdése biotechnológiai tárgyú szabadalmi bejelentéseknél Dr. Győrffy Béla, Egis Nyrt., Budapest Neutrokin α - jelentős kereskedelmi érdekek
Iparilag alkalmazható szekvenciák, avagy az ipari alkalmazhatóság kérdése biotechnológiai tárgyú szabadalmi bejelentéseknél Dr. Győrffy Béla, Egis Nyrt., Budapest Neutrokin α - jelentős kereskedelmi érdekek
Gabonacsíra- és amarant fehérjék funkcionális jellemzése modell és komplex rendszerekben
 Budapesti Műszaki és Gazdaságtudományi Egyetem Biokémiai és Élelmiszertechnológiai Tanszék Gabonacsíra- és amarant fehérjék funkcionális jellemzése modell és komplex rendszerekben c. PhD értekezés Készítette:
Budapesti Műszaki és Gazdaságtudományi Egyetem Biokémiai és Élelmiszertechnológiai Tanszék Gabonacsíra- és amarant fehérjék funkcionális jellemzése modell és komplex rendszerekben c. PhD értekezés Készítette:
13. RNS szintézis és splicing
 13. RNS szintézis és splicing 1 Visszatekintés: Az RNS típusai és szerkezete Hírvivő RNS = mrns (messenger RNA = mrna) : fehérjeszintézis pre-mrns érett mrns (intronok kivágódnak = splicing) Transzfer
13. RNS szintézis és splicing 1 Visszatekintés: Az RNS típusai és szerkezete Hírvivő RNS = mrns (messenger RNA = mrna) : fehérjeszintézis pre-mrns érett mrns (intronok kivágódnak = splicing) Transzfer
A biomassza alapú falufűtőművek létesítésének társadalomföldrajzi kérdései a Hernád-völgy településein
 Tóth Tamás 1 Tóth József Barnabás 2 A biomassza alapú falufűtőművek létesítésének társadalomföldrajzi kérdései a Hernád-völgy településein Summary Village heating plants operating in the EU have directly
Tóth Tamás 1 Tóth József Barnabás 2 A biomassza alapú falufűtőművek létesítésének társadalomföldrajzi kérdései a Hernád-völgy településein Summary Village heating plants operating in the EU have directly
ACTA AGRONOMICA ÓVÁRIENSIS
 ACTA AGRONOMICA ÓVÁRIENSIS VOLUME 50. NUMBER 2. Mosonmagyaróvár 2008 2 UNIVERSITY OF WEST HUNGARY Faculty of Agricultural and Food Sciences Mosonmagyaróvár Hungary NYUGAT-MAGYARORSZÁGI EGYETEM Mezôgazdaság-
ACTA AGRONOMICA ÓVÁRIENSIS VOLUME 50. NUMBER 2. Mosonmagyaróvár 2008 2 UNIVERSITY OF WEST HUNGARY Faculty of Agricultural and Food Sciences Mosonmagyaróvár Hungary NYUGAT-MAGYARORSZÁGI EGYETEM Mezôgazdaság-
Genetikai panel kialakítása a hazai tejhasznú szarvasmarha állományok hasznos élettartamának növelésére
 Genetikai panel kialakítása a hazai tejhasznú szarvasmarha állományok hasznos élettartamának növelésére Dr. Czeglédi Levente Dr. Béri Béla Kutatás-fejlesztés támogatása a megújuló energiaforrások és agrár
Genetikai panel kialakítása a hazai tejhasznú szarvasmarha állományok hasznos élettartamának növelésére Dr. Czeglédi Levente Dr. Béri Béla Kutatás-fejlesztés támogatása a megújuló energiaforrások és agrár
Molekuláris biológiai módszerek alkalmazása a maláriát okozó paraziták elterjedésének és prevalenciájának vizsgálatában
 TÉMA ÉRTÉKELÉS TÁMOP-4.2.1/B-09/1/KMR-2010-0003 (minden téma külön lapra) 2010. június 1. 2012. május 31. 1. Az elemi téma megnevezése Molekuláris biológiai módszerek alkalmazása a maláriát okozó paraziták
TÉMA ÉRTÉKELÉS TÁMOP-4.2.1/B-09/1/KMR-2010-0003 (minden téma külön lapra) 2010. június 1. 2012. május 31. 1. Az elemi téma megnevezése Molekuláris biológiai módszerek alkalmazása a maláriát okozó paraziták
ADATBÁNYÁSZAT I. ÉS OMICS
 Az élettudományi-klinikai felsőoktatás gyakorlatorientált és hallgatóbarát korszerűsítése a vidéki képzőhelyek nemzetközi versenyképességének erősítésére TÁMOP-4.1.1.C-13/1/KONV-2014-0001 ADATBÁNYÁSZAT
Az élettudományi-klinikai felsőoktatás gyakorlatorientált és hallgatóbarát korszerűsítése a vidéki képzőhelyek nemzetközi versenyképességének erősítésére TÁMOP-4.1.1.C-13/1/KONV-2014-0001 ADATBÁNYÁSZAT
10. CSI. A molekuláris biológiai technikák alkalmazásai
 10. CSI. A molekuláris biológiai technikák alkalmazásai A DNS mint azonosító 3 milliárd bázispár az emberi DNS-ben (99.9%-ban azonos) 0.1%-nyi különbség elegendő az egyedek megkülönböztetéséhez Genetikai
10. CSI. A molekuláris biológiai technikák alkalmazásai A DNS mint azonosító 3 milliárd bázispár az emberi DNS-ben (99.9%-ban azonos) 0.1%-nyi különbség elegendő az egyedek megkülönböztetéséhez Genetikai
A HUMÁN GENOM PROJEKT Sasvári-Székely Mária* Semmelweis Egyetem, Orvosi Vegytani, Molekuláris Biológiai és Pathobiokémiai Intézet
 A HUMÁN GENOM PROJEKT Sasvári-Székely Mária* Semmelweis Egyetem, Orvosi Vegytani, Molekuláris Biológiai és Pathobiokémiai Intézet *Levelezési cím: Dr. Sasvári-Székely Mária, Semmelweis Egyetem, Orvosi
A HUMÁN GENOM PROJEKT Sasvári-Székely Mária* Semmelweis Egyetem, Orvosi Vegytani, Molekuláris Biológiai és Pathobiokémiai Intézet *Levelezési cím: Dr. Sasvári-Székely Mária, Semmelweis Egyetem, Orvosi
Bogácsi-Szabó Erika. Ph.D. értekezés tézisei. Témavezető: Prof. Dr. Raskó István. Szegedi Tudományegyetem, Molekuláris és sejtbiológiai Ph.D.
 Populációgenetikai és diagnosztikai célú mitokondriális DNS és autoszómális marker vizsgálatok Magyarországon feltárt régészeti csontleletekből és modern mintákból Bogácsi-Szabó Erika Ph.D. értekezés tézisei
Populációgenetikai és diagnosztikai célú mitokondriális DNS és autoszómális marker vizsgálatok Magyarországon feltárt régészeti csontleletekből és modern mintákból Bogácsi-Szabó Erika Ph.D. értekezés tézisei
On The Number Of Slim Semimodular Lattices
 On The Number Of Slim Semimodular Lattices Gábor Czédli, Tamás Dékány, László Ozsvárt, Nóra Szakács, Balázs Udvari Bolyai Institute, University of Szeged Conference on Universal Algebra and Lattice Theory
On The Number Of Slim Semimodular Lattices Gábor Czédli, Tamás Dékány, László Ozsvárt, Nóra Szakács, Balázs Udvari Bolyai Institute, University of Szeged Conference on Universal Algebra and Lattice Theory
USER MANUAL Guest user
 USER MANUAL Guest user 1 Welcome in Kutatótér (Researchroom) Top menu 1. Click on it and the left side menu will pop up 2. With the slider you can make left side menu visible 3. Font side: enlarging font
USER MANUAL Guest user 1 Welcome in Kutatótér (Researchroom) Top menu 1. Click on it and the left side menu will pop up 2. With the slider you can make left side menu visible 3. Font side: enlarging font
2. Local communities involved in landscape architecture in Óbuda
 Év Tájépítésze pályázat - Wallner Krisztina 2. Közösségi tervezés Óbudán Óbuda jelmondata: Közösséget építünk, ennek megfelelően a formálódó helyi közösségeket bevonva fejlesztik a közterületeket. Békásmegyer-Ófaluban
Év Tájépítésze pályázat - Wallner Krisztina 2. Közösségi tervezés Óbudán Óbuda jelmondata: Közösséget építünk, ennek megfelelően a formálódó helyi közösségeket bevonva fejlesztik a közterületeket. Békásmegyer-Ófaluban
kpis(ppk20) kpis(ppk46)
 Table S1. C. elegans strains used in this study Strains carrying transgenes Strain Transgene Genotype Reference IT213 tcer-1 prom:tcer-1orf:gfp:tcer-1 3'utr unc-119(ed3) III; kpis(ppk6) This study IT283
Table S1. C. elegans strains used in this study Strains carrying transgenes Strain Transgene Genotype Reference IT213 tcer-1 prom:tcer-1orf:gfp:tcer-1 3'utr unc-119(ed3) III; kpis(ppk6) This study IT283
T-helper Type 2 driven Inflammation Defines Major Subphenotypes of Asthma
 T-helper Type 2 driven Inflammation Defines Major Subphenotypes of Asthma Prescott G. Woodruff, M.D., M.P.H., Barmak Modrek, Ph.D., David F. Choy, B.S., Guiquan Jia, M.D., Alexander R. Abbas, Ph.D., Almut
T-helper Type 2 driven Inflammation Defines Major Subphenotypes of Asthma Prescott G. Woodruff, M.D., M.P.H., Barmak Modrek, Ph.D., David F. Choy, B.S., Guiquan Jia, M.D., Alexander R. Abbas, Ph.D., Almut
Erdészettudományi Közlemények
 Erdészettudományi Közlemények 2. évfolyam 1. szám 2012 73 80 oldal AZ EZÜSTHÁRS FATERMÉSI TÁBLÁJÁNAK MÓDOSÍTÁSA Peszlen Roland József és Veperdi Gábor Nyugat-magyarországi Egyetem, Erdőmérnöki Kar, Erdővagyon-gazdálkodási
Erdészettudományi Közlemények 2. évfolyam 1. szám 2012 73 80 oldal AZ EZÜSTHÁRS FATERMÉSI TÁBLÁJÁNAK MÓDOSÍTÁSA Peszlen Roland József és Veperdi Gábor Nyugat-magyarországi Egyetem, Erdőmérnöki Kar, Erdővagyon-gazdálkodási
Magyar vakcsiga (Bythiospeum hungaricum (Soós, 1927)) egy szisztematikai probléma vizsgálata genetikai módszerekkel
 Földtan, őslénytan, flóra, fauna, természetvédelem www.mecsek.gportal.hu 2013.07.01. Szerkesztő: Fazekas Imre E-mail: fazekas.hu@gmail.com Magyar vakcsiga (Bythiospeum hungaricum (Soós, 1927)) egy szisztematikai
Földtan, őslénytan, flóra, fauna, természetvédelem www.mecsek.gportal.hu 2013.07.01. Szerkesztő: Fazekas Imre E-mail: fazekas.hu@gmail.com Magyar vakcsiga (Bythiospeum hungaricum (Soós, 1927)) egy szisztematikai
A genetikai lelet értelmezése monogénes betegségekben
 A genetikai lelet értelmezése monogénes betegségekben Tory Kálmán Semmelweis Egyetem, I. sz. Gyermekklinika A ~20 ezer fehérje-kódoló gén a 23 pár kromoszómán A kromoszómán található bázisok száma: 250M
A genetikai lelet értelmezése monogénes betegségekben Tory Kálmán Semmelweis Egyetem, I. sz. Gyermekklinika A ~20 ezer fehérje-kódoló gén a 23 pár kromoszómán A kromoszómán található bázisok száma: 250M
ACTA ACADEMIAE PAEDAGOGICAE AGRIENSIS
 Separatum ACTA ACADEMIAE PAEDAGOGICAE AGRIESIS OVA SERIES TOM. XXII. SECTIO MATEMATICAE TÓMÁCS TIBOR Egy rekurzív sorozat tagjainak átlagáról EGER, 994 Egy rekurzív sorozat tagjainak átlagáról TÓMÁCS TIBOR
Separatum ACTA ACADEMIAE PAEDAGOGICAE AGRIESIS OVA SERIES TOM. XXII. SECTIO MATEMATICAE TÓMÁCS TIBOR Egy rekurzív sorozat tagjainak átlagáról EGER, 994 Egy rekurzív sorozat tagjainak átlagáról TÓMÁCS TIBOR
DOKTORI (Ph.D.) ÉRTEKEZÉS TÉZISEI VESZPRÉMI EGYETEM GEORGIKON MEZGAZDASÁGTUDOMÁNYI KAR KESZTHELY
 DOKTORI (Ph.D.) ÉRTEKEZÉS TÉZISEI VESZPRÉMI EGYETEM GEORGIKON MEZGAZDASÁGTUDOMÁNYI KAR KESZTHELY NÖVÉNYTERMESZTÉSI ÉS KERTÉSZETI TUDOMÁNYOK DOKTORI ISKOLA ALTERNARIA FAJOK ÖSSZEHASONLÍTÓ ELEMZÉSE KÉSZÍTETTE:
DOKTORI (Ph.D.) ÉRTEKEZÉS TÉZISEI VESZPRÉMI EGYETEM GEORGIKON MEZGAZDASÁGTUDOMÁNYI KAR KESZTHELY NÖVÉNYTERMESZTÉSI ÉS KERTÉSZETI TUDOMÁNYOK DOKTORI ISKOLA ALTERNARIA FAJOK ÖSSZEHASONLÍTÓ ELEMZÉSE KÉSZÍTETTE:
A BÜKKI KARSZTVÍZSZINT ÉSZLELŐ RENDSZER KERETÉBEN GYŰJTÖTT HIDROMETEOROLÓGIAI ADATOK ELEMZÉSE
 KARSZTFEJLŐDÉS XIX. Szombathely, 2014. pp. 137-146. A BÜKKI KARSZTVÍZSZINT ÉSZLELŐ RENDSZER KERETÉBEN GYŰJTÖTT HIDROMETEOROLÓGIAI ADATOK ELEMZÉSE ANALYSIS OF HYDROMETEOROLIGYCAL DATA OF BÜKK WATER LEVEL
KARSZTFEJLŐDÉS XIX. Szombathely, 2014. pp. 137-146. A BÜKKI KARSZTVÍZSZINT ÉSZLELŐ RENDSZER KERETÉBEN GYŰJTÖTT HIDROMETEOROLÓGIAI ADATOK ELEMZÉSE ANALYSIS OF HYDROMETEOROLIGYCAL DATA OF BÜKK WATER LEVEL
Leica SmartRTK, az aktív ionoszféra kezelésének záloga (I. rész)
 Leica SmartRTK, az aktív ionoszféra kezelésének záloga (I. rész) Aki egy kicsit is nyomon követi a GNSS technológia aktualitásait, az egyre gyakrabban találkozhat különböző cikkekkel, értekezésekkel, melyek
Leica SmartRTK, az aktív ionoszféra kezelésének záloga (I. rész) Aki egy kicsit is nyomon követi a GNSS technológia aktualitásait, az egyre gyakrabban találkozhat különböző cikkekkel, értekezésekkel, melyek
AZ ERDÕ NÖVEKEDÉSÉNEK VIZSGÁLATA TÉRINFORMATIKAI ÉS FOTOGRAMMETRIAI MÓDSZEREKKEL KARSZTOS MINTATERÜLETEN
 Tájökológiai Lapok 5 (2): 287 293. (2007) 287 AZ ERDÕ NÖVEKEDÉSÉNEK VIZSGÁLATA TÉRINFORMATIKAI ÉS FOTOGRAMMETRIAI MÓDSZEREKKEL KARSZTOS MINTATERÜLETEN ZBORAY Zoltán Honvédelmi Minisztérium Térképészeti
Tájökológiai Lapok 5 (2): 287 293. (2007) 287 AZ ERDÕ NÖVEKEDÉSÉNEK VIZSGÁLATA TÉRINFORMATIKAI ÉS FOTOGRAMMETRIAI MÓDSZEREKKEL KARSZTOS MINTATERÜLETEN ZBORAY Zoltán Honvédelmi Minisztérium Térképészeti
A DNS szerkezete. Genom kromoszóma gén DNS genotípus - allél. Pontos méretek Watson genomja. J. D. Watson F. H. C. Crick. 2 nm C G.
 1955: 46 emberi kromoszóma van 1961: mrns 1975: DNS szekvenálás 1982: gén-bank adatbázisok 1983: R (polymerase chain reaction) Mérföldkövek 1 J. D. Watson F. H.. rick 2008 1953 2003 Watson genomja DNS
1955: 46 emberi kromoszóma van 1961: mrns 1975: DNS szekvenálás 1982: gén-bank adatbázisok 1983: R (polymerase chain reaction) Mérföldkövek 1 J. D. Watson F. H.. rick 2008 1953 2003 Watson genomja DNS
Az örökítőanyag. Az élőlények örökítőanyaga minden esetben nukleinsav (DNS,RNS) (1)Griffith, (2)Avery, MacLeod and McCarty (3)Hershey and Chase
 SZTE, Orv. Biol. Int., Mol- és Sejtbiol. Gyak., VIII. Az örökítőanyag Az élőlények örökítőanyaga minden esetben nukleinsav (DNS,RNS) (1)Griffith, (2)Avery, MacLeod and McCarty (3)Hershey and Chase Ez az
SZTE, Orv. Biol. Int., Mol- és Sejtbiol. Gyak., VIII. Az örökítőanyag Az élőlények örökítőanyaga minden esetben nukleinsav (DNS,RNS) (1)Griffith, (2)Avery, MacLeod and McCarty (3)Hershey and Chase Ez az
APÁKGYERMEKGONDOZÁSI SZABADSÁGON-AVAGY EGY NEM HAGYOMÁNYOS ÉLETHELYZET MEGÍTÉLÉSE A FÉRFIAKSZEMSZÖGÉBŐL
 APÁKGYERMEKGODOZÁSI SZABADSÁGO-AVAGY EGY EM HAGYOMÁYOS ÉLETHELYZET MEGÍTÉLÉSE A FÉRFIAKSZEMSZÖGÉBŐL PROF: HABIL BECSIK ADREA 1 - DR. JUHÁSZ TÍMEA 2 Összefoglalás: em mondhatjuk, hogy ma még sok férfi megy
APÁKGYERMEKGODOZÁSI SZABADSÁGO-AVAGY EGY EM HAGYOMÁYOS ÉLETHELYZET MEGÍTÉLÉSE A FÉRFIAKSZEMSZÖGÉBŐL PROF: HABIL BECSIK ADREA 1 - DR. JUHÁSZ TÍMEA 2 Összefoglalás: em mondhatjuk, hogy ma még sok férfi megy
FAMILY STRUCTURES THROUGH THE LIFE CYCLE
 FAMILY STRUCTURES THROUGH THE LIFE CYCLE István Harcsa Judit Monostori A magyar társadalom 2012-ben: trendek és perspektívák EU összehasonlításban Budapest, 2012 november 22-23 Introduction Factors which
FAMILY STRUCTURES THROUGH THE LIFE CYCLE István Harcsa Judit Monostori A magyar társadalom 2012-ben: trendek és perspektívák EU összehasonlításban Budapest, 2012 november 22-23 Introduction Factors which
