pleics-06 Amp pleics-13 Amp/Zeo N-His 10/ 3 x
|
|
|
- Gréta Tamás
- 6 évvel ezelőtt
- Látták:
Átírás
1 Protease Sequence primers Vector Name AR Tag/Flag cleavage Tag and linker pleics-01 Amp N-His 6 TEV Extra SM His6-SSGVDLGT-TEV site-1 T7 Promoter pleics-01-seq-r pleics-02 Amp N-GST TEV Extra SM GST-SS-TEV Site-1 pgex5 pgex3 pleics-03 Kan N-His 6 TEV Extra SM His6-SSGVDLGT-TEV site-1 T7 Promoter T7 terminator pleics-04 Kan N-GST TEV Extra SM GST-SS-TEV Site-1 pgex5 T7 terminator pleics-05 Amp C-His 6 TEV Extra ENLYFQ TEV site-1-le-his6 T7 Promoter T7 terminator N-His 6 for first gene TEV Extra SM His6-SSGVDLGT-TEV site-1 pleics-06-f1-seq pleics-06-r1-seq pleics-06 Amp C- His 6/S for Extra ENLYFQ TEV site-1-le-his6-lesg-s-stsaa pleics-06-f2-seq T7 terminator second gene pleics-07 Kan N-His 6/S TEV Extra GA His6-SSGLVPRGSGM-S-PDLGTGS-TEV site-2 T7 Promoter T7 terminator pleics-08 Kan N-GST/ N- TEV Extra GA GST-DGSTSGSG-His6-SAGLVPRGSTAIGM-S- pgex5 T7 terminator His 6/S PDLGTGS-TEV site-2 pleics-09 Amp N-Trx N- His 6/S TEV Extra GA Trx-GSGSGHM-His6-SSGLVPRGSGM-S- PDLGTGS-TEV site-2 pleics-09-seq-f T7 terminator pleics-10 Amp N-MBP TEV Extra GA MBP-SNNNNNNNNNNLG-TEV site-2 pleics-10-seq-f pleics-10-seq-r pleics-11 Amp N-His 6 TEV Extra GA His6-DYDIPTT-TEV site-2 pleics-11-seq-f pleics-11-seq-r pleics-12 Amp/Neo N-His 10/ 3 x TEV Extra GA His10-DYKDDDDKDYKDDDDKDYKDDDDK- pleics-12-seq-f pleics-12-seq-r Flag GS-TEV site-2 pleics-13 Amp/Zeo N-His 10/ 3 x Flag TEV Extra GA His10-DYKDDDDKDYKDDDDKDYKDDDDK- GS-TEV site-2 pleics-12-seq-f pleics-12-seq-r pleics-14 Amp N-GST TEV Extra GA GST-GS-TEV site-2 pgex5 pgex3 pleics-15 Kan/Neo N-TAP TEV Extra GA TAP-CPGGS-TEV site-2 pleics-15-seq-f T7 Promoter pleics-16 Amp N-GST TEV Extra GA GST-DLVPRGS-TEV site-2 pgex5 pleics-11-seq-r pleics-17 Kan N-GST/His 6 TEV Extra GA GST-GSTSGGGGSNNNPPTPTPSSGSG-His6- pleics-17-seq-f T7 terminator SAALEVLFQGPGYQDP-TEV site-2 pleics-18 Kan N-MBP TEV Extra GA MBP-SNNNNNNNNNNPMS-TEV site-2 pleics-10-seq-f T7 terminator pleics-19 Amp N-HA TEV Extra GA HA(MYPYDVPDYA)-LMAMEAR-TEV site-2 pleics-19-seq-f pleics-19-seq-r pleics-20 Amp N-myc TEV Extra GA Myc(ASMQALISEEDL)- LMAMEAR-TEV site-2 pleics-19-seq-f pleics-19-seq-r
2 Vector Name AR Tag/Flag Protease cleavage pleics-21 Kan/Neo N-EGFP TEV Extra GA pleics-22 Kan/Neo N-EGYP TEV Extra GA pleics-23 Kan/Neo N-mCherry TEV Extra GA pleics-24 Kan/Neo N-ECFP TEV Extra GA pleics-25 Kan/Neo N-mEGFP (A206K) TEV Extra GA pleics-26 Kan/Neo N-mEYFP (A206K) TEV Extra GA pleics-27 Kan/Neo N-mECFP (A206K) TEV Extra GA pleics-29 Kan/Neo C-EGFP pleics-30 Kan/Neo C-mCherry TEV, Extra ENLYFQ TEV, Extra ENLYFQ Tag and linker EGFP-SGLRSR-TEV site-2 EGFP-SGLRS EYFP-SGLRSR-TEV site-2 EYFP-SGLRS mcherry-sglrsr-tev site-2 mcherry-sglrs ECFP-SGLRSR-TEV site-2 ECFP-SGLRS megfp-sglrsr-tev site-2 megfp-sglrs meyfp-sglrsr-tev site-2 meyfp-sglrs mecfp-sglrsr-tev site-2 mecfp-sglrs L-TEV site-2- EFMQSTVPRARDPPVAT -EGFP EFMQSTVPRARDPPVAT -EGFP L-TEV site-2- EFMQSTVPRARDPPVAT -mcherry EFMQSTVPRARDPPVAT -mcherry Sequence primers pleics-21-seq-f pleics-21-seq-r pleics-21-seq-f pleics-21-seq-r pleics-23-seq-f pleics-21-seq-r pleics-21-seq-f pleics-21-seq-r pleics-21-seq-f pleics-21-seq-r pleics-21-seq-f pleics-21-seq-r pleics-21-seq-f pleics-21-seq-r pleics-29-seq-f pleics-29-seq-r pleics-29-seq-f pleics-30-seq-r
3 Secretion Protease Sequence primers Vector Name AR Tag/Flag Signal cleavage Tag and linker pleics-31 Amp C-His 6 No TEV Extra ENLYFQ TEV site-1-le-his6 5 AOX1-F 3 AOX1-R pleics-32 Amp C-His 6 PHO1 TEV Extra ENLYFQ TEV site-1-le-his6 5 AOX1-F 3 AOX1-R pleics-33 Amp C-His 6 No TEV Extra ENLYFQ TEV site-1-le-his6 5 AOX1-F 3 AOX1-R pleics-34 Amp C-His 6 α-factor TEV Extra ENLYFQ TEV site-1-le-his6 5 AOX1-F 3 AOX1-R Protease Sequence primers Vector Name AR Tag/Flag cleavage Tag and linker pleics-35 Kan GAL4-BD T7 Promoter pleics-35-seq-r pleics-36 Amp GAL4-AD T7 Promoter pleics-36-seq-r pleics-37 Amp RFP pleics-37-seq-f pleics-72-seq-r pleics-38 Amp pleics-37-seq-f pleics-72-seq-r pleics-39 Amp N-His 10-3 x Flag TEV Extra GA at N- N terminal: His10- T7 Promoter pleics-39-seq-r C-Halo terminal DYKDDDDKDYKDDDDKDYKDDDDK-GS-TEV site-2 pleics-40 Amp N-His 10-3 x Flag C-hBirA TEV Extra GA at N- terminal C terminal: EFELRLE-Halo N terminal: His10- DYKDDDDKDYKDDDDKDYKDDDDK-GS-TEV site-2 C terminal: LE-hBirA T7 Promoter pleics-40-seq-r pleics-41 Amp N-His 6 TEV Extra SM His6-SSGVDLGT-TEV site-1 pleics-41-seq-f pleics-41-seq-r pleics-42 Amp C-His 6 TEV Extra ENLYFQ TEV site-1-le-his6 pleics-41-seq-f pleics-41-seq-r pleics-43 Amp N-His 6/Mistic TEV Extra SM His6-PG-Mistic-PWSSGVDLGT-TEV site-1 pleics-43-seq-f pleics-01-seq-r pleics-44 Kan N-His 6/Mistic TEV Extra SM His6-PG-Mistic-PWSSGVDLGT-TEV site-1 pleics-43-seq-f T7 terminator pleics-45 Amp N-His 6/mEGFP (A206K) TEV Extra GA His6-PG- megfp-sglrsr-tev site-2 His6-mEGFP-SGLRS pleics-45-seq-f pleics-45-seq-r pleics-46 Kan N-GB1/His6 TEV Extra GA GB1-His6-GS-TEV site-2 T7 Promoter T7 terminator pleics-12-seq-f pleics-11-seq-r pleics-47 Amp N-His 10/ 3 x Flag TEV Extra GA His10-DYKDDDDKDYKDDDDKDYKDDDDK- GS-TEV site-2 pleics-48 Amp pleics-48-seq-f pleics-11-seq-r
4 pleics-49 Amp C-His 4/3 x Flag TEV, Extra ENLYFQ TEV-2-His4- T7 Promoter pleics-12-seq-r DYKDDDDKDYKDDDDKDYKDDDDK-GSEN pleics-50 Amp C-GST TEV, Extra ENLYFQ TEV site-1-le-gst T7 Promoter pleics-50-seq-r Protease Sequence primers Vector Name AR Tag/Flag cleavage Tag and linker pleics-51 Kan N-FlAsH TEV Extra SM His6-PG-CCPGCC-PWSSGVDLGT-TEV site-1 T7 Promoter T7 terminator pleics-52 Kan N-FlAsH/ megfp1-128 TEV Extra SM His6-PG-CCPGCC-PW-mEGFP pleics-52-seq-f T7 terminator PWSSGVDLGT-TEV site-1 pleics-53 Kan N-FlAsH/ megfp129- TEV Extra SM His6-PG-CCPGCC-PW-mEGFP pleics-53-seq-f T7 terminator 238 PWSSGVDLGT-TEV site-1 pleics-54 Kan N-mEGFP1-128 TEV Extra SM His6-PG-mEGFP1-128-PWSSGVDLGT-TEVsite-1 pleics-52-seq-f T7 terminator pleics-55 Kan N-mEGFP TEV Extra SM His6-PG-mEGFP PWSSGVDLGT-TEVsite-1 pleics-53-seq-f T7 terminator pleics-56 Amp Super-folded EGFP (1- TEV Extra SM 10) pleics-57 Kan N-mEGFP11 TEV Extra SM His6-PG-mEGFP11-PWSSGVDLGT-TEV site-1 T7 Promoter T7 terminator pleics-58 Amp megfp1-128 TEV Extra SM pleics-59 Amp megfp TEV Extra SM pleics-60 Kan N-FlAsH /Super-folded EGFP (1-10) TEV Extra SM His6-PG-CCPGCC-PW-super-folded EGFP1-10- PWSSGVDLGT-TEV site-1 pleics-60-seq-f T7 terminator pleics-61 Amp N-FlAsH TEV Extra SM His6-PG-CCPGCC-PWSSGVDLGT-TEV site-1 T7 Promoter pleics-01-seq-r pleics-63 Amp C- Super-folded megfp TEV Extra TEV site-1-le-super-folded megfp-le-his6 T7 Promoter pleics-63-seq-r /His6 ENLYFQ pleics-64 Amp C- megfp /His6 TEV Extra TEV site-1-le-megfp-le-his6 T7 Promoter pleics-63-seq-r ENLYFQ pleics-65 Amp C- Super-folded TEV Extra TEV site-1-le-super-folded megfp(1-10)-le-his6 T7 Promoter pleics-63-seq-r megfp1-10 /His6 ENLYFQ pleics-66 Amp C- Super-folded megfp11 /His6 TEV Extra ENLYFQ TEV site-1-le-super-folded megfp(11)-le-his6 T7 Promoter T7 terminator pleics-67 Amp His6-PG- mcherry-sglrsr-tev site-2 N-His 6/mCherry TEV Extra GA pleics-67-seq-f pleics-45-seq-r His6-mEGFP-SGLRS pleics-68 Amp His6-PG- meyfp-sglrsr-tev site-2 TEV Extra GA pleics-68-seq-f pleics-45-seq-r
5 N-His 6/mEYFP (A206K) His6-mEGFP-SGLRS pleics-69 Amp N-GST PreScission Protease Extra GP GST-SD-PreScission Site-LGS pgex5 pgex3 pleics-72 Amp EGFP pleics-72-seq-f pleics-72-seq-r pleics-73 Amp EGFP pleics-73-seq-f pleics-73-seq-r pleics-74 Amp 5xGAL4/mCD8 pleics-74-seq-f pleics-74-seq-r pleics-75 Amp 5xGAL4/mCD8 pleics-74-seq-f pleics-74-seq-r pleics-76 Amp 5xGAL4/myr pleics-76-seq-f pleics-74-seq-r pleics-77 Amp pleics-72-seq-f pleics-12-seq-r pleics-78 Amp pleics-72-seq-f pleics-12-seq-r pleics-79 Amp 5xGAL4 pleics-74-seq-f pleics-79-seq-r pleics-80 Amp copgfp/ha pleics-80-seq-f pleics-80-seq-r pleics-81 Amp LexA pleics-81-seq-f pleics-81-seq-r pleics-82 Amp VP16 pleics-82-seq-f pleics-82-seq-r pleics-83 Amp VP16 pleics-83-seq-f pleics-83-seq-r pleics-84 Amp GAL4 pleics-83-seq-f pleics-83-seq-r pleics-85 Amp Luciferase pleics-85-seq-f pleics-85-seq-r pleics-86 Amp Luciferase pleics-85-seq-f pleics-85-seq-r pleics-87 Amp Luciferase pleics-85-seq-f pleics-85-seq-r pleics-88 Amp EGFP and mcherry pleics-73-seq-f pleics-72-seq-r pleics-89 Amp N-His 6/S/TEV-Halo TEV Extra GA His6-SSGLVPRGSGM-S-PDLGTGS-TEVsite-2- Halo-HG pleics-89-seq-f pleics-91-seq-r pleics-90 Amp C-Halo-TEV- His 6 TEV, Extra ENLYFQ GS-Halo-TEV site-1-le-his6 T7 Promoter pleics-90-seq-r pleics-91 Amp N-GB1/His6 TEV Extra GA GB1-His6-GS-TEV site-2 T7 Promoter pleics-91-seq-r pleics-92 Amp N-GST/His 6 TEV Extra GA GST-GSTSGGGGSNNNPPTPTPSSGSG-His6- pleics-17-seq-f pleics-91-seq-r SAALEVLFQGPGYQDP-TEV site-2 pleics-93 Amp N-His 6/S TEV Extra GA His6-SSGLVPRGSGM-S-PDLGTGS-TEV site-2 T7 Promoter pleics-91-seq-r pleics-94 Amp C-GFP pleics-94-seq-f pleics-94-seq-r
6 CRISPR Effector Cleavage PAM grna Sequence primers Vector Name AR Selection Sequence pleics-95 Amp EGFP Cas9 Nick (D10A) 3 -NGG All-in-one pleics-95-seq-f pleics-95-seq-r pleics-96 Amp Puromycin Cas9 Nick (D10A) 3 -NGG All-in-one pleics-95-seq-f pleics-95-seq-r pleics-97 Amp Puromycin Cas9 Cut 3 -NGG All-in-one pleics-95-seq-f pleics-95-seq-r pleics-98 Kan Cas9 grna pleics-98-seq-f pleics-98-seq-r pleics-99 Amp mcherry Cas9 Nick (D10A) 3 -NGG All-in-one pleics-95-seq-f pleics-95-seq-r pleics-100 Amp mcherry Cas9 Cut 3 -NGG All-in-one pleics-95-seq-f pleics-95-seq-r pleics-101 Amp megfp Cas9 Cut 3 -NGG All-in-one pleics-95-seq-f pleics-95-seq-r pleics-102 Amp Neomycin Cas9 Nick (D10A) 3 -NGG All-in-one pleics-95-seq-f pleics-95-seq-r pleics-103 Amp Neomycin Cas9 Cut 3 -NGG All-in-one pleics-95-seq-f pleics-95-seq-r pleics-104 Amp Zeocin Cas9 Nick (D10A) 3 -NGG All-in-one pleics-95-seq-f pleics-95-seq-r pleics-105 Amp Zeocin Cas9 Cut 3 -NGG All-in-one pleics-95-seq-f pleics-95-seq-r pleics-106 Amp Puromycin Cas9 All-in-one, Cut 3 -NGG Lentivirus pleics-106-seq-f pleics-106-seq-r pleics-107 Amp Puromycin Cas9 grna Lentivirus pleics-107-seq-f pleics-107-seq-r pleics-108 Amp blasticidin Cas9 Cas9 3 -NGG Lentivirus pleics-109 Amp Puromycin Cas9 All-in-one, Inducible Cas9 3 -NGG Lentivirus pleics-106-seq-f pleics-106-seq-r pleics-110 Amp Puromycin Cas9 All-in-one, Inducible Cas9 3 -NGG All-in-one pleics-95-seq-f pleics-95-seq-r pleics-111 Amp Neomycin Cas9 All-in-one, Inducible Cas9 3 -NGG All-in-one pleics-95-seq-f pleics-95-seq-r pleics-112 Amp Puromycin Cas9 Nick (D10A) 3 -NGAG All-in-one pleics-95-seq-f pleics-95-seq-r pleics-113 Amp Puromycin Cas9 Cut 3 -NGAG All-in-one pleics-95-seq-f pleics-95-seq-r pleics-114 Amp mcherry Cas9 Nick (D10A) 3 -NGAG All-in-one pleics-95-seq-f pleics-95-seq-r pleics-115 Amp mcherry Cas9 Cut 3 -NGAG All-in-one pleics-95-seq-f pleics-95-seq-r pleics-116 Amp Neomycin Cas9 Nick (D10A) 3 -NGAG All-in-one pleics-95-seq-f pleics-95-seq-r pleics-117 Amp Neomycin Cas9 Cut 3 -NGAG All-in-one pleics-95-seq-f pleics-95-seq-r pleics-118 Amp Puromycin Cpf1 Staggered Cut 5 -TTN All-in-one pleics-95-seq-f pleics-95-seq-r pleics-119 Amp Neomycin Cpf1 Staggered Cut 5 -TTN All-in-one pleics-95-seq-f pleics-95-seq-r pleics-120 Amp megfp Cpf1 Staggered Cut 5 -TTN All-in-one pleics-95-seq-f pleics-95-seq-r pleics-121 Amp mcherry Cpf1 Staggered Cut 5 -TTN All-in-one pleics-95-seq-f pleics-95-seq-r pleics-122 Amp Puromycin High-fidelity Cas9 Cut 3 -NGG All-in-one pleics-95-seq-f pleics-95-seq-r pleics-123 Amp Neomycin High-fidelity Cas9 Cut 3 -NGG All-in-one pleics-95-seq-f pleics-95-seq-r pleics-124 Amp megfp High-fidelity Cas9 Cut 3 -NGG All-in-one pleics-95-seq-f pleics-95-seq-r pleics-125 Amp mcherry High-fidelity Cas9 Cut 3 -NGG All-in-one pleics-95-seq-f pleics-95-seq-r pleics-126 Amp Puromycin High-fidelity Cas9 Cut 3 -NGG All-in-one pleics-95-seq-f pleics-95-seq-r
7 pleics-127 Amp Neomycin High-fidelity Cas9 Cut 3 -NGG All-in-one pleics-95-seq-f pleics-95-seq-r pleics-128 Amp megfp High-fidelity Cas9 Cut 3 -NGG All-in-one pleics-95-seq-f pleics-95-seq-r pleics-129 Amp mcherry High-fidelity Cas9 Cut 3 -NGG All-in-one pleics-95-seq-f pleics-95-seq-r pleics-140 Amp megfp Cas9 Nick (D10A), direct mutations 3 -NGG All-in-one pleics-95-seq-f pleics-95-seq-r pleics-141 Amp mcherry Cas9 of C to T or G to A Nick (D10A), direct mutations 3 -NGG All-in-one pleics-95-seq-f pleics-95-seq-r pleics-142 Amp Puromycin Cas9 of C to T or G to A Nick (D10A), direct mutations 3 -NGG All-in-one pleics-95-seq-f pleics-95-seq-r pleics-143 Amp Neomycin Cas9 of C to T or G to A Nick (D10A), direct mutations 3 -NGG All-in-one pleics-95-seq-f pleics-95-seq-r pleics-144 Kan megfp Cas9 of C to T or G to A Cut 3 -NGG All-in-one pleics-144-seq-f pleics-145 Kan Hygromycin mcherry Cas9 Cut 3 -NGG All-in-one pleics-144-seq-f Hygromycin pleics-146 Kan megfp Cas9 Nick (D10A) 3 -NGG All-in-one pleics-144-seq-f Hygromycin pleics-147 Kan mcherry Cas9 Nick (D10A) 3 -NGG All-in-one pleics-144-seq-f pleics-148 Kan Hygromycin megfp High-fidelity Cas9 Cut 3 -NGG All-in-one pleics-144-seq-f pleics-149 Kan Hygromycin mcherry High-fidelity Cas9 Cut 3 -NGG All-in-one pleics-144-seq-f pleics-150 Kan Hygromycin Hygromycin High-fidelity Cas9 Cut 3 -NGG All-in-one pleics-144-seq-f pleics-151 Tet pleics-151-seq-f pleics-151-seq-r
8 Vector Name AR Tag/Flag Protease cleavage Tag and linker Sequence primers N-His10-3xFlag C-EGFP-PEX2 TEV Extra GA N-Term-His10-DYKDDDDKDYKDDDDKDYKDDDDK-GS-TEV site-2 C-Term-EF-EGFP-GRAKGSGSTSGSG-PEX2 T7 Promoter 131-EGFP-Seq-R pleics-131 Amp/Neo N-His10-3xFlag TEV N-Term-His10-DYKDDDDKDYKDDDDKDYKDDDDK-GS-TEV site-2 T7 Promoter 131-APEX2-Seq-R C-APEX2 Extra GA C-Term-GRAKGSGSTSGSG-PEX2 C-EGFP -APEX2 C-Term-EF-EGFP-GRAKGSGSTSGSG-PEX2 CMV-P-F 131-EGFP-Seq-R C-APEX2 C-Term-GRAKGSGSTSGSG-PEX2 CMV-P-F 131-APEX2-Seq-R N-His10-3xFlag C-mCherry-APEX2 TEV Extra GA N-Term-His10-DYKDDDDKDYKDDDDKDYKDDDDK-GS-TEV site-2 C-Term-EF-mCherry-GRAKGSGSTSGSG-PEX2 T7 Promoter 132-mCherry-Seq-R pleics-132 Amp/Neo N-His10-3xFlag TEV N-Term-His10-DYKDDDDKDYKDDDDKDYKDDDDK-GS-TEV site-2 T7 Promoter 131-APEX2-Seq-R C-APEX2 Extra GA C-Term-GRAKGSGSTSGSG-PEX2 C-mCherry-APEX2 C-Term-EF-mCherry-GRAKGSGSTSGSG-PEX2 CMV-P-F 132-mCherry-Seq-R C-APEX2 C-Term-GRAKGSGSTSGSG-PEX2 CMV-P-F 131-APEX2-Seq-R N-His10-3xFlag C-EGFP -minisog TEV Extra GA N-Term-His10-DYKDDDDKDYKDDDDKDYKDDDDK-GS-TEV site-2 C-Term-EF-EGFP-GRAKGSGSTSGSG-miniSOG T7 Promoter 131-EGFP-Seq-R pleics-133 Amp/Neo N-His10-3xFlag TEV N-Term-His10-DYKDDDDKDYKDDDDKDYKDDDDK-GS-TEV site-2 T7 Promoter 133-miniSOG-Seq-R C-miniSOG Extra GA C-Term-GRAKGSGSTSGSG-miniSOG C-EGFP-miniSOG C-Term-EF-EGFP-GRAKGSGSTSGSG-miniSOG CMV-P-F 131-EGFP-Seq-R C-miniSOG C-Term-GRAKGSGSTSGSG-PEX2 CMV-P-F 133-miniSOG-Seq-R N-His10-3xFlag C-mCherry-miniSOG TEV Extra GA N-Term-His10-DYKDDDDKDYKDDDDKDYKDDDDK-GS-TEV site-2 C-Term-EF-mCherry-GRAKGSGSTSGSG-miniSOG T7 Promoter 132-mCherry-Seq-R pleics-134 Amp/Neo N-His10-3xFlag TEV N-Term-His10-DYKDDDDKDYKDDDDKDYKDDDDK-GS-TEV site-2 T7 Promoter 133-miniSOG-Seq-R C-miniSOG Extra GA C-Term-GRAKGSGSTSGSG-miniSOG C-mCherry-miniSOG C-Term-EF-mCherry-GRAKGSGSTSGSG-miniSOG CMV-P-F 132-mCherry-Seq-R C-miniSOG C-Term-GRAKGSGSTSGSG-miniSOG CMV-P-F 133-miniSOG-Seq-R pleics-135 Amp/Puro pleics-135-seq-f pleics-135-seq-r pleics-136 Amp/Neo N-His10-3xFlag TEV N-Term-His10-DYKDDDDKDYKDDDDKDYKDDDDK-GS-TEV site-2 T7 Promoter 131-EGFP-Seq-R C-mEGFP Extra GA C-Term-EF-mEGFP pleics-137 Amp/Neo N-His10-3xFlag TEV N-Term-His10-DYKDDDDKDYKDDDDKDYKDDDDK-GS-TEV site-2 T7 Promoter 132-mCherry-Seq-R C-mCherry Extra GA C-Term-EF-mCherry pleics-138 Amp/Neo N-mEGFP (A206K) TEV Extra GA megfp-sglrsr-tev site-2 megfp-sglrs pleics-21-seq-f pleics-12-seq-r pleics-139 Amp/Neo N-mCherry TEV Extra GA mcherry-sglrsr-tev site-2 mcherry-sglrs pleics-23-seq-f pleics-12-seq-r
9 His6-Tag: HHHHHH GST-Tag: MSPILGYWKIKGLVQPTRLLLEYLEEKYEEHLYERDEGDKWRNKKFELGLEFPNLPYYIDGDVKLTQSMAIIRYIADKHNMLGGCPKE RAEISMLEGAVLDIRYGVSRIAYSKDFETLKVDFLSKLPEMLKMFEDRLCHKTYLNGDHVTHPDFMLYDALDVVLYMDPMCLDAFPK LVCFKKRIEAIPQIDKYLKSSKYIAWPLQGWQATFGGGDHPPKS S-Tag: KETAAAKFERQHMDS Trx-Tag: MSDKIIHLTDDSFDTDVLKADGAILVDFWAEWCGPCKMIAPILDEIADEYQGKLTVAKLNIDQNPGTAPKYGIRGIPTLLLFKNGEVAA TKVGALSKGQLKEFLDANLA TAP-Tag: KRRWKKNFIAVSAANRFKKISSSGALGSGSGTMDEKTTGWRGGHVVEGLAGELEQLRARLEHHPQGQREPSGGCKLG MBP-Tag: MKTEEGKLVIWINGDKGYNGLAEVGKKFEKDTGIKVTVEHPDKLEEKFPQVAATGDGPDIIFWAHDRFGGYAQSGLLAEITPDKAFQD KLYPFTWDAVRYNGKLIAYPIAVEALSLIYNKDLLPNPPKTWEEIPALDKELKAKGKSALMFNLQEPYFTWPLIAADGGYAFKYENGK YDIKDVGVDNAGAKAGLTFLVDLIKNKHMNADTDYSIAEAAFNKGETAMTINGPWAWSNIDTSKVNYGVTVLPTFKGQPSKPFVGVL SAGINAASPNKELAKEFLENYLLTDEGLEAVNKDKPLGAVALKSYEEELAKDPRIAATMENAQKGEIMPNIPQMSAFWYAVRTAVINA ASGRQTVDEALKDAQTNSS EGFP:
10 MVSKGEELFTGVVPILVELDGDVNGHKFSVSGEGEGDATYGKLTLKFICTTGKLPVPWPTLVTTLTYGVQCFSRYPDHMKQHDFFKSA MPEGYVQERTIFFKDDGNYKTRAEVKFEGDTLVNRIELKGIDFKEDGNILGHKLEYNYNSHNVYIMADKQKNGIKVNFKIRHNIEDGS VQLADHYQQNTPIGDGPVLLPDNHYLSTQSALSKDPNEKRDHMVLLEFVTAAGITLGMDELYK EYFP: MVSKGEELFTGVVPILVELDGDVNGHKFSVSGEGEGDATYGKLTLKFICTTGKLPVPWPTLVTTFGYGLQCFARYPDHMKQHDFFKS AMPEGYVQERTIFFKDDGNYKTRAEVKFEGDTLVNRIELKGIDFKEDGNILGHKLEYNYNSHNVYIMADKQKNGIKVNFKIRHNIEDG SVQLADHYQQNTPIGDGPVLLPDNHYLSYQSALSKDPNEKRDHMVLLEFVTAAGITLGMDELYK mcherry: MVSKGEEDNMAIIKEFMRFKVHMEGSVNGHEFEIEGEGEGRPYEGTQTAKLKVTKGGPLPFAWDILSPQFMYGSKAYVKHPADIPDY LKLSFPEGFKWERVMNFEDGGVVTVTQDSSLQDGEFIYKVKLRGTNFPSDGPVMQKKTMGWEASSERMYPEDGALKGEIKQRLKLK DGGHYDAEVKTTYKAKKPVQLPGAYNVNIKLDITSHNEDYTIVEQYERAEGRHSTGGMDELYK ECFP: MVSKGEELFTGVVPILVELDGDVNGHKFSVSGEGEGDATYGKLTLKFICTTGKLPVPWPTLVTTLTWGVQCFSRYPDHMKQHDFFKS AMPEGYVQERTIFFKDDGNYKTRAEVKFEGDTLVNRIELKGIDFKEDGNILGHKLEYNYISHNVYITADKQKNGIKANFKIRHNIEDGS VQLADHYQQNTPIGDGPVLLPDNHYLSTQSALSKDPNEKRDHMVLLEFVTAAGITLGMDELYK Mistic: MFCTFFEKHHRKWDILLEKSTGVMEAMKVTSEEKEQLSTAIDRMNEGLDAFIQLYNESEIDEPLIQLDDDTAELMKQARDMYGQEKL NEKLNTIIKQILSISVSEEGEKE GB1: MQYKLILNGKTLKGETTTEAVDAATAEKVFKQYANDNGVDGEWTYDDATKTFTVTEGS Halo: EIGTGFPFDPHYVEVLGERMHYVDVGPRDGTPVLFLHGNPTSSYVWRNIIPHVAPTHRCIAPDLIGMGKSDKPDLGYFFDDHVRFMDA FIEALGLEEVVLVIHDWGSALGFHWAKRNPERVKGIAFMEFIRPIPTWDEWPEFARETFQAFRTTDVGRKLIIDQNVFIEGTLPMGVVRP LTEVEMDHYREPFLNPVDREPLWRFPNELPIAGEPANIVALVEEYMDWLHQSPVPKLLFWGTPGVLIPPAEAARLAKSLPNCKAVDIGP GLNLLQEDNPDLIGSEIARWLSTLEISG
11 hbira: MKDNTVPLKLIALLANGEFHSGEQLGETLGMSRAAINKHIQTLRDWGVDVFTVPGKGYSLPEPIQLLNAKQILGQLDGGSVAVLPVIDS TNQYLLDRIGELKSGDACIAEYQQAGRGRRGRKWFSPFGANLYLSMFWRLEQGPAAAIGLSLVIGIVMAEVLRKLGADKVRVKWPND LYLQDRKLAGILVELTGKTGDAAQIVIGAGINMAMRRVEESVVNQGWITLQEAGINLDRNTLAAMLIRELRAALELFEQEGLAPYLSR WEKLDNFINRPVKLIIGDKEIFGISRGIDKQGALLLEQDGIIKPWMGGEISLRSAEK PEX2: GKSYPTVSADYQDAVEKAKKKLRGFIAEKRCAPLMLRLAFHSAGTFDKGTKTGGPFGTIKHPAELAHSANNGLDIAVRLLEPLKAEFPI LSYADFYQLAGVVAVTGGPKVPFHPGREDKPEPPPEGRLPDPTKGSDHLRDVFGKAMGLTDQDIVALSGGHTIGAAHKERSGFEGPW TSNPLIFDNSYFTELLSGEKEGLLQLPSDKALLSDPVFRPLVDKYAADEDAFFADYAEAHQKLSELGFADA minisog: MEKSFVITDPRLPDNPIIFASDGFLELTEYSREEILGRNGRFLQGPETDQATVQKIRDAIRDQREITVQLINYTKSGKKFWNLLHLQPMRD QKGELQYFIGVQLDG TEV site-1: ENLYFQS TEV site-2: ENLYFQGA PreScission Protease Site: LEVLFQGP
12 Primer sequence: T7 Promoter 5 -TAATACGACTCACTATAGGG-3 T7 Terminator 5 -GCTAGTTATTGCTCAGCGG-3 pgex5 5 - GGGCTGGCAAGCCACGTTTGGTG-3 pgex3 5 - CCGGGAGCTGCATGTGTCAGAGG-3 5 AOX1-F 5 -GACTGGTTCCAATTGACAAGC-3 3 AOX1-R 5 -GCAAATGGCATTCTGACATCC-3 pleics-01-seq-r 5 -ATTAACATTAGTGGTGGTGGT-3 pleics-06-seq-f1 5 -CTAGAAATAATTTTGTTTAA-3 pleics-06-seq-r1 5 -GTACAATACGATTACTTTCT-3 pleics-06-seq-f2 5 -GAAAGTAATCGTATTGTACA-3 pleics-09-seq-f 5 -CCGCTGCTGCTAAATTCGAA-3 pleics-10-seq-f 5 -AGACTAATTCGAGCTCGAACA-3 pleics-10-seq-r 5 -CCCAGTCACGACGTTGTAAAACG-3 pleics-11-seq-f 5 -GAAACCATGTCGTACTACCAT-3 pleics-11-seq-r 5 -AACCTCTACAAATGTGGTAT-3 pleics-12-seq-f 5 - GGAGACCCAAGCTTGGTACC-3 pleics-12-seq-r 5 -AAGGCACAGTCGAGGCTGA-3 pleics-15-seq-r 5 -TACCATGGACGAGAA-3
13 pleics-17-seq-f 5 -TGGTTCTGGTCATCACCAT-3 pleics-19-seq-f 5 -CTGTACGGAAGTGTTACTT-3' pleics-19-seq-r 5 -CATCACAAATTTCACAAATA-3 pleics-21-seq-f 5 -CACATGGTCCTGCTGGAGTT-3 pleics-21-seq-r 5 -GCTGATTATGATCAGTTAT-3 pleics-23-seq-f 5 -CTACACCATCGTGGAACAGTA-3 pleics-29-seq-f 5 -CAAATGGGCGGTAGGCGTGTA-3 pleics-29-seq-r 5 -TCCAGCTCGACCAGGATGGGCA-3 pleics-30-seq-r 5 - TACCGTCGACTGCATGAATT-3 pleics-35-seq-r 5 - CTCAAGACCCGTTTAGAGG -3 pleics-36-seq-r 5 -ATGCACAGTTGAAGTGAACTTG -3 pleics-37-seq-f 5 - ACGCTTTGCCTGACCCTGCTTGCTCAACTC -3 pleics-39-seq-r 5 - ATCGCGCGGACCAACATCGACGTAGT -3 pleics-40-seq-r 5 - AGCTGCTCGCCAGAGTGGAACTCGCCGTT -3 pleics-41-seq-f 5 -TCCCCAACACCTAATAA-3 pleics-41-seq-r 5 -CCCTATATATGGATCCAA-3 pleics-43-seq-f 5 -AAACAGATTTTATCCATCT-3 pleics-45-seq-f 5 -GCAAAGACCCCAACGAGAA-3 pleics-45-seq-r 5 -CGGCGTTTCACTTCTGAGTT-3 pleics-48-seq-f 5 -GATAACCATCTCGCAAAT- 3
14 pleics-50-seq-r 5 - CCAAAAGAAGTCGAGTGGGT -3 pleics-52-seq-f 5 -CACCCTGGTGAACCGCATCGA-3 pleics-53-seq-f 5 -CACTCTCGGCATGGACGAGCT-3 pleics-60-seq-f 5 -CGACAACCACTACCTGAGCA-3 pleics-63-seq-r 5 -GGCCGTTTACGTCGCCGTCCA -3 pleics-67-seq-f 5 - ATCACCTCCCACAACGAGGA-3 pleics-68-seq-f 5 - TCCAAACTGAGCAAAGACCCCAA-3 pleics-71-seq-r 5 -GTAAAGTGTAAGTTGGTATTA-3 pleics-72-seq-f 5 - GCTCTAGAGCCTCTGCTAA -3 pleics-72-seq-r 5 -GGCAATATGGTGGAAAAT -3 pleics-73-seq-f 5 -CTAGGTCGATGCAGGATAA -3 pleics-74-seq-f 5 -GATTGGACTTCGCCTGTGAT -3 pleics-74-seq-r 5 -TTCCATAGGTTGGAATCTA -3 pleics-76-seq-f 5 -AAGCATAACAACGGTGGCTC -3 pleics-79-seq-r 5 -GTTGCATCACCTTCACCCTCT -3 pleics-80-seq-f 5 - AGTTTCCAAAAACGAGGAGGATTTGATATTCAC -3 pleics-80-seq-r 5 - AATATGGTGGAAAATAACATATAGACAAAC -3 pleics-81-seq-f 5 -GTTGCCAGAAAATAGCGAGTT-3 pleics-81-seq-r 5 -CCTAAGAGTCACTTTAA-3 pleics-82-seq-f 5 - TACGGCGCTCTGGATAT-3
15 pleics-82-seq-r 5 -GTAACGCCAGGGTTTTCCCA-3 pleics-83-seq-f 5 - CCCACTGCTTAACTGGCTTA-3 pleics-83-seq-r 5 -CCAATTATGTCACACCACAG-3 pleics-85-seq-f 5 - CGATAGTACTAACATACGCT -3 pleics-85-seq-r 5 -TTGGCGTCTTCCATGGTGGC-3 pleics-89-seq-f 5 -CTGCCTAACTGCAAGGCTGTGGACAT-3 pleics-90-seq-r 5 -TGCCATCGCGCGGACCAACATCGACGTAGT-3 pleics-91-seq-r 5 - TCACTTCTGAGTTCGGCATGG -3 pleics-94-seq-f 5 -CGGCATCGCAGCTTGGATACA-3 pleics-94-seq-r 5 - GGAATTTACGTAGCGGCCGC -3 pleics-95-seq-f 5 -GAGCCTATGGAAAAACGCCA-3 pleics-95-seq-r 5 - TCAGCCAGGCGGGCCATTTA -3 pleics-98-seq-f 5 -TTACGTGCCGATCAAGTCAA-3 pleics-98-seq-r 5 - AGCAGCAGATTACGCGCAG -3 pleics-106-seq-f 5 - GCAACAGACATACAAACTAA -3 pleics-106-seq-r 5 - GATGTGCGCTCTGCCCACTGA -3 pleics-107-seq-f 5 - AAGAATCCTGGCTGTGGAAA-3 pleics-107-seq-r 5 - CACTCCTTTCAAGACCTAGA EGFP-Seq-R 5 -TGAACTTGTGGCCGTTTACGT APEX2-Seq-R 5 -ATGAAGCCTCTGAGCTTCTTCTTCGCCTTCTCAA-3
16 CMV-P-F 132-mCherry-Seq-R 133-miniSOG-Seq-R pleics-135-seq-f pleics-135-seq-r pleics-144-seq-f pleics-151-seq-f pleics-151-seq-r 5 -GTCTATATAAGCAGAGCTCTCTGGCTAA-3 5 -ATGTGCACCTTGAAGCGCAT-3 5 -TCTCTGGAATACTCGGTCAGCTCCA-3 5 -GTACGGTGGGAGGCCTATATAA TGAGGAGTGAATTCCTCGAGGA-3 5 -GCAATTAATGTGAGTTAGCT-3 5 -TGGCAAATATTCTGAAATGAGCTGTTGACA-3 5 -CACGATGATCGTGCCGTGATCGAAATCCAG-3 Family A vectors homology region: N-ter: GTATTTTCAGGGCGCC... C-ter: GACGGAGCTCGAATTTCA... Family B vectors homology region: N-ter: AGGAGATATACATATG... C-ter-His or GST tag: GAAGTACAGGTTCTC... C-ter-no tag: GAAGTACAGGTTCTCTCA... Family C vectors homology region: N-ter: TACTTCCAATCCATG... C-ter: TATCCACCTTTACTGTCA...
17 Family D vectors homology region (with TEV site): Family E vectors homology region (without TEV eite): Family F vectors homology region: Family G vectors homology region: Family H vectors homology region: Family I vectors homology region: N-ter: GTATTTTCAGGGCGCC... C-ter: GTCGACTGCAGAATTtca... N-ter: TCCGGACTCAGATCT... C-ter: GTCGACTGCAGAATTtca... N-ter: CGAAACGAGGAATTCTCGATG... C-ter: CAGGTTCTCACTAGT... N-ter: GCTCGAGAATTCTTCG... C-ter: CAGGTTCTCACTAGT... N-ter: TACGTAGAATTCTCGATG... C-ter: CAGGTTCTCACTAGT... N-ter: TACGTAGAATTCTCG... C-ter: CAGGTTCTCACTAGT...
18 Family J vectors homology region (with TEV site): Family K vectors homology region (without TEV site): Family 29T vector homology region (with TEV site): Family 29 vector homology region (without TEV site): Family 35 vector homology region: Family 36 vector homology region: N-ter: GTATTTTCAGGGCGCC... C-ter: CAATGCCAATAGGATATCtca... N-ter: TCCGGACTCAGATCT... C-ter: GTGGTGGTGGTGCTCGAGtca... N-ter: TAGCGCTACCGGACTCAGATCTCGAGATG... C-ter: CTGAAAATACAGGTTCTCGAG... N-ter: CTACCGGACTCAGATCTCGAGATG... C-ter: TACCGTCGACTGCATGAATTC... N-ter: ATGGCCATGGAGGCCGAATTC... C-ter: TCGACGGATCCCCGGGAATTC... N-ter: GCCATGGAGGCCAGTGAATTC... C-ter: ATGCCCACCCGGGTGGAATTC...
19 Family 37 vector homology region: N-ter: CTACTCTAGAGCTAGCGAATTC... C-ter: GGATCCGATTTAAATTCGAATTCtca... Family 39C-Tag vector homology region: N-ter: AGGGAGACCCAAGCTTGGTACCATG... C-ter: TCGAGACGGAGCTCGAATTC Family 39NC-Tag vector homology region: N-ter: GTATTTTCAGGGCGCC... C-ter: TCGAGACGGAGCTCGAATTC Family 40C-Tag vector homology region: N-ter: AGGGAGACCCAAGCTTGGTACCATG... C-ter: ACGGTGTTGTCCTTCATCTCGAG Family 40NC-Tag vector homology region: N-ter: GTATTTTCAGGGCGCC... C-ter: ACGGTGTTGTCCTTCATCTCGAG Family 49 vector homology region: N-ter: ACCCAAGCTTGGTACCATG... C-ter: AAAATACAGGTTCTCGAG...
20 Family 69 vector homology region: N-ter: TTCCAGGGGCCCCTGGGATCC... C-ter: CTCGAGTCGACCCGGGAATTCtca... Family 72 vector homology region: N-ter: CATCATTTTGGCAAAGAATTCATG... C-ter: GGAGGGAGAGGGGCGGAATTCtca... Family 73 vector homology region: N-ter: ACCTGTGGGTACCCGCTCGAGATG... C-ter: TATCGATACCGTCGACCTCGAGtca... Family 74 vectors homology region: N-ter: TCTGCTACCACAGCCGCGGATCC... C-ter: TCCTTCACAAAGATCCTCTAGAtca... Family 76 vector homology region: N-ter: CATCATCAGACCACGCGGATTC... C-ter: TCCTTCACAAAGATCCTCTAGAtca... Family 77 vector homology region: N-ter: CATCATTTTGGCAAAACC... C-ter: GACGGAGCTCGAATTtca...
21 Family 79 vector homology region: N-ter: TTCAGGCGGCCGCGGCTCGAGC... C-ter: TTCTCCTTTACTCATGGATCCtca... Family 80 vector homology region: N-ter: TCCAAGTTTGGTCTAGAGCTAGCATG... C-ter: CCGCGGATCCGATTTAAATTCGAAtca... Family 81 vector homology region: N-ter: CTGGAATTCCCGGGGATC... C-ter: CTGCAGGTCGACGGATCC... Family 82 vector homology region: N-ter: GGTGGGATCGATTGGATC... C-ter: CTCGGTACCCGGGGATCC... Family 85 vector homology region: N-ter: TATCGATAGGTACCGAGCTC... C-ter: ATCGCAGATCTCGAGCCCGGG... Family 89vector homology region: N-ter: ATTTCCGGTCATGGGATG... C-ter: GACGGAGCTCGAATTTCA...
22 Family 90 vector homology region: N-ter: AGGAGATATACATATG... C-ter: CAGTACCGATTTCGGATCC... Family 94 vector homology region: N-ter:TAGCTAGTTAATTAAGGATCC... C-ter: CGTACCACCACACTGGGATCC... Family 95 vector homology region: Family 98 vector homology region: Family 118 vector homology region: Forward Primer:GNNNNNNNNNNNNNNNNNNNNgttttagagctagaaatagca Reverse Primer: nnnnnnnnnnnnnnnnnnnncggtgtttcgtcctttccaca N: 20-nt guild sequence; n: Reverse complementary of N. Forward Primer:GNNNNNNNNNNNNNNNNNNNNgttttagagctagaaatagca Reverse Primer: nnnnnnnnnnnnnnnnnnnncctatagtgagtcgtatta N: 20-nt guild sequence; n: Reverse complementary of N. Forward Primer: NNNNNNNNNNNNNNNNNNNNNNNNtttttttagagctagaaatagcaag Reverse Primer: nnnnnnnnnnnnnnnnnnnnnnnnatctacacttagtagaaattggt
23 N: 24-nt guild sequence; n: Reverse complementary of N. Family 131-A homology region (Tag: N-His10-3xFlag, C-EGFP/mCherry-APEX2/miniSOG): N-ter:AGGGCGCGATATCTAAGCTAGC... C-ter: GCCCTTGCTCACCATGAATTC... Family 131-B homology region (Tag: N-His10-3xFlag, C-APEX2/miniSOG Tag): N-ter:AGGGCGCGATATCTAAGCTAGC... C-ter: GTCGAGCCCGAGCCCTTGGCGCGCCC... Family 131-C homology region (Tag: C-EGFP/mCherry-APEX2/miniSOG Tag): Family 131-D homology region (Tag: C-APEX2/miniSOG Tag): Family 135 vector homology region: N-ter: TATAGGGAGACCCAAGCTTGGTACCATG... C-ter: GCCCTTGCTCACCATGAATTC... N-ter:TATAGGGAGACCCAAGCTTGGTACCATG... C-ter: GTCGAGCCCGAGCCCTTGGCGCGCCC... N-ter: GTCGAGTTAATTTGTTAACATG... C-ter: TGGTTAGCAGAGGGTTAACTCA...
24 Family 136 vector homology region: N-ter: GTATTTTCAGGGCGCC... C-ter: GCCCTTGCTCACCATGAATTC... Family 144 vector homology region: Family 151 vector homology region: Forward Primer:GNNNNNNNNNNNNNNNNNNNNgttttagagctagaaatagca Reverse Primer: nnnnnnnnnnnnnnnnnnnncaatcgctatgtcgactctat N: 20-nt guild sequence; n: Reverse complementary of N. N-ter: TTCACACAGGAAACAGAATTC... C-ter: TCCGCTAGTCCGAGGCCTCGAG...
Supporting Information
 Supporting Information Plasmid Construction DNA cloning was carried out according to standard protocols. The sequence of all plasmids was confirmed by DNA sequencing and amplified using the E. coli strain
Supporting Information Plasmid Construction DNA cloning was carried out according to standard protocols. The sequence of all plasmids was confirmed by DNA sequencing and amplified using the E. coli strain
pjnc-pgluc Codon-optimized Gaussia luciferase (pgluc) Vector backbone: JM83 pjnc-pgluc is resistant to ampicillin and neomycin High or low copy:
 pjnc-pgluc Gene/Insert name: Codon-optimized Gaussia luciferase (pgluc) Vector backbone: pcmv-jnc Vector type: Mammalian cells Backbone size w/o insert (bp): 5375 Bacterial resistance: Ampicillin and neomycin
pjnc-pgluc Gene/Insert name: Codon-optimized Gaussia luciferase (pgluc) Vector backbone: pcmv-jnc Vector type: Mammalian cells Backbone size w/o insert (bp): 5375 Bacterial resistance: Ampicillin and neomycin
University of Bristol - Explore Bristol Research
 Al-Salihi, S. A. A., Scott, T. A., Bailey, A. M., & Foster, G. D. (2017). Improved vectors for Agrobacterium mediated genetic manipulation of Hypholoma spp. and other homobasidiomycetes. Journal of Microbiological
Al-Salihi, S. A. A., Scott, T. A., Bailey, A. M., & Foster, G. D. (2017). Improved vectors for Agrobacterium mediated genetic manipulation of Hypholoma spp. and other homobasidiomycetes. Journal of Microbiological
Modular Optimization of Hemicellulose-utilizing Pathway in. Corynebacterium glutamicum for Consolidated Bioprocessing of
 [Supplementary materials] Modular Optimization of Hemicellulose-utilizing Pathway in Corynebacterium glutamicum for Consolidated Bioprocessing of Hemicellulosic Biomass Sung Sun Yim 1, Jae Woong Choi 1,
[Supplementary materials] Modular Optimization of Hemicellulose-utilizing Pathway in Corynebacterium glutamicum for Consolidated Bioprocessing of Hemicellulosic Biomass Sung Sun Yim 1, Jae Woong Choi 1,
Supplemental Information. RNase H1-Dependent Antisense Oligonucleotides. Are Robustly Active in Directing RNA Cleavage
 YMTHE, Volume 25 Supplemental Information RNase H1-Dependent Antisense Oligonucleotides Are Robustly Active in Directing RNA Cleavage in Both the Cytoplasm and the Nucleus Xue-Hai Liang, Hong Sun, Joshua
YMTHE, Volume 25 Supplemental Information RNase H1-Dependent Antisense Oligonucleotides Are Robustly Active in Directing RNA Cleavage in Both the Cytoplasm and the Nucleus Xue-Hai Liang, Hong Sun, Joshua
Trinucleotide Repeat Diseases: CRISPR Cas9 PacBio no PCR Sequencing MFMER slide-1
 Trinucleotide Repeat Diseases: CRISPR Cas9 PacBio no PCR Sequencing 2015 MFMER slide-1 Fuch s Eye Disease TCF 4 gene Fuchs occurs in about 4% of the US population. Leads to deteriorating vision without
Trinucleotide Repeat Diseases: CRISPR Cas9 PacBio no PCR Sequencing 2015 MFMER slide-1 Fuch s Eye Disease TCF 4 gene Fuchs occurs in about 4% of the US population. Leads to deteriorating vision without
Glyma10g15850 aagggatccattctggaaccatatcttgctgtg ttgggtacccttggatgcaggatgacacg AtMKK6, AT5g56580
 Supplemental table 1. Soybean MAPK, MAPKK, and MAPKKK genes and primers for VIGS cloning Gene_ID Forward Primer Reverse Primer Arabidopsis Ortholog Glyma04g03210 aagggatcctgcagcaagaaccagttcat ttgggtaccctaatggatgcgcattaggataaag
Supplemental table 1. Soybean MAPK, MAPKK, and MAPKKK genes and primers for VIGS cloning Gene_ID Forward Primer Reverse Primer Arabidopsis Ortholog Glyma04g03210 aagggatcctgcagcaagaaccagttcat ttgggtaccctaatggatgcgcattaggataaag
T-helper Type 2 driven Inflammation Defines Major Subphenotypes of Asthma
 T-helper Type 2 driven Inflammation Defines Major Subphenotypes of Asthma Prescott G. Woodruff, M.D., M.P.H., Barmak Modrek, Ph.D., David F. Choy, B.S., Guiquan Jia, M.D., Alexander R. Abbas, Ph.D., Almut
T-helper Type 2 driven Inflammation Defines Major Subphenotypes of Asthma Prescott G. Woodruff, M.D., M.P.H., Barmak Modrek, Ph.D., David F. Choy, B.S., Guiquan Jia, M.D., Alexander R. Abbas, Ph.D., Almut
SUPPLEMENTAL DATA. Structure and specificity of the Type VI secretion system ClpV-TssC interaction in enteroaggregative Escherichia coli.
 SUPPLEMENTAL DATA Structure and specificity of the Type VI secretion system ClpV-TssC interaction in enteroaggregative Escherichia coli. B. Douzi, Y.R. Brunet, S. Spinelli, V. Lensi, P. Legrand, S. Blangy,
SUPPLEMENTAL DATA Structure and specificity of the Type VI secretion system ClpV-TssC interaction in enteroaggregative Escherichia coli. B. Douzi, Y.R. Brunet, S. Spinelli, V. Lensi, P. Legrand, S. Blangy,
SOLiD Technology. library preparation & Sequencing Chemistry (sequencing by ligation!) Imaging and analysis. Application specific sample preparation
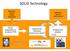 SOLiD Technology Application specific sample preparation Application specific data analysis library preparation & emulsion PCR Sequencing Chemistry (sequencing by ligation!) Imaging and analysis SOLiD
SOLiD Technology Application specific sample preparation Application specific data analysis library preparation & emulsion PCR Sequencing Chemistry (sequencing by ligation!) Imaging and analysis SOLiD
Expression analysis of PIN genes in root tips and nodules of Lotus japonicus
 Article Expression analysis of PIN genes in root tips and nodules of Lotus japonicus Izabela Sańko-Sawczenko 1, Dominika Dmitruk 1, Barbara Łotocka 1, Elżbieta Różańska 1 and Weronika Czarnocka 1, * 1
Article Expression analysis of PIN genes in root tips and nodules of Lotus japonicus Izabela Sańko-Sawczenko 1, Dominika Dmitruk 1, Barbara Łotocka 1, Elżbieta Różańska 1 and Weronika Czarnocka 1, * 1
Nan Wang, Qingming Dong, Jingjing Li, Rohit K. Jangra, Meiyun Fan, Allan R. Brasier, Stanley M. Lemon, Lawrence M. Pfeffer, Kui Li
 Supplemental Material IRF3-dependent and NF- B-independent viral induction of the zinc-finger antiviral protein Nan Wang, Qingming Dong, Jingjing Li, Rohit K. Jangra, Meiyun Fan, Allan R. Brasier, Stanley
Supplemental Material IRF3-dependent and NF- B-independent viral induction of the zinc-finger antiviral protein Nan Wang, Qingming Dong, Jingjing Li, Rohit K. Jangra, Meiyun Fan, Allan R. Brasier, Stanley
Red light-regulated reversible nuclear localization of proteins in mammalian cells and zebrafish
 Red light-regulated reversible nuclear localization of proteins in mammalian cells and zebrafish Hannes M. Beyer,1,2,3,, Samuel Juillot,1,2,3, Kathrin Herbst,4,5, Sophia L. Samodelov 1,2,3, Konrad Müller
Red light-regulated reversible nuclear localization of proteins in mammalian cells and zebrafish Hannes M. Beyer,1,2,3,, Samuel Juillot,1,2,3, Kathrin Herbst,4,5, Sophia L. Samodelov 1,2,3, Konrad Müller
Supporting Information
 Supporting Information Paired design of dcas9 as a systematic platform for the detection of featured nucleic acid sequences in pathogenic strains Yihao Zhang 1,2,8, Long Qian 4,8, Weijia Wei 1,3,7,8, Yu
Supporting Information Paired design of dcas9 as a systematic platform for the detection of featured nucleic acid sequences in pathogenic strains Yihao Zhang 1,2,8, Long Qian 4,8, Weijia Wei 1,3,7,8, Yu
AKCIÓS KATALÓGUS 2013 / 3
 Minden gépünkre 1+2 év kiterjesztett garanciát biztosítunk internetes regisztrálást követően. Az akció érvényes: 2013. szeptember 1-jétől visszavonásig, illetve a készlet erejéig. Az esetleges nyomdai
Minden gépünkre 1+2 év kiterjesztett garanciát biztosítunk internetes regisztrálást követően. Az akció érvényes: 2013. szeptember 1-jétől visszavonásig, illetve a készlet erejéig. Az esetleges nyomdai
Forensic SNP Genotyping using Nanopore MinION Sequencing
 Forensic SNP Genotyping using Nanopore MinION Sequencing AUTHORS Senne Cornelis 1, Yannick Gansemans 1, Lieselot Deleye 1, Dieter Deforce 1,#,Filip Van Nieuwerburgh 1,#,* 1 Laboratory of Pharmaceutical
Forensic SNP Genotyping using Nanopore MinION Sequencing AUTHORS Senne Cornelis 1, Yannick Gansemans 1, Lieselot Deleye 1, Dieter Deforce 1,#,Filip Van Nieuwerburgh 1,#,* 1 Laboratory of Pharmaceutical
a) bevételi főösszegét 397.168 ezer forintban, b) kiadási főösszegét 397.168 ezer forintban
 Somlóvásárhely Község Önkormányzata Képviselő-testületének 11/2015. (V.5.) önkormányzati rendelete a 2014. évi költségvetéséről szóló 2/2014. (II. 14.) önkormányzati rendelete módosításáról Somlóvásárhely
Somlóvásárhely Község Önkormányzata Képviselő-testületének 11/2015. (V.5.) önkormányzati rendelete a 2014. évi költségvetéséről szóló 2/2014. (II. 14.) önkormányzati rendelete módosításáról Somlóvásárhely
Suppl. Materials. Polyhydroxyalkanoate (PHA) Granules Have no Phospholipids. Germany
 Suppl. Materials Polyhydroxyalkanoate (PHA) Granules Have no Phospholipids Stephanie Bresan 1, Anna Sznajder 1, Waldemar Hauf 2, Karl Forchhammer 2, Daniel Pfeiffer 3 and Dieter Jendrossek 1 1 Institute
Suppl. Materials Polyhydroxyalkanoate (PHA) Granules Have no Phospholipids Stephanie Bresan 1, Anna Sznajder 1, Waldemar Hauf 2, Karl Forchhammer 2, Daniel Pfeiffer 3 and Dieter Jendrossek 1 1 Institute
Supporting Information
 Supporting Information Cell-free GFP simulations Cell-free simulations of degfp production were consistent with experimental measurements (Fig. S1). Dual emmission GFP was produced under a P70a promoter
Supporting Information Cell-free GFP simulations Cell-free simulations of degfp production were consistent with experimental measurements (Fig. S1). Dual emmission GFP was produced under a P70a promoter
KITTEK. www.sprinthungaria.hu. Cikkszám Terméknév Kiszerelés Nettó lista ár. S9912651G1 S99 1,03 l Unisoft kitt 1,350 kg 6 / 1 db 3430
 KITTEK S9912651G1 S99 1,03 l Unisoft kitt 1,350 kg 6 / 1 db 3430 S9976661G1 S99 3,85 l Unisoft kitt 5,000 kg 4 / 1 db 11950 KIFUTÓ! S9012651G1 S90 984ml Flexibilis gyors kitt 1,800 kg 6 / 1 db 3195 S9612651G1
KITTEK S9912651G1 S99 1,03 l Unisoft kitt 1,350 kg 6 / 1 db 3430 S9976661G1 S99 3,85 l Unisoft kitt 5,000 kg 4 / 1 db 11950 KIFUTÓ! S9012651G1 S90 984ml Flexibilis gyors kitt 1,800 kg 6 / 1 db 3195 S9612651G1
!"#$%" &' # ' $'## '$ '# # ( ) * )!"+,+,, - Y ' ()* +,- B &'. &'.+ +,?0>1! * 1! * E )! 5(61 78 $9 : ;> )1! 8 E & < ;7 6* ;> => 4 B ; A&B
 !"#$%" &' # ' $'## '$ '# # ( ) * )!"+,+,, - Y ' ()* +,- B &'. &'.+ +,?0>1! * 1! * E 2 1 3 4 )! 5(61 78 $9 : ;> )1! 8 E & < ;7 6* ;> => *? @$ 4 B ; A&B V C9 ; &' 2;> V2+ 1! @ $ O * @ $ ; > P -./ 1! : ;>78
!"#$%" &' # ' $'## '$ '# # ( ) * )!"+,+,, - Y ' ()* +,- B &'. &'.+ +,?0>1! * 1! * E 2 1 3 4 )! 5(61 78 $9 : ;> )1! 8 E & < ;7 6* ;> => *? @$ 4 B ; A&B V C9 ; &' 2;> V2+ 1! @ $ O * @ $ ; > P -./ 1! : ;>78
Fehérje expressziós rendszerek. Gyógyszerészi Biotechnológia
 Fehérje expressziós rendszerek Gyógyszerészi Biotechnológia Expressziós rendszerek Cél: rekombináns fehérjék előállítása nagy tisztaságban és nagy mennyiségben kísérleti ill. gyakorlati (therapia) felhasználásokra
Fehérje expressziós rendszerek Gyógyszerészi Biotechnológia Expressziós rendszerek Cél: rekombináns fehérjék előállítása nagy tisztaságban és nagy mennyiségben kísérleti ill. gyakorlati (therapia) felhasználásokra
Involvement of ER Stress in Dysmyelination of Pelizaeus-Merzbacher Disease with PLP1 Missense Mutations Shown by ipsc-derived Oligodendrocytes
 Stem Cell Reports, Volume 2 Supplemental Information Involvement of ER Stress in Dysmyelination of Pelizaeus-Merzbacher Disease with PLP1 Missense Mutations Shown by ipsc-derived Oligodendrocytes Yuko
Stem Cell Reports, Volume 2 Supplemental Information Involvement of ER Stress in Dysmyelination of Pelizaeus-Merzbacher Disease with PLP1 Missense Mutations Shown by ipsc-derived Oligodendrocytes Yuko
Dr. Ottó Szabolcs Országos Onkológiai Intézet
 Nemzeti Kutatási és Fejlesztési Program 1. Főirány: Életminőség javítása Nemzeti Onkológiai Kutatás-Fejlesztési Konzorcium a daganatos halálozás csökkentésére 1/48/2001. Részjelentés: 200. November 0.-2004.
Nemzeti Kutatási és Fejlesztési Program 1. Főirány: Életminőség javítása Nemzeti Onkológiai Kutatás-Fejlesztési Konzorcium a daganatos halálozás csökkentésére 1/48/2001. Részjelentés: 200. November 0.-2004.
University of Bristol - Explore Bristol Research
 Tan, B. G., Wellesley, F. C., Savery, N. J., & Szczelkun, M. D. (2016). Length heterogeneity at conserved sequence block 2 in human mitochondrial DNA acts as a rheostat for RNA polymerase POLRMT activity.
Tan, B. G., Wellesley, F. C., Savery, N. J., & Szczelkun, M. D. (2016). Length heterogeneity at conserved sequence block 2 in human mitochondrial DNA acts as a rheostat for RNA polymerase POLRMT activity.
Akt1 Akt kinase activity Creb signaling CCTTACAGCCCTCAAGTACTCATTC GGCGTACTCCATGACAAAGCA Arc Actin binding
 Additional File 1 (Table S1): Description and primer sequences of 80 candidate genes tested as potential synaptic plasticity formation genes in qrt-pcr array. Abbreviations: LTP: long-term potentiation,
Additional File 1 (Table S1): Description and primer sequences of 80 candidate genes tested as potential synaptic plasticity formation genes in qrt-pcr array. Abbreviations: LTP: long-term potentiation,
Példa: Aktuális könyvtár tartalmának fájlba mentése, melynek neve az aktuális dátum és idő: ls l > `date+%f_%h-%m`.txt
 Beépített változók (paraméterezés): $# - paraméterek száma $1...9 - az adott paraméter értéke $0 - az adott héjprogram neve $* - az összes paraméter egyben Idézőjelek típusai: ' ' - belső szöveg stringként
Beépített változók (paraméterezés): $# - paraméterek száma $1...9 - az adott paraméter értéke $0 - az adott héjprogram neve $* - az összes paraméter egyben Idézőjelek típusai: ' ' - belső szöveg stringként
aktuális Csőtisztítás - ellenőrzés www.rothenberger.hu ROSCOPE PIPE 25 / 22 kamera klt. Csövek, üregek vizsgálatához d 32-110 mm-ig
 Akciós ajánlatok 20 január 1-20 április 30-ig Csőtisztítás - ellenőrzés R 550 és R 600 DUGULÁSELHÁRÍTÓ GÉP Ø 20-1 és Ø 20-150 mm csövekhez, spirál és szerszámkészlettel Mobil, praktikus, gyorsan, egyszerűen
Akciós ajánlatok 20 január 1-20 április 30-ig Csőtisztítás - ellenőrzés R 550 és R 600 DUGULÁSELHÁRÍTÓ GÉP Ø 20-1 és Ø 20-150 mm csövekhez, spirál és szerszámkészlettel Mobil, praktikus, gyorsan, egyszerűen
Manuscript Title: Identification of a thermostable fungal lytic polysaccharide monooxygenase and
 1 2 3 4 5 Journal name: Applied Microbiology and Biotechnology Manuscript Title: Identification of a thermostable fungal lytic polysaccharide monooxygenase and evaluation of its effect on lignocellulosic
1 2 3 4 5 Journal name: Applied Microbiology and Biotechnology Manuscript Title: Identification of a thermostable fungal lytic polysaccharide monooxygenase and evaluation of its effect on lignocellulosic
Lakossági Általános Szerződési Feltételek. 9. Melléklet. Akciók
 Lakossági Általános Szerződési Feltételek 9. Melléklet Akciók 1. vezetékes telefon, TV, Akció megnevezése Igénybevevők köre Akció tartama Területi hatálya Feltételek Jogkövetkezmény Telepítési díjkedvezmény
Lakossági Általános Szerződési Feltételek 9. Melléklet Akciók 1. vezetékes telefon, TV, Akció megnevezése Igénybevevők köre Akció tartama Területi hatálya Feltételek Jogkövetkezmény Telepítési díjkedvezmény
Architecture of the Trypanosome RNA Editing Accessory Complex, MRB1
 Architecture of the Trypanosome RNA Editing Accessory Complex, MRB1 Michelle L. Ammerman, Kurtis M. Downey, Hassan Hashimi, John C. Fisk, Danielle L. Tomasello, Drahomíra Faktorová, Lucie Hanzálková, Tony
Architecture of the Trypanosome RNA Editing Accessory Complex, MRB1 Michelle L. Ammerman, Kurtis M. Downey, Hassan Hashimi, John C. Fisk, Danielle L. Tomasello, Drahomíra Faktorová, Lucie Hanzálková, Tony
HIVATALOS ÉRTESÍTÕ. 51. szám. A MAGYAR KÖZLÖNY MELLÉKLETE 2010. június 28., hétfõ. Tartalomjegyzék. III. Utasítások, jogi iránymutatások
 HIVATALOS ÉRTESÍTÕ A MAGYAR KÖZLÖNY MELLÉKLETE 2010. június 28., hétfõ 51. szám Tartalomjegyzék III. Utasítások, jogi iránymutatások 7/2010. (VI. 28.) KIM utasítás a Közigazgatási és Igazságügyi Minisztérium
HIVATALOS ÉRTESÍTÕ A MAGYAR KÖZLÖNY MELLÉKLETE 2010. június 28., hétfõ 51. szám Tartalomjegyzék III. Utasítások, jogi iránymutatások 7/2010. (VI. 28.) KIM utasítás a Közigazgatási és Igazságügyi Minisztérium
(A F) H&E staining of the duodenum in the indicated genotypes at E16.5 (A, D), E18.5
 Fig. S1. Brg1 ablation leads to abnormal villous structure in the duodenum in the perinatal period (A F) H&E staining of the duodenum in the indicated genotypes at E16.5 (A, D), E18.5 (B, E), and P0.5
Fig. S1. Brg1 ablation leads to abnormal villous structure in the duodenum in the perinatal period (A F) H&E staining of the duodenum in the indicated genotypes at E16.5 (A, D), E18.5 (B, E), and P0.5
Seven gene deletions in seven days: Fast generation of Escherichia coli strains tolerant to acetate and osmotic stress Supplementary Figure S1.
 Seven gene deletions in seven days: Fast generation of Escherichia coli strains tolerant to acetate and osmotic stress Sheila I. Jensen 1*, Rebecca M. Lennen 1, Markus J. Herrgård 1, Alex T. Nielsen 1*
Seven gene deletions in seven days: Fast generation of Escherichia coli strains tolerant to acetate and osmotic stress Sheila I. Jensen 1*, Rebecca M. Lennen 1, Markus J. Herrgård 1, Alex T. Nielsen 1*
AKCIÓS KATALÓGUS 2011 / 1
 Minden gépünkre 1+2 év kiterjesztett garanciát biztosítunk internetes regisztrálást követően. Az akció érvényes: 2011. április 1-től visszavonásig, illetve a készlet erejéig. A feltüntetett árak bruttó
Minden gépünkre 1+2 év kiterjesztett garanciát biztosítunk internetes regisztrálást követően. Az akció érvényes: 2011. április 1-től visszavonásig, illetve a készlet erejéig. A feltüntetett árak bruttó
BIZONYÍTOTT MINŐSÉG. KIVÁLÓ ÁR.
 BIZONYÍTOTT MINŐSÉG. KIVÁLÓ ÁR. MUNKAFÉNYSZÓRÓK EGYFUNKCIÓS LÁMPÁK MULTIFUNKCIÓS LÁMPÁK HELLA VALUEFIT Bizonyított minőség. Kiváló ár. A HELLA a HELLA VALUEFIT keretében olyan specifikus árukészletet kínál,
BIZONYÍTOTT MINŐSÉG. KIVÁLÓ ÁR. MUNKAFÉNYSZÓRÓK EGYFUNKCIÓS LÁMPÁK MULTIFUNKCIÓS LÁMPÁK HELLA VALUEFIT Bizonyított minőség. Kiváló ár. A HELLA a HELLA VALUEFIT keretében olyan specifikus árukészletet kínál,
Supplemental Table S1. Overview of MYB transcription factor genes analyzed for expression in red and pink tomato fruit.
 Supplemental Table S1. Overview of MYB transcription factor genes analyzed for expression in red and pink tomato fruit. MYB Primer pairs TC AtMYB Forward Reverse TC199266 MYB12 AGGCTCTTGGAGGTCGTTACC CAACTCTTTCCGCATCTCAATAATC
Supplemental Table S1. Overview of MYB transcription factor genes analyzed for expression in red and pink tomato fruit. MYB Primer pairs TC AtMYB Forward Reverse TC199266 MYB12 AGGCTCTTGGAGGTCGTTACC CAACTCTTTCCGCATCTCAATAATC
RINGIER AXEL SPRINGER HUNGARY RATE CARD PRICE LIST
 RINGIER AXEL SPRINGER HUNGARY RATE CARD PRICE LIST 1/1 3 670 000 B2 3 880 000 B3 3 600 000 B4 4 200 000 2/1 6 100 000 2 x 1/2 3 300 000 2/3 2 550 000 Junior 3 150 000 1/2 2 300 000 1/3 1 690 000 1/4 1
RINGIER AXEL SPRINGER HUNGARY RATE CARD PRICE LIST 1/1 3 670 000 B2 3 880 000 B3 3 600 000 B4 4 200 000 2/1 6 100 000 2 x 1/2 3 300 000 2/3 2 550 000 Junior 3 150 000 1/2 2 300 000 1/3 1 690 000 1/4 1
Limitations and challenges of genetic barcode quantification
 Limitations and challenges of genetic barcode quantification Lars hielecke, im ranyossy, ndreas Dahl, Rajiv iwari, Ingo Roeder, Hartmut eiger, Boris Fehse, Ingmar lauche and Kerstin ornils SUPPLEMENRY
Limitations and challenges of genetic barcode quantification Lars hielecke, im ranyossy, ndreas Dahl, Rajiv iwari, Ingo Roeder, Hartmut eiger, Boris Fehse, Ingmar lauche and Kerstin ornils SUPPLEMENRY
Az elektromágneses spektrum
 Az elektromágneses spektrum 400 nm 750 nm Hőmérsékleti sugárzás 1 Minden test anyagi minőségétől független, csak a test hőmérséklete által meghatározott spektrumú elektromágneses sugárzást bocsát ki, melyet
Az elektromágneses spektrum 400 nm 750 nm Hőmérsékleti sugárzás 1 Minden test anyagi minőségétől független, csak a test hőmérséklete által meghatározott spektrumú elektromágneses sugárzást bocsát ki, melyet
A HP ProLiant szerverek következő generációja
 A HP ProLiant szerverek következő generációja Megbízhatóság a változó gazdasági környezetben Almási Gábor ISS termék manager 2009 április 2008 Hewlett-Packard Development Company, L.P. The information
A HP ProLiant szerverek következő generációja Megbízhatóság a változó gazdasági környezetben Almási Gábor ISS termék manager 2009 április 2008 Hewlett-Packard Development Company, L.P. The information
Adatközpontok felügyelete
 Adatközpontok felügyelete A PlateSpin termékcsalád Hargitai Zsolt Sales Support Manager zsolt.hargitai@novell.hu Szolgáltatás-orientált adatközpont 2 Adatközpontok felügyelete 3 Miről lesz szó? 1 Fizikai
Adatközpontok felügyelete A PlateSpin termékcsalád Hargitai Zsolt Sales Support Manager zsolt.hargitai@novell.hu Szolgáltatás-orientált adatközpont 2 Adatközpontok felügyelete 3 Miről lesz szó? 1 Fizikai
Az LPCB tanúsítja, hogy
 Adószám: 11365073-2-42 MULTI ALARM BIZTONSÁGTECHNIKAI FEJLESZTŐ, KERESKEDELMI ÉS SZOLGÁLTATÓ ZRT. Bejegyezte: Fővárosi Bíróság, mint Cégbíróság: Cg.01-10-044636 Bank: RAIFFEISEN BANK 12072507-00328591-00100001
Adószám: 11365073-2-42 MULTI ALARM BIZTONSÁGTECHNIKAI FEJLESZTŐ, KERESKEDELMI ÉS SZOLGÁLTATÓ ZRT. Bejegyezte: Fővárosi Bíróság, mint Cégbíróság: Cg.01-10-044636 Bank: RAIFFEISEN BANK 12072507-00328591-00100001
Slade Pyro-Tex JS HP Hőcserélő tömítés bemutató
 Slade Pyro-Tex JS HP Hőcserélő tömítés bemutató Két gyártott termékvonal 1. Zsinóros tömítés (3300G) Szivattyúk Szelepek (Örökös Cseregarancia ) Termékismertető Egy típus valamennyi szelep és szivattyú
Slade Pyro-Tex JS HP Hőcserélő tömítés bemutató Két gyártott termékvonal 1. Zsinóros tömítés (3300G) Szivattyúk Szelepek (Örökös Cseregarancia ) Termékismertető Egy típus valamennyi szelep és szivattyú
Koffer-program Bosch elektromos kéziszerszámok számára
 686 erákofferek Bosch tartozékok elektros kézieráokhoz 09/0 offer-prora Bosch elektros kézieráok áára kívül praktikus erákoffer ütésálló űanból () va féből () Az elektros kézierához illetve sok hellyel
686 erákofferek Bosch tartozékok elektros kézieráokhoz 09/0 offer-prora Bosch elektros kézieráok áára kívül praktikus erákoffer ütésálló űanból () va féből () Az elektros kézierához illetve sok hellyel
Lakossági Általános Szerződési Feltételek. 9. Melléklet. Akciók
 Lakossági Általános Szerződési Feltételek 9. Melléklet Akciók 1. vezetékes tel efon, TV, Akció megnevezése Igénybevevők köre Akció tartama Területi hatálya Feltételek Jogkövetkezmény Telepítési díjkedvezmény
Lakossági Általános Szerződési Feltételek 9. Melléklet Akciók 1. vezetékes tel efon, TV, Akció megnevezése Igénybevevők köre Akció tartama Területi hatálya Feltételek Jogkövetkezmény Telepítési díjkedvezmény
tccattaattcgacagaccagagttaaataatccttgtatgccattgtgatcacatctacagttcagattttgtatttca
 Figure Legends for Supplementary Materials: Fig. 1. Nucleotide sequence of the zebrafish lumican (zlum) gene. Exons are indicated by uppercase letters, and introns are indicated by lowercase letters. The
Figure Legends for Supplementary Materials: Fig. 1. Nucleotide sequence of the zebrafish lumican (zlum) gene. Exons are indicated by uppercase letters, and introns are indicated by lowercase letters. The
(11) Lajstromszám: E 008 257 (13) T2 EURÓPAI SZABADALOM SZÖVEGÉNEK FORDÍTÁSA
 !HU00000827T2! (19) HU (11) Lajstromszám: E 008 27 (13) T2 MAGYAR KÖZTÁRSASÁG Szellemi Tulajdon Nemzeti Hivatala EURÓPAI SZABADALOM SZÖVEGÉNEK FORDÍTÁSA (21) Magyar ügyszám: E 0 727848 (22) A bejelentés
!HU00000827T2! (19) HU (11) Lajstromszám: E 008 27 (13) T2 MAGYAR KÖZTÁRSASÁG Szellemi Tulajdon Nemzeti Hivatala EURÓPAI SZABADALOM SZÖVEGÉNEK FORDÍTÁSA (21) Magyar ügyszám: E 0 727848 (22) A bejelentés
Heterológ fehérjék expressziójára alkalmas rendszer. fejlesztése. című doktori értekezés tézisei. Készítette: Szamecz Béla
 Heterológ fehérjék expressziójára alkalmas rendszer fejlesztése című doktori értekezés tézisei Készítette: Szamecz Béla Témavezető: Dr. Dorgai László Bay Zoltán Alkalmazott Kutatási Közalapítvány Biotechnológiai
Heterológ fehérjék expressziójára alkalmas rendszer fejlesztése című doktori értekezés tézisei Készítette: Szamecz Béla Témavezető: Dr. Dorgai László Bay Zoltán Alkalmazott Kutatási Közalapítvány Biotechnológiai
Bosch tartozékok elektromos kéziszerszámokhoz 09/10
 Elszívás Áttekintés 657 Elszívás A tartozék program külső elszíváshoz Bosch porszívókkal, ipari elektromos kéziszerszámok használatakor. Gyémánt-fúrógépek Ütvefúrógépek Falhorony- és résmarók GBH 2-20
Elszívás Áttekintés 657 Elszívás A tartozék program külső elszíváshoz Bosch porszívókkal, ipari elektromos kéziszerszámok használatakor. Gyémánt-fúrógépek Ütvefúrógépek Falhorony- és résmarók GBH 2-20
HUMAN IMMUNODEFICIENCY VIRUS (HIV) ÉS AIDS
 HUMAN IMMUNODEFICIENCY VIRUS (HIV) ÉS AIDS Dr. Mohamed Mahdi MD. MPH. Department of Infectology and Pediatric Immunology University of Debrecen 2010 Történelmi tények a HIV-ről 1981: Első megjelenés San
HUMAN IMMUNODEFICIENCY VIRUS (HIV) ÉS AIDS Dr. Mohamed Mahdi MD. MPH. Department of Infectology and Pediatric Immunology University of Debrecen 2010 Történelmi tények a HIV-ről 1981: Első megjelenés San
Supplemental Information. Investigation of Penicillin Binding Protein. (PBP)-like Peptide Cyclase and Hydrolase
 Cell Chemical Biology, Volume upplemental Information Investigation of Penicillin Binding Protein (PBP)-like Peptide Cyclase and ydrolase in urugamide on-ribosomal Peptide Biosynthesis Yongjun Zhou, Xiao
Cell Chemical Biology, Volume upplemental Information Investigation of Penicillin Binding Protein (PBP)-like Peptide Cyclase and ydrolase in urugamide on-ribosomal Peptide Biosynthesis Yongjun Zhou, Xiao
IES TM Evaluating Light Source Color Rendition
 IES TM-30-15 Evaluating Light Source Color Rendition "Original" "CRI = 80" Desaturated "CRI = 80" Saturated More metrics Color Fidelity Color Discrimination Color Preference Metrics/Measures R f (IES TM-30-15)
IES TM-30-15 Evaluating Light Source Color Rendition "Original" "CRI = 80" Desaturated "CRI = 80" Saturated More metrics Color Fidelity Color Discrimination Color Preference Metrics/Measures R f (IES TM-30-15)
KockaKobak Országos Matematikaverseny 3. osztály
 KockaKobak Országos Matematikaverseny 3. osztály 2012. november 12. Feladatok: BALOG MARIANNA, általános iskolai tanár IZSÁK DÁVID, általános iskolai tanár Lektorok: GERŐ MÁRIA, általános iskolai tanár
KockaKobak Országos Matematikaverseny 3. osztály 2012. november 12. Feladatok: BALOG MARIANNA, általános iskolai tanár IZSÁK DÁVID, általános iskolai tanár Lektorok: GERŐ MÁRIA, általános iskolai tanár
A prion fehérjecsalád citoprotektív és toxikus funkciói
 SZEGEDI TUDOMÁNYEGYETEM ÁLTALÁNOS ORVOSTUDOMÁNYI KAR MULTIDISZCIPLINÁRIS ORVOSTUDOMÁNYOK DOKTORI ISKOLA A prion fehérjecsalád citoprotektív és toxikus funkciói Tézisfüzet Nyeste Antal Témavezető: Dr. Welker
SZEGEDI TUDOMÁNYEGYETEM ÁLTALÁNOS ORVOSTUDOMÁNYI KAR MULTIDISZCIPLINÁRIS ORVOSTUDOMÁNYOK DOKTORI ISKOLA A prion fehérjecsalád citoprotektív és toxikus funkciói Tézisfüzet Nyeste Antal Témavezető: Dr. Welker
Programhely Csatorna Csomag Műhold Transzponder Frekvencia Polarizáció Formátum SymbolRate FEC Tematika Encryption
 A frekvencialista segítséget nyújt az előfizetett -csomag csatornáinak újrahangolásában. Amennyiben a mediaboxot a UPC Direct biztosította az Ön számára, a a legtöbb esetben automatikusan frissülnek a
A frekvencialista segítséget nyújt az előfizetett -csomag csatornáinak újrahangolásában. Amennyiben a mediaboxot a UPC Direct biztosította az Ön számára, a a legtöbb esetben automatikusan frissülnek a
4. gyakorlat. Mosószóda nátrium-karbonát-tartalmának meghatározása potenciometrikus titrálással
 4. gyakorlat Mosószóda nátrium-karbonát-tartalmának meghatározása potenciometrikus titrálással Név, osztály:... Mérés dátuma:... Feladat Ön egy mosószóda kiszerelő üzemben dolgozik. A beérkezett nátrium-karbonátot
4. gyakorlat Mosószóda nátrium-karbonát-tartalmának meghatározása potenciometrikus titrálással Név, osztály:... Mérés dátuma:... Feladat Ön egy mosószóda kiszerelő üzemben dolgozik. A beérkezett nátrium-karbonátot
ÉSREMEK L. csak1 NYERŐÁR
 ó n g 0 5 ób g mgm p n GO MGYRORZ L R M GZ ÉRM L O JL h h d nn űu g ú d n m 3 3 hn óg m ó ó g m n g ő p g b 5 5 0 5 85 h n ú pő n m bő pő m u m ó pdó n m pp 3 0 ÉDLM ZÉLÉ LL ROZDO» «h d M n h h d nn űu
ó n g 0 5 ób g mgm p n GO MGYRORZ L R M GZ ÉRM L O JL h h d nn űu g ú d n m 3 3 hn óg m ó ó g m n g ő p g b 5 5 0 5 85 h n ú pő n m bő pő m u m ó pdó n m pp 3 0 ÉDLM ZÉLÉ LL ROZDO» «h d M n h h d nn űu
E F O P
 E g y ü t t m z k ö d é s i a j á n l a t K ö z ö s é r t é k e i n k s o k s z í n z t á r s a d a l o m E F O P - 1.3.4-1 6 P á l y á z a t i t e r v e z e t 2. 0 ( F o r r á s : w w w. p a l y a z a
E g y ü t t m z k ö d é s i a j á n l a t K ö z ö s é r t é k e i n k s o k s z í n z t á r s a d a l o m E F O P - 1.3.4-1 6 P á l y á z a t i t e r v e z e t 2. 0 ( F o r r á s : w w w. p a l y a z a
SRP-270 SOROZAT ELECT RO-MECHANI CS
 Elakadás és ferdülésmentes technológia Többszörös interface kártya SRP-270 SOROZAT ELECT RO-MECHANI CS Blokknyomtató család Generálimportõr: FELHASZNÁLÓI ÉS PROGRAMOZÓI KÉZIKÖNYV A gyártó fenntartja a
Elakadás és ferdülésmentes technológia Többszörös interface kártya SRP-270 SOROZAT ELECT RO-MECHANI CS Blokknyomtató család Generálimportõr: FELHASZNÁLÓI ÉS PROGRAMOZÓI KÉZIKÖNYV A gyártó fenntartja a
Minden gépünkre 1+2 év kiterjesztett garanciát biztosítunk, internetes regisztrálást követően. www.hitachi-powertools.hu
 Minden gépünkre 1+2 év kiterjesztett garanciát biztosítunk, internetes regisztrálást követően. www.hitachi-powertools.hu Az akció érvényes: 2010. június 1-től visszavonásig, illetve a készlet erejéig.
Minden gépünkre 1+2 év kiterjesztett garanciát biztosítunk, internetes regisztrálást követően. www.hitachi-powertools.hu Az akció érvényes: 2010. június 1-től visszavonásig, illetve a készlet erejéig.
Az orvosi biotechnológiai mesterképzés megfeleltetése az Európai Unió új társadalmi kihívásainak a Pécsi Tudományegyetemen és a Debreceni Egyetemen
 Az orvosi biotechnológiai mesterképzés megfeleltetése az Európai Unió új társadalmi kihívásainak a Pécsi Tudományegyetemen és a Debreceni Egyetemen Azonosító szám: TÁMOP-4.1.2-08/1/A-2009-0011 Az orvosi
Az orvosi biotechnológiai mesterképzés megfeleltetése az Európai Unió új társadalmi kihívásainak a Pécsi Tudományegyetemen és a Debreceni Egyetemen Azonosító szám: TÁMOP-4.1.2-08/1/A-2009-0011 Az orvosi
Frekvencia tartományok. Számítógépes Hálózatok és Internet Eszközök. Frekvencia tartományok rádió kommunikációhoz
 Frekvencia tartományok Számítógépes Hálózatok és Internet Eszközök 2007 5. Fizikai réteg Médium közös használata, példa: ADSL LF (Low Frequency) = LW (Langwelle) = hosszúhullám MF (Medium Frequency) =
Frekvencia tartományok Számítógépes Hálózatok és Internet Eszközök 2007 5. Fizikai réteg Médium közös használata, példa: ADSL LF (Low Frequency) = LW (Langwelle) = hosszúhullám MF (Medium Frequency) =
csak csak4 csak3 csak1 sak csak2 NYERŐÁR
 mn W FM K F v n d m n d K v d 3 p d 3p 0 d 0 0 0 ó vn g 0 p mb B x M hnő po pő 3 0 3 30 CLgnd 0 Mpo á E gán po á v p g őn gbó M 8 m 08 Nop nú ó K ú ó ú á mn nop n m o C pá bnph ó ü önbö ő m bn 8 0 P dá
mn W FM K F v n d m n d K v d 3 p d 3p 0 d 0 0 0 ó vn g 0 p mb B x M hnő po pő 3 0 3 30 CLgnd 0 Mpo á E gán po á v p g őn gbó M 8 m 08 Nop nú ó K ú ó ú á mn nop n m o C pá bnph ó ü önbö ő m bn 8 0 P dá
Ft 5000 Ft 5000 Ft Ft Ft 5000 Ft 5000 Ft 5000 Ft 5000 Ft Ft 5000 Ft Ft Ft 5000 Ft 5000 Ft 5000 Ft 5000 Ft
 Az Előadóművészi Jogvédő Iroda Egyesület Elnöke által a Színművészek Jogdíjbizottsága javaslatára 2017. május 8. napján megítélt szociális támogatások Igénylő neve Jogcím Összeg Megjegyzés A.K. szociális
Az Előadóművészi Jogvédő Iroda Egyesület Elnöke által a Színművészek Jogdíjbizottsága javaslatára 2017. május 8. napján megítélt szociális támogatások Igénylő neve Jogcím Összeg Megjegyzés A.K. szociális
Programhely Csatorna Csomag Műhold Transzponder Frekvencia Polarizáció Formátum SymbolRate FEC Tematika Encryption
 Amennyiben a mediaboxot a UPC Direct biztosította az Ön számára, a a legtöbb esetben automatikusan frissülnek a készülék stand-by Amennyiben a mediaboxot nem a UPC Direct biztosította az Ön számára, a
Amennyiben a mediaboxot a UPC Direct biztosította az Ön számára, a a legtöbb esetben automatikusan frissülnek a készülék stand-by Amennyiben a mediaboxot nem a UPC Direct biztosította az Ön számára, a
 Távközlési szoftverek Ziegler Gábor, BME-T T T A távközlési szoftverek jelentősége Távközlőhálózat: műszaki eszközök komplex rendszere, mely f öldraj zilag t á v ol eső pont ok közöt t I nf ormá c ió c
Távközlési szoftverek Ziegler Gábor, BME-T T T A távközlési szoftverek jelentősége Távközlőhálózat: műszaki eszközök komplex rendszere, mely f öldraj zilag t á v ol eső pont ok közöt t I nf ormá c ió c
Gyártmányválaszték 8.5 kiadás. Vezérlés 2012-2013
 Gyártmányválaszték 8.5 kiadás Vezérlés 01-01 Tartalomjegyzék 1 > Mozgatás Újdonságokkal > Vezérlés Újdonságokkal > Előkészítés Újdonságokkal 4 > Csatlakozás Újdonságokkal 5 > Vákuum > Vezérlés Újdonságot
Gyártmányválaszték 8.5 kiadás Vezérlés 01-01 Tartalomjegyzék 1 > Mozgatás Újdonságokkal > Vezérlés Újdonságokkal > Előkészítés Újdonságokkal 4 > Csatlakozás Újdonságokkal 5 > Vákuum > Vezérlés Újdonságot
A cell-based screening system for RNA Polymerase I inhibitors
 Electronic Supplementary Material (ESI) for MedChemComm. This journal is The Royal Society of Chemistry 2019 Supporting Information A cell-based screening system for RNA Polymerase I inhibitors Xiao Tan,
Electronic Supplementary Material (ESI) for MedChemComm. This journal is The Royal Society of Chemistry 2019 Supporting Information A cell-based screening system for RNA Polymerase I inhibitors Xiao Tan,
Lindab vékonyfalú profilok méretezése DimRoof statikai szoftverrel
 indab Profil oktatási program 010 indab vékonyfalú profilok méretezése DimRoof statikai szoftverrel indab Kft. 1 1. A statikai tervezés eszközei a indabnál indab vékonyfalú acélszelvények (burkolati lemezek
indab Profil oktatási program 010 indab vékonyfalú profilok méretezése DimRoof statikai szoftverrel indab Kft. 1 1. A statikai tervezés eszközei a indabnál indab vékonyfalú acélszelvények (burkolati lemezek
Új temékek az UD-GenoMed Kft. kínálatában!
 Új temékek az UD-GenMed Kft. kínálatában! Műanyag termékek: SARSTEDT műanyag termékek teljes választéka Egyszer használats labratóriumi műanyag eszközök szövet és sejttenyésztéshez Vérvételi és diagnsztikai
Új temékek az UD-GenMed Kft. kínálatában! Műanyag termékek: SARSTEDT műanyag termékek teljes választéka Egyszer használats labratóriumi műanyag eszközök szövet és sejttenyésztéshez Vérvételi és diagnsztikai
AZ ÁRPA SZÁRAZSÁGTŰRÉSÉNEK VIZSGÁLATA: QTL- ÉS ASSZOCIÁCIÓS ANALÍZIS, MARKER ALAPÚ SZELEKCIÓ, TILLING
 Hagyomány és haladás a növénynemesítésben AZ ÁRPA SZÁRAZSÁGTŰRÉSÉNEK VIZSGÁLATA: QTL- ÉS ASSZOCIÁCIÓS ANALÍZIS, MARKER ALAPÚ SZELEKCIÓ, BÁLINT ANDRÁS FERENC 1, SZIRA FRUZSINA 1, ANDREAS BÖRNER 2, KERSTIN
Hagyomány és haladás a növénynemesítésben AZ ÁRPA SZÁRAZSÁGTŰRÉSÉNEK VIZSGÁLATA: QTL- ÉS ASSZOCIÁCIÓS ANALÍZIS, MARKER ALAPÚ SZELEKCIÓ, BÁLINT ANDRÁS FERENC 1, SZIRA FRUZSINA 1, ANDREAS BÖRNER 2, KERSTIN
AKCIÓS KATALÓGUS 2013 / 1
 Minden gépünkre 1+2 év kiterjesztett garanciát biztosítunk internetes regisztrálást követően. Az akció érvényes: 2013. január 2-tól visszavonásig, illetve a készlet erejéig. A forintárfolyam jelentős változása
Minden gépünkre 1+2 év kiterjesztett garanciát biztosítunk internetes regisztrálást követően. Az akció érvényes: 2013. január 2-tól visszavonásig, illetve a készlet erejéig. A forintárfolyam jelentős változása
«DURAK HAZELNUTS» PRESENTATION
 «DURAK HAZELNUTS» PRESENTATION PRODUCT RANGE CAPACITIES p.a. HAZELNUT KERNELS RAW : 80.000 MT ------------------------------------------------- ROASTINGCAPACITY : 30.000 MT, out of which: ROASTED WHOLE
«DURAK HAZELNUTS» PRESENTATION PRODUCT RANGE CAPACITIES p.a. HAZELNUT KERNELS RAW : 80.000 MT ------------------------------------------------- ROASTINGCAPACITY : 30.000 MT, out of which: ROASTED WHOLE
Zárójelentés. Állati rotavírusok összehasonlító genomvizsgálata. c. OTKA kutatási programról. Bányai Krisztián (MTA ATK ÁOTI)
 Zárójelentés Állati rotavírusok összehasonlító genomvizsgálata c. OTKA kutatási programról Bányai Krisztián (MTA ATK ÁOTI) 2012 1 Az Állati rotavírusok összehasonlító genomvizsgálata c. programban azt
Zárójelentés Állati rotavírusok összehasonlító genomvizsgálata c. OTKA kutatási programról Bányai Krisztián (MTA ATK ÁOTI) 2012 1 Az Állati rotavírusok összehasonlító genomvizsgálata c. programban azt
kpis(ppk20) kpis(ppk46)
 Table S1. C. elegans strains used in this study Strains carrying transgenes Strain Transgene Genotype Reference IT213 tcer-1 prom:tcer-1orf:gfp:tcer-1 3'utr unc-119(ed3) III; kpis(ppk6) This study IT283
Table S1. C. elegans strains used in this study Strains carrying transgenes Strain Transgene Genotype Reference IT213 tcer-1 prom:tcer-1orf:gfp:tcer-1 3'utr unc-119(ed3) III; kpis(ppk6) This study IT283
THE LITERARY WORKS OF ŚRĪMANTA ŚAṄKARADEVA AND MAHĀPURUṢA MĀDHAVADEVA
 THE LITERARY WORKS OF ŚRĪMANTA ŚAṄKARADEVA AND MAHĀPURUṢA MĀDHAVADEVA Uploaded by The Literature Branch of Śrīmanta Śaṅkaradeva Saṅgha [15 th June/2012] BARGĪT The Śaṅkarī Classical (BHAKTI) songs Composed
THE LITERARY WORKS OF ŚRĪMANTA ŚAṄKARADEVA AND MAHĀPURUṢA MĀDHAVADEVA Uploaded by The Literature Branch of Śrīmanta Śaṅkaradeva Saṅgha [15 th June/2012] BARGĪT The Śaṅkarī Classical (BHAKTI) songs Composed
A kábelszerelés világából Bevizsgált FAM szerszámok Bázis termékcsalád. Brutto-árlista 2008. Érvényes 2008.01.01-től. ... megoldások, melyek meggyőzik
 HU A kábelszerelés világából Bevizsgált FAM szerszámok Bázis termékcsalád Brutto-árlista 2008 Érvényes 2008.01.01-től Kábelvágó ollók Kábelvágó Kábelvágó Vágópisztoly 20 02 10 35-ig 550 1 32,99 20 02 11
HU A kábelszerelés világából Bevizsgált FAM szerszámok Bázis termékcsalád Brutto-árlista 2008 Érvényes 2008.01.01-től Kábelvágó ollók Kábelvágó Kábelvágó Vágópisztoly 20 02 10 35-ig 550 1 32,99 20 02 11
Immunológia alapjai előadás MHC. szerkezete és genetikája, és az immunológiai felismerésben játszott szerepe. Antigén bemutatás.
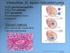 Immunológia alapjai 5-6. előadás MHC szerkezete és genetikája, és az immunológiai felismerésben játszott szerepe. Antigén bemutatás. Antigén felismerés Az ellenanyagok és a B sejt receptorok natív formában
Immunológia alapjai 5-6. előadás MHC szerkezete és genetikája, és az immunológiai felismerésben játszott szerepe. Antigén bemutatás. Antigén felismerés Az ellenanyagok és a B sejt receptorok natív formában
United Kingdom Dr Diószeghy Csaba PhD,FCEM,FERC,FFICM
 Emergency Medicine in the United Kingdom Dr Diószeghy Csaba PhD,FCEM,FERC,FFICM Consultant in Emergency Medicine and Intensive Care East Surrey Hospital (Brighton Medical School) Redhill, Surrey United
Emergency Medicine in the United Kingdom Dr Diószeghy Csaba PhD,FCEM,FERC,FFICM Consultant in Emergency Medicine and Intensive Care East Surrey Hospital (Brighton Medical School) Redhill, Surrey United
BIOFIZIKA. Metodika- 2. Liliom Károly. MTA TTK Enzimológiai Intézet liliom@enzim.hu
 BIOFIZIKA 2012 11 12 Metodika- 2 Liliom Károly MTA TTK Enzimológiai Intézet liliom@enzim.hu A biofizika előadások temakkája 1. 09-03 Biofizika: fizikai szemlélet, modellalkotás, biometria 2. 09-10 SZÜNET
BIOFIZIKA 2012 11 12 Metodika- 2 Liliom Károly MTA TTK Enzimológiai Intézet liliom@enzim.hu A biofizika előadások temakkája 1. 09-03 Biofizika: fizikai szemlélet, modellalkotás, biometria 2. 09-10 SZÜNET
Kábelszerelés szerszámai
 KH8 Préselési tartomány: 1,5-10 mm 2 KH14 Préselési tartomány: 6-16 mm 2 KH16 Préselési tartomány: 1,5-16 mm 2 KH120 Benyomótüskés méretpontosító csavar, méretjelölés a fejen Préselési tartomány: 10-95
KH8 Préselési tartomány: 1,5-10 mm 2 KH14 Préselési tartomány: 6-16 mm 2 KH16 Préselési tartomány: 1,5-16 mm 2 KH120 Benyomótüskés méretpontosító csavar, méretjelölés a fejen Préselési tartomány: 10-95
MikroLAN-Industria Elektronikai Kft. POSTAI CSOMAGKÜLDÉS: ( 52 ) 414-777, ( 52 ) 315-774 1149 Budapest, Nagy Lajos király útja 112.
 CD lemezek: Adathordozók Hologramos, jogdíjas termékek. Emtec-BASF CD-R 80 min. / 700 MB 52x cake box. 10 darab kerek műanyag dobozban Emtec-BASF CD-R 80 min. / 700 MB 52x vékony tok CD-R 80 min. / 700
CD lemezek: Adathordozók Hologramos, jogdíjas termékek. Emtec-BASF CD-R 80 min. / 700 MB 52x cake box. 10 darab kerek műanyag dobozban Emtec-BASF CD-R 80 min. / 700 MB 52x vékony tok CD-R 80 min. / 700
Akciós árlista 2015.07.11.
 M O T O R O L A J S Z E M É L Y A U T Ó K H O Z VS GAS SAE 15W-40, CCMC G4, API SG 1 fém 10861626 1 133 Ft 1 439 Ft 1 439 Ft HPX SAE 20W-50, ACEA A3/B2, API SJ/CF, 1 műa. 15129318 1 351 Ft 1 716 Ft 1 716
M O T O R O L A J S Z E M É L Y A U T Ó K H O Z VS GAS SAE 15W-40, CCMC G4, API SG 1 fém 10861626 1 133 Ft 1 439 Ft 1 439 Ft HPX SAE 20W-50, ACEA A3/B2, API SJ/CF, 1 műa. 15129318 1 351 Ft 1 716 Ft 1 716
Tankönyvrendelés a 2012/13-as tanévre a leendő 11. A osztály számára
 a leendő 11. A osztály számára A tan ára tájékoztató jellegű, a kiadó fenntartja az árváltoztatás jogát. megjelölt tanet és tanulást segítő kiadványokat, melyeket 2012. augusztusában a leendő 11. B osztály
a leendő 11. A osztály számára A tan ára tájékoztató jellegű, a kiadó fenntartja az árváltoztatás jogát. megjelölt tanet és tanulást segítő kiadványokat, melyeket 2012. augusztusában a leendő 11. B osztály
NEVEZÉSI LAP / ENTRY FORM MARATON - váltó / MARATHON - relay
 NEVEZÉSI LAP / ENTRY FORM MARATON - váltó / MARATHON - relay RAJTSZÁM / START NUMBER: (A rajtszámot a rendezőség tölti ki. / To be entered by the organisers:) Kérjük a nevezési lapot NYOMTATOTT BETŰVEL,
NEVEZÉSI LAP / ENTRY FORM MARATON - váltó / MARATHON - relay RAJTSZÁM / START NUMBER: (A rajtszámot a rendezőség tölti ki. / To be entered by the organisers:) Kérjük a nevezési lapot NYOMTATOTT BETŰVEL,
Lele Zsolt. MTA Kísérleti Orvostudományi Kutatóintézet
 A hal mint modellállat a kutatásban Lele Zsolt MTA Kísérleti Orvostudományi Kutatóintézet A hal mint modellállat a kutatásban Halfajták A hal mint modellállat a kutatásban Halfajták Gazdaságilag jelentıs
A hal mint modellállat a kutatásban Lele Zsolt MTA Kísérleti Orvostudományi Kutatóintézet A hal mint modellállat a kutatásban Halfajták A hal mint modellállat a kutatásban Halfajták Gazdaságilag jelentıs
Gyógyszerek és DNS mutációk kimutatása vérből
 Gyógyszerek és DNS mutációk kimutatása vérből AKI kíváncsi kémikus kutatótábor Kolostyák Zsuzsanna, Békés Márta, Varga Bálint 2010. Július 2. Enzimek; metabolizmus - A legtöbb biokémiai lépést enzimek
Gyógyszerek és DNS mutációk kimutatása vérből AKI kíváncsi kémikus kutatótábor Kolostyák Zsuzsanna, Békés Márta, Varga Bálint 2010. Július 2. Enzimek; metabolizmus - A legtöbb biokémiai lépést enzimek
Felona st. 6 typefaces Latin Extended-A & Sᴍᴀʟʟ Cᴀᴘꜱ 802 ɢʟʏᴘʜꜱ. ;)Ù}HFLåORTXYWyzv7Snr]jo{ by defharo
![Felona st. 6 typefaces Latin Extended-A & Sᴍᴀʟʟ Cᴀᴘꜱ 802 ɢʟʏᴘʜꜱ. ;)Ù}HFLåORTXYWyzv7Snr]jo{ by defharo Felona st. 6 typefaces Latin Extended-A & Sᴍᴀʟʟ Cᴀᴘꜱ 802 ɢʟʏᴘʜꜱ. ;)Ù}HFLåORTXYWyzv7Snr]jo{ by defharo](/thumbs/101/149858952.jpg) Felona st. 6 typefaces Latin Extended-A & Sᴍᴀʟʟ Cᴀᴘꜱ 802 ɢʟʏᴘʜꜱ ;)Ù}HFLåORTXYWyzv7Snr]jo{ by defharo FAMILY A A A A ᴀ ᵃaaaaₐ A A A A ᴀ ᵃaaaaₐ EXTRA LIGHT LIGHT A A A A ᴀ ᵃaaaaₐ A A A A ᴀ ᵃaaaaₐ REGULAR
Felona st. 6 typefaces Latin Extended-A & Sᴍᴀʟʟ Cᴀᴘꜱ 802 ɢʟʏᴘʜꜱ ;)Ù}HFLåORTXYWyzv7Snr]jo{ by defharo FAMILY A A A A ᴀ ᵃaaaaₐ A A A A ᴀ ᵃaaaaₐ EXTRA LIGHT LIGHT A A A A ᴀ ᵃaaaaₐ A A A A ᴀ ᵃaaaaₐ REGULAR
TRANSZGENIKUS BIOMARKER ZEBRADÁNIÓ (DANIO RERIO) VONALAK ALKALMAZÁSÁNAK LEHETŐSÉGEI AZ ÖKOTOXIKOLÓGIÁBAN
 TRANSZGENIKUS BIOMARKER ZEBRADÁNIÓ (DANIO RERIO) VONALAK ALKALMAZÁSÁNAK LEHETŐSÉGEI AZ ÖKOTOXIKOLÓGIÁBAN Csenki-Bakos Zsolt, Csenki-Bakos Katalin, Kovács Balázs, Ferincz Árpád és Urbányi Béla Zebradánió
TRANSZGENIKUS BIOMARKER ZEBRADÁNIÓ (DANIO RERIO) VONALAK ALKALMAZÁSÁNAK LEHETŐSÉGEI AZ ÖKOTOXIKOLÓGIÁBAN Csenki-Bakos Zsolt, Csenki-Bakos Katalin, Kovács Balázs, Ferincz Árpád és Urbányi Béla Zebradánió
MELLÉKLET / ANNEX. EU MEGFELELŐSÉGI NYILATKOZAT-hoz for EU DECLARATION OF CONFORMITY
 TRACON BUDAPEST KFT. TRACON BUDAPEST LTD. 2120 DUNAKESZI, PALLAG U. 23. TEL.: (27) 540-000, FAX: (27) 540-005 WWW.TRACON.HU EU MEGFELELŐSÉGI NYILATKOZAT, a 79/1997. (XII. 31.) IKIM számú és a 374/2012.
TRACON BUDAPEST KFT. TRACON BUDAPEST LTD. 2120 DUNAKESZI, PALLAG U. 23. TEL.: (27) 540-000, FAX: (27) 540-005 WWW.TRACON.HU EU MEGFELELŐSÉGI NYILATKOZAT, a 79/1997. (XII. 31.) IKIM számú és a 374/2012.
Karosszéria védelem. Kőfelverődés elleni védelem Üregvédelem Alvázvédelem
 Karosszéria védelem Kőfelverődés elleni védelem Üregvédelem Alvázvédelem Karosszéria védelem További szolgáltatások az autósok részére Három felhasználási területre kínálunk termékeket: A használt és az
Karosszéria védelem Kőfelverődés elleni védelem Üregvédelem Alvázvédelem Karosszéria védelem További szolgáltatások az autósok részére Három felhasználási területre kínálunk termékeket: A használt és az
Vektoralgebra feladatlap 2018 január 20.
 1. Adott az ABCD tetraéder, határozzuk meg: a) AB + BD + DC b) AD + CB + DC c) AB + BC + DA + CD Vektoralgebra feladatlap 018 január 0.. Adott az ABCD tetraéder. Igazoljuk, hogy AD + BC = BD + AC, majd
1. Adott az ABCD tetraéder, határozzuk meg: a) AB + BD + DC b) AD + CB + DC c) AB + BC + DA + CD Vektoralgebra feladatlap 018 január 0.. Adott az ABCD tetraéder. Igazoljuk, hogy AD + BC = BD + AC, majd
Többfunkciós termosztatikus Cirkulációs szelep - MTCV
 Többfunkciós termosztatikus Cirkulációs szelep - MTCV Alkalmazás 1. ábra Alapváltozat - A. ábra* Segédenergia nélküli automatikus fertőtlenítési funkcióval - B * A hőmérő külön opció 3. ábra Elektronikus
Többfunkciós termosztatikus Cirkulációs szelep - MTCV Alkalmazás 1. ábra Alapváltozat - A. ábra* Segédenergia nélküli automatikus fertőtlenítési funkcióval - B * A hőmérő külön opció 3. ábra Elektronikus
a NAT /2013 nyilvántartási számú akkreditált státuszhoz
 Nemzeti Akkreditáló Testület RÉSZLETEZÕ OKIRAT a NAT-1-1406/2013 nyilvántartási számú akkreditált státuszhoz A BIOMI Biotechnológiai Szolgáltató Kft. Vizsgálólaboratórium (2100 Gödöllõ, Szent-Györgyi Albert
Nemzeti Akkreditáló Testület RÉSZLETEZÕ OKIRAT a NAT-1-1406/2013 nyilvántartási számú akkreditált státuszhoz A BIOMI Biotechnológiai Szolgáltató Kft. Vizsgálólaboratórium (2100 Gödöllõ, Szent-Györgyi Albert
TRANSZGENIKUS BIOMARKER ZEBRADÁNIÓ (DANIO RERIO) VONALAK POTENCIÁLIS ALKALMAZÁSA A TOXIKOLÓGIÁBAN
 TRANSZGENIKUS BIOMARKER ZEBRADÁNIÓ (DANIO RERIO) VONALAK POTENCIÁLIS ALKALMAZÁSA A TOXIKOLÓGIÁBAN Csenki Zsolt, Bakos Katalin, Kovács Róbert, Ferincz Árpád, Czimmerer Zsolt, Reining Márta, Urbányi Béla
TRANSZGENIKUS BIOMARKER ZEBRADÁNIÓ (DANIO RERIO) VONALAK POTENCIÁLIS ALKALMAZÁSA A TOXIKOLÓGIÁBAN Csenki Zsolt, Bakos Katalin, Kovács Róbert, Ferincz Árpád, Czimmerer Zsolt, Reining Márta, Urbányi Béla
Termékkatalógus. Metylan - MESTERIEN. EGYSZERÛEN.
 Termékkatalógus 2008 Metylan - MESTERIEN. EGYSZERÛEN. Javasolt fogyasztói árlista Érvényes: 2008. február 15-tôl. Metylan tapétaragasztók és segédanyagok Termék Cikkszám EAN-kód Száll. egys. tartalma Jav.
Termékkatalógus 2008 Metylan - MESTERIEN. EGYSZERÛEN. Javasolt fogyasztói árlista Érvényes: 2008. február 15-tôl. Metylan tapétaragasztók és segédanyagok Termék Cikkszám EAN-kód Száll. egys. tartalma Jav.
Markerless Escherichia coli rrn Deletion Strains for Genetic Determination of Ribosomal Binding Sites
 Markerless Escherichia coli rrn Deletion Strains for Genetic Determination of Ribosomal Binding Sites Selwyn Quan *, Ole Skovgaard, Robert E. McLaughlin, Ed T. Buurman,1, and Catherine L. Squires * Department
Markerless Escherichia coli rrn Deletion Strains for Genetic Determination of Ribosomal Binding Sites Selwyn Quan *, Ole Skovgaard, Robert E. McLaughlin, Ed T. Buurman,1, and Catherine L. Squires * Department
LiveWire I-Wrench nyomatékkulcs gyártáshoz. LiveWire I-Wrench HU 0513. Okos és rugalmas.
 ivewire I-Wrench nyomatékkulcs gyártáshoz ivewire I-Wrench HU 0513 kos és rugalmas. ivewire I-Wrench nyomatékkulcs Vezeték nélküli. Rugalmas. Az Ön produktivitása - az Ön szabadsága. A Cleco ivewire I-Wrench
ivewire I-Wrench nyomatékkulcs gyártáshoz ivewire I-Wrench HU 0513 kos és rugalmas. ivewire I-Wrench nyomatékkulcs Vezeték nélküli. Rugalmas. Az Ön produktivitása - az Ön szabadsága. A Cleco ivewire I-Wrench
NYOMÁSTARTÁS ÉS VÍZMINŐSÉG Cikkszám Megnevezés Nettó ár (EUR)
 Statico tágulási tartályok fűtésre, hűtésre Statico SD - 3 bar 7101000 SD 8.3 tágulási tartály 8 liter PS=3bar P0=1,0bar 74,06 7101001 SD 12.3 tágulási tartály 12 liter PS=3bar P0=1,0bar 77,32 7101002
Statico tágulási tartályok fűtésre, hűtésre Statico SD - 3 bar 7101000 SD 8.3 tágulási tartály 8 liter PS=3bar P0=1,0bar 74,06 7101001 SD 12.3 tágulási tartály 12 liter PS=3bar P0=1,0bar 77,32 7101002
