Genetikai térképezés, gén azonosítás
|
|
|
- Gréta Siposné
- 8 évvel ezelőtt
- Látták:
Átírás
1 Genetikai térképezés, gén azonosítás, Karsai Ildikó
2 Növénynemesítés feladatai Növénytermesztés hatékonyságának és a termésbiztonság növelése speciális termesztési rendszerek biztosítása (herbicid tolerancia) biotikus stressz tolerancia növelése környezeti adaptáció és abiotikus stressz tolerancia javítása. Víz (WUE) és nitrogén hasznosítás (NUE) javítása, fagyállóság és szárazságtűrés javítása Funkcionális élelmiszer alapanyag előállítására alkalmas növényfajta Bioenergetikai célra alkalmas növények nemesítése Technológiai rendszerekre adaptált és/vagy nemesített fajták (gyógyszer alapanyag, oltóanyag) sejt fermentorokban szántóföldi növénytermesztésben
3 Gén azonosítás Adott tulajdonság fenotípusos megnyilvánulásában meghatározó szerepet játszó genetikai komponensek, szabályozó mechanizmusok, biokémiai folyamatok megértése. diagnosztikai markerek, módszerek kidolgozása természetes variabilitás tesztelése vad és termesztett fajtákon belül, valamint a rokon fajok csoportjában hasznos gének, gén allélek beépítése a nemesítési alapanyagokba marker szelekcióval egybekötött hagyományos keresztezések során gén transzformációs technikák alkalmazásával
4 Minőségi és mennyiségi tulajdonságok Azok a tulajdonságokat amelyek mértékegységgel nem, vagy csak nehezen mérhetők, kialakulásuk kevéssé függ a környezet hatásától, minőségi tulajdonságoknak nevezzük. Megnyilvánulásukat egy vagy néhány gén határozza meg. Pl: betegség ellenállóság A mértékegységgel mérhető tulajdonságok, amelyek kialakulásában a környezet hatása jelentős szerepet játszik: mennyiségi tulajdonságok. Kifejeződésük általában sok gén kölcsönhatásának eredménye pl: termőképesség
5 Gén azonosítás főbb módszerei Marker kapcsoltsági térképek Két szülős térképező populációk Széles genetikai bázist képviselő fajtakör Jelölt gén megközelítése Genom pozíció függő stratégiák: pozicionális klónozás, deléciós vonalak Összehasonlító genomikai stratégiák: modell növények Mesterséges genetikai variáció (indukált mutációs populációk) Gén csapdázás: T-DNS, transzpozon Gén expressziós mintázatok elemzése: Differential Display DNS microarray, DNS chip
6 Gén azonosítás főbb módszerei Marker kapcsoltsági térképek Két szülős térképező populációk Széles genetikai bázist képviselő fajtakör Jelölt gén megközelítése Genom pozíció függő stratégiák: pozicionális klónozás, deléciós vonalak Összehasonlító genomikai stratégiák: modell növények Mesterséges genetikai variáció (indukált mutációs populációk) Gén csapdázás: T-DNS, transzpozon Gén expressziós mintázatok elemzése: Differential Display DNS microarray, DNS chip
7 Marker kapcsoltsági térképek Térképező populáció Molekuláris marker technikák Számítógépes térképező programok
8 Térképező populáció Két homozigóta szülő keresztezéséből származó utódok a térképező populáció, amelynek minden egyedéről meghatározható, hogy a szülőktől milyen kombinációt örökölt két vizsgált allélpárra, P vagy R típust. Ennek ismeretében a rekombináció gyakorisága meghatározható akár közvetlenül (r=r/p+r), akár LOD számítással. Ezt az r (rekombinaciós gyakoriság) értéket genetikai térképezésre használhatjuk.
9 Marker kapcsoltsági térképek Térképező populáció Keresztezési partnerek kiválasztása: távoli vagy speciális Populáció típusa: hasadó F2 populáció homozigóta populációk (DH, RIL, SSD) egyéb speciális populációk (NIL, egy kromoszómára rekombináns stb.) Populáció mérete DNS izolálás markeres vizsgálatok Genom szintű különbségek
10 Öntermékenyülő növények térképezési populációinak főbb típusai
11 Genetikai markerek Marker kapcsoltsági térképek A genetikai markerek az vizsgált szervezetek közötti genetikai különbségeket teszik láthatóvá. Morfológiai marker Biokémiai markerek Molekuláris markerek Számuk korlátozott megnyilvánulásuk Környezeti hatástól és Fejlődési állapottól függő Nukleotid azaz DNS szintű variabilitás vizsgálatára alkalmasak. Számuk korlátlan, megnyilvánulásuk nem függ a környezeti hatásoktól. Az ideális DNS markerre jellemző a nagyfokú polimorfizmus koddomináns öröklődés Gyakori előfordulás Könnyű hozzáférhetőség Reprodukálhatóság Kódoló Genom Nem kódoló Morfológiai marker Biokémiai marker Molekuláris marker
12 Oregon Wolf Barley Morfológiai markerek
13 Genetikai markerek Marker kapcsoltsági térképek A genetikai markerek az vizsgált szervezetek közötti genetikai különbségeket teszik láthatóvá. Morfológiai marker Biokémiai markerek Molekuláris markerek Számuk korlátozott megnyilvánulásuk Környezeti hatástól és Fejlődési állapottól függő Nukleotid azaz DNS szintű variabilitás vizsgálatára alkalmasak. Számuk korlátlan, megnyilvánulásuk nem függ a környezeti hatásoktól. Az ideális DNS markerre jellemző a nagyfokú polimorfizmus koddomináns öröklődés Gyakori előfordulás Könnyű hozzáférhetőség Reprodukálhatóság Kódoló Genom Nem kódoló Morfológiai marker Biokémiai marker Molekuláris marker
14 Biokémiai markerek Izoenzimek: Az emzimek allél tíusait különbözteti meg Elektroforézis vagy speciális festés segítségével tehetők láthatóvá
15 Genetikai markerek Marker kapcsoltsági térképek A genetikai markerek az vizsgált szervezetek közötti genetikai különbségeket teszik láthatóvá. Morfológiai marker Biokémiai markerek Molekuláris markerek Számuk korlátozott megnyilvánulásuk környezeti hatástól és fejlődési állapottól függő Nukleotid azaz DNS szintű variabilitás vizsgálatára alkalmasak. Számuk korlátlan, megnyilvánulásuk nem függ a környezeti hatásoktól. Az ideális DNS markerre jellemző a nagyfokú polimorfizmus koddomináns öröklődés Gyakori előfordulás Könnyű hozzáférhetőség Reprodukálhatóság Kódoló Genom Nem kódoló Morfológiai marker Biokémiai marker Molekuláris marker
16 Monomorf és polimorf markerek
17 Marker kapcsoltsági térképek Molekuláris marker technikák Hibridizáción alapuló RFLP PCR alapú markerek ismeretlen kromoszómális elhelyezkedésű: RAPD, AFLP ismert kromoszómális elhelyezkedésű: SSR, STS funkcionális markerek: EST, SNP, Tillling HTP marker technikák (DArT) Szekvencia alapú Új generációs szekvenálás (Illumina platform)
18 Marker kapcsoltsági térképek RFLP (Restriction Fragment Lenght Polimorphism) A DNS molekula restrikciós endonukleázzal történő emésztésén és Southern-hibridizáción alapuló módszer. A hasító helyeken történt pont mutáció (SNP) vagy inszerció és deléció (INDEL) okozta különbségek mutathatók ki. Restrikciós endonukleázok: bakteriális eredetű enzimek 4-10 nukleotid hosszúságú palindróma szekcvenciát ismernek fel nagyon specifikusak az adott DNS-szekvenciára; (ha akár csak egy bázisnyi különbség van a hasítási helyen, már nem történik meg a hasítás) a hasítással ragadós (EcoRI) vagy tompa (SmaI; HaeIII) vég jön létre
19 RFLP Lépések: a vad típusú és az ismeretlen DNS hasítása restrikciós endonukleázokkal (ha a mutáció a restrikciós hasítóhelyen volt, akkor ott az enzim nem képes hasítani) a mintát gélre vinni, elektromos erőtérben megfuttatni blottolás membránra hibridizáció általában radioaktív próbával Autoradiográfia Előnye: Nem szükséges a templát DNS szekvenciájának ismerete A heterozigóták megkülönböztetését biztosító kodomináns módszer Megbízható, jól ismételhető Hátránya: Egyes fajokban a restrikciós hasítóhelyek megfelelő szintű polimorfizmusának hiánya Nagy mennyiségű DNS-t igényel Southern hibridizációhoz jelölt próba szükséges Hosszadalmas és költséges
20 Marker kapcsoltsági térképek Molekuláris marker technikák RFLP PCR alapú markerek ismeretlen kromoszómális elhelyezkedésű: RAPD, AFLP ismert kromoszómális elhelyezkedésű: SSR, STS funkcionális markerek: EST, SNP, Tillling HTP marker technikák (DArT) Új generációs szekvenálás (Illumina platform)
21 PCR (polymerase chain reaction) Akár 1 sejtből származó, egyetlen nukleinsav molekula megsokszorozására enzimatikus úton, élő szervezet (baktérium, élesztő) igénybevétele nélkül..95c. DNS szálak kitekerednek..denaturáció.. Hűtés primerek olvadáspontja alá 1-2 fokkal Primerek bekötődése hibridizáció 72 C DNS polimeráz a primerektől építi a DNS láncot polimerizáció DNS Nukleotidok Primerek DNS polimeráz puffer
22 PCR (polymerase chain reaction) PCR készülék PCR alapú markerek Előnye: Gyors, kis mennyiségű DNS-t igényel, nem kell jelölt próbát alkalmazni Hátránya: Megbizhatósága függ az alkalmazott markertípustól Horizontális gél elektroforézis Agaróz gélelektroforézis EtBr festés
23 Molekuláris marker technikák ismeretlen kromoszómális elhelyezkedésű: RAPD (Randomly Amplyfied Polymorphic DNA), Nagyfokú polimorfizmus Marker kapcsoltsági térképek Reprodukálhatósága függ a kisérleti körülményektől A termék szekvenálása után specifikus primer tervezhető (SCAR) Kodominánsá tehető CAPS (Cleaved Amplified Polymorphic Secuence) A random amplifikáció során kapott monomorf mintázat (azonos méret, de különböző szekvencia) restrikciós hasitással polimorffá tehető. Kodomináns AFLP (Amplified Fragment Lenght Polimorphism) A genomi DNS restrikciós emésztésén és a fragmantumok szelektív PCR amplifikációján alapul. Sok fragmentumot eredményez, domináns markereket eredményez, lokusz-specifikus markerek fejlesztése nemhéz.
24 Marker kapcsoltsági térképek Molekuláris marker technikák ismert kromoszómális elhelyezkedésű: SSR (Simple Sequence Repeat) Fajtól függően a genom 10-90% monoton ismétlődő nukleotid motívumokat tartalmaz (AC, AG, CG, TA, AG/TC, AC/TG) A határszekvenciákra tervezett primerekkel hossz polimorfizmus mutatható ki. Jól használható genotipizálásra, ujjlenyomat meghatározásra. STS (Sequence-Tagged-Site) Egyedi szekvenciájú rövid DNS szakasz Kromoszómális elhelyezkedése ismert Igen gyors és megbízható eszközzé vált a genetikai vizsgálatokban, mely térképezésre és markerszerlekcióra egyaránt használható
25 Markerek (M1 ) DNS markerek, mint ujjlenyomatok M1 N1 N2 N5 N10 N15 M2 M3 A szülő B szülő A x B keresztezés utódpopulációjának növényegyedei (N1 )
26 Marker kapcsoltsági térképek Molekuláris marker technikák funkcionális markerek: EST (Expressed sequence Tagg) cdns-ek részei RNS izolálás > cdns klónozás > szekvenálás > EST adatbázis > primer tervezés Összehasonlító térképezés SNP (Single nucleotide polymorphysms) DNS szekvencia variáció, mely akkor jön létre, ha egy nukleotid a genomban megváltozik (pl.: AAGCCTA AAGCTTA ) tulajdonképpen pontmutáció Csak akkor tekinthetjük a variációt SNP-nek, ha a populáció legalább 1%-ában megjelenik Allélvariációk kimutatása Szekvencia ismeret szükséges Tillling (Targeting Induced Local Lesions IN Genomes) Mutáns populáció előállítás HTTP mutáns azonosítás
27 esnp detektálás Árpában több, mint 420,000 EST áll rendelkezésre 8 különböző genotípusból EST jelentős többségében kimutatható SNP a fajták között 10,985 esnp azonosítottak 3,334 génben Elsődleges szempont, az eredmények egységesíthetősége az ÁRPA1 Gén-Chip (kapcsolat a gén expressziós mintázatokkal), valamint a BAC- könyvtár alapú fizikai térkép információkkal
28 Marker kapcsoltsági térképek Molekuláris marker technikák HTP marker technikák Speciális műszerezettséget igényel Multiplex módszerek Rövid idő alatt több száz vagy ezer marker Szolgáltatás jellegűek DArT (Diversity Arrays Technology) hybridizáción alapuló marker technológia Nem igényel szekvencia információkat Nagy mennyiségű adat létrehozása Új generációs szekvenálás Illumina platform,
29 Az Illumina Bead Array az SNP genotipizálás nagyhatékonyságú rendszere Bead Array leolvasó (SNP-re) Oligo pool assays (OPA) 1,536 SNP tesztelhető párhuzamosan Typical Data in GenCall Display
30 Marker kapcsoltsági térképek A növénynemesítésben és a genetikában az egyik alapvető cél, hogy a tulajdonságokat meghatározó gének kromoszómális lokalizációját, és egymáshoz viszonyított sorrendjüket meg tudjuk határozni. A géntérképezés során a gének egymástól való távolságát, a genomban való viszonylagos elhelyezkedésüket határozzuk meg anélkül, hogy valóban megszekvenálnánk a DNS-t. Ezt oly módon érik el, hogy az együtt öröklődő tulajdonságok (koszegregáció) gyakoriságát figyelik, tehát a Mendeli független öröklődéstől való eltérést, és ebből következtetnek a gének egymástól való távolságára a kromoszómán. Amint készen áll a térkép, képesek vagyunk egy ismert gén vagy marker alapján megmondani egy másik gén helyét a genomban.
31 Rekombináció Molekuláris rekombináció: DNS törése és újraegyesülése (nem feltétlenül homológ szakaszoké) Genetikai rekombináció: új (nem-szülői) allélkombinációk létrejötte, mely a meiózis profázisában történő nem testvér kromatidák közötti molekuláris rekombináció eredménye (crossing over)
32 Két tulajdonság együttes öröklődésmenete P1 (AABB) x P2 (aabb) F1 AaBb (AB ab haplotípus: kapcsolt allélek sora) nem rekombináns nem rekombináns rekombináns F1 gamétái: AB Ab ab ab ha A és B független ha A és B kapcsolt ha szorosan kapcsoltak ha teljesen kapcsoltak
33 Testcross rekombináció gyakorisága A testcross megmutatja az F1 szülőben keletkező 4-féle gaméta arányait, mert a tester szülő mindig ab-t ad. F1 x tester: AaBbx aabb utódok: AaBb Aabb aabb aabb(4 kül fenotípus)
34 Rekombináció gyakorisága közeli és távoli gének esetén
35 Térképtávolság r = rekombináns utódok gyakorisága rekombináns gaméták száma/ összes gaméta d = térkép távolság Kis távolságok (10 cm) esetén d=r A gének közötti, rekombinációs gyakoriság felhasználásával számolható a térképtávolság. Mértékegysége a centimorgan (cm). Egy cm távolság van két gén között, ahol a gaméták 1%-a rekombináns, vagyis a szülőitől eltérő kombinációt hordoz.
36
37 Morgan d = r (r 0,5) Használatos függvények Haldane-függvény C=1 (tfh. nincs interferencia) d = -½ ln(1-2r) r = ½ (1-e-2d) Kosambi-függvény C=2r (ha r=0,5 => nincs interferencia, ha r=0 => teljes interferencia) d = ¼ ln[(1+2r)/(1-2r)] r= ½ e4d-1/e4d+1
38 Marker kapcsoltsági térképek Számítógépes térképező programok marker térkép készítés (MAPMAKER, GMENDEL, JOINMAP) A LOD - dal kapott r (rekombinációs) gyakoriságokat (megfelelő lötyögéssel) a térképező programok a legvalószínűbb géntérképekké rakják össze. Az r értékekből levezetett d értékeket (Haldane függvény) a kétfaktoros térképezési logika (additivitások) alapján rendezik.
39 Marker kapcsoltsági térkép elkészítése 8> make chromosome chrom2h chrom4h chrom5h chromosomes defined: chrom2h chrom4h chrom5h 9> seq hvm36 bmag125 sequence #1= hvm36 bmag125 10> anchor chrom2h HvM36 - anchor locus on chrom2h Bmag125 - anchor locus on chrom2h chromosome chrom2h anchor(s): HvM36 Bmag125
40 Marker kapcsoltsági térkép elkészítése 8> make chromosome chrom2h chrom4h chrom5h chromosomes 16> assign defined: chrom2h chrom4h chrom5h HvM36 - anchor locus on chrom2h...cannot re-assign 9> seq hvm36 Bmag125 bmag125 - anchor locus on chrom2h...cannot re-assign Bmac30 - anchor locus on chrom4h...cannot re-assign sequence HvM67 #1= hvm36 - bmag125 anchor locus on chrom4h...cannot re-assign Bmag337 - anchor locus on chrom5h...cannot re-assign 10> anchor ABG702 chrom2h - anchor locus on chrom5h...cannot re-assign HvM36 ABG8 - anchor locus - assigned on chrom2h to chrom2h at LOD 20.1 Bmag125 ABC311 - anchor locus - assigned on chrom2h to chrom2h at LOD 24.4 chromosome OPH121 chrom2h - assigned anchor(s): to HvM36 chrom2h Bmag125 at LOD 28.6 OPB162 - assigned to chrom2h at LOD 23.8 ABG318 - assigned to chrom2h at LOD 22.9 ABG358 - assigned to chrom2h at LOD 13.8 ABG459 - assigned to chrom2h at LOD 24.4
41 Marker kapcsoltsági térkép elkészítése 8> make chromosome chrom2h chrom4h chrom5h chromosomes 16> assign defined: chrom2h chrom4h chrom5h HvM36 18> - three anchor point locus on chrom2h...cannot re-assign 9> seq hvm36 Bmag125 bmag125 - anchor locus on chrom2h...cannot re-assign Bmac30 - anchor locus on chrom4h...cannot re-assign sequence HvM67 #1= hvm36 bmag125 'triple - anchor error locus detection' chrom4h...cannot is on. re-assign Bmag337 - anchor locus on chrom5h...cannot re-assign 10> anchor ABG702 chrom2h - anchor locus on chrom5h...cannot re-assign HvM36 ABG8 - anchor count locus - assigned on markers chrom2h to chrom2h at LOD 20.1 Bmag125 ABC311 - anchor locus - assigned on chrom2h to chrom2h at LOD 24.4 chromosome OPH121 chrom2h - assigned anchor(s): to HvM36 chrom2h Bmag125 at LOD 28.6 OPB162 - assigned to chrom2h at LOD 23.8 ABG318 - assigned to chrom2h at LOD 22.9 ABG358 - assigned to chrom2h at LOD 13.8 ABG459 - assigned to chrom2h at LOD 24.4 Linkage Groups at min LOD 3.00, max Distance 37.2 Triplet criteria: LOD 3.00, Max-Dist 37.2, #Linkages 2 counting linked triplets in 2 linkage groups log-likelihood differences a-b-c b-a-c a-c-b 1: ABG8 ABC311 OPH : ABG8 ABC311 HvM : ABG8 ABC311 OPB : ABG8 ABC311 ABG : ABG8 ABC311 ABG : ABG8 ABC311 ABG : ABG8 ABC311 Bmc222a : ABG8 ABC311 OPP102b : ABG8 ABC311 OPA : ABG8 ABC311 OPQ
42 Marker kapcsoltsági térkép elkészítése 8> make chromosome chrom2h chrom4h chrom5h chromosomes 16> assign defined: chrom2h chrom4h chrom5h HvM36 18> - three anchor point locus on chrom2h...cannot re-assign 9> seq hvm36 Bmag125 bmag125 - anchor locus on chrom2h...cannot re-assign Bmac30 - Placing anchor at locus log-likelihood on chrom4h...cannot threshold re-assign sequence HvM67 #1= hvm36 bmag125 'triple - Start: anchor error 9 locus detection' chrom4h...cannot is on. re-assign Npt-2: (10) Bmag337 - No anchor unique placements locus on chrom5h...cannot for 6 remaining markers re-assign 10> anchor ABG702 chrom2h - anchor locus on chrom5h...cannot re-assign HvM36 ABG8 - anchor count locus Map: - assigned on markers chrom2h to chrom2h at LOD 20.1 Markers Distance Bmag125 ABC311 - anchor locus - assigned 9 on Bmc222a chrom2h to chrom2h 21.5 cm at LOD 24.4 chromosome OPH121 chrom2h - assigned anchor(s): 5 OPB162 to HvM36 chrom2h 8.2 Bmag125 cm at LOD ABG cm OPB162 - assigned 10 OPP102b to chrom2h 6.3 cm at LOD 23.8 ABG318 - assigned 11 OPA192 to chrom2h 10.6 cm at LOD 22.9 ABG358 - assigned 12 OPQ10 to chrom2h at LOD 13.8 ABG459 - assigned to chrom2h at LOD 24.4 Linkage Groups at min LOD 3.00, max Distance 37.2 Triplet criteria: LOD 3.00, Max-Dist 37.2, #Linkages 2 counting linked triplets in 2 linkage groups log-likelihood differences a-b-c b-a-c a-c-b 1: ABG8 ABC311 OPH : ABG8 ABC311 HvM : ABG8 ABC311 OPB : ABG8 ABC311 ABG : ABG8 ABC311 ABG : ABG8 ABC311 ABG : ABG8 ABC Bmc222a cm 6 markers 0.00 log-likelihood= : ABG8 ABC311 OPP102b Markers placed relative to above map: 9: ABG8 ABC OPA : ABG8 ABC311 OPQ :-21-:--8-:-11-:--6-:-11-: 8 2.*.:.**.:...:...:...:...: *.:.**.:...:...:...:...: :.**.:..*.:...:...:...: :..*.:.**.:...:...:...: :...:.**.:..*.:...:...: :...:.**.:..*.:...:...: Placing at log-likelihood threshold Start: Npt-4: 9 5 (2) Npt-4: 9 5 (4) Npt-4: (3)
43 Marker kapcsoltsági térkép elkészítése 8> make chromosome chrom2h chrom4h chrom5h chromosomes 16> assign defined: chrom2h chrom2h framework: chrom4h chrom5h HvM36 18> - three anchor point locus on chrom2h...cannot re-assign 9> seq hvm36 Bmag125 bmag125 - anchor locus on chrom2h...cannot re-assign Bmac30 - Placing anchor at locus log-likelihood on chrom4h...cannot threshold re-assign sequence #1= hvm36 Markers HvM67 bmag125 'triple - Start: anchor error 9 locus detection' Distance 12 chrom4h...cannot is on. re-assign Npt-2: (10) Bmag No anchor ABG358 unique placements locus on 20.6 chrom5h...cannot for cm 6 remaining markers re-assign 10> anchor ABG702 chrom2h - anchor locus on chrom5h...cannot re-assign HvM36 ABG8 - anchor count locus - 5 Map: assigned OPB162 on markers chrom2h to chrom2h 2.4 cm at LOD 20.1 Markers Distance Bmag125 ABC311 - anchor locus 4 - assigned HvM36 9 on Bmc222a chrom2h to chrom2h cm cm at LOD 24.4 chromosome OPH121 chrom2h - assigned anchor(s): 5 OPB162 to HvM36 chrom2h 8.2 Bmag125 cm at LOD 28.6 OPB162-3 assigned OPH121 1 ABG8 to chrom2h cm cm 10 OPP102b 6.3 cm at LOD 23.8 ABG assigned ABC311 OPA192 to chrom2h cm cm at LOD 22.9 ABG358 - assigned 12 OPQ10 to chrom2h at LOD 13.8 ABG459 - assigned to chrom2h at LOD 24.4 Linkage Groups at min LOD 3.00, max Distance 37.2 Triplet criteria: LOD 3.00, Max-Dist 37.2, #Linkages 2 counting linked triplets in 2 linkage groups log-likelihood differences a-b-c b-a-c a-c-b 1: ABG8 ABC311 OPH : ABG8 ABC311 HvM : ABG8 ABC311 OPB : ABG8 ABC311 ABG : ABG8 ABC311 ABG : ABG8 ABC311 ABG : ABC Bmc222a cm 6 markers 0.00 log-likelihood= ABG cm 8: ABG8 ABC311 OPP102b Markers placed relative to above map: 9: 10 ABG8 OPP102b ABC OPA192 cm : ABG8 ABC311 OPQ OPA cm 12 OPQ :-21-:--8-:-11-:--6-:-11-: 8 2.*.:.**.:...:...:...:...: *.:.**.:...:...:...:...: :.**.:..*.:...:...:...: :..*.:.**.:...:...:...: :...:.**.:..*.:...:...: cm 9 markers :...:.**.:..*.:...:...: Placing at log-likelihood threshold Start: log-likelihood= Npt-4: 9 5 (2) Npt-4: 9 5 (4) Npt-4: (3)
44 DK1DK2DK3DK4DK7DK8DK9 DK10 DK11 DK12 DK13 DK14 DK15 DK16 DK17 DK18 DK19 DK20 DK21 DK22 DK24 DK25 DK26 DK27 DK28 DK29 DK30 DK31 DK32 DK35 DK36 DK37 DK38 DK39 *mst102 A B A A A B B B A A B B A B B B A B B B A B B A B B A A B A A B A B *Bmac144d A B A A A B B B A B B A B B B B B B B B A B B A B B A A B A A B A B TCGA96 A B A A A B B B A B B A B B B A B B B B A B B A B B A A B A A B A B *MWG938 A B A A B B B B A B B A B B B A B B B B A B B A B B A A B A A B A B AGAT166/162 A B A A B B B B A B B A B B B A B B B B A B B A B B A A B A A B A B GCTC58 A B A A B B B B A B B A B B B A B A B B A B B A B B A A B A B B B B OPK16 A B A A B B B B A B B A B B B A B A B B A B B A B B A A B A B B B B *abc156.1 A B A A B B B B A B B A B B B A B A B B A B B A B B A A B A B B B B *BMAC213 A B A A B B B B A B B A B B B A B A B B A B B A B B A A B A B B B B GAAT76 A B A A B B B B A B B A B B B A B A B B A B B A B B A A B A B A B B *OPE171 A B A A B B B B A B B - B B B A B A B B A B B A B B B A B A B A B B GAAT414 A B A B B B B B A B B A B B B A B A B B A B B A B B B A B A B A B A *OPO18 A B A B B B B B A B B A B B B A B A B B A B B A B B B A B A B A B A *OPB16-1 A B A B B B B B A A B A B B B A B A A A A B B A B B B A B A B A B A *Bmag211 A B A B B B B B B A B A B B B A B A A A A B B A B B B A B A B A B A *OPS16 A B A B B B B B B A B A B B B A B A A A A B B A B B A A B A B A B A GCGA428 A B A B B B B B B A B A B B B A B A A A A B B A B B A A B A B A B A *Ebmac560a A B A B B B B B B A B A B B B A B A A A A B B A B B A A B A B A B A *OPB41 A B A B B B B B B A B A B B B A B A A A A B B A B B A A B A B A B A GAAT550 A B A B B B B B B A B A B B B A B A A A A B B A B B A A B A B A B A AGGT290 A B A B B B B B B A B A B B B B B A A A A B B A B B A A B A B A B A TCGA119 A B A B B B B B B A B A B B B B B A A A A B B A B B A A B A B A B A *OPT152 A B A B B B B B B A B A B B B B B A A A A B B A B B A A B A B A B A GAAT510 A B A B B B B B B A B A B B B B B A A B A B A A A A A B A A B B A A AGAT51 A B A B A A A B B A B A B B B B B B A B B B A A A A A B A A A B A B *Bmac144a A B A B A A A A B A B A B B B B B B A B B B A A A A A B A A A B A B GCGA269/270 A B A B A A A A B A B A B B B B B B A B B B A A A A A B A A A B A B *ABC152b A B A B A A A A B A B A B B B B B B A B B B A A A A A B A A A B A B *HvHVA1 A B B B A A A A B A B A B B B B B B A B B B B A A A A B A A A B B B *BCD265c A B B B A A A A B A B A B B B B B B A B B B B A A A A B A A A B B B GCGT132 A B B B A A A A B A B A B B B B B B B B B B B A A2012. A október A B 3. A A A B B B *OPP142 A B B B A A A A B A B A B B B B B B B B B B B A AMészáros A A Klára B A A A B B A
45 mst102 0 Bmac144d 4 TCGA96 6 MWG938 8 AGAT GCTC58 15 OPK16 17 abc Bmac GAAT76 24 OPE GAAT OPO18 31 OPB Bmag OPS16 50 GCGA Ebmac560a 54 OPB GAAT AGGT TCGA OPT GAAT AGAT51 99 Bmac144a 100 GCGA ABC152b 109 HvHVA1 125 BCD265c 126 GCGT OPP OPX H Bmac222a 0 ABG459 1 ABG HvM36 25 ABC GAGT ABG8 31 GCGA52 37 GAGA72 39 TCTC GAAT OPA AGAT AGAT GAGA GAGT62 58 GCGT abg459a 101 Bmac Bmac144b 108 bcd175b 109 Bmag GAGT TCGT TCAT TCTC Bmag003c 127 GCAT OPG H OPD07 0 OPS141 6 OPO GATC GCGA TCAT GAGA TCTC AGTC GAAT GCAT OPI AGAT OPS Bmag384b 75 TCTC GCGT OPK HvM60 99 AGAT AGAT GAAT AGGT GCGT OPH ABG4 152 TCAT Bmag GCAT TCTC OPS Hvm GAGA OPH Hvm GCGT TCAT GCAT GCGA TCGA ABC AGTC GAAT H HvPhyA 0 GCAT76 8 TCAT300 TCAT77 9 AGAT HvPhyB 21 CAB 22 Bmac30 24 AGAT Bmag Bmag173a 33 Bmac OPJ15 40 TCAT OPU AGAT46 59 ABG abg54 64 AGAT GCGT GCAT AGAT HvM67 85 OPS VRN-H2 HvSnf2 92 AGGT Hdamyb 100 4H OPT151 0 GCAT50 7 Bmag136b 8 GAGA556 9 Bmag GATC AGTC AGGA GAGA AGAT42 32 GAGA58 33 Bmag abc AGAT TCTC Bmag223 mr 41 GATC abg459b 51 OPH GCGT GCGA TCAT abc306d 90 OPK TCTC ABG AGTC Bmag381b 112 gms HvPhyC VRN-H1 117 abc TCGT GCAT ABC H BMAC316 0 AGGT256 2 TCTC386 9 AGAT ABC152c 23 Bmag Bmag21a 51 GAGA GCGT Bmag OPH Bmag HvCry1a HvCry2 GCGA48 71 AGGA62 72 OPI OPA abc306b 80 abc306c 81 abc AGGT Bmag003b 89 ebmac TCTC OPT14 95 GCTC GAGA AGGT TCAT GCAT AGAT wg464a GAGA GAGA TCTC TCAT GCGT H Hvm4 0 Bmac222b 9 GCAT TCAT TCTC abg TCTC abc152a 16 GCGT69 25 AGTC TCAT Bmag21c 32 GCAT Ebmac TCGT TCGA VRN-H3 50 KSuA1a 52 abc306a 59 TCGT TCGT64 62 AGGT64 65 OPE Bmac TCGT Bmag OPR OPS mst101b 88 Bmag AGAT AGAT GCGT OPQ TCTC OPB AGGT GCTC GAAT H B1 B3 B7 B9 B10 B12 B13 B5 B3 B3 B7 B7 B8 B15 B14 B16 B18 B19 B4 B5 B6 B1 0 B1 2 B1 4 B4 B5 B7 B11 B13 B18 B1 B3 B4 B8 B1 0 B1 2 B3 B6 B5 B9 B12 B10 Dicktoo Kompolti korai marker térkép (246 lókusz)
46 Oregon Wolfe Barley Map Centromere Morphological marker ABG Bmag ABG HVCMA 44.3 Bmac X Bmac047B 65.9 DAK MWG nud 81.6 lks Bmag WG380B Ris ABC ABG461A WG380A X HVM ThA (7H) ABG DAK ABG X Pox 42.4 MWG949B 55.8 Hot EBmac ABG X Bmag0113E 77.9 Bmac0144F 84.1 vrs Bmag MWG MWG844E X KFP MWG882A ABG EBmac cnx zeo X MWG X wst X MWG949A (2H) BCD ABC171A 26.4 X ABG alm 62.2 Bmac ABC BCD Bmac Bmag HVM X ABG X Pub MWG HVM ABC ABC (3H) MWG MWG HVM CDO CDO hvknox Dhn ABC HVM X Bmag X Bmac ABG X KFP MWG652B EBmac X Hsh HVM X ABG (4H) Rh 0.0 Act8A 8.8 kazmil 15.6 MWG837B 21.2 MWG837A 24.5 Bmac X BCD X Bmag ABG X ABC Bmac0144A 85.0 Bmag0113A 88.3 MWG706A 98.2 Blp MWG ABC X WMC1E MWG ABG387A (1H) Bmac MWG MWG652A 28.8 MWG602B 33.0 X ABG X HVM rob 70.5 Bmag X Bmac0218C 88.1 ABG MWG X MWG Tef X Bmac X (6H) MWG ABC ABG ABG Bmac0273B 38.2 Bmac KFP ABC srh 88.3 Bmag0113D ABG003B MWG X ABG ABG ABC Bmag0113C MWG602A (5H)
47 Marker kapcsoltsági térképek alkalmazási területei Összehasonlító térképezés fajon belül különböző populációk között, rokon fajok között Genom szerveződés makro és mikro kolinearitás kromoszóma átrendeződések intergénikus és génklaszter szakaszok azonosítása
48 Egyedi térképek fajra jellemző konszenzus térkép Stein et al. TAG(2007)114:823
49 A búza kromoszómák BIN térképe Qi et al. Genetics 2004
50 P92201D5-2/P91193D1-10 Ajana/WAWHT2074 Cadoux/Reeves EGA Blanco/Millewa B Marker kapcsoltsági térkép kromoszóma fizikai térképe wpt-5195* wpt-0643 wpt-6575 wpt-5587 wpt-3459 wpt-5934 wpt-6627* wpt-4916* wpt-0100* wpt-8235* gwm614b* wpt-8404* wpt-1041* wmc25* gwm319* wpt-3569* wpt-1650* wpt-1068* wpt-7937* wpt-0775* wpt-8294* wpt-0395* wpt-1494* wpt-2249* wpt-2907* wpt-0434* wpt-9131* barc183* wpt-0615* wpt-1127* wpt-5440* wpt-6144* wpt-0079* gwm630* wpt-9350* cfd56* AF112966* wpt-7625* gwm47* wmc477* gwm120* wpt-3109* wpt-9336* wpt-7350* wpt-4701 wpt-2266* gwm526c* barc1147* barc B gwm614 wpt-1663 wpt-5567 wpt-4527 cfd238 wmc25a barc318 wpt-9423 wpt-9402 wpt-1489 wpt-8072 wpt-4301 wpt-0462 wpt-3561 wpt-6932 wpt-3983 wpt-5707 wpt-2314 wpt-6199 wpt-8583 wpt-6477 wpt-4125 wpt-9644 wpt-7757 wpt-0408 wpt-0615 wpt-5672 wpt-8492 barc183b* gwm46 barc7 barc55 wmc474 cfa2278 wpt-0335 wpt-0709 wpt-7750 gwm374 gwm319 gwm630b gwm271 gdm14a gwm120a gwm191a* wpt-1140* wpt-7200 wpt-3132 wpt-0950 wpt-7404 wpt-5128* gwm120b wpt-4199 gdm61* B wpt-4527 cfd238 wpt-8004 wpt-8326 wpt-9423 wpt-9402 wpt-1489 wpt-8072 wpt-5707 wpt-3983 wpt-4997 gwm429 barc13 wpt-6477 wpt-0615 wpt-7750 wpt-2430* wpt-8569* B wpt-6575* wpt-5587* wpt-3459* wpt-5934* wpt-6970* wpt-6627* wpt-5195* wpt-0643* wpt-6805* wpt-0100* wpt-8760* wpt-2410* wpt-3565* gwm614* wpt-6223* barc35* barc297a* wpt-2106* wpt-8004* wpt-8326* wmc154 wpt-5374 wpt-3561 wpt-5707 wpt-7757 wpt-5672 wpt-5556 wpt-4125 barc55 wpt-6278 barc1114 cfa2043b gwm120 wpt-4199* wpt-2430 wpt-8569 wpt-0094 wpt-7200 wpt-3132 wpt-8460 wpt-5680 wpt-5242 wpt-0694 stm509acag wpt-4701 wpt-7004 wpt-4210 wpt-0047 barc1147 wpt-2135 wpt-3378 barc159 wpt-4559 wpt-6970 wpt-7123 wpt-9288 wpt-6311 wpt-8737 wpt-4916 wpt-9423 wpt-5587 wpt-5934 wpt-6575 wpt-9859 wpt-7995 wpt-9220 wpt-3459 wpt-3188 wpt-1549 wpt-1549 wpt-2249 wpt-2397 wpt-3807 wpt-5287 wpt-1650 wpt-8074 wpt-0408 wpt-0615 wpt-1489 wpt-1494 wpt-3188 wpt-3983 wpt-4701 wpt-4737 wpt-4997 wpt-5249 wpt-5556 wpt-5597 wpt-7348 wpt-7610 wpt-8072 wpt-8294 wpt-8460 wpt-8720 wpt-9402 wpt-6144 wpt-6053 wpt-6627 wpt-7408 wpt-7757 wpt-8070 wpt BS-3 2BS-1 2BL-6 Chromosome 2B
51 Térképegység (cm) és megabázis (Mb) 1 cm 1Mb 1 Mb = 106bázis A rekombinációs gyakoriság változó genomon belül is, ezt jellemzi a rekombinációs ráta, amely 1 körül változik. Vannak rekombinációs hot-spotok, míg máshol, pl. a centroméra környékén szinte nincs rekombináció.
52 Marker kapcsoltsági térképek alkalmazási területei Mennyiségi tulajdonságok lókuszainak (QTL) elemzése lókuszok számának és helyének azonosítása Adott lókusz szerepének elemzése; QTL dinamika, tulajdonság komponensek vizsgálata Gének azonosítása, fenotípusos hatásuk vizsgálata finom térképezés, pozicionális klónozás modell növény ismert génszekvenciája alapján Allél gyakoriságok vizsgálata szélesebb genetikai bázison Marker szelekció
53 QTL elemzés fő módszerei Sigle-marker analízis: nincs szükség kapcsoltsági térképre. Egy marker és az adott tulajdonság kapcsolatát vizsgálja A linearis regresszió determinációs koefficiense (R2) megadja, hogy az adott marker a fenotípusos variancia hány százalékét magyarázza. Hátránya: a marker és a QTL közti rekombináció esetén alulbecsli a kapcsoltságot Nagy populáció méret csökkenti ezt a hatást Single intervall mapping (SIM): Két szomszédos marker közötti intervallumot vizsgál az egész kromoszóma mentén. Kiküszöböli a rekombinációból származó torzítást. Composit interval mappiung (CIM): egyesíti az intervallum térképezést a lineáris regresszió analízissel és a szomszédos markerpárokat további markerekkel egészíti ki.
54 Adott tulajdonsággal kapcsolt marker
55 QTL elemzés marker kapcsoltsági térkép alkalmazásával QTL azonosítás a teljes marker kapcsoltsági térképre kiterjedve Markerek közötti intervallumok vizsgálata Az adott QTL hatásának nagyságát a LOD (likelihood ratio) értékkel jellemezzük LOD= lg(qtl csúcs jelenlétének valószínűsége/annak a valószínűsége, hogy nincs QTL hatás) LOD küszöbérték: genom méret marker típus és sűrűség populáció típusa és méret Fenotípusos vizsgálatok ismételhetőség, pontosság Fenomika
56 QTL elemzés marker kapcsoltsági térkép alkalmazásával Fenotípusos jellemzés Hasadó populáció Genotípusos jellemzés szülők AAABBAB ABBABABB BBBAA A B Adat mátrix *mst1011 ABAAABBBABBBABBBBBABBABBA *mst1012 ABBAABAABBAABBBBBBABBBBBB *mst102 AAABBBAABBABBBABBBBABBABB *wg4642 ABBAABAABBAABBBBBBABBBABB *bcd1752 ABBAAABBBA---ABBABBBABABAA *wg541 AA-BBBA---ABBABBABBABABAABA *v8h *uv16h *v16h *uv24h
57 QTL elemzés marker kapcsoltsági térkép alkalmazásával ============================================================= QTL-Map for peak 3: Confidence Interval: Left Boundary= OPS31-Snf Right Boundary= Snf2-Hdamyb (off end) INTERVAL LENGTH QTL-POS GENETICS WEIGHT Snf2-Hdamyb free chi^2= (1 D.F.) log-likelihood= mean= sigma^2= variance-explained= 93.8% =============================================================
58 QTL elemzés marker kapcsoltsági térkép alkalmazásával cm Weight R 2 LOD OPJ15-ABG cm % % % % * % ** ============================================================= % ** % *** QTL-Map 14.0 for peak 3: 37.8% *** Confidence Interval: 38.2% Left Boundary= **** OPS31-Snf % **** Right Boundary= Snf2-Hdamyb (off end) % **** % **** % **** INTERVAL % LENGTH QTL-POS *** GENETICS WEIGHT Snf2-Hdamyb % *** free ABG366-abg cm % *** % *** chi^2= (1 29.1% D.F.) log-likelihood= ** mean= sigma^2= variance-explained= abg54-opr % cm % ============================================================= % % 1.992
59 LOD values QTL elemzés marker kapcsoltsági térkép alkalmazásával cm Weight R 2 LOD OPJ15-ABG cm % % H25.0% H 5H % * % ** ============================================================= % ** % *** QTL-Map 14.0 for peak 3: 37.8% *** Confidence Interval: 38.2% Left Boundary= **** OPS31-Snf % **** Right Boundary= Snf2-Hdamyb (off end) % **** % **** % **** 30 INTERVAL % LENGTH QTL-POS *** GENETICS WEIGHT Snf2-Hdamyb % *** free ABG366-abg cm % *** % *** chi^2= (1 D.F.) log-likelihood= % ** mean= sigma^2= variance-explained= abg54-opr % cm % recombination distance (cm) ============================================================= % % HD16h uv HD16h vern HD24h uv HD24h vern
60 DH vonalak száma Kalászolási idő, mint vizsgált tulajdonság Populáció egyedei közti változékonyság Kompolti X átl =77 nap Dicktoo 16h + hő ciklus Kalás zolás i idő
61 Dicktoo x Kompolti korai DH vonalak HvSnf2 ZCCTH 4H árpa kromoszóma 0 HvPhyA 8 GCAT76 9 TCAT300 TCAT77 16 AGAT HvPhyB 22 CAB 24 Bmac30 29 AGAT Bmag Bmag173a 39 Bmac OPJ15 41 TCAT OPU AGAT46 62 ABG abg54 65 AGAT GCGT GCAT AGAT HvM67 86 OPS VRN-H2 HvSnf2 AGGT650 Hdamyb QTL elemzés vernalizálatlan vernalizált kezelés
62 Pozicionális klónozás Alapja: a QTL hatás 1 cm-nál kisebb marker intervallumhoz köthető Finomtérképezés, nagy populáció méret bevonásával A génhez közeli rekombinációt hordozó egyedek azonosítása, fenotípusos jellemzése A kromoszóma régióhoz köthető átfedő BAC klónok azonosítása, térképezése A keresett gént tartalmazó BAC klón szekvenálása A BAC klónon talált gén(ek) funkciójának tisztázása Gén expresszió transzformáció
63 Pozicionális klónozás A kalászos gabonafélék vernalizációs igényéért felelős VRN-2 gén azonosításának menete Yan et al., Science 303(2004):
64 Pozicionális klónozás A kalászos gabonafélék vernalizációs igényéért felelős VRN-2 gén azonosításának menete Yan et al., Science 303(2004):
65 Pozicionális klónozás A kalászos gabonafélék vernalizációs igényéért felelős VRN-2 gén azonosításának menete Yan et al., Science 303(2004):
66 Deléciós vonalak alkalmazása búza fagytűrésének vizsgálatában
67 Gén azonosítás főbb módszerei Marker kapcsoltsági térképek Két szülős térképező populációk Széles genetikai bázist képviselő fajtakör Jelölt gén megközelítése Genom pozíció függő stratégiák: pozicionális klónozás, deléciós vonalak Összehasonlító genomikai stratégiák: modell növények Mesterséges genetikai variáció (indukált mutációs populációk) Gén csapdázás: T-DNS, transzpozon Gén expressziós mintázatok elemzése: Differential Display DNS microarray, DNS chip
68 Széles genetikai bázisú fajtakör asszociációs genetika Hagyományos két-szülős térképező populációk hátrányai: Előállításuk idő és munka igényes Lókuszonként csak két allél hatásának vizsgálatára van mód A két szülő közti hasonlóság korlátozza az egyes gének azonosítási lehetőségeit Adott populációban azonosított QTL csúcshoz szorosan kapcsolt marker nem polimorf a nemesítési anyagban Gén (allél) kölcsönhatások vizsgálata limitált
69 Gén azonosítás főbb módszerei Marker kapcsoltsági térképek Két szülős térképező populációk Széles genetikai bázist képviselő fajtakör Jelölt gén megközelítése Genom pozíció függő stratégiák: pozicionális klónozás, deléciós vonalak Összehasonlító genomikai stratégiák: modell növények Mesterséges genetikai variáció (indukált mutációs populációk) Gén csapdázás: T-DNS, transzpozon Gén expressziós mintázatok elemzése: Differential Display DNS microarray, DNS chip
70 Összehasonlító genomikai stratégiák Ortológ lokusz: Különböző fajokban ugyanabból az ősi lokuszból származó hasonló szekvenciájú és funkciójú géneket hordozó lokusz. Paralóg lokusz: Ősi lokusz duplikációjából származó lokusz. Szinténia: Két különböző fajban a gének és markerek hasonló sorrendje. Mikroszinténia: Szekvencia szintű szinténia. A homológ szekvenciájú gének hasonló sorrendje, szekvenciája és orientációja a genomban.
71 Gu et al Plant Phys
72 Stein et al. TAG(2007)114:823
73 Cockram et al. JEXB(2007)58:1231
74 Gén azonosítás főbb módszerei Marker kapcsoltsági térképek Két szülős térképező populációk Széles genetikai bázist képviselő fajtakör Jelölt gén megközelítése Genom pozíció függő stratégiák: pozicionális klónozás, deléciós vonalak Összehasonlító genomikai stratégiák: modell növények Mesterséges genetikai variáció (indukált mutációs populációk) Gén csapdázás: T-DNS, transzpozon Gén expressziós mintázatok elemzése: Differential Display DNS microarray, DNS chip
75 Mutáció A mutáció az örökítő anyag spontán, maradandó megváltozása, amelynek során új tulajdonság keletkezik. A Hugo De Vries által a múlt század elején bevezetett fogalmat ma szűkebb értelemben a génen belüli bázissorrend-változásra vonatkoztatjuk. Tágabb értelemben mutációnak tekinthető a kromoszómaszerkezetben, ill. a kromoszómák számában bekövetkezett minden változás, ideértve a poliploidiát is.
76 A génmutációk (pontmutációk) egy gén bázissorendjének változását jelentik. Ennek hatása ritkán jelenik meg azonnal a fenotípusban, csak ha domináns a mutáció. Bázis csere (tranzicio, transzverzió) Keret eltolódás (Frameshift) Hatásuk: same sence azonos aminósav missense aminósav csere nonsense stop kodon kialakulása Kromoszómamutáció: A kromoszóma egyes részei változnak meg. Ez lehet szerkezeti változás, például a kromoszómák törlődése (deléció), megfordulása (inverzió) kicserélődése (transzlokáció), megduplázódása (duplikáció). Másrészt lehet számbeli, melyen belül megkülönböztetünk poliploidiát (kromoszómagarnitúra a normális egész számú többszöröse) és aneuploidiát (csak egy kromoszómánál van eltérés).
77 Mutagenezis kémiai (EMS, ENU, DEB, HNO 2, ) besugárzás (R, a-g, gyors neutron, UV) inszerció [T-DNS és/v. (retro)transzpozon] kimérikus (RNS-DNS) oligonukleotidok véletlen, azonosítás?? letalitás géncsendesítés (antiszensz, RNSi)
78 TILLING (Targeting Induced Local Lesions IN Genomes) (EMS) mutagenezis + genomika = HTP azonosítás Szelekció a genotípus alapján ( reverz genetika) Lépései mutáns populáció előállítása HTP mutáns azonosítás értékelés
79 TILLING mutáns populáció előállítása
80 TILLING mutáns populáció előállítása
81 TILLING Mutáció kimutatása
82 TILLING a módszer Heteroduplex formációk
83 TILLING a módszer A mutációk kimutatása IR 700 IR 800 IR 700 IR 800
84 TILLING a módszer LI-COR 4300 DNA Analyzer
85 TILLING Elválasztás IR 700 IR 800
86 TILLING
87 ECOTILLING TILLING a természetben létező ökotípusokkal vagy populációkkal és TILLING mesterségesen fenntartott genetikai forrásokkal, pl. fajta- és mutánsgyűjtemények, izogén vonalak (NIL), hasadó és térképezési populációk (BSA), fajtajelöltek,...
88 ecotilling
89 TILLING alkalmazások allél(sorozatok) azonosítása (funkc. genomika, SNP) többszörös mutánsok azonosítása HTP szelekció (MAS, GAS, SAS ) pedigrévizsgálat rokonsági körök igazolása (kukorica) (fejlődés)rendszertan genetikai térképezés (génizolálás)
90 Ajánlott irodalom Balázs E. és Duduts D.: Molekuláris növénybiológia Dudits D., Heszky L.: Növényi biotechnológia és géntechnológia Heszky L., Fésüs L., Hornok L.: Mezőgazdasági biotechnológia B.C.Y. Collard1,4,, M.Z.Z. Jahufer2, J.B. Brouwer3 & E.C.K. Pang (2005) An introduction to markers, quantitative trait loci (QTL) mapping and marker-assisted selection for crop improvement: The basic concepts. Euphytica 142: DOI: /s
91 Köszönöm a figyelmet!
Genetikai térképezés, gén azonosítás Mészáros Klára, Karsai Ildikó meszarosk@mail.mgki.hu
 Genetikai térképezés, gén azonosítás, Karsai Ildikó meszarosk@mail.mgki.hu Növénynemesítés feladatai Termésbiztonság növelése, nagy szezonális szélsőségek csökkentése Biotikus és abiotikus stresszrezisztencia
Genetikai térképezés, gén azonosítás, Karsai Ildikó meszarosk@mail.mgki.hu Növénynemesítés feladatai Termésbiztonság növelése, nagy szezonális szélsőségek csökkentése Biotikus és abiotikus stresszrezisztencia
Gén térképezés, génizolálás Dr. Karsai Ildikó, tudományos tanácsadó karsai.ildiko@agrar.mta.hu
 Gén térképezés, génizolálás Dr. Karsai Ildikó, tudományos tanácsadó karsai.ildiko@agrar.mta.hu Karsai Ildikó Gén azonosítás tulajdonságok fenotípusos megnyilvánulásában meghatározó szerepet játszó genetikai
Gén térképezés, génizolálás Dr. Karsai Ildikó, tudományos tanácsadó karsai.ildiko@agrar.mta.hu Karsai Ildikó Gén azonosítás tulajdonságok fenotípusos megnyilvánulásában meghatározó szerepet játszó genetikai
Többgénes jellegek. 1. Klasszikus (poligénes) mennyiségi jellegek. 2.Szinte minden jelleg több gén irányítása alatt áll
 Többgénes jellegek Többgénes jellegek 1. 1. Klasszikus (poligénes) mennyiségi jellegek Multifaktoriális jellegek: több gén és a környezet által meghatározott jellegek 2.Szinte minden jelleg több gén irányítása
Többgénes jellegek Többgénes jellegek 1. 1. Klasszikus (poligénes) mennyiségi jellegek Multifaktoriális jellegek: több gén és a környezet által meghatározott jellegek 2.Szinte minden jelleg több gén irányítása
DNS-szekvencia meghatározás
 DNS-szekvencia meghatározás Gilbert 1980 (1958) Sanger 3-1 A DNS-polimerázok jellemzői 5'-3' polimeráz aktivitás 5'-3' exonukleáz 3'-5' exonukleáz aktivitás Az új szál szintéziséhez kell: templát DNS primer
DNS-szekvencia meghatározás Gilbert 1980 (1958) Sanger 3-1 A DNS-polimerázok jellemzői 5'-3' polimeráz aktivitás 5'-3' exonukleáz 3'-5' exonukleáz aktivitás Az új szál szintéziséhez kell: templát DNS primer
Hátterükben egyetlen gén áll, melynek általában számottevő a viselkedésre gyakorolt hatása, öröklési mintázata jellegzetes.
 Múlt órán: Lehetséges tesztfeladatok: Kitől származik a variáció-szelekció paradigma, mely szerint az egyéni, javarészt öröklött különbségek között a társadalmi harc válogat? Fromm-Reichmann Mill Gallton
Múlt órán: Lehetséges tesztfeladatok: Kitől származik a variáció-szelekció paradigma, mely szerint az egyéni, javarészt öröklött különbségek között a társadalmi harc válogat? Fromm-Reichmann Mill Gallton
Az Agrármérnöki MSc szak tananyagfejlesztése TÁMOP-4.1.2-08/1/A-2009-0010 projekt
 Az Agrármérnöki MSc szak tananyagfejlesztése TÁMOP-4.1.2-08/1/A-2009-0010 projekt ÁLLATGENETIKA Debreceni Egyetem Nyugat-magyarországi Egyetem Pannon Egyetem A projekt az Európai Unió támogatásával, az
Az Agrármérnöki MSc szak tananyagfejlesztése TÁMOP-4.1.2-08/1/A-2009-0010 projekt ÁLLATGENETIKA Debreceni Egyetem Nyugat-magyarországi Egyetem Pannon Egyetem A projekt az Európai Unió támogatásával, az
HAPMAP -2010 Nemzetközi HapMap Projekt. SNP GWA Haplotípus: egy kromoszóma szegmensen lévő SNP mintázat
 HAPMAP -2010 Nemzetközi HapMap Projekt A Nemzetközi HapMap Project célja az emberi genom haplotípus* térképének(hapmap; haplotype map) megszerkesztése, melynek segítségével katalogizálni tudjuk az ember
HAPMAP -2010 Nemzetközi HapMap Projekt A Nemzetközi HapMap Project célja az emberi genom haplotípus* térképének(hapmap; haplotype map) megszerkesztése, melynek segítségével katalogizálni tudjuk az ember
Biomassza alapú bioalkohol előállítási technológia fejlesztése metagenomikai eljárással
 Biomassza alapú bioalkohol előállítási technológia fejlesztése metagenomikai eljárással Kovács Zoltán ügyvezető DEKUT Debreceni Kutatásfejlesztési Közhasznú Nonprofit Kft. Problémadefiníció Első generációs
Biomassza alapú bioalkohol előállítási technológia fejlesztése metagenomikai eljárással Kovács Zoltán ügyvezető DEKUT Debreceni Kutatásfejlesztési Közhasznú Nonprofit Kft. Problémadefiníció Első generációs
ADATBÁNYÁSZAT I. ÉS OMICS
 Az élettudományi-klinikai felsőoktatás gyakorlatorientált és hallgatóbarát korszerűsítése a vidéki képzőhelyek nemzetközi versenyképességének erősítésére TÁMOP-4.1.1.C-13/1/KONV-2014-0001 ADATBÁNYÁSZAT
Az élettudományi-klinikai felsőoktatás gyakorlatorientált és hallgatóbarát korszerűsítése a vidéki képzőhelyek nemzetközi versenyképességének erősítésére TÁMOP-4.1.1.C-13/1/KONV-2014-0001 ADATBÁNYÁSZAT
Kromoszómák, Gének centromer
 Kromoszómák, Gének A kromoszóma egy hosszú DNS szakasz, amely a sejt életének bizonyos szakaszában (a sejtosztódás előkészítéseként) tömörödik, így fénymikroszkóppal láthatóvá válik. A kromoszómák két
Kromoszómák, Gének A kromoszóma egy hosszú DNS szakasz, amely a sejt életének bizonyos szakaszában (a sejtosztódás előkészítéseként) tömörödik, így fénymikroszkóppal láthatóvá válik. A kromoszómák két
10. CSI. A molekuláris biológiai technikák alkalmazásai
 10. CSI. A molekuláris biológiai technikák alkalmazásai A DNS mint azonosító 3 milliárd bázispár az emberi DNS-ben (99.9%-ban azonos) 0.1%-nyi különbség elegendő az egyedek megkülönböztetéséhez Genetikai
10. CSI. A molekuláris biológiai technikák alkalmazásai A DNS mint azonosító 3 milliárd bázispár az emberi DNS-ben (99.9%-ban azonos) 0.1%-nyi különbség elegendő az egyedek megkülönböztetéséhez Genetikai
Conserved ortholog set (COS) markerek térképezése Aegilops kromoszómákon
 Conserved ortholog set (COS) markerek térképezése Aegilops kromoszómákon Rövid tanulmányút 2011. 01.03-03. 30., John Inn Centre, Dept. of Crop Genetics, Norwich Research Park, Norwich NR4 7UH, UK Supervisor:
Conserved ortholog set (COS) markerek térképezése Aegilops kromoszómákon Rövid tanulmányút 2011. 01.03-03. 30., John Inn Centre, Dept. of Crop Genetics, Norwich Research Park, Norwich NR4 7UH, UK Supervisor:
Molekuláris genetikai vizsgáló. módszerek az immundefektusok. diagnosztikájában
 Molekuláris genetikai vizsgáló módszerek az immundefektusok diagnosztikájában Primer immundefektusok A primer immundeficiencia ritka, veleszületett, monogénes öröklődésű immunhiányos állapot. Családi halmozódást
Molekuláris genetikai vizsgáló módszerek az immundefektusok diagnosztikájában Primer immundefektusok A primer immundeficiencia ritka, veleszületett, monogénes öröklődésű immunhiányos állapot. Családi halmozódást
NÖVÉNYGENETIKA. Az Agrármérnöki MSc szak tananyagfejlesztése TÁMOP /1/A
 NÖVÉNYGENETIKA Az Agrármérnöki MSc szak tananyagfejlesztése TÁMOP-4.1.2-08/1/A-2009-0010 A citológia és a genetika társtudománya Citogenetika A kromoszómák eredetét, szerkezetét, genetikai funkcióját,
NÖVÉNYGENETIKA Az Agrármérnöki MSc szak tananyagfejlesztése TÁMOP-4.1.2-08/1/A-2009-0010 A citológia és a genetika társtudománya Citogenetika A kromoszómák eredetét, szerkezetét, genetikai funkcióját,
Genomika. Mutációk (SNP-k) és vizsgálatuk egyszerű módszerekkel. DNS szekvenálási eljárások. DNS ujjlenyomat (VNTR)
 Genomika (A genom, génállomány vizsgálata) Mutációk (SNP-k) és vizsgálatuk egyszerű módszerekkel DNS szekvenálási eljárások DNS ujjlenyomat (VNTR) DNS chipek statikus és dinamikus információk vizsgálata
Genomika (A genom, génállomány vizsgálata) Mutációk (SNP-k) és vizsgálatuk egyszerű módszerekkel DNS szekvenálási eljárások DNS ujjlenyomat (VNTR) DNS chipek statikus és dinamikus információk vizsgálata
Engedélyszám: 18211-2/2011-EAHUF Verziószám: 1. 2460-06 Humángenetikai vizsgálatok követelménymodul szóbeli vizsgafeladatai
 1. feladat Ismertesse a gyakorlaton lévő szakasszisztens hallgatóknak a PCR termékek elválasztása céljából végzett analitikai agaróz gélelektroforézis során használt puffert! Az ismertetés során az alábbi
1. feladat Ismertesse a gyakorlaton lévő szakasszisztens hallgatóknak a PCR termékek elválasztása céljából végzett analitikai agaróz gélelektroforézis során használt puffert! Az ismertetés során az alábbi
A PKU azért nem hal ki, mert gyógyítják, és ezzel növelik a mutáns allél gyakoriságát a Huntington kór pedig azért marad fenn, mert csak későn derül
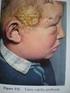 1 Múlt órán: Genetikai alapelvek, monogénes öröklődés Elgondolkodtató feladat Vajon miért nem halnak ki az olyan mendeli öröklődésű rendellenességek, mint a Phenylketonuria, vagy a Huntington kór? A PKU
1 Múlt órán: Genetikai alapelvek, monogénes öröklődés Elgondolkodtató feladat Vajon miért nem halnak ki az olyan mendeli öröklődésű rendellenességek, mint a Phenylketonuria, vagy a Huntington kór? A PKU
10. Genomika 2. Microarrayek és típusaik
 10. Genomika 2. 1. Microarray technikák és bioinformatikai vonatkozásaik Microarrayek és típusaik Korrelált génexpresszió mint a funkcionális genomika eszköze 2. Kombinált megközelítés a funkcionális genomikában
10. Genomika 2. 1. Microarray technikák és bioinformatikai vonatkozásaik Microarrayek és típusaik Korrelált génexpresszió mint a funkcionális genomika eszköze 2. Kombinált megközelítés a funkcionális genomikában
2011. január április 10. IPK Gatersleben (Németország) május 17. Kruppa Klaudia
 2011. január 10. 2011. április 10. IPK Gatersleben (Németország) Gatersleben (G-life) Country State District Town Administration Germany Saxony-Anhalt Salzlandkreis Seeland Basic statistics Area 16.00
2011. január 10. 2011. április 10. IPK Gatersleben (Németország) Gatersleben (G-life) Country State District Town Administration Germany Saxony-Anhalt Salzlandkreis Seeland Basic statistics Area 16.00
A Multi Locus Sequence Typing (MLST) alkalmazhatósága az élelmiszermikrobiológiában
 A Multi Locus Sequence Typing (MLST) alkalmazhatósága az élelmiszermikrobiológiában Sipos Rita, Lukács Alena, Simon Janka, Szántó-Egész Réka, Micsinai Adrienn 2100 Gödöllő, Szent-Györgyi Albert út 4. info@biomi.hu,
A Multi Locus Sequence Typing (MLST) alkalmazhatósága az élelmiszermikrobiológiában Sipos Rita, Lukács Alena, Simon Janka, Szántó-Egész Réka, Micsinai Adrienn 2100 Gödöllő, Szent-Györgyi Albert út 4. info@biomi.hu,
MUTÁCIÓK. A mutáció az örökítő anyag spontán, maradandó megváltozása, amelynek során új genetikai tulajdonság keletkezik.
 MUTÁCIÓK A mutáció az örökítő anyag spontán, maradandó megváltozása, amelynek során új genetikai tulajdonság keletkezik. Pontmutáció: A kromoszóma egy génjében pár nukleotidnál következik be változás.
MUTÁCIÓK A mutáció az örökítő anyag spontán, maradandó megváltozása, amelynek során új genetikai tulajdonság keletkezik. Pontmutáció: A kromoszóma egy génjében pár nukleotidnál következik be változás.
Szakmai zárójelentés. 1. Strukturális genomika Az RPS13 gén szekvenciájához homológ régió deléciója M. sativa-ban
 Szakmai zárójelentés 1. Strukturális genomika 1. 1. Az RPS13 gén szekvenciájához homológ régió deléciója M. sativa-ban A diploid lucerna (Medicago sativa) genetikai térképének megszerkesztése során csoportunk
Szakmai zárójelentés 1. Strukturális genomika 1. 1. Az RPS13 gén szekvenciájához homológ régió deléciója M. sativa-ban A diploid lucerna (Medicago sativa) genetikai térképének megszerkesztése során csoportunk
Diagnosztikai célú molekuláris biológiai vizsgálatok
 Diagnosztikai célú molekuláris biológiai vizsgálatok Dr. Patócs Attila, PhD MTA-SE Molekuláris Medicina Kutatócsoport, Semmelweis Egyetem II. sz. Belgyógyászati Klinika Laboratóriumi Medicina Intézet Genetikai
Diagnosztikai célú molekuláris biológiai vizsgálatok Dr. Patócs Attila, PhD MTA-SE Molekuláris Medicina Kutatócsoport, Semmelweis Egyetem II. sz. Belgyógyászati Klinika Laboratóriumi Medicina Intézet Genetikai
A FISH technika alkalmazása az előnemesítésben
 Linc Gabriella A FISH technika alkalmazása az előnemesítésben Czuczor Gergely Bencés Gimnázium és Kollégium Győr, 2016. április 13. www.meetthescientist.hu 1 26 - 1997-98 vendégkutató - növény genetikai
Linc Gabriella A FISH technika alkalmazása az előnemesítésben Czuczor Gergely Bencés Gimnázium és Kollégium Győr, 2016. április 13. www.meetthescientist.hu 1 26 - 1997-98 vendégkutató - növény genetikai
Populációgenetikai. alapok
 Populációgenetikai alapok Populáció = egyedek egy adott csoportja Az egyedek eltérnek egymástól morfológiailag, de viselkedésüket tekintve is = genetikai különbségek Fenotípus = külső jellegek morfológia,
Populációgenetikai alapok Populáció = egyedek egy adott csoportja Az egyedek eltérnek egymástól morfológiailag, de viselkedésüket tekintve is = genetikai különbségek Fenotípus = külső jellegek morfológia,
Molekuláris biológiai eljárások alkalmazása a GMO analitikában és az élelmiszerbiztonság területén
 Molekuláris biológiai eljárások alkalmazása a GMO analitikában és az élelmiszerbiztonság területén Dr. Dallmann Klára A molekuláris biológia célja az élőlények és sejtek működésének molekuláris szintű
Molekuláris biológiai eljárások alkalmazása a GMO analitikában és az élelmiszerbiztonság területén Dr. Dallmann Klára A molekuláris biológia célja az élőlények és sejtek működésének molekuláris szintű
Tudománytörténeti visszatekintés
 GENETIKA I. AZ ÖRÖKLŐDÉS TÖRVÉNYSZERŰSÉGEI Minek köszönhető a biológiai sokféleség? Hogyan történik a tulajdonságok átörökítése? Tudománytörténeti visszatekintés 1. Keveredés alapú öröklődés: (1761-1766,
GENETIKA I. AZ ÖRÖKLŐDÉS TÖRVÉNYSZERŰSÉGEI Minek köszönhető a biológiai sokféleség? Hogyan történik a tulajdonságok átörökítése? Tudománytörténeti visszatekintés 1. Keveredés alapú öröklődés: (1761-1766,
A bioinformatika gyökerei
 A bioinformatika gyökerei 1944: Avery a transforming principle a DNS 1952: Hershey és Chase perdöntő bizonyíték: a bakteriofágok szaporodásakor csak a DNS jut be a sejtbe 1953: Watson és Crick a DNS szerkezete
A bioinformatika gyökerei 1944: Avery a transforming principle a DNS 1952: Hershey és Chase perdöntő bizonyíték: a bakteriofágok szaporodásakor csak a DNS jut be a sejtbe 1953: Watson és Crick a DNS szerkezete
GENOMIKA TÖBBFÉLE MAKROMOLEKULA VIZSGÁLATA EGYIDŐBEN
 GENOMIKA TÖBBFÉLE MAKROMOLEKULA VIZSGÁLATA EGYIDŐBEN Strukturális genomika Genomkönyvtárak DNS szekvenálás Genom programok Polimorfizmusok RFLP DNS könyvtár készítés humán genom 1. Emésztés RE-kal Emberi
GENOMIKA TÖBBFÉLE MAKROMOLEKULA VIZSGÁLATA EGYIDŐBEN Strukturális genomika Genomkönyvtárak DNS szekvenálás Genom programok Polimorfizmusok RFLP DNS könyvtár készítés humán genom 1. Emésztés RE-kal Emberi
A genetikai lelet értelmezése monogénes betegségekben
 A genetikai lelet értelmezése monogénes betegségekben Tory Kálmán Semmelweis Egyetem, I. sz. Gyermekklinika A ~20 ezer fehérje-kódoló gén a 23 pár kromoszómán A kromoszómán található bázisok száma: 250M
A genetikai lelet értelmezése monogénes betegségekben Tory Kálmán Semmelweis Egyetem, I. sz. Gyermekklinika A ~20 ezer fehérje-kódoló gén a 23 pár kromoszómán A kromoszómán található bázisok száma: 250M
Evolúcióbiológia. Biológus B.Sc tavaszi félév
 Evolúcióbiológia Biológus B.Sc. 2011. tavaszi félév A biológiában minden csak az evolúció fényében válik érthetővé Theodosius Dobzhansky : Nothing in biology makes sense except in the light of evolution.
Evolúcióbiológia Biológus B.Sc. 2011. tavaszi félév A biológiában minden csak az evolúció fényében válik érthetővé Theodosius Dobzhansky : Nothing in biology makes sense except in the light of evolution.
Fehérje expressziós rendszerek. Gyógyszerészi Biotechnológia
 Fehérje expressziós rendszerek Gyógyszerészi Biotechnológia Expressziós rendszerek Cél: rekombináns fehérjék előállítása nagy tisztaságban és nagy mennyiségben kísérleti ill. gyakorlati (therapia) felhasználásokra
Fehérje expressziós rendszerek Gyógyszerészi Biotechnológia Expressziós rendszerek Cél: rekombináns fehérjék előállítása nagy tisztaságban és nagy mennyiségben kísérleti ill. gyakorlati (therapia) felhasználásokra
BIOLÓGIA HÁZIVERSENY 1. FORDULÓ BIOKÉMIA, GENETIKA BIOKÉMIA, GENETIKA
 BIOKÉMIA, GENETIKA 1. Nukleinsavak keresztrejtvény (12+1 p) 1. 2. 3. 4. 5. 6. 7. 8. 9. 10. 11. 12. 1. A nukleinsavak a.-ok összekapcsolódásával kialakuló polimerek. 2. Purinvázas szerves bázis, amely az
BIOKÉMIA, GENETIKA 1. Nukleinsavak keresztrejtvény (12+1 p) 1. 2. 3. 4. 5. 6. 7. 8. 9. 10. 11. 12. 1. A nukleinsavak a.-ok összekapcsolódásával kialakuló polimerek. 2. Purinvázas szerves bázis, amely az
Klónozás: tökéletesen egyforma szervezetek csoportjának előállítása, vagyis több genetikailag azonos egyed létrehozása.
 Növények klónozása Klónozás Klónozás: tökéletesen egyforma szervezetek csoportjának előállítása, vagyis több genetikailag azonos egyed létrehozása. Görög szó: klon, jelentése: gally, hajtás, vessző. Ami
Növények klónozása Klónozás Klónozás: tökéletesen egyforma szervezetek csoportjának előállítása, vagyis több genetikailag azonos egyed létrehozása. Görög szó: klon, jelentése: gally, hajtás, vessző. Ami
Genetika 3 ea. Bevezetés
 Genetika 3 ea. Mendel törvényeinek a kiegészítése: Egygénes öröklődés Többtényezős öröklődés Bevezetés Mendel által vizsgált tulajdonságok: diszkrétek, két különböző fenotípus Humán tulajdonságok nagy
Genetika 3 ea. Mendel törvényeinek a kiegészítése: Egygénes öröklődés Többtényezős öröklődés Bevezetés Mendel által vizsgált tulajdonságok: diszkrétek, két különböző fenotípus Humán tulajdonságok nagy
AZ ÁRPA SZÁRAZSÁGTŰRÉSÉNEK VIZSGÁLATA: QTL- ÉS ASSZOCIÁCIÓS ANALÍZIS, MARKER ALAPÚ SZELEKCIÓ, TILLING
 Hagyomány és haladás a növénynemesítésben AZ ÁRPA SZÁRAZSÁGTŰRÉSÉNEK VIZSGÁLATA: QTL- ÉS ASSZOCIÁCIÓS ANALÍZIS, MARKER ALAPÚ SZELEKCIÓ, BÁLINT ANDRÁS FERENC 1, SZIRA FRUZSINA 1, ANDREAS BÖRNER 2, KERSTIN
Hagyomány és haladás a növénynemesítésben AZ ÁRPA SZÁRAZSÁGTŰRÉSÉNEK VIZSGÁLATA: QTL- ÉS ASSZOCIÁCIÓS ANALÍZIS, MARKER ALAPÚ SZELEKCIÓ, BÁLINT ANDRÁS FERENC 1, SZIRA FRUZSINA 1, ANDREAS BÖRNER 2, KERSTIN
Az orvosi biotechnológiai mesterképzés megfeleltetése az Európai Unió új társadalmi kihívásainak a Pécsi Tudományegyetemen és a Debreceni Egyetemen
 Az orvosi biotechnológiai mesterképzés megfeleltetése az Európai Unió új társadalmi kihívásainak a Pécsi Tudományegyetemen és a Debreceni Egyetemen Azonosító szám: TÁMOP-4.1.2-08/1/A-2009-0011 Az orvosi
Az orvosi biotechnológiai mesterképzés megfeleltetése az Európai Unió új társadalmi kihívásainak a Pécsi Tudományegyetemen és a Debreceni Egyetemen Azonosító szám: TÁMOP-4.1.2-08/1/A-2009-0011 Az orvosi
Johann Gregor Mendel Az olmüci (Olomouc) és bécsi egyetem diákja Brünni ágostonrendi apát (nem szovjet tudós) Tudatos és nagyon alapos kutat
 10.2.2010 genmisk1 1 Áttekintés Mendel és a mendeli törvények Mendel előtt és körül A genetika törvényeinek újbóli felfedezése és a kromoszómák Watson és Crick a molekuláris biológoa központi dogmája 10.2.2010
10.2.2010 genmisk1 1 Áttekintés Mendel és a mendeli törvények Mendel előtt és körül A genetika törvényeinek újbóli felfedezése és a kromoszómák Watson és Crick a molekuláris biológoa központi dogmája 10.2.2010
A GABONAFÉLÉK EGYEDFEJLŐDÉSÉT ÉS KALÁSZOLÁSÁT MEGHATÁROZÓ GENETIKAI KOMPONENSEK TANULMÁNYOZÁSA
 A martonvásári agrárkutatások hatodik évtizede A GABONAFÉLÉK EGYEDFEJLŐDÉSÉT ÉS KALÁSZOLÁSÁT MEGHATÁROZÓ GENETIKAI KOMPONENSEK TANULMÁNYOZÁSA KARSAI ILDIKÓ 1, MÉSZÁROS KLÁRA 2, KŐSZEGI BÉLA 3, PATRICK
A martonvásári agrárkutatások hatodik évtizede A GABONAFÉLÉK EGYEDFEJLŐDÉSÉT ÉS KALÁSZOLÁSÁT MEGHATÁROZÓ GENETIKAI KOMPONENSEK TANULMÁNYOZÁSA KARSAI ILDIKÓ 1, MÉSZÁROS KLÁRA 2, KŐSZEGI BÉLA 3, PATRICK
Ph.D. értekezés tézisei GENOMIKAI ÉS FENOMIKAI MEGKÖZELÍTÉSEK KOMBINÁLÁSA AZ ÁRPA SZÁRAZSÁGTŰRÉSÉNEK JELLEMZÉSÉBEN. Cseri András.
 Ph.D. értekezés tézisei GENOMIKAI ÉS FENOMIKAI MEGKÖZELÍTÉSEK KOMBINÁLÁSA AZ ÁRPA SZÁRAZSÁGTŰRÉSÉNEK JELLEMZÉSÉBEN Cseri András Témavezetők: Prof. Dr. Dudits Dénes Dr. Törjék Ottó Biológia Doktori Iskola
Ph.D. értekezés tézisei GENOMIKAI ÉS FENOMIKAI MEGKÖZELÍTÉSEK KOMBINÁLÁSA AZ ÁRPA SZÁRAZSÁGTŰRÉSÉNEK JELLEMZÉSÉBEN Cseri András Témavezetők: Prof. Dr. Dudits Dénes Dr. Törjék Ottó Biológia Doktori Iskola
Intelligens Rendszerek Elmélete. Párhuzamos keresés genetikus algoritmusokkal
 Intelligens Rendszerek Elmélete Dr. Kutor László Párhuzamos keresés genetikus algoritmusokkal http://mobil.nik.bmf.hu/tantargyak/ire.html login: ire jelszó: IRE0 IRE / A természet általános kereső algoritmusa:
Intelligens Rendszerek Elmélete Dr. Kutor László Párhuzamos keresés genetikus algoritmusokkal http://mobil.nik.bmf.hu/tantargyak/ire.html login: ire jelszó: IRE0 IRE / A természet általános kereső algoritmusa:
A növény inváziójában szerepet játszó bakteriális gének
 A növény inváziójában szerepet játszó bakteriális gének merisztéma korai szimbiotikus zóna késői szimbiotikus zóna öregedési zóna gyökér keresztmetszet NODULÁCIÓ növényi jel Rhizobium meliloti rhizobium
A növény inváziójában szerepet játszó bakteriális gének merisztéma korai szimbiotikus zóna késői szimbiotikus zóna öregedési zóna gyökér keresztmetszet NODULÁCIÓ növényi jel Rhizobium meliloti rhizobium
Molekuláris biológiai módszerek m. hibridizációs s technikák
 Molekuláris biológiai módszerek m II: közvetlen k szekvencia analízis, hibridizációs s technikák Tordai Attila OVSZ Molekuláris Diagnosztikai Labor Bp., 2011. április 21. Labordiagnosztikai szinten tartó
Molekuláris biológiai módszerek m II: közvetlen k szekvencia analízis, hibridizációs s technikák Tordai Attila OVSZ Molekuláris Diagnosztikai Labor Bp., 2011. április 21. Labordiagnosztikai szinten tartó
A termesztett búza diploid őseinek molekuláris citogenetikai elemzése: pachytén- és fiber-fish.
 OTKA K67808 zárójelentés 2012. A termesztett búza diploid őseinek molekuláris citogenetikai elemzése: pachytén- és fiber-fish. A fluoreszcens in situ hibridizáció (FISH) olyan technikai fejlettséget ért
OTKA K67808 zárójelentés 2012. A termesztett búza diploid őseinek molekuláris citogenetikai elemzése: pachytén- és fiber-fish. A fluoreszcens in situ hibridizáció (FISH) olyan technikai fejlettséget ért
BÁLINT András Beszámoló az AGRISAFE által támogatott tanulmányútról 2008 november 2009 február. 1. Az IPK bemutatása 2.
 BÁLINT András Beszámoló az AGRISAFE által támogatott tanulmányútról 2008 november 2009 február 1. Az IPK bemutatása 2. A TILLING módszer Hol található az IPK? Gatersleben Általános adatok az IPK-ról Leibniz
BÁLINT András Beszámoló az AGRISAFE által támogatott tanulmányútról 2008 november 2009 február 1. Az IPK bemutatása 2. A TILLING módszer Hol található az IPK? Gatersleben Általános adatok az IPK-ról Leibniz
A szamóca érése során izolált Spiral és Spermidin-szintáz gén jellemzése. Kiss Erzsébet Kovács László
 A szamóca érése során izolált Spiral és Spermidin-szintáz gén jellemzése Kiss Erzsébet Kovács László Bevezetés Nagy gazdasági gi jelentıségük k miatt a gyümölcs lcsök, termések fejlıdésének mechanizmusát
A szamóca érése során izolált Spiral és Spermidin-szintáz gén jellemzése Kiss Erzsébet Kovács László Bevezetés Nagy gazdasági gi jelentıségük k miatt a gyümölcs lcsök, termések fejlıdésének mechanizmusát
Mangalica specifikus DNS alapú módszer kifejlesztés és validálása a MANGFOOD projekt keretében
 Mangalica specifikus DNS alapú módszer kifejlesztés és validálása a MANGFOOD projekt keretében Szántó-Egész Réka 1, Mohr Anita 1, Sipos Rita 1, Dallmann Klára 1, Ujhelyi Gabriella 2, Koppányné Szabó Erika
Mangalica specifikus DNS alapú módszer kifejlesztés és validálása a MANGFOOD projekt keretében Szántó-Egész Réka 1, Mohr Anita 1, Sipos Rita 1, Dallmann Klára 1, Ujhelyi Gabriella 2, Koppányné Szabó Erika
A Hardy-Weinberg egyensúly. 2. gyakorlat
 A Hardy-Weinberg egyensúly 2. gyakorlat A Hardy-Weinberg egyensúly feltételei: nincs szelekció nincs migráció nagy populációméret (nincs sodródás) nincs mutáció pánmixis van allélgyakoriság azonos hímekben
A Hardy-Weinberg egyensúly 2. gyakorlat A Hardy-Weinberg egyensúly feltételei: nincs szelekció nincs migráció nagy populációméret (nincs sodródás) nincs mutáció pánmixis van allélgyakoriság azonos hímekben
Poligénes v. kantitatív öröklődés
 1. Öröklődés komplexebb sajátosságai 2. Öröklődés molekuláris alapja Poligénes v. kantitatív öröklődés Azok a tulajdonságokat amelyek mértékegységgel nem, vagy csak nehezen mérhetők, kialakulásuk kevéssé
1. Öröklődés komplexebb sajátosságai 2. Öröklődés molekuláris alapja Poligénes v. kantitatív öröklődés Azok a tulajdonságokat amelyek mértékegységgel nem, vagy csak nehezen mérhetők, kialakulásuk kevéssé
Példák a független öröklődésre
 GENETIKAI PROBLÉMÁK Példák a független öröklődésre Az amelogenesis imperfecta egy, a fogzománc gyengeségével és elszíneződésével járó öröklődő betegség, a 4-es kromoszómán lévő enam gén recesszív mutációja
GENETIKAI PROBLÉMÁK Példák a független öröklődésre Az amelogenesis imperfecta egy, a fogzománc gyengeségével és elszíneződésével járó öröklődő betegség, a 4-es kromoszómán lévő enam gén recesszív mutációja
A Leuce szekcióba tartozó hazai nyárak dunántúli természetes eredetű állományainak populációgenetikai vizsgálata
 Nyugat-magyarországi Egyetem Erdőmérnöki Kar Doktori (PhD) értekezés tézisei A Leuce szekcióba tartozó hazai nyárak dunántúli természetes eredetű állományainak populációgenetikai vizsgálata Benke Attila
Nyugat-magyarországi Egyetem Erdőmérnöki Kar Doktori (PhD) értekezés tézisei A Leuce szekcióba tartozó hazai nyárak dunántúli természetes eredetű állományainak populációgenetikai vizsgálata Benke Attila
Szakmai zárójelentés
 Szakmai zárójelentés Kései szárazságtűrésben szerepet játszó génjelöltek asszociációs vizsgálata árpában EcoTILLING módszerrel című OTKA pályázat (OTKA azonosító: 72366) Kutatásaink elsődleges célja a
Szakmai zárójelentés Kései szárazságtűrésben szerepet játszó génjelöltek asszociációs vizsgálata árpában EcoTILLING módszerrel című OTKA pályázat (OTKA azonosító: 72366) Kutatásaink elsődleges célja a
A genomikai oktatás helyzete a Debreceni Egyetemen
 A genomikai oktatás helyzete a Debreceni Egyetemen Bálint Bálint L. GNTP Oktatás és Tudásmenedzsment Munkabizottság, 2009. június 10. Tények Debreceni Egyetemről 21000 nappali és 33000 összes hallgató
A genomikai oktatás helyzete a Debreceni Egyetemen Bálint Bálint L. GNTP Oktatás és Tudásmenedzsment Munkabizottság, 2009. június 10. Tények Debreceni Egyetemről 21000 nappali és 33000 összes hallgató
A kromoszómák kialakulása előtt a DNS állomány megkettőződik. A két azonos információ tartalmú DNS egymás mellé rendeződik és egy kromoszómát alkot.
 Kromoszómák, Gének A kromoszóma egy hosszú DNS szakasz, amely a sejt életének bizonyos szakaszában (a sejtosztódás előkészítéseként) tömörödik, így fénymikroszkóppal láthatóvá válik. A kromoszómák két
Kromoszómák, Gének A kromoszóma egy hosszú DNS szakasz, amely a sejt életének bizonyos szakaszában (a sejtosztódás előkészítéseként) tömörödik, így fénymikroszkóppal láthatóvá válik. A kromoszómák két
12/4/2014. Genetika 7-8 ea. DNS szerkezete, replikáció és a rekombináció. 1952 Hershey & Chase 1953!!!
 Genetika 7-8 ea. DNS szerkezete, replikáció és a rekombináció 1859 1865 1869 1952 Hershey & Chase 1953!!! 1879 1903 1951 1950 1944 1928 1911 1 1. DNS szerkezete Mi az örökítő anyag? Friedrich Miescher
Genetika 7-8 ea. DNS szerkezete, replikáció és a rekombináció 1859 1865 1869 1952 Hershey & Chase 1953!!! 1879 1903 1951 1950 1944 1928 1911 1 1. DNS szerkezete Mi az örökítő anyag? Friedrich Miescher
A géntechnológiát megalapozó felfedezések
 2010. december BIOTECHNOLÓGIA Rova tvezető: Dr. Heszky László akadémikus A géntechnológia genetikai alapjai c. I. fejezet 1-5. részében azokat a tudományos eredményeket mutattuk be, melyek bizonyítják,
2010. december BIOTECHNOLÓGIA Rova tvezető: Dr. Heszky László akadémikus A géntechnológia genetikai alapjai c. I. fejezet 1-5. részében azokat a tudományos eredményeket mutattuk be, melyek bizonyítják,
Intelligens Rendszerek Elmélete. Párhuzamos keresés genetikus algoritmusokkal. A genetikus algoritmus működése. Az élet információ tárolói
 Intelligens Rendszerek Elmélete dr. Kutor László Párhuzamos keresés genetikus algoritmusokkal http://mobil.nik.bmf.hu/tantargyak/ire.html login: ire jelszó: IRE07 IRE 5/ Természetes és mesterséges genetikus
Intelligens Rendszerek Elmélete dr. Kutor László Párhuzamos keresés genetikus algoritmusokkal http://mobil.nik.bmf.hu/tantargyak/ire.html login: ire jelszó: IRE07 IRE 5/ Természetes és mesterséges genetikus
Bakteriális identifikáció 16S rrns gén szekvencia alapján
 Bakteriális identifikáció 16S rrns gén szekvencia alapján MOHR ANITA SIPOS RITA, SZÁNTÓ-EGÉSZ RÉKA, MICSINAI ADRIENN 2100 Gödöllő, Szent-Györgyi Albert út 4. info@biomi.hu, www.biomi.hu TÖRZS AZONOSÍTÁS
Bakteriális identifikáció 16S rrns gén szekvencia alapján MOHR ANITA SIPOS RITA, SZÁNTÓ-EGÉSZ RÉKA, MICSINAI ADRIENN 2100 Gödöllő, Szent-Györgyi Albert út 4. info@biomi.hu, www.biomi.hu TÖRZS AZONOSÍTÁS
MUTÁCIÓK. A mutáció az örökítő anyag spontán, maradandó megváltozása, amelynek során új genetikai tulajdonság keletkezik.
 MUTÁCIÓK A mutáció az örökítő anyag spontán, maradandó megváltozása, amelynek során új genetikai tulajdonság keletkezik. Pontmutáció: A kromoszóma egy génjében pár nukleotidnál következik be változás.
MUTÁCIÓK A mutáció az örökítő anyag spontán, maradandó megváltozása, amelynek során új genetikai tulajdonság keletkezik. Pontmutáció: A kromoszóma egy génjében pár nukleotidnál következik be változás.
5. Molekuláris biológiai technikák
 5. Molekuláris biológiai technikák DNS szaporítás kémcsőben és élőben. Klónozás, PCR, cdna, RT-PCR, realtime-rt-pcr, Northern-, Southernblotting, génexpresszió, FISH 5. Molekuláris szintű biológiai technikák
5. Molekuláris biológiai technikák DNS szaporítás kémcsőben és élőben. Klónozás, PCR, cdna, RT-PCR, realtime-rt-pcr, Northern-, Southernblotting, génexpresszió, FISH 5. Molekuláris szintű biológiai technikák
Dr. Máthéné Dr. Szigeti Zsuzsanna és munkatársai
 Kar: TTK Tantárgy: CITOGENETIKA Kód: AOMBCGE3 ECTS Kredit: 3 A tantárgyat oktató intézet: TTK Mikrobiális Biotechnológiai és Sejtbiológiai Tanszék A tantárgy felvételére ajánlott félév: 3. Melyik félévben
Kar: TTK Tantárgy: CITOGENETIKA Kód: AOMBCGE3 ECTS Kredit: 3 A tantárgyat oktató intézet: TTK Mikrobiális Biotechnológiai és Sejtbiológiai Tanszék A tantárgy felvételére ajánlott félév: 3. Melyik félévben
MOLEKULÁRIS GENETIKA A LABORATÓRIUMI MEDICINÁBAN. Laboratóriumi Medicina Intézet 2017.
 MOLEKULÁRIS GENETIKA A LABORATÓRIUMI MEDICINÁBAN Laboratóriumi Medicina Intézet 2017. 1 Történeti áttekintés Humán genom projekt 20-25 000 gén azonosítása 1,800 betegséghez köthető gén 1000 genetikai teszt
MOLEKULÁRIS GENETIKA A LABORATÓRIUMI MEDICINÁBAN Laboratóriumi Medicina Intézet 2017. 1 Történeti áttekintés Humán genom projekt 20-25 000 gén azonosítása 1,800 betegséghez köthető gén 1000 genetikai teszt
Szelekció. Szelekció. A szelekció típusai. Az allélgyakoriságok változása 3/4/2013
 Szelekció Ok: több egyed születik, mint amennyi túlél és szaporodni képes a sikeresség mérése: fitnesz Szelekció Ok: több egyed születik, mint amennyi túlél és szaporodni képes a sikeresség mérése: fitnesz
Szelekció Ok: több egyed születik, mint amennyi túlél és szaporodni képes a sikeresség mérése: fitnesz Szelekció Ok: több egyed születik, mint amennyi túlél és szaporodni képes a sikeresség mérése: fitnesz
PÉCSI TUDOMÁNYEGYETEM. Agrobacterium rezisztencia térképezése szőlőben. Kuczmog Anett
 PÉCSI TUDOMÁNYEGYETEM Biológia Doktori Iskola Genetika program Agrobacterium rezisztencia térképezése szőlőben PhD értekezés Kuczmog Anett Témavezető: Dr. Putnoky Péter egyetemi tanár PÉCS, 2012. 1. BEVEZETÉS
PÉCSI TUDOMÁNYEGYETEM Biológia Doktori Iskola Genetika program Agrobacterium rezisztencia térképezése szőlőben PhD értekezés Kuczmog Anett Témavezető: Dr. Putnoky Péter egyetemi tanár PÉCS, 2012. 1. BEVEZETÉS
Kappelmayer János. Malignus hematológiai megbetegedések molekuláris háttere. MOLSZE IX. Nagygyűlése. Bük, 2005 szeptember
 Kappelmayer János Malignus hematológiai megbetegedések molekuláris háttere MOLSZE IX. Nagygyűlése Bük, 2005 szeptember 29-30. Laboratóriumi vizsgálatok hematológiai malignómákban Általános laboratóriumi
Kappelmayer János Malignus hematológiai megbetegedések molekuláris háttere MOLSZE IX. Nagygyűlése Bük, 2005 szeptember 29-30. Laboratóriumi vizsgálatok hematológiai malignómákban Általános laboratóriumi
Nagy Emese: Polimorfizmus és rokonsági körök vizsgálata kukoricában (Zea mays) Témavezetők: Cs. L. Marton G Gyulai
 Nagy Emese: Polimorfizmus és rokonsági körök vizsgálata kukoricában (Zea mays) Témavezetők: Cs. L. Marton G Gyulai BEVEZETÉS A molekuláris biológiai és genetikai módszerek gyors fejlődése egyre inkább
Nagy Emese: Polimorfizmus és rokonsági körök vizsgálata kukoricában (Zea mays) Témavezetők: Cs. L. Marton G Gyulai BEVEZETÉS A molekuláris biológiai és genetikai módszerek gyors fejlődése egyre inkább
DOKTORI (PHD) ÉRTEKEZÉS CERNÁK ISTVÁN
 DOKTORI (PHD) ÉRTEKEZÉS CERNÁK ISTVÁN KESZTHELY 2008 2 PANNON EGYETEM GEORGIKON MEZŐGAZDASÁGTUDOMÁNYI KAR Növénytudományi és Biotechnológia Tanszék Növénytermesztés és Kertészeti Tudományok Doktori Iskola
DOKTORI (PHD) ÉRTEKEZÉS CERNÁK ISTVÁN KESZTHELY 2008 2 PANNON EGYETEM GEORGIKON MEZŐGAZDASÁGTUDOMÁNYI KAR Növénytudományi és Biotechnológia Tanszék Növénytermesztés és Kertészeti Tudományok Doktori Iskola
A SZIE, MKK GENETIKA ÉS BIOTECHNOLÓGIAI INTÉZETÉNEK EREDMÉNYEI A NÖVÉNYNEMESÍTÉS TUDOMÁNYOKBAN ÉS A NEMESÍTÉS UTÁNPÓTLÁS NEVELÉSBEN
 A SZIE, MKK GENETIKA ÉS BIOTECHNOLÓGIAI INTÉZETÉNEK EREDMÉNYEI A NÖVÉNYNEMESÍTÉS TUDOMÁNYOKBAN ÉS A NEMESÍTÉS UTÁNPÓTLÁS NEVELÉSBEN Heszky László és Kiss Erzsébet Keszthely 2013 PARADIGMAVÁLTÁS AZ ÉLETTUDOMÁNYOKBAN
A SZIE, MKK GENETIKA ÉS BIOTECHNOLÓGIAI INTÉZETÉNEK EREDMÉNYEI A NÖVÉNYNEMESÍTÉS TUDOMÁNYOKBAN ÉS A NEMESÍTÉS UTÁNPÓTLÁS NEVELÉSBEN Heszky László és Kiss Erzsébet Keszthely 2013 PARADIGMAVÁLTÁS AZ ÉLETTUDOMÁNYOKBAN
Mutáció detektáló módszerek
 Mutáció detektáló módszerek Molekuláris genetikai vizsgáló módszerek 2014.03.19. Bármilyen eltérés a referencia szekvenciától Lehet Egy bázispárnyi szubsztitúció, deléció, inzerció Kromoszóma deléció,
Mutáció detektáló módszerek Molekuláris genetikai vizsgáló módszerek 2014.03.19. Bármilyen eltérés a referencia szekvenciától Lehet Egy bázispárnyi szubsztitúció, deléció, inzerció Kromoszóma deléció,
Gabonafélék hidegadaptálódását befolyásoló gének térképezése és molekuláris markerezése
 Gabonafélék hidegadaptálódását befolyásoló gének térképezése és molekuláris markerezése Doktori értekezés Tóth Balázs Gödöllő 2003. 1 Doktori iskola: Vezetője: Tudományága: Program: Programvezető: Témavezető:
Gabonafélék hidegadaptálódását befolyásoló gének térképezése és molekuláris markerezése Doktori értekezés Tóth Balázs Gödöllő 2003. 1 Doktori iskola: Vezetője: Tudományága: Program: Programvezető: Témavezető:
Tipizálási módszerek alkalmazása methicillin-rezisztens Staphylococcus aureus (MRSA) törzsek molekuláris epidemiológiai vizsgálatai során
 Tipizálási módszerek alkalmazása methicillin-rezisztens Staphylococcus aureus (MRSA) törzsek molekuláris epidemiológiai vizsgálatai során Ungvári Erika, Tóth Ákos Magyar Infektológiai és Klinikai Mikrobiológiai
Tipizálási módszerek alkalmazása methicillin-rezisztens Staphylococcus aureus (MRSA) törzsek molekuláris epidemiológiai vizsgálatai során Ungvári Erika, Tóth Ákos Magyar Infektológiai és Klinikai Mikrobiológiai
NÖVÉNYNEMESÍTÉS. Az Agrármérnöki MSc szak tananyagfejlesztése TÁMOP /1/A
 NÖVÉNYNEMESÍTÉS Az Agrármérnöki MSc szak tananyagfejlesztése TÁMOP-4.1.2-08/1/A-2009-0010 Előadás áttekintése A növénynemesítés és molekuláris biológia története A biotechnológia jelentősége a növénynemesítésben
NÖVÉNYNEMESÍTÉS Az Agrármérnöki MSc szak tananyagfejlesztése TÁMOP-4.1.2-08/1/A-2009-0010 Előadás áttekintése A növénynemesítés és molekuláris biológia története A biotechnológia jelentősége a növénynemesítésben
Kvantitatív genetikai alapok április
 Kvantitatív genetikai alapok 2018. április A vizsgálható tulajdonságok köre: egyed - szám Egyedek morfológiai tulajdonságai: testméretek, arányok, testtömeg Egyedek fiziológiai tulajdonságai: vérnyomás,
Kvantitatív genetikai alapok 2018. április A vizsgálható tulajdonságok köre: egyed - szám Egyedek morfológiai tulajdonságai: testméretek, arányok, testtömeg Egyedek fiziológiai tulajdonságai: vérnyomás,
R. W. Allard (1996) Nemesítési haladás
 Főbb trendek a növénynemesítésben Termesztett populáció növények heterogenitás R. W. Allard (1996) Tájfajta igen nagy Genetikai elszegényedés és a hasznos gének akkumulációja Régi fajtapopulációk nagy
Főbb trendek a növénynemesítésben Termesztett populáció növények heterogenitás R. W. Allard (1996) Tájfajta igen nagy Genetikai elszegényedés és a hasznos gének akkumulációja Régi fajtapopulációk nagy
Bioinformatika - egészséges környezet, egészséges élelmiszer
 CESCI - III. SZENTGOTTHÁRDI SZLOVÉN MAGYAR FÓRUM Bioinformatika - egészséges környezet, egészséges élelmiszer Pannon Bio-Innováció Kft Taller János, PhD ügyvezető Szentgotthárd, 2017. május 23. 1 Pannon
CESCI - III. SZENTGOTTHÁRDI SZLOVÉN MAGYAR FÓRUM Bioinformatika - egészséges környezet, egészséges élelmiszer Pannon Bio-Innováció Kft Taller János, PhD ügyvezető Szentgotthárd, 2017. május 23. 1 Pannon
Orvosi Genomika. 1. Trendek a modern orvostudományban. 2. Genomika és modern orvostudomány
 1 1. ORVOSI GENOMIKA Orvosi Genomika 1. Trendek a modern orvostudományban DIA 1 Az emberi genom megfejtése óta eltelt 10 évben rendkívül gyors fejlődés ment végbe a genomikai technikákban, ill. a genomikai
1 1. ORVOSI GENOMIKA Orvosi Genomika 1. Trendek a modern orvostudományban DIA 1 Az emberi genom megfejtése óta eltelt 10 évben rendkívül gyors fejlődés ment végbe a genomikai technikákban, ill. a genomikai
Szakkifejezések III.
 a hónap témája BIOTECHNOLÓGIA ROVA TVEZETŐ: Dr. Heszky László akadémikus Az előző 11-16. részekben a transzgénikus (GM) fajták nemesítésének alternatív módszereit mutattuk be. A korábbi I-II. fejezetekhez
a hónap témája BIOTECHNOLÓGIA ROVA TVEZETŐ: Dr. Heszky László akadémikus Az előző 11-16. részekben a transzgénikus (GM) fajták nemesítésének alternatív módszereit mutattuk be. A korábbi I-II. fejezetekhez
Evolúció. Dr. Szemethy László egyetemi docens Szent István Egyetem VadVilág Megőrzési Intézet
 Evolúció Dr. Szemethy László egyetemi docens Szent István Egyetem VadVilág Megőrzési Intézet Mi az evolúció? Egy folyamat: az élőlények tulajdonságainak változása a környezethez való alkalmazkodásra Egy
Evolúció Dr. Szemethy László egyetemi docens Szent István Egyetem VadVilág Megőrzési Intézet Mi az evolúció? Egy folyamat: az élőlények tulajdonságainak változása a környezethez való alkalmazkodásra Egy
BEVEZETÉS CÉLKITŰZÉS
 BEVEZETÉS A molekuláris biológiai és genetikai módszerek gyors fejlődése egyre inkább tért hódít a növénynemesítés különböző területein, így a kukoricanemesítésben is. A növényi fenotípusos jellemzők és
BEVEZETÉS A molekuláris biológiai és genetikai módszerek gyors fejlődése egyre inkább tért hódít a növénynemesítés különböző területein, így a kukoricanemesítésben is. A növényi fenotípusos jellemzők és
Prenatalis diagnosztika lehetőségei mikor, hogyan, miért? Dr. Almássy Zsuzsanna Heim Pál Kórház, Budapest Toxikológia és Anyagcsere Osztály
 Prenatalis diagnosztika lehetőségei mikor, hogyan, miért? Dr. Almássy Zsuzsanna Heim Pál Kórház, Budapest Toxikológia és Anyagcsere Osztály Definíció A prenatális diagnosztika a klinikai genetika azon
Prenatalis diagnosztika lehetőségei mikor, hogyan, miért? Dr. Almássy Zsuzsanna Heim Pál Kórház, Budapest Toxikológia és Anyagcsere Osztály Definíció A prenatális diagnosztika a klinikai genetika azon
Evolúció. Dr. Szemethy László egyetemi docens Szent István Egyetem VadVilág Megőrzési Intézet
 Evolúció Dr. Szemethy László egyetemi docens Szent István Egyetem VadVilág Megőrzési Intézet Mi az evolúció? Egy folyamat: az élőlények tulajdonságainak változása a környezethez való alkalmazkodásra Egy
Evolúció Dr. Szemethy László egyetemi docens Szent István Egyetem VadVilág Megőrzési Intézet Mi az evolúció? Egy folyamat: az élőlények tulajdonságainak változása a környezethez való alkalmazkodásra Egy
Algoritmusok Tervezése. 9. Előadás Genetikus Algoritmusok Dr. Bécsi Tamás
 Algoritmusok Tervezése 9. Előadás Genetikus Algoritmusok Dr. Bécsi Tamás Biológiai háttér (nagyvonalúan) A sejt genetikai információit hordozó DNS általában kromoszómának nevezett makromolekulákba van
Algoritmusok Tervezése 9. Előadás Genetikus Algoritmusok Dr. Bécsi Tamás Biológiai háttér (nagyvonalúan) A sejt genetikai információit hordozó DNS általában kromoszómának nevezett makromolekulákba van
Genetikai panel kialakítása a hazai tejhasznú szarvasmarha állományok hasznos élettartamának növelésére
 Genetikai panel kialakítása a hazai tejhasznú szarvasmarha állományok hasznos élettartamának növelésére Dr. Czeglédi Levente Dr. Béri Béla Kutatás-fejlesztés támogatása a megújuló energiaforrások és agrár
Genetikai panel kialakítása a hazai tejhasznú szarvasmarha állományok hasznos élettartamának növelésére Dr. Czeglédi Levente Dr. Béri Béla Kutatás-fejlesztés támogatása a megújuló energiaforrások és agrár
Algaközösségek ökológiai, morfológiai és genetikai diverzitásának összehasonlítása szentély jellegű és emberi használatnak kitett élőhelykomplexekben
 Algaközösségek ökológiai, morfológiai és genetikai diverzitásának összehasonlítása szentély jellegű és emberi használatnak kitett élőhelykomplexekben Duleba Mónika Környezettudományi Doktori Iskola I.
Algaközösségek ökológiai, morfológiai és genetikai diverzitásának összehasonlítása szentély jellegű és emberi használatnak kitett élőhelykomplexekben Duleba Mónika Környezettudományi Doktori Iskola I.
Humán genom variációk single nucleotide polymorphism (SNP)
 Humán genom variációk single nucleotide polymorphism (SNP) A genom ~ 97 %-a két különböző egyedben teljesen azonos ~ 1% különbség: SNP miatt ~2% különbség: kópiaszámbeli eltérés, deléciók miatt 11-12 millió
Humán genom variációk single nucleotide polymorphism (SNP) A genom ~ 97 %-a két különböző egyedben teljesen azonos ~ 1% különbség: SNP miatt ~2% különbség: kópiaszámbeli eltérés, deléciók miatt 11-12 millió
Nemesítési haladás. Főbb trendek a növénynemesítésben. R. W. Allard (1996) Genetikai elszegényedés és a hasznos gének akkumulációja.
 Főbb trendek a növénynemesítésben R. W. Allard (1996) Termesztett növények Tájfajta Régi fajtapopulációk populáció heterogenitás igen nagy nagy Genetikai elszegényedés és a hasznos gének akkumulációja
Főbb trendek a növénynemesítésben R. W. Allard (1996) Termesztett növények Tájfajta Régi fajtapopulációk populáció heterogenitás igen nagy nagy Genetikai elszegényedés és a hasznos gének akkumulációja
A DNS szerkezete. Genom kromoszóma gén DNS genotípus - allél. Pontos méretek Watson genomja. J. D. Watson F. H. C. Crick. 2 nm C G.
 1955: 46 emberi kromoszóma van 1961: mrns 1975: DNS szekvenálás 1982: gén-bank adatbázisok 1983: R (polymerase chain reaction) Mérföldkövek 1 J. D. Watson F. H.. rick 2008 1953 2003 Watson genomja DNS
1955: 46 emberi kromoszóma van 1961: mrns 1975: DNS szekvenálás 1982: gén-bank adatbázisok 1983: R (polymerase chain reaction) Mérföldkövek 1 J. D. Watson F. H.. rick 2008 1953 2003 Watson genomja DNS
Az evolúció folyamatos változások olyan sorozata, melynek során bizonyos populációk öröklődő jellegei nemzedékről nemzedékre változnak.
 Evolúció Az evolúció folyamatos változások olyan sorozata, melynek során bizonyos populációk öröklődő jellegei nemzedékről nemzedékre változnak. Latin eredetű szó, jelentése: kibontakozás Időben egymást
Evolúció Az evolúció folyamatos változások olyan sorozata, melynek során bizonyos populációk öröklődő jellegei nemzedékről nemzedékre változnak. Latin eredetű szó, jelentése: kibontakozás Időben egymást
A humán mitokondriális genom: Evolúció, mutációk, polimorfizmusok, populációs vonatkozások. Egyed Balázs ELTE Genetikai Tanszék
 A humán mitokondriális genom: Evolúció, mutációk, polimorfizmusok, populációs vonatkozások Egyed Balázs ELTE Genetikai Tanszék Endoszimbiotikus gén-transzfer (Timmis et al., 2004, Nat Rev Gen) Endoszimbiotikus
A humán mitokondriális genom: Evolúció, mutációk, polimorfizmusok, populációs vonatkozások Egyed Balázs ELTE Genetikai Tanszék Endoszimbiotikus gén-transzfer (Timmis et al., 2004, Nat Rev Gen) Endoszimbiotikus
TEMATIKA Biokémia és molekuláris biológia IB kurzus (bb5t1301)
 Biokémia és molekuláris biológia I. kurzus (bb5t1301) Tematika 1 TEMATIKA Biokémia és molekuláris biológia IB kurzus (bb5t1301) 0. Bevezető A (a biokémiáról) (~40 perc: 1. heti előadás) A BIOkémia tárgya
Biokémia és molekuláris biológia I. kurzus (bb5t1301) Tematika 1 TEMATIKA Biokémia és molekuláris biológia IB kurzus (bb5t1301) 0. Bevezető A (a biokémiáról) (~40 perc: 1. heti előadás) A BIOkémia tárgya
A HUMÁNGENETIKA LEGÚJABB EREDMÉNYEI Péterfy Miklós
 A HUMÁNGENETIKA LEGÚJABB EREDMÉNYEI Péterfy Miklós Összefoglalás A humángenetika korunk egyik legdinamikusabban fejlődő tudományága. Ennek a fejlődésnek legfőbb mozgatórugója az, hogy a humángenetika,
A HUMÁNGENETIKA LEGÚJABB EREDMÉNYEI Péterfy Miklós Összefoglalás A humángenetika korunk egyik legdinamikusabban fejlődő tudományága. Ennek a fejlődésnek legfőbb mozgatórugója az, hogy a humángenetika,
molekuláris (RAPD, SSR, AFLP és SCAR) jellemzése.
 1 Különböző nyár, búza és kender genotípusok molekuláris (RAPD, SSR, AFLP és SCAR) jellemzése. Doktori értekezés Törjék Ottó Gödöllő 2001. 2 Doktori iskola: Vezetője: Tudományága: Program: Programvezető:
1 Különböző nyár, búza és kender genotípusok molekuláris (RAPD, SSR, AFLP és SCAR) jellemzése. Doktori értekezés Törjék Ottó Gödöllő 2001. 2 Doktori iskola: Vezetője: Tudományága: Program: Programvezető:
Genetika. Tartárgyi adatlap: tantárgy adatai
 Genetika Előadás a I. éves Génsebészet szakos hallgatók számára Tartárgyi adatlap: tantárgy adatai 2.1. Tantárgy címe Genetika 2.2. Előadás felelőse Dr. Mara Gyöngyvér, docens 2.3. Egyéb oktatási tevékenységek
Genetika Előadás a I. éves Génsebészet szakos hallgatók számára Tartárgyi adatlap: tantárgy adatai 2.1. Tantárgy címe Genetika 2.2. Előadás felelőse Dr. Mara Gyöngyvér, docens 2.3. Egyéb oktatási tevékenységek
A géntechnológia genetikai alapjai (I./3.)
 Az I./2. rész (Gének és funkciójuk) rövid összefoglalója A gének a DNS információt hordozó szakaszai, melyekben a 4 betű (ATCG) néhány ezerszer, vagy százezerszer ismétlődik. A gének önálló programcsomagként
Az I./2. rész (Gének és funkciójuk) rövid összefoglalója A gének a DNS információt hordozó szakaszai, melyekben a 4 betű (ATCG) néhány ezerszer, vagy százezerszer ismétlődik. A gének önálló programcsomagként
A Hardy Weinberg-modell gyakorlati alkalmazása
 1 of 6 5/16/2009 2:59 PM A Hardy Weinberg-modell gyakorlati alkalmazása A genotípus-gyakoriság megoszlásának vizsgálata 1. ábra. A Hardy Weinberg-egyensúlyi genotípus-gyakoriságok az allélgyakoriság Számos
1 of 6 5/16/2009 2:59 PM A Hardy Weinberg-modell gyakorlati alkalmazása A genotípus-gyakoriság megoszlásának vizsgálata 1. ábra. A Hardy Weinberg-egyensúlyi genotípus-gyakoriságok az allélgyakoriság Számos
PÉCSI TUDOMÁNYEGYETEM. Természetes és mesterséges agrobaktérium rezisztencia vizsgálata szőlőben. Galambos Anikó
 PÉCSI TUDOMÁNYEGYETEM Biológiai Doktori Iskola Genetika program Természetes és mesterséges agrobaktérium rezisztencia vizsgálata szőlőben PhD értekezés tézisei Galambos Anikó Témavezető: Dr. Putnoky Péter
PÉCSI TUDOMÁNYEGYETEM Biológiai Doktori Iskola Genetika program Természetes és mesterséges agrobaktérium rezisztencia vizsgálata szőlőben PhD értekezés tézisei Galambos Anikó Témavezető: Dr. Putnoky Péter
Biológiai feladatbank 12. évfolyam
 Biológiai feladatbank 12. évfolyam A pedagógus neve: A pedagógus szakja: Az iskola neve: Műveltségi terület: Tantárgy: A tantárgy cél és feladatrendszere: Tantárgyi kapcsolatok: Osztály: 12. Felhasznált
Biológiai feladatbank 12. évfolyam A pedagógus neve: A pedagógus szakja: Az iskola neve: Műveltségi terület: Tantárgy: A tantárgy cél és feladatrendszere: Tantárgyi kapcsolatok: Osztály: 12. Felhasznált
Mendeli genetika, kapcsoltság 26
 Mendeli genetika, kapcsoltság 26 6. MENDELI GENETIKA. KAPCSOLT- SÁG ÉS GÉNTÉRKÉPEK. Mendel szabályai. Az örökl dés típusai. Független kombinálódás. Kapcsoltság, crossing over és géntérképek. egyformák.
Mendeli genetika, kapcsoltság 26 6. MENDELI GENETIKA. KAPCSOLT- SÁG ÉS GÉNTÉRKÉPEK. Mendel szabályai. Az örökl dés típusai. Független kombinálódás. Kapcsoltság, crossing over és géntérképek. egyformák.
Számítógépes döntéstámogatás. Genetikus algoritmusok
 BLSZM-10 p. 1/18 Számítógépes döntéstámogatás Genetikus algoritmusok Werner Ágnes Villamosmérnöki és Információs Rendszerek Tanszék e-mail: werner.agnes@virt.uni-pannon.hu BLSZM-10 p. 2/18 Bevezetés 1950-60-as
BLSZM-10 p. 1/18 Számítógépes döntéstámogatás Genetikus algoritmusok Werner Ágnes Villamosmérnöki és Információs Rendszerek Tanszék e-mail: werner.agnes@virt.uni-pannon.hu BLSZM-10 p. 2/18 Bevezetés 1950-60-as
6. Az öröklődés alapjai
 6. z öröklődés alapjai GENETIK z örökítő anyag szerveződésével és funkciójával, az élőlények tulajdonságinak átöröklődésével foglalkozó tudomány. genesis: : születés, teremtés, keletkezés, származás Elméletek
6. z öröklődés alapjai GENETIK z örökítő anyag szerveződésével és funkciójával, az élőlények tulajdonságinak átöröklődésével foglalkozó tudomány. genesis: : születés, teremtés, keletkezés, származás Elméletek
