HÍRLEVÉL. ZöldGMO. I. évfolyam 1. szám OKTÓBER
|
|
|
- Jenő Somogyi
- 8 évvel ezelőtt
- Látták:
Átírás
1 ZöldGMO HÍRLEVÉL I. évfolyam 1. szám OKTÓBER A kiadvány a TÁMOP / projekt keretében, az Európai Unió támogatásával, az Európai Szociális Alap és az Európai Regionális Fejlesztési Alap társfinanszírozásával valósult meg. 1
2 ZöldGMO HÍRLEVÉL Kiadja: MTA Szegedi Biológiai Központ Felelős szerkesztő: Dudits Dénes I. évfolyam 1. szám OKTÓBER Dudits Dénes: Génizolálási stratégiák: a búzagének funkcionális elemei Búzagének kémcsőben Amint azt az 1. ábra szemlélteti, a gének misztikus világa a 80-as évekig rejtve maradt a búzanemesítő előtt, aki ha összehasonlít egy tar és egy szálkás kalászt, arra következtethet, hogy különböző gének hatásának eredménye ez a szembetűnő különbség. A törpeségi gén jelenlétét feltételezheti a rövid és vastag szalmájú fajták esetében. Mutánsok hirtelen fellépő tulajdonságai a gének hibáira utalnak. A keresztezést követően tapasztalt hasadási arányokból öröklődési törvényszerűségeket lehetett felismerni, domináns és recesszív tulajdonságokat megkülönböztetni. A több évszázados gyakorlat szerint a növények nemesítője a felszínen megjelenő tulajdonságokat értékeli, amikor kiválasztja a számára előnyös sajátosságokkal rendelkező növényegyedeket. A fajta-előállítás során génekben gondolkodik és a hasznos gének felhasználására törekszik, bár nincs közvetlen információja a gének jelenlétéről és működésük mikéntjéről. A 90-es évektől az intuitív, tapasztalati nemesítés a rekombináns DNS módszereknek köszönhetően mindinkább támaszkodhat a génekkel kapcsolatos DNS-szintű molekuláris adatokra, és ezzel hatékonyabbá és megbízhatóbbá válik a kedvező génkombinációk kialakítása. Egyrészt mód nyílik agronómiai értékekkel bíró gének izolálására, a DNS-molekulák kémcsőben történő átalakítására, majd a búzába történő visszaépítésére, másrészt a nemesítés szempontjából fontos tulajdonságok génjeihez kapcsolt DNS-markerek azonosítására. Mind a transzgenikus tenyészanyagok (GM-növények) létrehozása, mind a DNS-markerekkel végzett szelekció a nemesítés precizitásának növeléséhez vezet. Így a génmanipuláció művészetét mindinkább a pontos tervezés, a mérnöki tudatosság váltja fel. Napjainkban szemtanúi, részesei vagyunk a molekuláris nemesítés térhódításának, és az elkövetkező években lehet majd értékelni, hogy az új, DNS alapú technológiák milyen versenyelőnyt nyújthatnak a születő fajták nemesítésében, amikor nem csak a helyi, hanem a globális tényezők is szerephez jutnak a vetőmagpiacon. A rekombináns DNS-módszerek kidolgozását a fágokkal, baktériumokkal végzett kutatások a 70-es években tették lehetővé. A DNS-láncot szekvenciaspecifikusan feldaraboló restrikciós enzimek mellett a DNS-fragmenteket összekapcsoló ligázok szükségesek új DNS-molekulák létrehozásához. Az in vitro kialakított DNS-molekulák baktériumokban, élesztőben felszaporíthatók, és a több millió kolónia között azonosíthatók a kívánt gént hordozó sejtek. Ezekben a mikroorganizmusokban megsokszorozott DNS-molekulák nukleotidsorrendjének meghatározását teszik lehetővé a modern DNS-szekvenátorok. Egyes gének vagy a teljes genom DNS-szekvenciájának ismerete kiindulási alapot ad a funkcionális elemek azonosításához, a kódolt fehérjék aminosav-sorrendjének, majd szerkezetének predikciójához. Néhány éves késéssel kezdték a molekuláris biológusok a rekombináns-dns-módszereket felhasználni növényi gének izolálására, és ezzel egy új korszak kezdődött nem csak az alapkutatásban, hanem az alkalmazott növénytudományokban, mint amilyen a növénynemesítés is. Lényegében a 80-as évek kezdetétől beszélhetünk a növényi génsebészetről, aminek köszönhetően alapjaiban új világ tárult fel a növények életjelenségeinek megismerésében. Magától értetődő, hogy a génekről és azok működéséről szerzett információk közvetlenül 2
3 felhasználhatók az agronómiai szempontból fontos tulajdonságok javítására. Búzagének izolálásáról szóló beszámoló korai közlemények a 80-as évek közepén jelentek meg. Metodikai könnyebbségből fakadóan elsők között a búza ribulóz-1,5-bifoszfátkarboxiláz (RUBISCO) fotoszintetikus fehérje-kisalegység génjét (BROGLIE és mtsai, 1983), valamint a gliadin és glutenin raktározott magfehérjét kódoló géneket sikerült megklónozni (ANDERSON és mtsai, 1984; FORDE és mtsai, 1985). Szintén az mrns-molekulák nagy példányszámú előfordulása segítette a búza hisztongénjeinek izolálását és részletes jellemzését (TABATA és mtsai, 1983). Ezekkel az úttörő kísérletekkel indult el az egyre terebélyesedő génsebészeti tevékenység, amely mára búzagenomikai programokhoz vezetett. A következő elemzések betekintést adnak egyrészt a búzagenom szerveződését feltáró molekuláris adatok rendszerébe, a búza génjeinek azonosítását szolgáló metodikai lehetőségek folyamatos bővülésének mikéntjébe. A búzagének funkcióit irányító szerkezeti elemek szerepének megértése feltételezi a kromatin szerveződéséből adódó szabályozási mechanizmusok figyelembevételét is. A búzagenom szerveződése mint evolúciós következmény A mindennapi kenyerünket adó fűféle, a búza igen bonyolult, máig sok vonatkozásban tisztázatlan, fejlődési utat tett meg, aminek folyamán jelenlegi génállománya három faj integrációjával jött létre, mint azt GILL és mtsai (2004) nyomán az 1. ábra szemlélteti. Hozzávetőlegesen 3 millió évvel ezelőtt egy közös őstől eredően jelentek meg a Triticum és Aegilops fajok, amelyek aztán többlépcsős poliploidizációs folyamat szereplőiként félmillió évvel ezelőtt először a tetraploid Triticum turgidum (AABB) faj kialakulásában vettek részt. A legújabb molekuláris evolúciós vizsgálatok alapján, amikor a gének nukleotidszekvenciájának összevetésével állítják fel a filogenetikai rokonsági fokokat, megerősítést nyert a T. urartu (AA) mint az A genom forrása a tetraploid búzában (HUANG és mtsai, 2002). A citoplazmatikus acetil-coa-karboxilázt és a 3-foszfoglicerin kinázt kódoló gének szekvenciahomológiája alapján a T. monococcum (A m A m ) és T. urartu (AA) közeli rokon fajoknak tekinthetők, továbbá a hexaploid (AABBDD) búza (T. aestivum) B genomja az A. speltoides, D genomja pedig az A. tauschii (DD) fajtól származik. Ez a második poliploidizáció kb. 8 ezer évvel ezelőtt következhetett be. Az egymástól különböző genomok összeépülésével megvalósult alloploidizáció hatalmas, 16 ezer Mb méretű búzagenom kialakulásához vezetett, ami 40-szer nagyobb, mint a rizs genomja. Maga a poliploidizáció, amely kromoszómaszegmensek duplikációját jelenti, gyors és visszafordíthatatlan genomváltozások kiváltásával járulhat hozzá az új faj kialakulásához. OZKAN és mtsai (2001) mind a kromoszóma-, mind a genomspecifikus DNS-vesztésnek kiemelt jelentőséget tulajdonítanak, hiszen ezek teszik lehetővé a szülői genomok gyors egymáshoz való adaptációját, például a diploid típusú meiotikus események réven. Hasonlóan fontos szerepet játszik a transzpozonelemek (TE) megsokszo- 1. ábra: A citológiai vizsgálatok mellett a molekuláris térképek és szekvenálási adatok is segítenek a hexaploid (2n=42) kenyérbúza kialakulásának mikéntjét feltárni. Az alloploidizációra épülő fejlődési út a Triticum urartu (AA) és az Ae. speltoides (SS) fajjal rokon, de ismeretlen faj (BB) kereszteződésével kezdődött. Az így létrejött tetraploid búza a (Tr. turgidum, AABB) az Ae. tauschii (DD) fajjal 8 ezer évvel ezelőtt kereszteződött kialakítva kenyérbúzánkat, amelynek genomja 16 ezer Mb nagyságú (GILL és mtsai, 2004). 3
4 rozódása is. Az A. tauschii genomszekvenálása alapján a D genomot kb. 90%-ban ismétlődő szekvenciák alkotják, és kb. 70%-ban ismert TEből áll (LI és mtsai, 2004). 2,5%-ra becsülhető az ismert gének száma, és további 5,9%-ban azonosíthatók a kis kópiaszámú, ismeretlen funkciójú szekvenciák. A diploid A. tauschii (DD) genomja kb. 4 ezer Mb nagyságú, így viszonylagos kisebb mérete révén előnyösen használható a genomkutatásban (ARUMUGANATHAN és EARLE, 1991). Tekintettel az ismétlődő szekvenciák igen nagy arányára, mind a molekuláris nemesítés, mind a funkcionális genomika szempontjából meghatározó jelentőségű a génekben gazdag régiók (gene-rich regions, GRRs) behatárolása az egyes búzakromoszómákon. Már a kromoszómadeléciós vonalakkal végzett fizikai géntérképezési adatok rámutattak arra, hogy a búza esetében is a gének többsége csoportosan, gyakran a kromoszómakarok végéhez közeli régiókban találhatók (GILL és mtsai, 1996 a, b). Ezzel az eloszlási mintázattal a rekombinációk gyakorisága is korrelál. Így mind a relatív génsűrűség, mind a rekombinációk száma növekszik a centromertől mért relatív távolsággal (AKHUNOV és mtsai, 2003). A legújabb fizikai térképezési kutatások több mint 3 ezer gén markerdeléciós töréspontokhoz viszonyított helyének kijelölését tették lehetővé, ezzel alapvető információval szolgáltak a búzagenom szerveződéséről. ERAYMAN és mtsai (2004) 18 nagyobb és 30 kisebb génekben gazdag régiót azonosítottak, amelyek a gének 94%-át hordozó kromoszómaszakaszoknak felelnek meg. Valamennyi nagy, génben gazdag régió a kromoszómavégeken található, ahol a kromoszóma-töréspontok gyakorisága is nagyobb. A szerzők ezerre becsülik a búza génjeinek számát, és Mb méretűre az általuk elfoglalt genomrészt. Hosszabb DNS-régiók nukleotidszekvenciájának meghatározásával részletes szerkezetvizsgálatokat és elemzéseket is el lehet végezni. Mint azt a 2. ábra bemutatja, GU és mtsai (2004) megszekvenálták a HMW-glutein gént hordozó kromoszómarégiót a T. turgidum A és B genomja, valamint az A. tauschii D genomja esetében. A 2. ábra szemlélteti az árpa D-hordein gént tartalmazó régiójának szerveződését is. 2. ábra: A nagy molekulasúlyú (HMW) glutenint kódoló genomikus régió szerveződése a három búza- és az árpagenomban. A gének sorrendje megőrződött az evolúció során, a gének közötti DNS-szakaszok viszont jelentősen átrendeződtek. (GU és mtsai, 2004) A kb kiterjedésű DNS-szakaszokon 6 gént lehetett azonosítani. Bár a gének sorrendje azonos a vizsgált genomok esetében, a nem működő gének nagy aránya (4 inaktív gén), valamint a gének közötti távolságok különbözősége jelentős retrotranszpozon aktivitást tükröznek. Ezzel magyarázható a gének közötti szakaszok nukleotidszekvenciájának eltérése az egyes búzagenomokban. A Fűfélék Törzsfejlődési Munkacsoport (Grass Phylogeny Working Group GPWG: www. virtualherbarium.org/gpwg által javasolt filogenetikai fa a fajok kialakulásának viszonylagos sorrendjét mutatja be (3. ábra; KELLOGG, 2001). 4
5 3. ábra: A fontosabb egyszikű gabonafélék rokonsági kapcsolatát mutatja be a törzsfejlődési fa, amely egy millió évvel ezelőtti ősből indul ki. A búza és árpa közel rokon fajoknak tekinthetők, amelyek 11 millió évvel ezelőtt különültek el (KELLOG, 2001) Az egyetlen elágazási ponton keresztül kapcsolódó fajok mint a búza és árpa, vagy a kukorica és cirok közeli rokonságúnak tekintendők. Az elágazási pontok számának növekedésével rendszertanilag távolodnak a fajok egymástól. GALE és DEVOS (1998) analízise szerint az árpa, búza és rizs közös őse millió évvel ezelőtt különült el attól a Panicoideae őstől, melyből a kukorica és cirok származtatható. A búza és az árpa szétválásának ideje millió évvel ezelőttre becsülhető (GILL és mtsai, 2004). Alapul véve a főbb gabonafélék rokonsági viszonyait, az összehasonlító genomika a megfelelőségek (syntheny) feltárásával segíti a genomok megismerését a fajok egyre bővülő körében. Jelenleg a legkimerítőbb adatbázisa a rizsgenomkutatásnak van. Ezért fontos értékelni, hogy milyen mértékben használhatók fel ezek az információk, pl. a búzagének vizsgálatához. A.3. ábrán látható törzsfa tanúsága szerint a búza és a rizs rokonsági kapcsolata közvetett, így csak részben ad lehetőséget a géntérképezési adatok kölcsönös figyelembevételére, mégis a nagy és komplex genomok megismerését, mint pl. a búzáé, előmozdíthatja kis genomú, diploid fajokkal történő összevetés. A keresztezésből származó hasadó populációkon végzett korai genetikai térképezések elsősorban az ismert fenotípusos bélyegek génjeinek, illetve a restrikciós fragmentek hosszát jellemző molekuláris markerek (RFLP markerek) kromoszómális elhelyezkedését határozták meg. Ezek a vizsgálatok olyan kromoszómarégiókat tártak fel, amelyeken belül a gének, markerek elrendeződése azonos volt rizsben és számos gabonaféle esetében, így a rizs szolgálhat referenciagenomként (GALE és DEVOS, 1998). A makroszinten megfigyelt génsorrendbeli megfelelőség igen egyértelmű pl., ha a rizs első kromoszómáját és a búza B genom harmadik kromoszómáját hasonlítjuk egymáshoz (4. ábra). 4. ábra: A rizs 1 és a búza 3 kromoszómák megfelelőségét (syntheny) mutatja, hogy 141 búza EST térképezhető a búza 3 kromoszómákra, melyeknek homológjai megtalálhatók a rizs 1 kromoszómán. Az ábra a búza 3B kromoszómán láthatók a deléciós pontok által behatárolt régiókat (bin) mutatja és megadja a rizsével homológ EST klónok számát (AKHUNOV és mtsai, 2003) A megegyezés kimutatható akár a búza-, akár a rizsgének ortológjait keressük a másik faj kromoszómáin, és a gének sorrendje is megegyezik. Ez a kolinearitás megerősíti az egyes gének azonosságát (AKHUNOV és mtsai, 2003). A rendelkezésre álló legújabb rizs- és búzagenom-összehasonlítások búzakromoszómánként adják meg azokat a kapcsoltsági régiókat, amelyek egy, vagy több rizskromoszómának felelnek meg (SORRELLS és mtsai, 2003; LA ROTA és mtsai, 2004). A DNS-szekvencia információkra alapozott genomösszehasonlítások a korábbi RFLP térképezési adatokhoz viszonyítva kisebb mértékű konzerváltságot és nagyobb evolúciós változékonyságot tártak fel a rizs és a búza rokonságát tekintve. Jelentős, kromoszómák közötti transzlokációs, inszerciós és deléciós eseményekre utal, hogy átlagosan az egykópiás gének 35%-a olyan búzakromoszóma-régiókban találhatók, amelyek nem homológjai a rizsrégióknak. A kolinearitás hiánya megnehezíti a rizs mint modellnövény használatát a búzagenomikában. LA ROTA és SORRELLS (2004) arra a következtetésre jutottak, hogy a rizs kromoszómái inkább hasonlítanak egy ősi Poaceae kromoszómakészlethez, mint a búzáéhoz. Természetesen azon búzagének esetében, amelyek konzervált kromoszómarégiókban helyezkednek el, a rizs genomszekvencia-adatok fontos támpontot jelentenek a gének izolálásához vagy molekuláris markerek azonosításához. 5
6 Genomikus klóntárak, szekvenálási programok A 16 ezer Mb méretű búzagenom megismerésének alapvető feltétele, hogy a sejtmagi DNS-t izolálás után darabonként lehessen kezelni, felszaporítani és vizsgálni. A növényi genomikus génkönyvtárakat leggyakrabban bakteriális mesterséges kromoszómák (bacterial artificial chromosome, BAC) felhasználásával hozták létre. Napjainkban a búzagenomikai kutatásokhoz több BAC génkönyvtár is rendelkezésre áll. Így ez ideig a T. monococcum (LIJAVETZKY és mtsai, 1999; STEIN és mtsai, 2000), a A. tauschii (MOULETT és mtsai, 1999, LI és mtsai, 2004), T. turgidum sp. durum (CENCI és mtsai, 2003) és T. aestivum (ALLOUIS és mtsai, 2003; WU és mtsai, 2004) fajokból készült BAC génkönyvtárakat használták fel ez ideig szekvenálási programokhoz. A kenyérbúza esetében a kromoszómaspecifikus BAC génkönyvtárakat is készítettek miután az egyes kromoszómákat szétválogatták (SAFAR és mtsai, 2004; JANDA és mtsai 2004). A BAC génkönyvtárak készítéséhez több száz kb nagyságú DNS-fragmentekre van szükség, amelyeket részleges HindIII emésztés után váltakozó irányú elektroforézissel (pulse-field gel electrophoresis) frakcionálnak, és a kb méretű fragmenteket ligálják be a HindIII enzimmel felnyitott vektormolekulákba, mint pl. pcld0541, vagy pyltac17 (MOULLET és mtsai, 1999; LIU és mtsai, 2000). A ligatumot E. coli sejtekbe elektroporálják, és szelektív táptalajon a búza DNS-t hordozó klónokat azonosítják. Az egyedi klónok vagy néhány száz BAC klónt tartalmazó keverékek mélyhűtve tárolhatók, és a belőlük izolált DNS felhasználásával búzagének azonosíthatók, akár hibridizációval, akár PCR amplifikációval. A több száz kb méretű egyedi BAC-klónok formájában felszaporított búza DNS-szakaszok megszekvenálásával jó betekintést nyerhetünk a búzagenom felépítését illetően, mint azt az előzőekben is bemutattuk. (WICKER és mtsai, 2001; LI és mtsai, 2004; GU és mtsai, 2004). A követezőkben a féltörpe búza kialakulásáért felelős genetikai tényezők példáján keresztül vázoljuk a genomika nyújtotta lehetőségeket. Az ún. Zöld forradalom sikerességében meghatározó szerepe volt a féltörpe búzafajták bevezetésének. Az Rht-1 gén jelenléte mérsékelt gibberellinsav-hatáshoz vezet a búzában, aminek következtében nagy termőképességű féltörpe változatok jöhetnek létre (GALE és GREGORY, 1977). A búza Rht-1 gén szerepét tekintve megfelel az Arabidopsis GA1 génnek, amely a gibberellinsav-válaszban szerepet játszó transzkripciós faktort kódol, és jellegzetes aminosav-motívuma van, aminek alapján meg lehetett találni a rizs, illetve búza megfelelő génjét (PENG és mtsai, 1999). Az Rht-1 gén egy mutáns allélnek felel meg, amelyben egyetlen bázisváltozás következtében stop kódon (TAG) alakult ki, így a transzkripciós faktor szintézise gátolt. Specifikus PCR primerek segítségével mind a B genomban, mind a D genomban azonosítható a mutáns gén, ezzel diagnosztizálhatók az egyes búzagenotípusok (ELLIS és mtsai, 2005). A leírt PCR markerekre alapozva WU és mtsai (2004) azonosították azt a BACklónt (BAC 179), amely az Rht-D1 gént hordozza. A 200 kb nagyságú régió megszekvenálása lehetővé tette a Rht-D1 gén megtalálásán túl a főbb strukturális jellemzők felismerését, illetve a rizs ortológ régióval való összehasonlítását. Az 5. ábra szemlélteti a BAC-klón második felén azonosított 5 gént, valamint a jelentős kiterjedésű retrotranszpozon szekvenciarészeket, melyeken belül meghatározó a Ty-1-kópia (Wis3), valamint Ty-3gypsy (Romani1) típusú mobil elemek előfordulása. 6
7 5. ábra: A búza törpeségét, gibberellin-érzéketlenségét meghatározó gént (Rht-D1) hordozó BAC klón (BAC1J9) szekvenciaadatai alapján 5 gén azonosítható a 200 kb hosszú szakasz végén. A transzpozonokat hordozó régiók jelentős kiterjedésűek. A búza és a rizs megfelelő DNS-szakaszait összehasonlítva a gének homológiája felismerhető, de két búzagénnek nincs meg a homológja rizsben (WU és mtsai, 2004) CDS5 szekvenciarész felel meg az Rht-D1 génnek nagy homológiát mutatva a rizs Os GA1 szekvenciával. Ezen a BAC-klónon 41,4 kb-ként lehetett gént azonosítani. Az 5. ábra azt is bemutatja, hogy a gibberellinre való érzéketlenségért és a növény magasságáért felelős kromoszómarégióban a gének sorrendje, transzkripciójuk iránya azonos mind a búzában, mind a rizsben. A két BAC-klón szekvenciájának összehasonlítása több kisebb kiterjedésű átrendeződést is feltárt. Így két búzagénnek (CDS3 és CDS4) nincs meg a homológja a rizsklónban. Egy aktív rizsgén megfelelője elveszett a búza ezen régiójából, és további 7 feltételezett rizsgén homológja sem azonosítható a búza BAC-klón szekvenciájában. Talán már az eddig bemutatott kísérleti eredmények, valamint a búzagenom szerveződését feltáró ismeretek is elég meggyőzően tanúsítják, hogy a genomikai megközelítés valóban egy teljesen új fejezet kezdetét jelenti a búzakutatásban, és ezzel alapjaiban módosul a búzanemesítés eszköztára. A kiemelt tudományos és gazdasági jelentőségnek köszönhetően egyre határozottabb szervezeti formát öltenek azok a nemzetközi együttműködések, amelyek végső célja a kenyérbúza genomjának megszekvenálása novemberében Washingtonban került sor A búzagenom-szekvenálási munkaértekezlet -re (Wheat Genome Sequencing Workshop). Az értekezletről készült beszámoló (GILL és mtsai, 2004) áttekinti a búzagenom szerveződésével kapcsolatos legújabb ismereteket, illetve a megfelelő genotípus és szekvenálási stratégia kiválasztásának szempontjait. Több megfontolás is alátámasztja azt a döntést, hogy a búzagenomszekvenálási programot a hexaploid kenyérbúzán, mégpedig Chinese Spring fajtán kívánják megvalósítani a Búza Genomszekvenálási Konzorcium (Wheat Genome Sequencing Consortium, WGSC) keretében (APPELS és mtsai, 2005). A program első 5 évét előkészítő szakasznak tekintve a célok között szerepel nagy pontosságú, átfogó fizikai térképek készítése, amelynek során az összetartozó BACklónok elrendezésén (BAC-contigs) túl, azok elhelyezése a részletes felbontású genetikai és deléciós térképeken mind a 21 búzakromoszóma esetében. Szükség van egyre növekvő számban a BAC-klónok megszekvenálására, elsődlegesen génekben gazdag kromoszómarégiókból. Mind a DNS metiláltsága, mind a DNS-fragmentek reasszociációs kinetikája alapján mód van a gént kódoló, kis kópiaszámú DNSdarabok feldúsítására (LI és mtsai, 2004). A programban szerepet kap az összehasonlító genomika, amely a rizs mellett az árpa szekvenciainformációkra is támaszkodhat. Bár a konzorcium nemzetközi, és nyitott minden érdekelt kutatási egység számára, az európai részvétel jelentősen kisebb, mint az amerikai aktivitás. Jelenleg nehéz a hazai laboratóriumok bekapcsolódásának lehetőségeit meghatározni. Ennek ellenére az ilyen törekvések elkerülhetetlenek, ha a magyar búzanemesítés élni kíván a genomika által nyújtott csúcstechnológiák előnyeivel. Génvadászat géntérképek segítségével A molekuláris növénynemesítés nyújtotta lehetőségek sorában kiemelt fontosságú, hogy a DNS alapú molekuláris markerek sorozata biztosíthatja a fenotípusos bélyegek és a gének közötti kapcsoltság követését. Még az olyan összetett agronómiai tulajdonságok, mint a termőképesség, vagy a szárazságtűrés, virágzási idő esetében is mód nyílik a mennyiségi bélyegek kialakításban meghatározó szerepet játszó kromoszómarégiók behatárolására (Quantitative Trait Loci, QTL). A genetikai variabilitás kimutatására leggyakrabban használt markerek közé tartoznak azok, amelyek követik a restrikciós fragmentek hosszában mutatkozó polimorfizmust (RFLP), az amplifikált fragmentek polimorfizmusát (AFLP) vagy az egyszerű nukleotidszekvencia-ismétlődéseket (Simple Sequence Repeats, SSRs). A búza esetében a molekuláris markerek genetikai térképezését, a kromoszómális lokalizációt segítik a Chinese Spring fajta deléciós vonalai. Az egyes allélek közötti különbségtételben, vagy a hexaploid búza 3 7
8 genomjában található gének megkülönböztetésében az egyes nukleotideltérések (Single Nucleotide Polymorphysm, SNPs) kimutatására lehet támaszkodni. A kalászos gabonafélék, és így a búza genetikai térképezésében elért legújabb eredményeket kiterjedt adatbázisok teszik közzé (MATTHEWS és mtsai, 2003; A hagyományos nemesítés a fenotípus értékelésére támaszkodik, és így bizonyos határon túl korlátozódik az értékelhető allélkombinációk számának növelése. A molekuláris markerekre épülő nemesítés (Marker-Assited Breeding, MAB) nagyobb számú szülői allél vizsgálhatóságát teszi lehetővé, és így növelhető a genetikai előrehaladás eredményessége. A 6. ábra a szülői allélszám és a genetikai javulás összefüggését szemlélteti a két nemesítési stratégia összehasonlításában (FRANCKI és APPLES, 2002 nyomán). Ahhoz, hogy a hazai nemesítési programokban a mainál jelentősebb szerepet kapjon a molekuláris markerek használata, infrastrukturális fejlesztések is szükségesek lennének, többek között a nagy mintaszám értékeléséhez elengedhetetlen automatizált rendszerek használata. A minták számát különösen megemeli, ha a molekuláris markerekre építjük viszszakeresztezési programokat vagy a vad fajokból történő génátépítést. A részleteiben egyre bővülő genetikai és molekuláris térképek mind nagyobb lehetőséget adnak agronómiai értéket hordozó gének izolálására. Így vált lehetővé pl. a levélrozsda-rezisztenciagének (Lr10, Lr21) klónozása (HUANG és mtsai, 2003; FEUILLET és mtsai, 2003). A térképek segítségével végzett génizolálás (map-based gene isolation) komplex műveletsorát az egyik vernalizációs gén, a VRN2 gén izolálására végzett kísérletek változatos bemutatásával ismerhetjük YAN és mtsai (2004) közleménye alapján. A téli búzafajták hosszabb ideig tartó alacsony hőmérsékletű (+4 5 o C) kezelést, vernalizációt igényelnek a szárba szökkenéshez, a kalászoláshoz. Két kulcsfontosságú gén szerepét számos vizsgálat igazolta. A VRN-1 gén az 5-ös kromoszóma hosszú karján helyezkedik el, és domináns a tavaszi típust illetően. Az általa kódolt fehérje az Arabidopsis APETALA1 (AP1) homológja, amely egy, a merisztéma kialakulásában szerepet játszó MADbox transzkripciós faktor (TREVASKIS és mtsai, 2003; YAN és mtsai, 2003). A VRN2 gén izolálásának menetét, valamint a genetikai és fizikai térkép elemeit a 7. ábra mutatja be. 6. ábra: Nagyobb genetikai előrehaladás biztosítható a szülői allélek számának növelésével, ha a fenotipizálás helyett DNS alapú molekuláris markerekre épül a szelekció (FRANCKI és APPELS, 2002) 7. ábra: A búza (Tr. monococcum) vernalizációs génjének (VRN2) klónozása genetikai és fizikai térképezéssel (részletek a szövegben) (YAN és mtsai, 2004) 8
9 A T. monococcum tavaszi (DV92) illetve, téli, vad típusú (G3116) változatainak keresztezéséből származó F2 növények jellemzésével az UCW22 (RFLP marker) és az UCW2.1 (PCR) markerek által határolt régióra lehetett lokalizálni a VRN2 gént. E két marker segítségével történt BAC klóntár átvizsgálása is, amely a DV92 tavaszi genotípusból készült. Az egyes BAC klónok sorba rendezésével elkészített fizikai térkép, továbbá az átfedő klónok megszekvenálásával sor kerülhetett potenciális gének megtalálására. Az in silico szekvencia-összehasonlítás 8 gént és 1 pszeudogént tárt fel ebben a régióban. Így a gének sűrűsége 55 kb-onként egy génre becsülhető és a genetikai és fizikai távolság aránya hozzávetőlegesen 1,7 Mb/cM. Mint a 7. ábra szemlélteti, ebben a régióban kimutatható a búzával való kolinearitás az árpa és rizs gének sorrendjét és irányát tekintve. A lehetséges gének között kettőt 76%-ban hasonlatosak találtak (ZCCT1 és ZCCT2), és az általuk kódolt fehérjék egy 43 aminosavat tartalmazó szakaszon azonosságot mutattak az Arabidopsis CONSTANS, virágzást szabályozó transzkripciós faktorral. A gének lehetséges szerepével kapcsolatosan fontos információ, hogy vernalizáció során működésük gátlódik, pedig a VRN1 mrns szintje megemelkedik. A ZCCT1 gén esetében transzgenikus búza előállításával igazolták a virágzás szabályozásában betöltött szerepét. Az RNS-interferenciát okozó génbeépítés eredményeként 40 nappal korábban virágoztak azok a búzanövények, amelyekben a génbeépítés következtében lecsökkent a ZCCT1 mrns mennyisége. Az itt bemutatott kísérletsorozat napjaink búzakutatásának élvonalát képviseli. A géntérképezéstől, a BAC klónok szekvenálásán át vezet az út a gének izolálásához. A gén birtokában mód nyílik a transzgenikus tenyészanyagok előállításával a funkció igazolására, illetve megbecsülhető a géntechnológiai megközelítés nemesítési felhasználhatósága és jelentősége. Az aktív gének másolatai cdns-klóntárak Bár az élő szervezetek minden egyes sejtjében az örökítő anyag elméletileg azonos DNS-molekulák formájában található meg, mégis sejtenként, szövetenként, szervenként dinamikusan változik a felhasznált genetikai információ, amely irányítja az életfolyamatokhoz szükséges molekulák szintézisét. A hírvivő mrns-populációk és az azokról készített DNS-másolatok (cdns-ek) hű képet adnak a sejtek aktuális anyagcsere-állapotáról, az éppen aktív gének köréről. A funkcionális genomika ezért kiemelt figyelmet fordít a genom egészét érintően a génkifejeződési mintázatok vizsgálatára, amelyet a DNS-csip-technológia tesz lehetővé. Így például a búza szemkeménységéért felelős gének megismerésére CLARKE és RAHMAN (2005) cdnsmicroarrayt használtak. Ezek a vizsgálatok rámutattak arra, hogy a puroindolint kódoló gén kifejeződése összefüggött a szemek lágyságával. Alternatív megközelítésként kópia DNS (cdns) szintézisére kerül sor, és cdns-klóntárak készülnek a búzanövény szerveiből. A klónozás egyes lépéseit ma már cégek (pl. Stratagene) által forgalmazott reagensek és előre kidolgozott műveletek teszik megbízhatóvá. A számos módszerleírás közül javasolható WILSON és mtsai (2004) közleményében megadott metodika, amellyel 35 cdns-klóntárat készítettek búzából. A nagy teljesítményű DNS-szekvenátorok megvalósíthatóvá teszik a nagyszámú, véletlenszerűen kiválasztott DNS-klón nukleotidsorrendjének a meghatározását. Így génkifejeződési markerek (expressed sequence tags, ESTs) tömege hozható létre az egyes sejtek, szervek működésének jellemzésére. A genomszekvenálási programokban is igen jelentős szerepe van az EST információknak úgy is mint molekuláris markerek, amelyek kromoszomális térképezése több laboratóriumban is folyamatban van (AKHUMOV és mtsai, 2003; QI és mtsai, 2004; HOSSAIN és mtsai, 2004). Tekintettel az EST szekvenálási projektek jelentőségére, a génkutatásban a gabonafélék, és így a búza bevonásával jelentős nemzeti és nemzetközi programok folyamatosan bővítik az elérhető adatbázisokat ( International Triticeae EST Cooperative-ITEC: pw.usda.govigenome; WEST). A cdns klónok szekvenálásából származó nukleotidsorrendre vonatkozó adatok kezelését számos informatikai program teszi lehetővé. Így a phrap algoritmus ( az egymással 90%-ban azonos EST-ket csoportokba (conting) rendezi. Az egyszer előforduló (singleton) szekvenciákat képviselő EST-kből, illetve a csoportokra jellemző szekvenciákból állítható össze az egyedi gének (unigenes) listája. A LAZO és mtsai (2004) által végzett szekvenálási programokban 41 cdns-bankból 116 ezer EST klónt szekvenáltak meg. Ezen EST-k alapján 19 ezer csoportot lehetett kialakítani, illetve 23 ezer egyedi szekvencia került azonosításra. Így 42 ezer egyedi gént képviselő ESTékhez kötődő funkciók megbecslésére is. A 8. ábra bemutatja a prediktált fehérjék csoportosítását a búza anyagcseréjében betöltött szerep szerint. 9
10 8. ábra: A kifejezett gének szekvenciájának (EST) ismeretében a Gene Orthology program segítségével megbecsülhető a kódolt fehérjék szerepe a növények anyagcseréjében (LAZO és mtsai, 2004) Az egyre növekvő cdns-klóntárak szinte a búzanövény valamennyi fontosabb szervéből, szövetéből izolált RNS-minták felhasználásával készülnek. Gyakran különböző stresszhatásoknak, mint szárazságnak, hidegnek vagy magas hőmérsékletnek kitett szervekből származó cdns-klóntárak szekvenálásával bővítik az adatok számát. Figyelemre méltó metodikai előrehaladást jelentenek azok a kísérletek, amelyek egy adott sejtcsoport felhasználásával hoznak létre cdns-klóntárakat. SPRUNCK és mtsai (2005) izolált búza petesejtekből, illetve zigótából származó cdns-klónok megszekvenálásával jutottak speciális EST-adatbázishoz. Az egyes EST-ek előfordulási gyakorisága információt nyújt a gén kifejeződési mintázatokról, működésének erősségéről az egyes növényi mintákban. Az EST adatok in silico vizsgálatával mód nyílik virtuális northern hibridizáció elvégzésére (WILSON és mtsai 2004). Az egyedi cdns-klónok felhasználásával készített DNS-csipek (microarray) alkalmasak a génkifejeződési mintázatok genomszintű vizsgálatára (WILSON és mtsai, 2004; CLARKE és RAHMAN, 2005). Génkeresés a fehérjék szekvenálásával: proteomika A funkcionális genomikai megközelítések széles körű elterjedése közepette nem szabad megfeledkezni arról az alapvető biológiai tényről, hogy a sejtekben lejátszódó anyagcsere-folyamatok, a külső jelek érzékelése és továbbítása fehérjék és enzimek közreműködésével valósulnak meg. A fehérjék szerkezeti változásai a transzlációjuk utáni módosítások, mint pl. foszforiláció/ defoszforiláció, a fehérjék kölcsönhatásai, fehérjekomplex kialakulása, szétesése, a fehérjék szabályozott degradációja mind igen fontos molekuláris események, amelyek közvetlenül felelősek a sejtek működéséért, osztódásáért, differenciálódásáért és ezeken keresztül a növények növekedéséért, a fejlődési program egyes fázisainak bekövetkezéséért. A növény életciklusának beteljesedése egyben az ember számára hasznos növényi termékek létrejöttéhez vezet, hiszen növényi szerveket fogyasztunk mint élelmiszereket, használjuk azokat ipari nyersanyagokként vagy akár mint gyógyszereket. A kenyérbúza esetében a kettős megtermékenyítésből származó embrió és endospermium jelenti a hasznosításra kerülő mezőgazdasági terméket. A búzaszemfehérje öszszetétele kiemelt jelentőségű a tápérték, a minőség szempontjából. Ezért érthető, hogy a búzával végzett proteomikai kísérletek is kiemelten az endospermium-fehérjékre koncentrálnak (VENSEL és mtsai, 2005). A nagyobb mennyiségben található főbb fehérjék mint a gliadinok és gluteinek elválaszthatók az albuminoktól és globulinoktól KCl bufferrel. Az utóbbiak esetében további szétválasztás lehetséges a fehérjék metanololdhatósága alapján. A fehérjemintázat vizsgálatának központi eleme a kétdimenziós elektroforézis (2DE), amelynek során először izoelektromos fókuszálással (IEF) történik a fehérjék szétválasztása, majd a második dimenzió SDS-akrilamid elektroforézissel alakítható ki. A 9. ábra a portok megjelenését követő 10
11 10. napon izolált endospermium proteomtérképét mutatja be VENSEL és mtsai (2005) nyomán. A 2DEal történő szétválasztás lehetővé tette 450 fehérje kimutatását, amelyek számozását, azonosítás szoftverekkel lehet pontossá tenni. 9. ábra: A tíznapos búza-endospermium fehérjéinek proteomtérképe (VENSEL és mtsai, 2005) A szerzők több mint 250 fehérjefoltot izoláltak, majd gélben történt tripszinemésztés után MALDI-TOF tömegspektroszkópiával elkészítették a peptidek tömegtérképét, ami lehetővé tette a fehérjék azonosítását. A 10 és 36 napos endospermium fehérjéinek összehasonlításából több általános következtetést lehetett levonni. Így a szénhidrát-anyagcsere enzimei mind a korai, mind a későbbi mintákban kimutathatók. A raktározott fehérjék, valamint a stressz- és védekezési fehérjék a 36 napos endospermiumra jellemzőek. Magától értetődő, hogy az azonosított fehérjék aminosav-sorrendjének ismeretében lehetőség van specifikus PCR primerek szintetizálására és a megfelelő gén izolálására. A bemutatott proteommegközelítést felhasználták korábban a diploid, tetraploid és hexaploid búzák összehasonlítására is (ISLAM és mtsai, 2003). Várható, hogy hasonlóan a kétszikű fajokkal mint Arabidopsis, Medicago végzett proteomkutatások fejlődéséhez, a búza esetében is a DNS-adatbázisok kiegészülnek fehérjeinformációkkal (AGRAWAL és mtsai 2005a/b). A búzagének funkcionális elemei A fehérjék aminosavsorrendjét meghatározó hírvivő RNS- (mrns-) molekulák szintézisének paramétereit a gének 5 végén található szabályozó régiók, az ún. promóterek határozzák meg elsődlegesen. Az érett mrns-molekulák az exonoknak megfelelő DNS-szakaszokat képviselik, miután a pre-mrnsmolekulákból a nem kódoló ún. intronszakaszoknak megfelelő molekularészek kivágódnak és sor kerül a kódoló mrns-molekulák összeillesztésére (splicing). Az RNS-prekurzorok érésének folyamatát részletesen bemutatja SOLYMOSY FERENC öszszefoglalója (1999). A búza esetében is a gént hordozó genomikus klónok megszekvenálásával, a funkcionális elemek azonosításával juthatunk el a gének szerkezetének feltárásához és a működésük jellegzetességeinek megismeréséhez. Mind agronómiai, mind géntechnológiai szempontból kiemelt figyelmet érdemelnek a promóterek különböző típusai, hiszen ezek az 5 végi DNSszakaszok kulcsszerepet játszanak a génaktivitás szabályozásában. A sejtek, szövetek működési paraméterei, és így a hasznosítandó növényi szervek piaci értéke sokban függ attól, hogy mely gének milyen sejtekben, a növény életciklusának melyik fázisában és milyen erősséggel fejeződnek ki. A transzgenikus technológia nemesítési felhasználása során is kiemelt feladat a beépített gén szabályozott működtetése. Gyakran az erős és minden sejtben és szövetben aktív ún. konstitutív promóterek használata indokolt, de előfordulhat, hogy a gén termékének túlzott és differenciálatlan felhalmozása kedvezőtlen, nem kívánt hatásokat eredményez. Általában célszerű szövet- vagy szervspecifikus promótereket izolálni és felhasználni a kívánt hatás biztosítására. Külön jelentőségűek az ún. indukálható promóterek, amelyek kémiai anyagok vagy környezeti tényezők, mint magasalacsony hőmérséklet, vízhiány, fény hatására aktivizálódnak. A promóterszakaszokban specifikus DNS-szekvenciamotívumok, ún. cisz-elemek ismerhetők fel, amelyek aktiváló fehérjékkel, ún. transzkripciós faktorokkal történő kölcsönhatáson keresztül hozzák működésbe a gének átírását végző apparátust. Sokszor a transzkripciós faktorok jelentik az egyes jelátviteli láncolatok utolsó elemét, amelyek szelektív génaktiválással biztosítják a kívánt fehérjék szintézisét és a sejtek optimális működését. Lényeges annak szem előtt tartása, hogy a promóterek sajátosságain túl a kromatin szerkezeti állapota is hozzájárul a génkifejeződési mintázat meghatározásához. Az említett általános 11
12 szempontok felsorolása után indokolt néhány búzagén konkrét példájával bemutatni a génműködés szabályozásának főbb jellemzőit. A gazdasági szempontból is fontos búzagének szerveződésének ismertetéséhez példaként a III. típusú keményítő szintázgént választottuk LI és mtsai (2000) közleménye alapján. A búzaszem endospermiumából szintetizálódó keményítő több enzim működésének terméke. Így az ADP-glükózpirofoszforiláz (AGP) egy heterotetramer enzim (2 kis és 2 nagy alegység), amely glükóz-1-foszfát és ATP felhasználásával ADP-glükóz képződését katalizálja. Ez a keményítő szintézisét elsődlegesen szabályozó enzimatikus lépés, mert az ADP-glükóz, glükóz donorként szolgál a keményítőláncok hosszabbításakor. A kukorica ortofoszfát-(p i ) gátlással szemben érzéketlen AGP nagy alegység génjét (Schrunken 2 gén) búzában kifejeztetve mintegy 40%-kal megnőtt a növényenkénti szemtömeg (SMIDANSKY és mtsai 2002). A keményítőszintetáz (starch synthase SS) az α-1-4-glikozidos kötéssel bővíti a nem elágazó láncot azáltal, hogy az ADP-glukózról a glükózt átviszi a meglévő α-1-4-glukánra mint akceptorra. A gabonafélékben a keményítőszemcsékhez kötött (GBSS) keményítőszintetázon kívül még három (SSI, SSII, SSIII) enzim működik, amelyek génjei közül a búza ss3 gén szerveződését a 10. ábra szemlélteti. A T. tauschii által képviselt D genomban ez a gén 10 kb hosszúságú és 16 kódoló exonrégiót tartalmaz, amelyeket 15 intron választ szét. Figyelmet érdemel a 3. exon rendkívüli mérete, hiszen 2703 bp kiterjedésű. A kódoló régió lezárásaként található a poliadenizációs szignál és stop kódon. Az ss3 gén által kódolt enzim 1628 aminosavból épül fel, és a becsült tömege D. A fehérje amino terminális végén egy tranzitpeptid található, amelyet 30 aminosavból álló ismétlődéseket (RM1, RM2, RM3) is tartalmazó variábilis régió követ. A fehérje középső részén található az SSIII enzimekre jellemző és fokozottan konzervált aminosav-szekvenciákból felépülő régió, amelyen az RM4, RM5, RM6 ismétlődő motívumok azonosíthatók. A C terminális végén található a katalitikus domén, amely hasonló szekvenciaelemeket hordoz a növényi SS enzimek különböző típusaiban. A búza ss3 gén aktivitása fokozatosan nő az endospermiumban a portokok megjelenését követő 12. napig. Ez a gén kifejeződik még levelekben és fiatal kalászokban. Az endospermium-specifikus búzagének működését szabályozó promóterrégiók jellegzetes szekvenciaeleme, LAMACCHIA és mtsai (2001) a nagy molekulatömegű gluteint kódoló Glu 1D-1 gén esetében tanulmányozták. Az 1.0 kb nagyságú 5 végű promóterszakaszon található jellegzetes cisz elemeket a 11. ábra mutatja be. 10. ábra: A búzakeményítő szintáz III. enzim főbb funkcionális elemei a konzervált (CM) és ismétlődő (RM) motívumokkal. A gén exon és intron szerveződése az Arabidopsis génekkel összehasonlítva. (LI és mtsai, 2000) 12
13 Hely: Szekvencia: Helyzet: ATG ATG +62 Kezdet TTATCA +1 TATA box CTATAAAAG -30 HMW enhancer lásd a szövegben? -186-tól -149-ig Részleges HMW enhancer TTTGCAAA -359 Glu-1A-2 deletion -394-től -311 N box TGAGTCA -530 E box TGTAAA -685 Fordított N box CTCATC phmwgus ábra: A búza nagy molekulasúlyú (HMW) gluteningén (Glu-1D-1) promóterének felépítése és a feltételezett transzkripciós faktor kötőhelyek, cisz-motívumok elhelyezkedése (LAMACCHIA és mtsai, 2001) A transzkripció kezdetét biztosító TTATCA motívum helyzetéhez viszonyítva tünteti fel az ábra a többi jellegzetes elem távolságát a kezdési ponttól. 62 bp távolságra 3 irányban található az első aminosavat, a metionint kódoló ATG triplet. Ebben a génszekvenciában az ún. TATA boxot CTATAAAAG motívum jelenti. Szerepe az, hogy kötőhelyként szolgál transzkripciós faktorok és az RNS-polimeráz-komplex számára. A nagy molekulatömegű (high molecule weight, HMW) prolaminokat kódoló gének promótereiben felismerhető az ún. prolaminbox, amely két konzervált motívumot tartalmaz: endospermium (E vagy EM) motívum: TGTAAAGT és a GCN4-szerű (N vagy GLM motívum: G(A/G)TGAGTCAT. HOMMONDA KOSACH és mtsai (1993) elektroforetikus mobilitás alapján a búza endospermium magifehérje-frakciójában azonosítottak két faktort, amelyek közül az ESBFI az E(EM) motívumhoz kötődött specifikusan, az ESBFII pedig a GLM motívumhoz kapcsolódott. Továbbá ALBANI és mtsai (1997) azonosítottak egy magspecifikus bázikus leucinzipper-fehérjét, amely a GLM motívumról aktiválta a transzkripciót. A HMV prolamin gének promóterei igen aktívak, ami lehetővé teszi, hogy a teljes búzaszemfehérje mennyiségének 2%-át a HMW prolamin adja. Az ilyen nagymértékű promóteraktivitás biztosításában szerepet játszik a HMW enhancer-motívum, amelyet THOMAS és FLAVELL 1990-ben írtak le. A promóterek funkcionális vizsgálatához jól használhatók a transzgenikus növények, amelyek hordozzák a tanulmányozandó szabályozó régiót és a hozzá kapcsolt riportergént mint pl. UidA (β -glukoronidáz, GUS) gént. A 12. ábra bemutatja, hogy a búza HMW prolamingén 1191 bp-t hordozó promótere erős, endospermium-specifikus kifejeződést biztosít a transzgenikus búza kifejlett szemtermésében. 12. ábra: A β-glukoronidáz (GUS) aktivitás kimutatása a transzgenikus növények szemtermésében, amelyekben a GUS gént a Glu 1D 1 gén promóterrégiója ( ) működteti. (LAMACCHIA és mtsai, 2001) 13
14 Felhasznált és ajánlott irodalom AGRAWAL, G. K., YONEKURA, M., IWAHASHI, Y., IWAHASHI, H., RAKWAL, R. (2005): System, trends and perspectives of proteomics in dicot plants. Part I.: Technologies in proteome establishment. J. Chromatography B, 815: (1-2): AGRAWAL, G. K., YONEKURA, M., IWAHASHI, Y., IWAHASHI, H., RAKWAL, R. (2005): System, trends and perspectives of proteomics in dicot plants. Part II.: Proteomes of the complex developmental stages. J. Chromatography B, 815: (1-2): AKHUNOV, E. D., GOODYEAR, A. W., GENG, S., QI, L.-L., ECHALIER, B., GILL, B. S., MIFTAHUDIN, GUSTAFSON, J. P., LAZO, G., CHAO, S., ANDERSON, O. D., LINKIEWICZ, A. M., DUBCOVSKY, J., LA ROTA, M., SORRELLS, M. E., ZHANG, D., NGUYEN, H. T., KALAVACHARLA, V., HOSSAIN, K., KIANIAN, S. F., PENG, J., LAPITAN, N. L. V., GONZALEZ-HERNANDEZ, J. L., ANDERSON, J. A., CHOI, D.-W., CLOSE, T. J., DILBIRLIGI, M., GILL, K. S., WALKER-SIMMONS, M. K., STEBER, C., MCGUIRE, P. E., QUALSET, C. O., DVORAK, J. (2003): Genome Research, 13: ALBANI, D., HAMMOND-KOSACK, M. C., SMITH, C., CONLAN, S., COLOT, V., HOLDSWORTH M., BEVAN, M. W. (1997): The wheat transcriptional activator SPA: a seed-specific bzip protein that recognizes the GCN4-like motif in the bifactorial endosperm box of prolamin genes. The Plant Cell, 9: ALLOUIS, S., MOORE, G., BELLEC, A., SHARP, R., FAIVRE, P., MONTIMER, K., PATEYRON, S., FOOTE, T., GRIFFITHS, S., CABOCHE, M., CHALHOUB, B. (2003): Construction and characterization of a hexaploid wheat (Triticum aestivum L.) BAC library from the reference germplasm Chinese Spring. Cereal Res. Comm. 31: ANDERSON, O. D., LITTS, J. C., GAUTIER, M.-F., GREENE, F. C. (1984): Nucleic acid sequence and chromosome assignment of wheat storage protein gene. Nucleic Acid Res., 12: APPELS, R., FEUILLET, C., GILL, B., BUELL, R., CHUMLEY, F., EVERSOLE, K., FRITZ, D. (2005): Draft white paper on the international wheat genome sequencing consortium. BROGLIE, R., CORUZZI, G., LAMPPA, G., KEITH, B., CHUA, N.-H. (1983): Structural analysis of nuclear genes coding coding for the precurzor to the samll subunit of wheat ribulose-1,5- bisphosphate carboxylase. Bio/Technology, 1: CENCI, A., CHANTRET, N., KONG, X., GU, Y. G., ANDERSON, O. D., FAHIMA, T., DISTELFELD, A., DUBCOVSKY, J. (2003): Construction and characterization of a half million clone BAC library of durum wheat (Triticum turgidum ssp. durum). Theor. Appl. Genet., 107: CLARKE, B., RAHMAN, S. (2005): A microarray analysis of wheat grain hardness. Theor. Appl. Genet., 110: DEVOS, K. M., GALE, M.D. (1997): Comparative genetics in the grasses. Plant Molecular Biology, 35: DEVOS, K. M., GALE, M. D. (2000): Genome relationship: the grass model in current research. The Plant Cell, 12: ELLIS, M.H., REBETZKE, G. J., AZANZA, F., RICHARDS, R. A., SPIELMEYER, W. (2005): Molecular mapping of gibberellinresponsive dwarfing genes in bread wheat. Theor. Appl. Genet., 111: ERAYMAN, M., SANDHU, D., SIDHU, D., DILBIRLIGI, M., BAENZIGER P. S., GILL, K. S. (2004): Demarcating the generich regions of the wheat genome. Nucleic Acid. Res., 32 (12): FEUILLET, C., TRAVELLA, S., STEIN, N., ALBAR, L., NUBLAT, A., KELLER, B. (2003): Map-based isolation of the leaf rust disease resistance gene Lr10 from the hexaploid wheat (Triticum aestivum L.) genome. Proc. Natl. Acad. Sci, USA, 100 (25): FORDE, J., MALPICA, J-M., HALFORD, N. G., SHEWRY, P. R., ANDERSON, O. D., GREENE, F. C., MIFLIN, B. J. (1985): The nucleotide sequence of a HMW subunit gene located on chromosome 1A of wheat (Triticum aestivum L.). Nucleic Acis Res., 13: FRANCKI, M., APPELS, R. (2002): Wheat functional genomics and engineering crop improvement. Genome Biology, 3 (5): GALE, D. M., DEVOS, K. M. (1998): Comparative genetics in the grasses. Proc. Natl. Acad. Sci. USA, 95: GALE, M. D., GREGORY, R. S. (1977): A rapid method for early generation selection of dwarf genotypes in wheat. Euphytica, 26: GILL, B. S., APPELS, R., BOTHA-OBERHOLSTER, A.-M., BUELL, C. R., BENNETZEN, J. L., CHALHOUB, B., CHUMLEY, F., DVOŘAK, J., IWANAGA, M., KELLER, B., LI, W., MCCOMBIE, W. R., OGIHARA, Y., QUETIER, F., SASAKI, T. (2004): A workshop report on wheat genome sequencing: International genome research on wheat consortium. Genetics, 168: GILL, K. S., GILL, B. S., ENDO, T. R., BOYKO, E. V. (1996a) Identification and high-density mapping of gene-rich regions in chromosomes of group 5 of wheat. Genetics, 143: GILL, K. S., GILL, B. S., ENDO, T. R., TAYLOR, T. (1996b): Identification and high-density mapping of gene-rich regions in chromosomes of group 1 of wheat. Genetics, 143: GU, Y. Q., COLEMANN-DERR, D., KONG, X., ANDERSON, O. D. (2004): Rapid genome evolution revealed by comparative sequence analysis of orthologous regions four Triticeae genomes. Plant Phys., 135: HAMMOND-KOSACK, M. C., HOLDSWORTH M. J., BEVAN M. W. (1993): In vivo footprinting of a low molecular weight glutenin gene (LMWG-1D1) in wheat endosperm. The EMBO J. 12: HOSSAIN, K. G., KALAVACHARLA, V., LAZO, G. R., HEGSTAD, J., WENTZ, M. J., KIANIAN, P. M. A., SIMONS, K., GEHLHAR, S., RUST, J. L., SYAMALA, R. R., OBEORI, K., BHAMIDIMARRI, S., KARUNADHARMA, P., CHAO, S., ANDERSON, O. D., QI, L. L., ECHALIER, B., GILL, B. S., LINKIEWICZ, A. M., RATNASIRI, A., DUBCOVSKY, J., AKHUNOV, E. D., DVOŘÁK, J., MIFTAHUDIN, ROSS, K., GUSTAFSON, J. P., RADHAWA, H. S., DILBIRLIGI, M., GILL, K. S., PENG, J. H., LAPITAN, N. L. V., GREENE, R. A., BERMUDEZ-KANDIANIS, C. E., SORRELLS, M. E., FERIL, O., PATHAN, M. S., NGUYEN, H. T., GONZALEZ-HERNANDEZ, J. L., CONLEY, E. J., ANDERSON, J. A., CHOI, D. W., FENTON, D., CLOSE, T. J., MCGUIRE, P. E., QUALSET, C. Q., KIANIAN, S. F. (2004): A Chromosome bin map of 2148 expressed sequence tag loci of wheat homoeologous group 7. Genetics, 168: HUANG, L., BROOKS, S. A., LI, W. L., FELLERS, J. P., TRICK, H. N., GILL, B. S. (2003): Map-based cloning of leaf rust resistance gene Lr21 from the large and polyploidy genome of bread wheat. Genetics, 164: HUANG, S., SIRIKHACHORNKIT, A., SU, X., FARIS, J., GILL, B., HASELKORN, R., GORNICKI, P. (2002): Genes encoding plastid acetyl-coa carboxylase and 3-phosphoglycerate kinase of the Triticum/Aegilops complex and the evolutionary history of polyploid wheat. Proc. Natl. Acad. Sci., 99 (12): ISLAM, N., TSUJIMOTO H., HIRANO, H. (2003): Proteome 14
15 analysis of diploid, tetraploid and hexaploid wheat: Towards understanding genome interaction in protein expression. Proteomics, 3: JANDA, J., BARTOŠ, J., ŠAFAŘ, J., KUBALÁKOVÁ, M., VALÁRIK, M., ČIHALÍKOVÁ, J., ŠIMKOVÁ, H., CABOCHE, M., SOURDILLE, P., BERNARD, M., CHALHOUB, B., DOLEŽEL, J. (2004): Construction of a subgenomic BAC library specific for chromosomes 1D, 4D and 6D of hexaploid wheat. Theor. Appl. Genet., KELLOGG, E. A. (2001): Evolutionary history of the grasses. Plant Phys. 125: LA ROTA, M., SORRELLS, M. E. (2004): Comparative DNA sequence analysis of mapped wheat ESTs reveals the complexity of genome relationships between rice and wheat. Funct. Integr. Genomics, 4: LAMACCHIA, C., SHEWRY, P. R., DI FONZO, N., FORSYTH, J. L., HARRIS, N., LAZZERI, P. A., NAPIER, J. A., HALFORD, N. G., BARCELO, P. (2001): Endosperm-specific activity of a storage protein gene promoter in transgenic wheat seed. J. Exp. Bot., 52 (355): LAZO, G. R., CHAO, S., HUMMEL, D. D., EDWARDS, H., CROSSMAN, C. C., LUI, N., MATTEWS, D. E., CAROLLO, V. L., HANE, D. L., YOU, F. M., BUTLER, G. E., MILLER, R. E., CLOSE, T. J., PENG, J. H., LAPITAN, N. L. V., GUSTAFSON, J. P., QI, L. L., ECHALIER, B., GILL, B. S., DILBIRLIGI, M., RADHAWA, H. S., GILL, K. S., GREENE, R. A., SORRELLS, M. E., AKHUNOV, E. D., DVOŘÁK, J., LINKIEWICZ, A. M., DUBCOVSKY, J., HOSSAIN, K. G., KALAVACHARLA, V., KIANIAN, S. F., MAHMOUD, A. A., MIFTAHUDIN, MA, X.-F., CONLEY, E. J., ANDERSON, J. A., PATHAN, M. S., NGUYEN, H. T., MCGUIRE, P. E., QUALSET, C. Q., ANDERSON, O. D. (2004): Development o fan expressed sequence tag (EST) resource for wheat (Triticum aestivum L.): EST generation, unigene analysis, probe selection and bioinformatics for a 16,000-locus bin-delineated map. Genetics, 168: LI, W., ZHANG, P., FELLERS, J. P., FRIEBE, B., GILL, B. S. (2004): Sequence composition, organization, and evolution of the core Triticeae genome. The Plant J., 40: LI, Z., MOUILLE, G., KOSAR-HASHEMI, B., RAHMAN, S., CLARKE, B., GALE, K. R., APPELS, R., MORELL, M. K. (2000): The structure and expression of the wheat starch synthase III Gene. Motifs in the expressed gene define the lineage of the starch synthase III gene family. Plant Phys., 123: LIJAVETZKY, D., MUZZI, G., WICKER, T., KELLER, B., WING, R., DUBCOVSKY, J. (1999): Construction and characterization of a bacterial artificial chromosome (BAC) library for the A genome of wheat. Genome, 42: LIU, Y.-G., NAGAKI, K., FUJITA, M., KAWAURA, K., UOZUMI, M., OGIHARA, Y. (2000): Development of an efficient maintenance and screening system for large-insert gwenomic DNA libraries of hexaploid wheat in a transformation-competent artificial chromosome (TAC) vector. The Plant J., 23 (5): MATTEWS, S., TSAI, R. C., KELLOGG, E. (2000): Phylogenetic structure in the grass family (Poaceae): evidence from the nuclear gene phytocrome B. Am. J. Bot., 87: MOULLET, O., ZHANG, H.-B., LAGUDAH, E. S. (1999): Construction and characterization of a large DNA insert library from the D genome of wheat. Theor. Appl. Genet., 99: OZKAN, H., LEVY, A. A., FELDMAN, M. (2001): Allopolyploidyinduced rapid genome evolution in the wheat (Aegilops- Triticum) group. The Plant Cell, 13: PENG, J., RICHARDS, D. E., HARTLEY, N. M., MURPHY, G. P., DEVOS, K. M., FLINTHAM, J. E., BEALES, J., FISH, L. J., WORLAND, A. J., PELICA, F., SUDHAKAR, D., CHROSTOU, P., SNAPE, J.W., GALE, M.D., HARBERD, N.P. (1999): Green revolution genes encode mutant gibberellin response modulators. Nature, 400: QI, L. L., ECHALIER, B., CHAO, S., LAZO, G. R., BUTLER, G. E., ANDERSON, O. D. AKHUNOV, E. D., DVOŘÁK, J., LINKIEWICZ, A. M., RATNASIRI, A., DUBCOVSKY, J., BERMUDEZ-KANDIANIS, C. E., GREENE, R. A., KANTETY, R., LA ROTA, C. M., MUNKVOLD, J. D., SORRELLS, S. F., SORRELLS, M. E., DILBIRLIGI, M., SIDHU, D., ERAYMAN, M., RADHAWA, H. S., SANDHU, D., BONDAREVA, S. N., GILL, K. S., MAHMOUD, A. A., MA, X.-F., MIFTAHUDIN, GUSTAFSON, J. P., CONLEY, E. J., NDUATI, V., GONZALEZ- HERNANDEZ, J. L., ANDERSON, J. A., PENG, J. H., LAPITAN, N. L. V., HOSSAIN, K. G., KALAVACHARLA, V., KIANIAN, S. F., PATHAN, M. S., ZHANG, D. S., NGUYEN, H. T., CHOI, D.-W., FENTON, R. D., CLOSE, T. J., MCGUIRE, P. E., QUALSET, C. O., GILL, B. S. (2004): A chromosome bin map of 16,000 expressed sequence tag loci and distribution of genes among the three genomes of polyploid wheat. Genetics, 168: ŠAFAŘ, J.,BARTOŠ, J., JANDA, J., BELLEC, A, KUBALÁKOVÁ, M., VALÁRIK, M., PATEYRON, S., WEISEROVÁ, J., TUŠKOVÁ, R., ČIHALIKOVÁ, J., VRÁNA, J., ŠIMKOVÁ, H., FAIVRE-RAMPANT, P., SOURDILLE, P., CABOCHE, M., BERNARD, M. DOLEŽEL, J., CHALHAUB, B. (2004): Dissecting large and complex genomes: flow sorting and BAC cloning of individual chromosomes from bread wheat. Plant Journal 39 (6): SMIDANSKY, E. D., CLANCY, M., MEYER, F. D., LANNING, S. P., BLAKE, N. K., TALBERT, L. E., GIROUX, M. J. (2002): Enhanced ADP-glucose pyrophosphorylase activity in wheat endosperm increases seed yield. Proc. Natl. Acad. Sci. 99 (3): SOLYMOSY, F. (1999): RNS-prekurzorok érése növényekben. Balázs, E., Dudits, D. szerk. Molekuláris Növénybiológia: Szemelvények, Akadémiai Kiadó, SORRELLS, M. E., LA ROTA, M., BERMUDEZ-KANDIANIS, C. E., GREENE, R. A., KANTETY, R., MUNKVOLD, J. D., MIFTAHUDIN, MAHMOUD, A., MA, X.,GUSTAFSON, P. J., QI, L. L., ECHALIER, B., GILL, B. S., MATTHEWS, D. E., LAZO, G. R., CHAO, S., ANDERSON, O. D., NGUYEN, H. T., PENG, J., LAPITAN, N. L. V., GONZALES-HERNANDES, J. L., ANDERSON, J. A., HOSSAIN, K., KALAVACHARLA, V., KIANIAN, S. F., CHOI, D.-W., CLOSE, T. J., DILBIRLIGI, M., GILL, K. S., STEBER, C., WALKER-SIMMONS, M. K., MCGUIRE, P. E., QUALSET, C. O. (2003): Comparative DNA sequence analysis of wheat and rice genomes. Genome Research, 13: SPRUNCK, S., BAUMANN, U., EDWARDS, K., LANGRIDGE, P., DRESSELHAUS, T. (2005): The transcript composition of egg cells changes significantly following fertilization in wheat (Triticum aestivum L.). The Plant J., 41: STEIN, N., FEUILLET, C., WICKER, T., SCHLAGENHAUF, E., KELLER, B. (2000): Subgenome chromosome walking in wheat a 450 kb physical contig in Triticum monococcum L. spans the Lr10 resistance locus in hexaploid wheat (Triticum aestivum L.). Proc. Natl. Acad. Sci., 97: TABATA, T., SASAKI, K., IWABUCHI, M. (1983): The structural organization and DNA sequence of a wheat histone H4 gene. Nucleic Acid Res., 11 (17): THOMAS, M. S., FLAVELL, R. B. (1990): Identification of an enhancer element for theendosperm-specific expression of high molecular weight glutein. The Plant Cell, 2: TREVASKIS, B., BAGNALL, D. J., ELLIS, M. H., PEACOCK, W. J., DENNIS, E. S. (2003): MADS box genes control vernalizationinduced flowering cereals. Proc. Natl. Acad. Sci. USA, 100 (22): 15
Conserved ortholog set (COS) markerek térképezése Aegilops kromoszómákon
 Conserved ortholog set (COS) markerek térképezése Aegilops kromoszómákon Rövid tanulmányút 2011. 01.03-03. 30., John Inn Centre, Dept. of Crop Genetics, Norwich Research Park, Norwich NR4 7UH, UK Supervisor:
Conserved ortholog set (COS) markerek térképezése Aegilops kromoszómákon Rövid tanulmányút 2011. 01.03-03. 30., John Inn Centre, Dept. of Crop Genetics, Norwich Research Park, Norwich NR4 7UH, UK Supervisor:
A termesztett búza diploid őseinek molekuláris citogenetikai elemzése: pachytén- és fiber-fish.
 OTKA K67808 zárójelentés 2012. A termesztett búza diploid őseinek molekuláris citogenetikai elemzése: pachytén- és fiber-fish. A fluoreszcens in situ hibridizáció (FISH) olyan technikai fejlettséget ért
OTKA K67808 zárójelentés 2012. A termesztett búza diploid őseinek molekuláris citogenetikai elemzése: pachytén- és fiber-fish. A fluoreszcens in situ hibridizáció (FISH) olyan technikai fejlettséget ért
AZ ALACSONY HŐMÉRSÉKLET HATÁSÁRA BEKÖVETKEZŐ REDOX ÉS GÉNEXPRESSZIÓS VÁLTOZÁSOK GABONAFÉLÉKBEN
 A martonvásári agrárkutatások hatodik évtizede AZ ALAONY HŐMÉRSÉKLET HATÁSÁRA BEKÖVETKEZŐ REDOX ÉS GÉNEXPRESSZIÓS VÁLTOZÁSOK GABONAFÉLÉKBEN KOY GÁBOR, VÁGÚJFALVI ATTILA, TÓTH BALÁZS, SZALAI GABRIELLA,
A martonvásári agrárkutatások hatodik évtizede AZ ALAONY HŐMÉRSÉKLET HATÁSÁRA BEKÖVETKEZŐ REDOX ÉS GÉNEXPRESSZIÓS VÁLTOZÁSOK GABONAFÉLÉKBEN KOY GÁBOR, VÁGÚJFALVI ATTILA, TÓTH BALÁZS, SZALAI GABRIELLA,
Búza tartalékfehérjék mozgásának követése a transzgénikus rizs endospermium sejtjeiben
 TÉMA ÉRTÉKELÉS TÁMOP-4.2.1/B-09/1/KMR-2010-0003 (minden téma külön lapra) 2010. június 1. 2012. május 31. 1. Az elemi téma megnevezése Búza tartalékfehérjék mozgásának követése a transzgénikus rizs endospermium
TÉMA ÉRTÉKELÉS TÁMOP-4.2.1/B-09/1/KMR-2010-0003 (minden téma külön lapra) 2010. június 1. 2012. május 31. 1. Az elemi téma megnevezése Búza tartalékfehérjék mozgásának követése a transzgénikus rizs endospermium
A szamóca érése során izolált Spiral és Spermidin-szintáz gén jellemzése. Kiss Erzsébet Kovács László
 A szamóca érése során izolált Spiral és Spermidin-szintáz gén jellemzése Kiss Erzsébet Kovács László Bevezetés Nagy gazdasági gi jelentıségük k miatt a gyümölcs lcsök, termések fejlıdésének mechanizmusát
A szamóca érése során izolált Spiral és Spermidin-szintáz gén jellemzése Kiss Erzsébet Kovács László Bevezetés Nagy gazdasági gi jelentıségük k miatt a gyümölcs lcsök, termések fejlıdésének mechanizmusát
2011. január április 10. IPK Gatersleben (Németország) május 17. Kruppa Klaudia
 2011. január 10. 2011. április 10. IPK Gatersleben (Németország) Gatersleben (G-life) Country State District Town Administration Germany Saxony-Anhalt Salzlandkreis Seeland Basic statistics Area 16.00
2011. január 10. 2011. április 10. IPK Gatersleben (Németország) Gatersleben (G-life) Country State District Town Administration Germany Saxony-Anhalt Salzlandkreis Seeland Basic statistics Area 16.00
Kromoszómák, Gének centromer
 Kromoszómák, Gének A kromoszóma egy hosszú DNS szakasz, amely a sejt életének bizonyos szakaszában (a sejtosztódás előkészítéseként) tömörödik, így fénymikroszkóppal láthatóvá válik. A kromoszómák két
Kromoszómák, Gének A kromoszóma egy hosszú DNS szakasz, amely a sejt életének bizonyos szakaszában (a sejtosztódás előkészítéseként) tömörödik, így fénymikroszkóppal láthatóvá válik. A kromoszómák két
DI-, TETRA- ÉS HEXAPLOID TRITICUM FAJOK GENOMJAINAK ELEMZÉSE ÉS AZOK ÖSSZEHASONLÍTÁSA FLUORESZCENS IN SITU HIBRIDIZÁCIÓVAL
 Hagyomány és haladás a növénynemesítésben DI-, TETRA- ÉS HEXAPLOID TRITICUM FAJOK GENOMJAINAK ELEMZÉSE ÉS AZOK ÖSSZEHASONLÍTÁSA FLUORESZCENS IN SITU HIBRIDIZÁCIÓVAL VARGA MÓNIKA, MOLNÁR ISTVÁN ÉS KOVÁCS
Hagyomány és haladás a növénynemesítésben DI-, TETRA- ÉS HEXAPLOID TRITICUM FAJOK GENOMJAINAK ELEMZÉSE ÉS AZOK ÖSSZEHASONLÍTÁSA FLUORESZCENS IN SITU HIBRIDIZÁCIÓVAL VARGA MÓNIKA, MOLNÁR ISTVÁN ÉS KOVÁCS
Hátterükben egyetlen gén áll, melynek általában számottevő a viselkedésre gyakorolt hatása, öröklési mintázata jellegzetes.
 Múlt órán: Lehetséges tesztfeladatok: Kitől származik a variáció-szelekció paradigma, mely szerint az egyéni, javarészt öröklött különbségek között a társadalmi harc válogat? Fromm-Reichmann Mill Gallton
Múlt órán: Lehetséges tesztfeladatok: Kitől származik a variáció-szelekció paradigma, mely szerint az egyéni, javarészt öröklött különbségek között a társadalmi harc válogat? Fromm-Reichmann Mill Gallton
MTA ATK Mezőgazdasági Intézete, Alkalmazott Genomikai Osztály, Martonvásár
 Fogyaszthatóak-e az ősibb búzafélék? Tönke, alakor, kamut a lisztérzékenység tükrében Juhász Angéla- Gell Gyöngyvér- Kovács Krisztina MTA ATK Mezőgazdasági Intézete, Alkalmazott Genomikai Osztály, Martonvásár
Fogyaszthatóak-e az ősibb búzafélék? Tönke, alakor, kamut a lisztérzékenység tükrében Juhász Angéla- Gell Gyöngyvér- Kovács Krisztina MTA ATK Mezőgazdasági Intézete, Alkalmazott Genomikai Osztály, Martonvásár
A búzaszem fejlődésében szerepet játszó gének azonosítása bioinformatikai és molekuláris módszerekkel. Doktori (Ph.D.) értekezés SZŰCS ATTILA
 A búzaszem fejlődésében szerepet játszó gének azonosítása bioinformatikai és molekuláris módszerekkel Doktori (Ph.D.) értekezés SZŰCS ATTILA MTA Szegedi Biológiai Központ, Növénybiológiai Intézet Témavezető:
A búzaszem fejlődésében szerepet játszó gének azonosítása bioinformatikai és molekuláris módszerekkel Doktori (Ph.D.) értekezés SZŰCS ATTILA MTA Szegedi Biológiai Központ, Növénybiológiai Intézet Témavezető:
Orvosi Genomtudomány 2014 Medical Genomics 2014. Április 8 Május 22 8th April 22nd May
 Orvosi Genomtudomány 2014 Medical Genomics 2014 Április 8 Május 22 8th April 22nd May Hét / 1st week (9. kalendariumi het) Takács László / Fehér Zsigmond Magyar kurzus Datum/ido Ápr. 8 Apr. 9 10:00 10:45
Orvosi Genomtudomány 2014 Medical Genomics 2014 Április 8 Május 22 8th April 22nd May Hét / 1st week (9. kalendariumi het) Takács László / Fehér Zsigmond Magyar kurzus Datum/ido Ápr. 8 Apr. 9 10:00 10:45
Biomassza alapú bioalkohol előállítási technológia fejlesztése metagenomikai eljárással
 Biomassza alapú bioalkohol előállítási technológia fejlesztése metagenomikai eljárással Kovács Zoltán ügyvezető DEKUT Debreceni Kutatásfejlesztési Közhasznú Nonprofit Kft. Problémadefiníció Első generációs
Biomassza alapú bioalkohol előállítási technológia fejlesztése metagenomikai eljárással Kovács Zoltán ügyvezető DEKUT Debreceni Kutatásfejlesztési Közhasznú Nonprofit Kft. Problémadefiníció Első generációs
Új genetikai stratégia kidolgozása az Arabidopsis stressz válaszát szabályzó gének azonosítására
 Új genetikai stratégia kidolgozása az Arabidopsis stressz válaszát szabályzó gének azonosítására Tézisfüzet Papdi Csaba Témavezető: Dr. Szabados László MTA Szegedi Biológiai Központ Növénybiológia Intézet
Új genetikai stratégia kidolgozása az Arabidopsis stressz válaszát szabályzó gének azonosítására Tézisfüzet Papdi Csaba Témavezető: Dr. Szabados László MTA Szegedi Biológiai Központ Növénybiológia Intézet
Többgénes jellegek. 1. Klasszikus (poligénes) mennyiségi jellegek. 2.Szinte minden jelleg több gén irányítása alatt áll
 Többgénes jellegek Többgénes jellegek 1. 1. Klasszikus (poligénes) mennyiségi jellegek Multifaktoriális jellegek: több gén és a környezet által meghatározott jellegek 2.Szinte minden jelleg több gén irányítása
Többgénes jellegek Többgénes jellegek 1. 1. Klasszikus (poligénes) mennyiségi jellegek Multifaktoriális jellegek: több gén és a környezet által meghatározott jellegek 2.Szinte minden jelleg több gén irányítása
A kromoszómák kialakulása előtt a DNS állomány megkettőződik. A két azonos információ tartalmú DNS egymás mellé rendeződik és egy kromoszómát alkot.
 Kromoszómák, Gének A kromoszóma egy hosszú DNS szakasz, amely a sejt életének bizonyos szakaszában (a sejtosztódás előkészítéseként) tömörödik, így fénymikroszkóppal láthatóvá válik. A kromoszómák két
Kromoszómák, Gének A kromoszóma egy hosszú DNS szakasz, amely a sejt életének bizonyos szakaszában (a sejtosztódás előkészítéseként) tömörödik, így fénymikroszkóppal láthatóvá válik. A kromoszómák két
Molekuláris biológiai eljárások alkalmazása a GMO analitikában és az élelmiszerbiztonság területén
 Molekuláris biológiai eljárások alkalmazása a GMO analitikában és az élelmiszerbiztonság területén Dr. Dallmann Klára A molekuláris biológia célja az élőlények és sejtek működésének molekuláris szintű
Molekuláris biológiai eljárások alkalmazása a GMO analitikában és az élelmiszerbiztonság területén Dr. Dallmann Klára A molekuláris biológia célja az élőlények és sejtek működésének molekuláris szintű
Oszvald Mária. A búza tartalékfehérjék tulajdonságainak in vitro és in vivo vizsgálata rizs modell rendszerben
 Budapesti Műszaki és Gazdaságtudományi Egyetem Alkalmazott Biotechnológia és Élelmiszertudományi Tanszék PHD ÉRTEKEZÉS TÉZISEI Készítette: Oszvald Mária A búza tartalékfehérjék tulajdonságainak in vitro
Budapesti Műszaki és Gazdaságtudományi Egyetem Alkalmazott Biotechnológia és Élelmiszertudományi Tanszék PHD ÉRTEKEZÉS TÉZISEI Készítette: Oszvald Mária A búza tartalékfehérjék tulajdonságainak in vitro
Szelekciós lehetőségek a búza minőség-orientált nemesítésében fehérjekémiai és DNS markerekkel
 Budapesti Műszaki és Gazdaságtudományi Egyetem, Vegyészmérnöki Kar Biokémiai és Élelmiszertechnológiai Tanszék Szelekciós lehetőségek a búza minőség-orientált nemesítésében fehérjekémiai és DNS markerekkel
Budapesti Műszaki és Gazdaságtudományi Egyetem, Vegyészmérnöki Kar Biokémiai és Élelmiszertechnológiai Tanszék Szelekciós lehetőségek a búza minőség-orientált nemesítésében fehérjekémiai és DNS markerekkel
A genetikai lelet értelmezése monogénes betegségekben
 A genetikai lelet értelmezése monogénes betegségekben Tory Kálmán Semmelweis Egyetem, I. sz. Gyermekklinika A ~20 ezer fehérje-kódoló gén a 23 pár kromoszómán A kromoszómán található bázisok száma: 250M
A genetikai lelet értelmezése monogénes betegségekben Tory Kálmán Semmelweis Egyetem, I. sz. Gyermekklinika A ~20 ezer fehérje-kódoló gén a 23 pár kromoszómán A kromoszómán található bázisok száma: 250M
Fehérje expressziós rendszerek. Gyógyszerészi Biotechnológia
 Fehérje expressziós rendszerek Gyógyszerészi Biotechnológia Expressziós rendszerek Cél: rekombináns fehérjék előállítása nagy tisztaságban és nagy mennyiségben kísérleti ill. gyakorlati (therapia) felhasználásokra
Fehérje expressziós rendszerek Gyógyszerészi Biotechnológia Expressziós rendszerek Cél: rekombináns fehérjék előállítása nagy tisztaságban és nagy mennyiségben kísérleti ill. gyakorlati (therapia) felhasználásokra
In situ hibridizáció különböző módszereinek adaptálása és továbbfejlesztése búza genetikai alapanyagok elemzésére
 OTKA F037331 Zárójelentés 2006. In situ hibridizáció különböző módszereinek adaptálása és továbbfejlesztése búza genetikai alapanyagok elemzésére Kísérleteinkben célul tűztük ki a transzgének kimutatására
OTKA F037331 Zárójelentés 2006. In situ hibridizáció különböző módszereinek adaptálása és továbbfejlesztése búza genetikai alapanyagok elemzésére Kísérleteinkben célul tűztük ki a transzgének kimutatására
10. Genomika 2. Microarrayek és típusaik
 10. Genomika 2. 1. Microarray technikák és bioinformatikai vonatkozásaik Microarrayek és típusaik Korrelált génexpresszió mint a funkcionális genomika eszköze 2. Kombinált megközelítés a funkcionális genomikában
10. Genomika 2. 1. Microarray technikák és bioinformatikai vonatkozásaik Microarrayek és típusaik Korrelált génexpresszió mint a funkcionális genomika eszköze 2. Kombinált megközelítés a funkcionális genomikában
Bakteriális identifikáció 16S rrns gén szekvencia alapján
 Bakteriális identifikáció 16S rrns gén szekvencia alapján MOHR ANITA SIPOS RITA, SZÁNTÓ-EGÉSZ RÉKA, MICSINAI ADRIENN 2100 Gödöllő, Szent-Györgyi Albert út 4. info@biomi.hu, www.biomi.hu TÖRZS AZONOSÍTÁS
Bakteriális identifikáció 16S rrns gén szekvencia alapján MOHR ANITA SIPOS RITA, SZÁNTÓ-EGÉSZ RÉKA, MICSINAI ADRIENN 2100 Gödöllő, Szent-Györgyi Albert út 4. info@biomi.hu, www.biomi.hu TÖRZS AZONOSÍTÁS
Juhász Angéla MTA ATK MI Alkalmazott Genomikai Osztály SZEKVENCIA ADATBÁZISOK
 Juhász Angéla MTA ATK MI Alkalmazott Genomikai Osztály SZEKVENCIA ADATBÁZISOK Fehérjét kódol? Tulajdonságai? -Hol lokalizálódik? -Oldható? -3D szerkezete? -Accession #? -Annotációja elérhető? Már benne
Juhász Angéla MTA ATK MI Alkalmazott Genomikai Osztály SZEKVENCIA ADATBÁZISOK Fehérjét kódol? Tulajdonságai? -Hol lokalizálódik? -Oldható? -3D szerkezete? -Accession #? -Annotációja elérhető? Már benne
A doktori értekezés tézisei. A növényi NRP fehérjék lehetséges szerepe a hiszton defoszforiláció szabályozásában, és a hőstressz válaszban.
 A doktori értekezés tézisei A növényi NRP fehérjék lehetséges szerepe a hiszton defoszforiláció szabályozásában, és a hőstressz válaszban. Bíró Judit Témavezető: Dr. Fehér Attila Magyar Tudományos Akadémia
A doktori értekezés tézisei A növényi NRP fehérjék lehetséges szerepe a hiszton defoszforiláció szabályozásában, és a hőstressz válaszban. Bíró Judit Témavezető: Dr. Fehér Attila Magyar Tudományos Akadémia
SOLiD Technology. library preparation & Sequencing Chemistry (sequencing by ligation!) Imaging and analysis. Application specific sample preparation
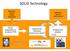 SOLiD Technology Application specific sample preparation Application specific data analysis library preparation & emulsion PCR Sequencing Chemistry (sequencing by ligation!) Imaging and analysis SOLiD
SOLiD Technology Application specific sample preparation Application specific data analysis library preparation & emulsion PCR Sequencing Chemistry (sequencing by ligation!) Imaging and analysis SOLiD
Egy 10,3 kb méretű, lineáris, a mitokondriumban lokalizált DNS-plazmidot izoláltunk a
 Egy 10,3 kb méretű, lineáris, a mitokondriumban lokalizált DNS-plazmidot izoláltunk a Fusarium proliferatum (Gibberella intermedia) ITEM 2337-es törzséből, és a plazmidot pfp1- nek neveztük el. Proteináz
Egy 10,3 kb méretű, lineáris, a mitokondriumban lokalizált DNS-plazmidot izoláltunk a Fusarium proliferatum (Gibberella intermedia) ITEM 2337-es törzséből, és a plazmidot pfp1- nek neveztük el. Proteináz
I. A sejttől a génekig
 Gén A gének olyan nukleinsav-szakaszok a sejtek magjainak kromoszómáiban, melyek a szervezet működését és növekedését befolyásoló fehérjék szabályozásához és előállításához szükséges információkat tartalmazzák.
Gén A gének olyan nukleinsav-szakaszok a sejtek magjainak kromoszómáiban, melyek a szervezet működését és növekedését befolyásoló fehérjék szabályozásához és előállításához szükséges információkat tartalmazzák.
NÖVÉNYÉLETTAN. Az Agrármérnöki MSc szak tananyagfejlesztése TÁMOP-4.1.2-08/1/A-2009-0010
 NÖVÉNYÉLETTAN Az Agrármérnöki MSc szak tananyagfejlesztése TÁMOP-4.1.2-08/1/A-2009-0010 Sejtfal szintézis és megnyúlás Környezeti tényezők hatása a növények növekedésére és fejlődésére Előadás áttekintése
NÖVÉNYÉLETTAN Az Agrármérnöki MSc szak tananyagfejlesztése TÁMOP-4.1.2-08/1/A-2009-0010 Sejtfal szintézis és megnyúlás Környezeti tényezők hatása a növények növekedésére és fejlődésére Előadás áttekintése
HAPMAP -2010 Nemzetközi HapMap Projekt. SNP GWA Haplotípus: egy kromoszóma szegmensen lévő SNP mintázat
 HAPMAP -2010 Nemzetközi HapMap Projekt A Nemzetközi HapMap Project célja az emberi genom haplotípus* térképének(hapmap; haplotype map) megszerkesztése, melynek segítségével katalogizálni tudjuk az ember
HAPMAP -2010 Nemzetközi HapMap Projekt A Nemzetközi HapMap Project célja az emberi genom haplotípus* térképének(hapmap; haplotype map) megszerkesztése, melynek segítségével katalogizálni tudjuk az ember
TDK lehetőségek az MTA TTK Enzimológiai Intézetben
 TDK lehetőségek az MTA TTK Enzimológiai Intézetben Vértessy G. Beáta egyetemi tanár TDK mind 1-3 helyezettek OTDK Pro Scientia különdíj 1 második díj Diákjaink Eredményei Zsűri különdíj 2 első díj OTDK
TDK lehetőségek az MTA TTK Enzimológiai Intézetben Vértessy G. Beáta egyetemi tanár TDK mind 1-3 helyezettek OTDK Pro Scientia különdíj 1 második díj Diákjaink Eredményei Zsűri különdíj 2 első díj OTDK
Mlo gén alapú lisztharmat rezisztencia: régi harc új megvilágításban Mlo gene based powdery mildew resistance: an old battle in a new light
 Vitányi Beáta 1 Nagy Katalin 2 Fekete Anna Katalin 3 Dudás Brigitta 4 Jenes Barnabás 5 Mlo gén alapú lisztharmat rezisztencia: régi harc új megvilágításban Mlo gene based powdery mildew resistance: an
Vitányi Beáta 1 Nagy Katalin 2 Fekete Anna Katalin 3 Dudás Brigitta 4 Jenes Barnabás 5 Mlo gén alapú lisztharmat rezisztencia: régi harc új megvilágításban Mlo gene based powdery mildew resistance: an
NÖVÉNYÉLETTAN. Az Agrármérnöki MSc szak tananyagfejlesztése TÁMOP /1/A
 NÖVÉNYÉLETTAN Az Agrármérnöki MSc szak tananyagfejlesztése TÁMOP-4.1.2-08/1/A-2009-0010 A növényi növekedés és fejlődés áttekintése Előadás áttekintése 1. A növekedés, differenciálódás és fejlődés fogalma
NÖVÉNYÉLETTAN Az Agrármérnöki MSc szak tananyagfejlesztése TÁMOP-4.1.2-08/1/A-2009-0010 A növényi növekedés és fejlődés áttekintése Előadás áttekintése 1. A növekedés, differenciálódás és fejlődés fogalma
ADATBÁNYÁSZAT I. ÉS OMICS
 Az élettudományi-klinikai felsőoktatás gyakorlatorientált és hallgatóbarát korszerűsítése a vidéki képzőhelyek nemzetközi versenyképességének erősítésére TÁMOP-4.1.1.C-13/1/KONV-2014-0001 ADATBÁNYÁSZAT
Az élettudományi-klinikai felsőoktatás gyakorlatorientált és hallgatóbarát korszerűsítése a vidéki képzőhelyek nemzetközi versenyképességének erősítésére TÁMOP-4.1.1.C-13/1/KONV-2014-0001 ADATBÁNYÁSZAT
Evolúcióbiológia. Biológus B.Sc tavaszi félév
 Evolúcióbiológia Biológus B.Sc. 2011. tavaszi félév A biológiában minden csak az evolúció fényében válik érthetővé Theodosius Dobzhansky : Nothing in biology makes sense except in the light of evolution.
Evolúcióbiológia Biológus B.Sc. 2011. tavaszi félév A biológiában minden csak az evolúció fényében válik érthetővé Theodosius Dobzhansky : Nothing in biology makes sense except in the light of evolution.
Az anafázis promoting complex (APC/C) katalitikus modulja Drosophila melanogasterben. Nagy Olga
 Ph.D. értekezés tézisei Az anafázis promoting complex (APC/C) katalitikus modulja Drosophila melanogasterben Nagy Olga Témavezető: Dr. Deák Péter MTA Szegedi Biológiai Kutatóközpont Biológia Doktori Iskola
Ph.D. értekezés tézisei Az anafázis promoting complex (APC/C) katalitikus modulja Drosophila melanogasterben Nagy Olga Témavezető: Dr. Deák Péter MTA Szegedi Biológiai Kutatóközpont Biológia Doktori Iskola
A FISH technika alkalmazása az előnemesítésben
 Linc Gabriella A FISH technika alkalmazása az előnemesítésben Czuczor Gergely Bencés Gimnázium és Kollégium Győr, 2016. április 13. www.meetthescientist.hu 1 26 - 1997-98 vendégkutató - növény genetikai
Linc Gabriella A FISH technika alkalmazása az előnemesítésben Czuczor Gergely Bencés Gimnázium és Kollégium Győr, 2016. április 13. www.meetthescientist.hu 1 26 - 1997-98 vendégkutató - növény genetikai
Genetikai panel kialakítása a hazai tejhasznú szarvasmarha állományok hasznos élettartamának növelésére
 Genetikai panel kialakítása a hazai tejhasznú szarvasmarha állományok hasznos élettartamának növelésére Dr. Czeglédi Levente Dr. Béri Béla Kutatás-fejlesztés támogatása a megújuló energiaforrások és agrár
Genetikai panel kialakítása a hazai tejhasznú szarvasmarha állományok hasznos élettartamának növelésére Dr. Czeglédi Levente Dr. Béri Béla Kutatás-fejlesztés támogatása a megújuló energiaforrások és agrár
avagy az ipari alkalmazhatóság kérdése biotechnológiai tárgyú szabadalmi bejelentéseknél Dr. Győrffy Béla, Egis Nyrt., Budapest
 Iparilag alkalmazható szekvenciák, avagy az ipari alkalmazhatóság kérdése biotechnológiai tárgyú szabadalmi bejelentéseknél Dr. Győrffy Béla, Egis Nyrt., Budapest Neutrokin α - jelentős kereskedelmi érdekek
Iparilag alkalmazható szekvenciák, avagy az ipari alkalmazhatóság kérdése biotechnológiai tárgyú szabadalmi bejelentéseknél Dr. Győrffy Béla, Egis Nyrt., Budapest Neutrokin α - jelentős kereskedelmi érdekek
NÖVÉNYGENETIKA. Az Agrármérnöki MSc szak tananyagfejlesztése TÁMOP /1/A
 NÖVÉNYGENETIKA Az Agrármérnöki MSc szak tananyagfejlesztése TÁMOP-4.1.2-08/1/A-2009-0010 A citológia és a genetika társtudománya Citogenetika A kromoszómák eredetét, szerkezetét, genetikai funkcióját,
NÖVÉNYGENETIKA Az Agrármérnöki MSc szak tananyagfejlesztése TÁMOP-4.1.2-08/1/A-2009-0010 A citológia és a genetika társtudománya Citogenetika A kromoszómák eredetét, szerkezetét, genetikai funkcióját,
Molekuláris genetikai vizsgáló. módszerek az immundefektusok. diagnosztikájában
 Molekuláris genetikai vizsgáló módszerek az immundefektusok diagnosztikájában Primer immundefektusok A primer immundeficiencia ritka, veleszületett, monogénes öröklődésű immunhiányos állapot. Családi halmozódást
Molekuláris genetikai vizsgáló módszerek az immundefektusok diagnosztikájában Primer immundefektusok A primer immundeficiencia ritka, veleszületett, monogénes öröklődésű immunhiányos állapot. Családi halmozódást
Sejtmag, magvacska magmembrán
 Sejtmag, magvacska magmembrán Láng Orsolya Semmelweis Egyetem, Genetikai, Sejt- és Immunbiológiai Intézet Kompartmentalizáció Prokaryóta Cytoplazma Eukaryóta Endomembrán Kromatin Plazma membrán Eredménye
Sejtmag, magvacska magmembrán Láng Orsolya Semmelweis Egyetem, Genetikai, Sejt- és Immunbiológiai Intézet Kompartmentalizáció Prokaryóta Cytoplazma Eukaryóta Endomembrán Kromatin Plazma membrán Eredménye
NÖVÉNYÉLETTAN. Az Agrármérnöki MSc szak tananyagfejlesztése TÁMOP /1/A
 NÖVÉNYÉLETTAN Az Agrármérnöki MSc szak tananyagfejlesztése TÁMOP-4.1.2-08/1/A-2009-0010 Gibberellinek és citokininek Előadás áttekintése 1. Gibberellinek: a növénymagasság és csírázás hormonjai 2. A gibberellinek
NÖVÉNYÉLETTAN Az Agrármérnöki MSc szak tananyagfejlesztése TÁMOP-4.1.2-08/1/A-2009-0010 Gibberellinek és citokininek Előadás áttekintése 1. Gibberellinek: a növénymagasság és csírázás hormonjai 2. A gibberellinek
A DNS szerkezete. Genom kromoszóma gén DNS genotípus - allél. Pontos méretek Watson genomja. J. D. Watson F. H. C. Crick. 2 nm C G.
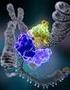 1955: 46 emberi kromoszóma van 1961: mrns 1975: DNS szekvenálás 1982: gén-bank adatbázisok 1983: R (polymerase chain reaction) Mérföldkövek 1 J. D. Watson F. H.. rick 2008 1953 2003 Watson genomja DNS
1955: 46 emberi kromoszóma van 1961: mrns 1975: DNS szekvenálás 1982: gén-bank adatbázisok 1983: R (polymerase chain reaction) Mérföldkövek 1 J. D. Watson F. H.. rick 2008 1953 2003 Watson genomja DNS
A GABONAFÉLÉK EGYEDFEJLŐDÉSÉT ÉS KALÁSZOLÁSÁT MEGHATÁROZÓ GENETIKAI KOMPONENSEK TANULMÁNYOZÁSA
 A martonvásári agrárkutatások hatodik évtizede A GABONAFÉLÉK EGYEDFEJLŐDÉSÉT ÉS KALÁSZOLÁSÁT MEGHATÁROZÓ GENETIKAI KOMPONENSEK TANULMÁNYOZÁSA KARSAI ILDIKÓ 1, MÉSZÁROS KLÁRA 2, KŐSZEGI BÉLA 3, PATRICK
A martonvásári agrárkutatások hatodik évtizede A GABONAFÉLÉK EGYEDFEJLŐDÉSÉT ÉS KALÁSZOLÁSÁT MEGHATÁROZÓ GENETIKAI KOMPONENSEK TANULMÁNYOZÁSA KARSAI ILDIKÓ 1, MÉSZÁROS KLÁRA 2, KŐSZEGI BÉLA 3, PATRICK
AZ ÁRPA SZÁRAZSÁGTŰRÉSÉNEK VIZSGÁLATA: QTL- ÉS ASSZOCIÁCIÓS ANALÍZIS, MARKER ALAPÚ SZELEKCIÓ, TILLING
 Hagyomány és haladás a növénynemesítésben AZ ÁRPA SZÁRAZSÁGTŰRÉSÉNEK VIZSGÁLATA: QTL- ÉS ASSZOCIÁCIÓS ANALÍZIS, MARKER ALAPÚ SZELEKCIÓ, BÁLINT ANDRÁS FERENC 1, SZIRA FRUZSINA 1, ANDREAS BÖRNER 2, KERSTIN
Hagyomány és haladás a növénynemesítésben AZ ÁRPA SZÁRAZSÁGTŰRÉSÉNEK VIZSGÁLATA: QTL- ÉS ASSZOCIÁCIÓS ANALÍZIS, MARKER ALAPÚ SZELEKCIÓ, BÁLINT ANDRÁS FERENC 1, SZIRA FRUZSINA 1, ANDREAS BÖRNER 2, KERSTIN
Evolúcióelmélet és az evolúció mechanizmusai
 Evolúcióelmélet és az evolúció mechanizmusai Az élet Darwini szemlélete Melyek az evolúció bizonyítékai a világban? EVOLÚCIÓ: VÁLTOZATOSSÁG Mutáció Horizontális géntranszfer Genetikai rekombináció Rekombináció
Evolúcióelmélet és az evolúció mechanizmusai Az élet Darwini szemlélete Melyek az evolúció bizonyítékai a világban? EVOLÚCIÓ: VÁLTOZATOSSÁG Mutáció Horizontális géntranszfer Genetikai rekombináció Rekombináció
A sikérfehérjék összetétele, hatásuk a sikér reológiai tulajdonságaira (Szemle)
 A sikérfehérjék összetétele, hatásuk a sikér reológiai tulajdonságaira (Szemle) Uri Csilla Tóth Árpád Sipos Péter Borbélyné Varga Mária Győri Zoltán Debreceni Egyetem Agrártudományi Centrum, Mezőgazdaságtudományi
A sikérfehérjék összetétele, hatásuk a sikér reológiai tulajdonságaira (Szemle) Uri Csilla Tóth Árpád Sipos Péter Borbélyné Varga Mária Győri Zoltán Debreceni Egyetem Agrártudományi Centrum, Mezőgazdaságtudományi
Tudománytörténeti visszatekintés
 GENETIKA I. AZ ÖRÖKLŐDÉS TÖRVÉNYSZERŰSÉGEI Minek köszönhető a biológiai sokféleség? Hogyan történik a tulajdonságok átörökítése? Tudománytörténeti visszatekintés 1. Keveredés alapú öröklődés: (1761-1766,
GENETIKA I. AZ ÖRÖKLŐDÉS TÖRVÉNYSZERŰSÉGEI Minek köszönhető a biológiai sokféleség? Hogyan történik a tulajdonságok átörökítése? Tudománytörténeti visszatekintés 1. Keveredés alapú öröklődés: (1761-1766,
MOLEKULÁRIS MODELL A FAGYÁLLÓSÁG ÉS A VERNALIZÁCIÓS IGÉNY KÖLCSÖNHATÁSÁNAK ÉRTELMEZÉSÉRE
 Hagyomány és haladás a növénynemesítésben MOLEKULÁRIS MODELL A FAGYÁLLÓSÁG ÉS A VERNALIZÁCIÓS IGÉNY KÖLCSÖNHATÁSÁNAK ÉRTELMEZÉSÉRE GALIBA GÁBOR 1,2, VÁGÚJFALVI ATTILA 1, CHENGXIA LI 3, SOLTÉSZ ALEXANDRA
Hagyomány és haladás a növénynemesítésben MOLEKULÁRIS MODELL A FAGYÁLLÓSÁG ÉS A VERNALIZÁCIÓS IGÉNY KÖLCSÖNHATÁSÁNAK ÉRTELMEZÉSÉRE GALIBA GÁBOR 1,2, VÁGÚJFALVI ATTILA 1, CHENGXIA LI 3, SOLTÉSZ ALEXANDRA
DNS-szekvencia meghatározás
 DNS-szekvencia meghatározás Gilbert 1980 (1958) Sanger 3-1 A DNS-polimerázok jellemzői 5'-3' polimeráz aktivitás 5'-3' exonukleáz 3'-5' exonukleáz aktivitás Az új szál szintéziséhez kell: templát DNS primer
DNS-szekvencia meghatározás Gilbert 1980 (1958) Sanger 3-1 A DNS-polimerázok jellemzői 5'-3' polimeráz aktivitás 5'-3' exonukleáz 3'-5' exonukleáz aktivitás Az új szál szintéziséhez kell: templát DNS primer
Génmódosítás: bioszféra
 bioszféra Génmódosítás: Nagy butaság volt politikusaink részérôl az alaptalan GMO-ellenesség alaptörvényben való rögzítése. A témával foglalkozó akadémikusok véleménye külföldön és Magyarországon egészen
bioszféra Génmódosítás: Nagy butaság volt politikusaink részérôl az alaptalan GMO-ellenesség alaptörvényben való rögzítése. A témával foglalkozó akadémikusok véleménye külföldön és Magyarországon egészen
Klónozás: tökéletesen egyforma szervezetek csoportjának előállítása, vagyis több genetikailag azonos egyed létrehozása.
 Növények klónozása Klónozás Klónozás: tökéletesen egyforma szervezetek csoportjának előállítása, vagyis több genetikailag azonos egyed létrehozása. Görög szó: klon, jelentése: gally, hajtás, vessző. Ami
Növények klónozása Klónozás Klónozás: tökéletesen egyforma szervezetek csoportjának előállítása, vagyis több genetikailag azonos egyed létrehozása. Görög szó: klon, jelentése: gally, hajtás, vessző. Ami
Genomadatbázisok Ld. Entrez Genome: Összes ismert genom, hierarchikus szervezésben (kromoszóma, térképek, gének, stb.)
 Genomika Új korszak, paradigmaváltás, forradalom: a teljes genomok ismeretében a biológia adatokban gazdag tudománnyá válik. Új kutatási módszerek, új szemlélet. Hajtóerõk: Genomszekvenálási projektek
Genomika Új korszak, paradigmaváltás, forradalom: a teljes genomok ismeretében a biológia adatokban gazdag tudománnyá válik. Új kutatási módszerek, új szemlélet. Hajtóerõk: Genomszekvenálási projektek
Áttekintés Az ALKOBEER projekt hét esztendeje Az alakor organikus nemesítése
 Áttekintés Az ALKOBEER projekt hét esztendeje Az alakor organikus nemesítése MTA Agrártudományi Kutatóközpont Kovács Géza - - Mikó Péter Áttekintés Áttekintés Az Az ALKOBEER ALKOBEER projekt projekt hét
Áttekintés Az ALKOBEER projekt hét esztendeje Az alakor organikus nemesítése MTA Agrártudományi Kutatóközpont Kovács Géza - - Mikó Péter Áttekintés Áttekintés Az Az ALKOBEER ALKOBEER projekt projekt hét
Hazai méhészeti genomikai és genetikai vizsgálatok
 AKÁCKÖRÚTON Hazai méhészeti genomikai és genetikai vizsgálatok Előző cikkünkben arról írtunk, milyen új eszköztárral rendelkezünk a XXI. században a genetikai vizsgálatok területén, és mit adhat a molekuláris
AKÁCKÖRÚTON Hazai méhészeti genomikai és genetikai vizsgálatok Előző cikkünkben arról írtunk, milyen új eszköztárral rendelkezünk a XXI. században a genetikai vizsgálatok területén, és mit adhat a molekuláris
BÁLINT András Beszámoló az AGRISAFE által támogatott tanulmányútról 2008 november 2009 február. 1. Az IPK bemutatása 2.
 BÁLINT András Beszámoló az AGRISAFE által támogatott tanulmányútról 2008 november 2009 február 1. Az IPK bemutatása 2. A TILLING módszer Hol található az IPK? Gatersleben Általános adatok az IPK-ról Leibniz
BÁLINT András Beszámoló az AGRISAFE által támogatott tanulmányútról 2008 november 2009 február 1. Az IPK bemutatása 2. A TILLING módszer Hol található az IPK? Gatersleben Általános adatok az IPK-ról Leibniz
A HUMÁN GENOM PROJEKT Sasvári-Székely Mária* Semmelweis Egyetem, Orvosi Vegytani, Molekuláris Biológiai és Pathobiokémiai Intézet
 A HUMÁN GENOM PROJEKT Sasvári-Székely Mária* Semmelweis Egyetem, Orvosi Vegytani, Molekuláris Biológiai és Pathobiokémiai Intézet *Levelezési cím: Dr. Sasvári-Székely Mária, Semmelweis Egyetem, Orvosi
A HUMÁN GENOM PROJEKT Sasvári-Székely Mária* Semmelweis Egyetem, Orvosi Vegytani, Molekuláris Biológiai és Pathobiokémiai Intézet *Levelezési cím: Dr. Sasvári-Székely Mária, Semmelweis Egyetem, Orvosi
Új tartalékfehérje gének izolálása a Bánkúti 1201 búzafajtából. doktori értekezés tézisei. készítette: Nagy Ila Julietta. témavezető: Dr Takács Imre
 Új tartalékfehérje gének izolálása a Bánkúti 1201 búzafajtából doktori értekezés tézisei készítette: Nagy Ila Julietta Biológia Doktori Iskola Doktori Iskola vezetője: Erdei Anna Kísérletes Növénybiológia
Új tartalékfehérje gének izolálása a Bánkúti 1201 búzafajtából doktori értekezés tézisei készítette: Nagy Ila Julietta Biológia Doktori Iskola Doktori Iskola vezetője: Erdei Anna Kísérletes Növénybiológia
A C. elegans TRA-1/GLI/Ci szex-determinációs faktor célgénjeinek meghatározása és analízise. Doktori értekezés tézisei.
 A C. elegans TRA-1/GLI/Ci szex-determinációs faktor célgénjeinek meghatározása és analízise Doktori értekezés tézisei Hargitai Balázs Eötvös Loránd Tudományegyetem Természettudományi Kar Biológia Doktori
A C. elegans TRA-1/GLI/Ci szex-determinációs faktor célgénjeinek meghatározása és analízise Doktori értekezés tézisei Hargitai Balázs Eötvös Loránd Tudományegyetem Természettudományi Kar Biológia Doktori
Egy új genetikai módszerrel azonosított Arabidopsis A4A hősokk faktor funkcionális jellemzése
 Egy új genetikai módszerrel azonosított Arabidopsis A4A hősokk faktor funkcionális jellemzése Tézisfüzet Immaculada Pérez Salamó Témavezető: Dr. Szabados László MTA Szegedi Biológiai Központ Növénybiológia
Egy új genetikai módszerrel azonosított Arabidopsis A4A hősokk faktor funkcionális jellemzése Tézisfüzet Immaculada Pérez Salamó Témavezető: Dr. Szabados László MTA Szegedi Biológiai Központ Növénybiológia
13. RNS szintézis és splicing
 13. RNS szintézis és splicing 1 Visszatekintés: Az RNS típusai és szerkezete Hírvivő RNS = mrns (messenger RNA = mrna) : fehérjeszintézis pre-mrns érett mrns (intronok kivágódnak = splicing) Transzfer
13. RNS szintézis és splicing 1 Visszatekintés: Az RNS típusai és szerkezete Hírvivő RNS = mrns (messenger RNA = mrna) : fehérjeszintézis pre-mrns érett mrns (intronok kivágódnak = splicing) Transzfer
Bioinformatika - egészséges környezet, egészséges élelmiszer
 CESCI - III. SZENTGOTTHÁRDI SZLOVÉN MAGYAR FÓRUM Bioinformatika - egészséges környezet, egészséges élelmiszer Pannon Bio-Innováció Kft Taller János, PhD ügyvezető Szentgotthárd, 2017. május 23. 1 Pannon
CESCI - III. SZENTGOTTHÁRDI SZLOVÉN MAGYAR FÓRUM Bioinformatika - egészséges környezet, egészséges élelmiszer Pannon Bio-Innováció Kft Taller János, PhD ügyvezető Szentgotthárd, 2017. május 23. 1 Pannon
Algaközösségek ökológiai, morfológiai és genetikai diverzitásának összehasonlítása szentély jellegű és emberi használatnak kitett élőhelykomplexekben
 Algaközösségek ökológiai, morfológiai és genetikai diverzitásának összehasonlítása szentély jellegű és emberi használatnak kitett élőhelykomplexekben Duleba Mónika Környezettudományi Doktori Iskola I.
Algaközösségek ökológiai, morfológiai és genetikai diverzitásának összehasonlítása szentély jellegű és emberi használatnak kitett élőhelykomplexekben Duleba Mónika Környezettudományi Doktori Iskola I.
Genetika 2. előadás. Bevezető
 Genetika 2. előadás Genetikai alapelvek: hogyan öröklődnek a tulajdonságok Mendeli genetika Bevezető Mi okozza a hasonlóságokat és különbségeket a családtagok között? Gének: biológiai információ alapegysége
Genetika 2. előadás Genetikai alapelvek: hogyan öröklődnek a tulajdonságok Mendeli genetika Bevezető Mi okozza a hasonlóságokat és különbségeket a családtagok között? Gének: biológiai információ alapegysége
A HLTF koordinált fehérje és DNS átrendezése és aktivitásának összehasonlítása a Bloom szindróma helikáz fehérjével
 A HLTF koordinált fehérje és DNS átrendezése és aktivitásának összehasonlítása a Bloom szindróma helikáz fehérjével Ph.D disszertáció tézisei Yathish Jagadheesh Achar Témavezető: Dr. Haracska Lajos Magyar
A HLTF koordinált fehérje és DNS átrendezése és aktivitásának összehasonlítása a Bloom szindróma helikáz fehérjével Ph.D disszertáció tézisei Yathish Jagadheesh Achar Témavezető: Dr. Haracska Lajos Magyar
A növény inváziójában szerepet játszó bakteriális gének
 A növény inváziójában szerepet játszó bakteriális gének merisztéma korai szimbiotikus zóna késői szimbiotikus zóna öregedési zóna gyökér keresztmetszet NODULÁCIÓ növényi jel Rhizobium meliloti rhizobium
A növény inváziójában szerepet játszó bakteriális gének merisztéma korai szimbiotikus zóna késői szimbiotikus zóna öregedési zóna gyökér keresztmetszet NODULÁCIÓ növényi jel Rhizobium meliloti rhizobium
A genomikai oktatás helyzete a Debreceni Egyetemen
 A genomikai oktatás helyzete a Debreceni Egyetemen Bálint Bálint L. GNTP Oktatás és Tudásmenedzsment Munkabizottság, 2009. június 10. Tények Debreceni Egyetemről 21000 nappali és 33000 összes hallgató
A genomikai oktatás helyzete a Debreceni Egyetemen Bálint Bálint L. GNTP Oktatás és Tudásmenedzsment Munkabizottság, 2009. június 10. Tények Debreceni Egyetemről 21000 nappali és 33000 összes hallgató
A proteomika új tudománya és alkalmazása a rákdiagnosztikában
 BIOTECHNOLÓGIAI FEJLESZTÉSI POLITIKA, KUTATÁSI IRÁNYOK A proteomika új tudománya és alkalmazása a rákdiagnosztikában Tárgyszavak: proteom; proteomika; rák; diagnosztika; molekuláris gyógyászat; biomarker;
BIOTECHNOLÓGIAI FEJLESZTÉSI POLITIKA, KUTATÁSI IRÁNYOK A proteomika új tudománya és alkalmazása a rákdiagnosztikában Tárgyszavak: proteom; proteomika; rák; diagnosztika; molekuláris gyógyászat; biomarker;
Zárójelentés. Állati rotavírusok összehasonlító genomvizsgálata. c. OTKA kutatási programról. Bányai Krisztián (MTA ATK ÁOTI)
 Zárójelentés Állati rotavírusok összehasonlító genomvizsgálata c. OTKA kutatási programról Bányai Krisztián (MTA ATK ÁOTI) 2012 1 Az Állati rotavírusok összehasonlító genomvizsgálata c. programban azt
Zárójelentés Állati rotavírusok összehasonlító genomvizsgálata c. OTKA kutatási programról Bányai Krisztián (MTA ATK ÁOTI) 2012 1 Az Állati rotavírusok összehasonlító genomvizsgálata c. programban azt
BIOLÓGIA HÁZIVERSENY 1. FORDULÓ BIOKÉMIA, GENETIKA BIOKÉMIA, GENETIKA
 BIOKÉMIA, GENETIKA 1. Nukleinsavak keresztrejtvény (12+1 p) 1. 2. 3. 4. 5. 6. 7. 8. 9. 10. 11. 12. 1. A nukleinsavak a.-ok összekapcsolódásával kialakuló polimerek. 2. Purinvázas szerves bázis, amely az
BIOKÉMIA, GENETIKA 1. Nukleinsavak keresztrejtvény (12+1 p) 1. 2. 3. 4. 5. 6. 7. 8. 9. 10. 11. 12. 1. A nukleinsavak a.-ok összekapcsolódásával kialakuló polimerek. 2. Purinvázas szerves bázis, amely az
Diagnosztikai célú molekuláris biológiai vizsgálatok
 Diagnosztikai célú molekuláris biológiai vizsgálatok Dr. Patócs Attila, PhD MTA-SE Molekuláris Medicina Kutatócsoport, Semmelweis Egyetem II. sz. Belgyógyászati Klinika Laboratóriumi Medicina Intézet Genetikai
Diagnosztikai célú molekuláris biológiai vizsgálatok Dr. Patócs Attila, PhD MTA-SE Molekuláris Medicina Kutatócsoport, Semmelweis Egyetem II. sz. Belgyógyászati Klinika Laboratóriumi Medicina Intézet Genetikai
Tipizálási módszerek alkalmazása methicillin-rezisztens Staphylococcus aureus (MRSA) törzsek molekuláris epidemiológiai vizsgálatai során
 Tipizálási módszerek alkalmazása methicillin-rezisztens Staphylococcus aureus (MRSA) törzsek molekuláris epidemiológiai vizsgálatai során Ungvári Erika, Tóth Ákos Magyar Infektológiai és Klinikai Mikrobiológiai
Tipizálási módszerek alkalmazása methicillin-rezisztens Staphylococcus aureus (MRSA) törzsek molekuláris epidemiológiai vizsgálatai során Ungvári Erika, Tóth Ákos Magyar Infektológiai és Klinikai Mikrobiológiai
Dr. Máthéné Dr. Szigeti Zsuzsanna és munkatársai
 Kar: TTK Tantárgy: CITOGENETIKA Kód: AOMBCGE3 ECTS Kredit: 3 A tantárgyat oktató intézet: TTK Mikrobiális Biotechnológiai és Sejtbiológiai Tanszék A tantárgy felvételére ajánlott félév: 3. Melyik félévben
Kar: TTK Tantárgy: CITOGENETIKA Kód: AOMBCGE3 ECTS Kredit: 3 A tantárgyat oktató intézet: TTK Mikrobiális Biotechnológiai és Sejtbiológiai Tanszék A tantárgy felvételére ajánlott félév: 3. Melyik félévben
A mutáns fenotípushoz szorosan kapcsolt markerek (1N1R és U212D) segítségével BAC (Bacterial Artifical Chromosome) klónokat azonosítottunk egy másik
 A szimbiotikus gümő kialakulásában résztvevő két gén azonosítása Medicago truncatulaból térképezésen alapuló génizolálással c. OTKA pályázat (T046645) zárójelentése A pályázat célja a Sinorhizobium meliloti
A szimbiotikus gümő kialakulásában résztvevő két gén azonosítása Medicago truncatulaból térképezésen alapuló génizolálással c. OTKA pályázat (T046645) zárójelentése A pályázat célja a Sinorhizobium meliloti
Bioinformatika előadás
 10. előadás Prof. Poppe László BME Szerves Kémia és Technológia Tsz. Bioinformatika proteomika Előadás és gyakorlat Genomika vs. proteomika A genomika módszereivel nem a tényleges fehérjéket vizsgáljuk,
10. előadás Prof. Poppe László BME Szerves Kémia és Technológia Tsz. Bioinformatika proteomika Előadás és gyakorlat Genomika vs. proteomika A genomika módszereivel nem a tényleges fehérjéket vizsgáljuk,
Az orvosi biotechnológiai mesterképzés megfeleltetése az Európai Unió új társadalmi kihívásainak a Pécsi Tudományegyetemen és a Debreceni Egyetemen
 Az orvosi biotechnológiai mesterképzés megfeleltetése az Európai Unió új társadalmi kihívásainak a Pécsi Tudományegyetemen és a Debreceni Egyetemen Azonosító szám: TÁMOP-4.1.2-08/1/A-2009-0011 Az orvosi
Az orvosi biotechnológiai mesterképzés megfeleltetése az Európai Unió új társadalmi kihívásainak a Pécsi Tudományegyetemen és a Debreceni Egyetemen Azonosító szám: TÁMOP-4.1.2-08/1/A-2009-0011 Az orvosi
A HUMÁNGENETIKA LEGÚJABB EREDMÉNYEI Péterfy Miklós
 A HUMÁNGENETIKA LEGÚJABB EREDMÉNYEI Péterfy Miklós Összefoglalás A humángenetika korunk egyik legdinamikusabban fejlődő tudományága. Ennek a fejlődésnek legfőbb mozgatórugója az, hogy a humángenetika,
A HUMÁNGENETIKA LEGÚJABB EREDMÉNYEI Péterfy Miklós Összefoglalás A humángenetika korunk egyik legdinamikusabban fejlődő tudományága. Ennek a fejlődésnek legfőbb mozgatórugója az, hogy a humángenetika,
TÁMOP /1/KONV
 A Pannon Növény-Biotechnológiai Egyesület és a Pannon Egyetem Georgikon Karának Doktori iskolája csatlakozva az Európai Növénytudományi Társaság (EPSO) szervezésében tartandó Növények Napjához, konferenciát
A Pannon Növény-Biotechnológiai Egyesület és a Pannon Egyetem Georgikon Karának Doktori iskolája csatlakozva az Európai Növénytudományi Társaság (EPSO) szervezésében tartandó Növények Napjához, konferenciát
NÖVÉNYGENETIKA. Az Agrármérnöki MSc szak tananyagfejlesztése TÁMOP /1/A
 NÖVÉNYGENETIKA Az Agrármérnöki MSc szak tananyagfejlesztése TÁMOP-4.1.2-08/1/A-2009-0010 Az előadás áttekintése A tantárgy keretében megtárgyalandó ismeretkörök A félév elfogadásának feltételei, követelmények
NÖVÉNYGENETIKA Az Agrármérnöki MSc szak tananyagfejlesztése TÁMOP-4.1.2-08/1/A-2009-0010 Az előadás áttekintése A tantárgy keretében megtárgyalandó ismeretkörök A félév elfogadásának feltételei, követelmények
Transzgénikus állatok előállítása
 Transzgénikus állatok előállítása A biotechnológia alapjai Pomázi Andrea Mezőgazdasági biotechnológia A gazdasági állatok és növények nemesítése új biotechnológiai eljárások felhasználásával. Cél: jobb
Transzgénikus állatok előállítása A biotechnológia alapjai Pomázi Andrea Mezőgazdasági biotechnológia A gazdasági állatok és növények nemesítése új biotechnológiai eljárások felhasználásával. Cél: jobb
A TATA-kötő fehérje asszociált faktor 3 (TAF3) p53-mal való kölcsönhatásának funkcionális vizsgálata
 Ph.D. ÉRTEKEZÉS TÉZISEI A TATA-kötő fehérje asszociált faktor 3 (TAF3) p53-mal való kölcsönhatásának funkcionális vizsgálata Buzás-Bereczki Orsolya Témavezetők: Dr. Bálint Éva Dr. Boros Imre Miklós Biológia
Ph.D. ÉRTEKEZÉS TÉZISEI A TATA-kötő fehérje asszociált faktor 3 (TAF3) p53-mal való kölcsönhatásának funkcionális vizsgálata Buzás-Bereczki Orsolya Témavezetők: Dr. Bálint Éva Dr. Boros Imre Miklós Biológia
Szakmai záró jelentés az OTKA PD83444 sz. pályázathoz
 Szakmai záró jelentés az OTKA PD83444 sz. pályázathoz A búza rokonsági köréhez tartozó vad fajok az agronómiai tulajdonságokért felelős gének olyan allél változatait hordozzák, amelyek a búzában nem fordulnak
Szakmai záró jelentés az OTKA PD83444 sz. pályázathoz A búza rokonsági köréhez tartozó vad fajok az agronómiai tulajdonságokért felelős gének olyan allél változatait hordozzák, amelyek a búzában nem fordulnak
Evolúció. Dr. Szemethy László egyetemi docens Szent István Egyetem VadVilág Megőrzési Intézet
 Evolúció Dr. Szemethy László egyetemi docens Szent István Egyetem VadVilág Megőrzési Intézet Mi az evolúció? Egy folyamat: az élőlények tulajdonságainak változása a környezethez való alkalmazkodásra Egy
Evolúció Dr. Szemethy László egyetemi docens Szent István Egyetem VadVilág Megőrzési Intézet Mi az evolúció? Egy folyamat: az élőlények tulajdonságainak változása a környezethez való alkalmazkodásra Egy
NÖVÉNYI GMO-król ALAPFOKON Dudits Dénes 1. Tartalom
 NÖVÉNYI GMO-król ALAPFOKON Dudits Dénes 1 Tartalom 1. Összefoglalás 2. Bevezetés 3. A genetikai módosítás (GM) alapfogalmai, története 4. Növényi gének kémcsőben: genom programok 4.1. A termőképességet,
NÖVÉNYI GMO-król ALAPFOKON Dudits Dénes 1 Tartalom 1. Összefoglalás 2. Bevezetés 3. A genetikai módosítás (GM) alapfogalmai, története 4. Növényi gének kémcsőben: genom programok 4.1. A termőképességet,
Az X kromoszóma inaktívációja. A kromatin szerkezet befolyásolja a génexpressziót
 Az X kromoszóma inaktívációja A kromatin szerkezet befolyásolja a génexpressziót Férfiak: XY Nők: XX X kromoszóma: nagy méretű több mint 1000 gén Y kromoszóma: kis méretű, kevesebb, mint 100 gén Kompenzációs
Az X kromoszóma inaktívációja A kromatin szerkezet befolyásolja a génexpressziót Férfiak: XY Nők: XX X kromoszóma: nagy méretű több mint 1000 gén Y kromoszóma: kis méretű, kevesebb, mint 100 gén Kompenzációs
Genomika. Mutációk (SNP-k) és vizsgálatuk egyszerű módszerekkel. DNS szekvenálási eljárások. DNS ujjlenyomat (VNTR)
 Genomika (A genom, génállomány vizsgálata) Mutációk (SNP-k) és vizsgálatuk egyszerű módszerekkel DNS szekvenálási eljárások DNS ujjlenyomat (VNTR) DNS chipek statikus és dinamikus információk vizsgálata
Genomika (A genom, génállomány vizsgálata) Mutációk (SNP-k) és vizsgálatuk egyszerű módszerekkel DNS szekvenálási eljárások DNS ujjlenyomat (VNTR) DNS chipek statikus és dinamikus információk vizsgálata
A Hardy-Weinberg egyensúly. 2. gyakorlat
 A Hardy-Weinberg egyensúly 2. gyakorlat A Hardy-Weinberg egyensúly feltételei: nincs szelekció nincs migráció nagy populációméret (nincs sodródás) nincs mutáció pánmixis van allélgyakoriság azonos hímekben
A Hardy-Weinberg egyensúly 2. gyakorlat A Hardy-Weinberg egyensúly feltételei: nincs szelekció nincs migráció nagy populációméret (nincs sodródás) nincs mutáció pánmixis van allélgyakoriság azonos hímekben
NÖVÉNYGENETIKA. Az Agrármérnöki MSc szak tananyagfejlesztése TÁMOP /1/A
 NÖVÉNYGENETIKA Az Agrármérnöki MSc szak tananyagfejlesztése TÁMOP-4.1.2-08/1/A-2009-0010 Faj- és nemzetségkeresztezések jelentősége -A termesztett növényekkel rokon kultúr- és vad fajok értékes génforrást
NÖVÉNYGENETIKA Az Agrármérnöki MSc szak tananyagfejlesztése TÁMOP-4.1.2-08/1/A-2009-0010 Faj- és nemzetségkeresztezések jelentősége -A termesztett növényekkel rokon kultúr- és vad fajok értékes génforrást
Egy Polycomb Response Element (PRE) in situ vizsgálata Drosophila melanogaster-ben génkonverzió segítségével. Kozma Gabriella
 Egy Polycomb Response Element (PRE) in situ vizsgálata Drosophila melanogaster-ben génkonverzió segítségével Kozma Gabriella Ph.D. tézisek Témavezető: Dr. Sipos László Genetikai Intézet MTA Szegedi Biológiai
Egy Polycomb Response Element (PRE) in situ vizsgálata Drosophila melanogaster-ben génkonverzió segítségével Kozma Gabriella Ph.D. tézisek Témavezető: Dr. Sipos László Genetikai Intézet MTA Szegedi Biológiai
Biológia egészségtan Általános iskola 7. osztály
 Általános iskola 7. osztály A tanuló értse az éghajlati övezetek kialakulásának okait és a biomok összetételének összefüggéseit az adott térségre jellemző környezeti tényezőkkel. Ismerje a globális környezetkárosítás
Általános iskola 7. osztály A tanuló értse az éghajlati övezetek kialakulásának okait és a biomok összetételének összefüggéseit az adott térségre jellemző környezeti tényezőkkel. Ismerje a globális környezetkárosítás
In Situ Hibridizáció a pathologiai diagnosztikában és ami mögötte van.
 In Situ Hibridizáció a pathologiai diagnosztikában és ami mögötte van. Kneif Józsefné PTE KK Pathologiai Intézet Budapest 2017. 05. 26 Kromoszóma rendellenesség kimutatás PCR technika: izolált nukleinsavak
In Situ Hibridizáció a pathologiai diagnosztikában és ami mögötte van. Kneif Józsefné PTE KK Pathologiai Intézet Budapest 2017. 05. 26 Kromoszóma rendellenesség kimutatás PCR technika: izolált nukleinsavak
Norvég Finanszírozási Mechanizmus által támogatott projekt HU-0115/NA/2008-3/ÖP-9 ÚJ TERÁPIÁS CÉLPONTOK AZONOSÍTÁSA GENOMIKAI MÓDSZEREKKEL
 Norvég Finanszírozási Mechanizmus által támogatott projekt HU-0115/NA/2008-3/ÖP-9 ÚJ TERÁPIÁS CÉLPONTOK AZONOSÍTÁSA GENOMIKAI MÓDSZEREKKEL KÖZÖS STRATÉGIA KIFEJLESZTÉSE MOLEKULÁRIS MÓDSZEREK ALKALMAZÁSÁVAL
Norvég Finanszírozási Mechanizmus által támogatott projekt HU-0115/NA/2008-3/ÖP-9 ÚJ TERÁPIÁS CÉLPONTOK AZONOSÍTÁSA GENOMIKAI MÓDSZEREKKEL KÖZÖS STRATÉGIA KIFEJLESZTÉSE MOLEKULÁRIS MÓDSZEREK ALKALMAZÁSÁVAL
3. Általános egészségügyi ismeretek az egyes témákhoz kapcsolódóan
 11. évfolyam BIOLÓGIA 1. Az emberi test szabályozása Idegi szabályozás Hormonális szabályozás 2. Az érzékelés Szaglás, tapintás, látás, íz érzéklés, 3. Általános egészségügyi ismeretek az egyes témákhoz
11. évfolyam BIOLÓGIA 1. Az emberi test szabályozása Idegi szabályozás Hormonális szabályozás 2. Az érzékelés Szaglás, tapintás, látás, íz érzéklés, 3. Általános egészségügyi ismeretek az egyes témákhoz
sejt működés jovo.notebook March 13, 2018
 1 A R É F Z S O I B T S Z E S R V E Z D É S I S E Z I N E T E K M O I B T O V N H C J W W R X S M R F Z Ö R E W T L D L K T E I A D Z W I O S W W E T H Á E J P S E I Z Z T L Y G O A R B Z M L A H E K J
1 A R É F Z S O I B T S Z E S R V E Z D É S I S E Z I N E T E K M O I B T O V N H C J W W R X S M R F Z Ö R E W T L D L K T E I A D Z W I O S W W E T H Á E J P S E I Z Z T L Y G O A R B Z M L A H E K J
ZÖLD BIOTECHNOLÓGIA. 6. évf. - 2010/2. február. http://www.zoldbiotech.hu
 ZÖLD BIOTECHNOLÓGIA 6. évf. - 2010/2. február http://www.zoldbiotech.hu Transzgenikus növények a jövõ biofermentorai? Oszvald Mária Növényélettani és Molekuláris Növénybiológiai Tanszék, ELTE, Budapest
ZÖLD BIOTECHNOLÓGIA 6. évf. - 2010/2. február http://www.zoldbiotech.hu Transzgenikus növények a jövõ biofermentorai? Oszvald Mária Növényélettani és Molekuláris Növénybiológiai Tanszék, ELTE, Budapest
Populációgenetikai. alapok
 Populációgenetikai alapok Populáció = egyedek egy adott csoportja Az egyedek eltérnek egymástól morfológiailag, de viselkedésüket tekintve is = genetikai különbségek Fenotípus = külső jellegek morfológia,
Populációgenetikai alapok Populáció = egyedek egy adott csoportja Az egyedek eltérnek egymástól morfológiailag, de viselkedésüket tekintve is = genetikai különbségek Fenotípus = külső jellegek morfológia,
Tudományközi beszélgetések
 VILÁGOSSÁG 2003/9 10. Tudományrendszer Tudományközi beszélgetések Molekuláris biológia A XXI. század tudományrendszere című nagyprojektje keretében tudományközti beszélgetések sorozatát indította el az
VILÁGOSSÁG 2003/9 10. Tudományrendszer Tudományközi beszélgetések Molekuláris biológia A XXI. század tudományrendszere című nagyprojektje keretében tudományközti beszélgetések sorozatát indította el az
Génkifejeződési vizsgálatok. Kocsy Gábor
 Génkifejeződési vizsgálatok MTA Mezőgazdasági Kutatóintézete Növényi Molekuláris Biológia Osztály A génkifejeződés A sejtmag géneket tartalmaz; (fehérjéket, RNSeket kódoló); A gének átíródnak mrns; Pre-mRNS
Génkifejeződési vizsgálatok MTA Mezőgazdasági Kutatóintézete Növényi Molekuláris Biológia Osztály A génkifejeződés A sejtmag géneket tartalmaz; (fehérjéket, RNSeket kódoló); A gének átíródnak mrns; Pre-mRNS
